低深度全基因组测序技术在复发性流产遗传学病因诊断中的应用
纪桢 李晓洲 王秀艳 刘蓝泽 孟凡荣 琚端

基金項目:国家自然科学基金资助项目(81901502);天津市医学重点学科(专科)建设项目(TJYXZDXK-031A)
作者单位:1天津市第五中心医院妇产科(邮编300450);2天津医科大学总医院妇产科遗传与产前诊断中心;3天津市女性生殖健康与优生重点实验室
作者简介:纪桢(1983),女,住院医师,主要从事妇科常见病、妇科内分泌疾病相关研究。E-mail:54623948@qq.com
△通信作者 E-mail:saijd@163.com
摘要:目的 应用低深度全基因组拷贝数变异分析(CNV-seq)技术研究胚胎染色体异常在复发性流产(RSA)及偶发流产(SA)中的差异。方法 采集158例RSA患者(RSA组)和244例SA患者(SA组)的流产组织进行CNV-seq检测,对可疑染色体异常的夫妇进行高分辨外周血染色体核型检测。结果 402例样本中有2例检测失败,检测成功率99.5%(400/402)。共检测出染色体异常238例(59.5%),包括染色体数目异常212例(89.1%),致病性拷贝数变异25例(10.5%),单亲二倍体1例(0.4%)。RSA组和SA组总体染色体异常、非整倍体、三倍体发生率差异均无统计学意义。RSA组致病性拷贝数变异在染色体异常中的构成比显著高于SA组(P<0.05)。高分辨外周血核型分析检测共发现4例平衡易位携带者。35~39岁年龄段中SA组胚胎染色体异常率高于RSA组(P<0.05)。2组早期流产中胚胎染色体异常率均明显高于中期流产;早期流产中,SA组的流产组织(POC)染色体异常率高于RSA组(P<0.05)。结论 CNV-seq可以对胚胎染色体数目异常和染色体片段重复/缺失进行精准诊断,在RSA和SA的遗传学病因诊断中同样重要,可为再生育指导提供依据。
关键词:流产,习惯性;全基因组测序;DNA拷贝数变异;遗传学;染色体畸变;核型分析
中图分类号:R394.2文献标志码:ADOI:10.11958/20231247
Application of low depth whole genome sequencing technology in the diagnosis of genetic etiology of recurrent spontaneous abortion
JI Zhen1, LI Xiaozhou2, 3, WANG Xiuyan2, 3, LIU Lanze1, MENG Fanrong2, 3, JU Duan2, 3△
1 Department of Gynecology and Obstetrics, Tianjin Fifth Central Hospital, Tianjin 300450, China; 2 Genetics and Prenatal Diagnosis Center of Obstetrics and Gynecology, Tianjin Medical University General Hospital;
3 Tianjin Key Laboratory of Female Reproductive Health and Eugenics
△Corresponding Author E-mail: saijd@163.com
Abstract: Objective To investigate the application value and significance of low depth genome-wide copy number variation sequencing (CNV-seq) in the diagnosis of genetic etiology of recurrent spontaneous abortion by comparing differences of chromosome abnormalities and copy number variation in recurrent spontaneous abortion (RSA) and sporadic abortion (SA). Methods A total of 402 aborted tissue from 158 RSA patients and 244 SA patients were collected for CNV-seq detection. The chromosome karyotypes in peripheral blood of couples with suspected chromosomal abnormality were detected with high resolution. Results In 402 samples, 2 cases were failed, and the detection success rate was 99.5% (400/402). A total of 238 (59.5%) chromosome abnormalities were detected in 400 samples, including 212 (89.1%) chromosome number abnormalities, 25 (10.5%) pathogenic copy number variation and 1 (0.4%) uniparental disomy. There were no significant differences in the overall incidence rate of chromosome abnormality, aneuploid abnormality and triploid between the two groups. The proportion of pathogenic copy number variation in chromosome abnormalities was significantly higher in the RSA group than that in the SA group (P<0.05). A total of 4 balanced translocation carriers were detected by high-resolution peripheral blood karyotype analysis. During the age range of 35-39, the rate of chromosomal abnormality was significantly higher in the SA group than that in the RSA group (P<0.05). In both the RSA group and the SA group, the chromosomal abnormality rate in the first trimester abortion was significantly higher than that in the second trimester abortion, while the rate of POCs chromosomal abnormalities was higher in the SA group than that in the RSA group in the first trimester abortion (P<0.05). Conclusion CNV-seq can accurately diagnose the numerical chromosomal abnormalities and the duplication/deletion of chromosome fragments in embryos, which is equally important in the genetic etiology diagnosis of RSA and SA, and can provide sufficient evidence for the guidance of reproduction.
Key words: abortion, habitual; whole genome sequencing; DNA copy number variations; genetics; chromosome aberrations; karyotyping
复发性流产(recurrent spontaneous abortion,RSA)是指连续发生的2次及以上的在妊娠28周之前的胎儿丢失,是常见的妊娠并发症,也是危害女性生殖健康的主要原因。据统计,育龄女性中RSA的发病率为1%~5%,并呈逐年上升趋势[1]。RSA的发病原因复杂,包括遗传、易栓、免疫、解剖、感染、内分泌等因素[1]。目前达成共识的是胚胎染色体异常是自然流产最主要的原因,占50%~70%[1-2]。基于二代测序技术(next generation sequencing,NGS)的低深度基因组拷贝数变异分析(copy number variation sequencing,CNV-seq)技术自2009年首次报道[3]以来已广泛应用于产前诊断及筛查领域,基于该技术分辨率高、准确性高、成本效益高以及覆盖范围广等特点,目前已经成为流产组织(products of conceptions,POCs)遗传学病因筛查的主要方法。既往对POCs遗传因素的研究多集中在早期流产患者中[4],且关于RSA患者和偶发性流产(sporadic abortion,SA)患者POCs遗传病因构成的差异尚存在争议[5]。本研究采用CNV-seq技术对158例RSA患者和244例SA患者的POCs进行检测,比较2组患者POCs中染色体数目异常以及拷贝数变异(copy number variation,CNV)的特点及差异,探讨CNV-seq在RSA患者遗传学病因诊断中的应用价值和意义。
1 对象与方法
1.1 研究对象 选取2020年4月—2022年5月在天津医科大学总医院及天津市第五中心医院就诊的158例RSA患者(RSA组)以及同期就诊的244例按年龄、流产孕周匹配的SA患者(SA组)为研究对象。纳入标准:(1)就诊时经超声确认胚胎或者胎儿停止发育,自然流产或者通过终止妊娠手术获得可检测的流产绒毛组织或者胎儿、胎盘组织且自愿进行POCs遗传学检测。(2)参照《复发性流产诊治专家共识(2022)》[1],RSA患者须符合RSA诊断标准。SA患者须符合就诊时为首次发生的28周之前的妊娠丢失。排除标准:(1)因内外科重大疾病或者意愿外妊娠而终止妊娠者。(2)无法获得可辨认的流产绒毛组织或者胎儿胎盘组织者。纳入研究对象年龄20~46岁,流产孕周5~28周。本研究经过天津医科大学总医院伦理委员会(IRB2023-WZ-128)及天津市第五中心医院伦理委员会(WZX-EC-KY2023025)批准。
1.2 CNV-seq检测 患者进行终止妊娠手术或者自然流产获得POCs后,取新鲜绒毛组织10~15 mg、胎儿皮肤组织或者胎盘组织0.5 cm×0.5 cm×0.5 cm,4 ℃保存,48 h内进行检测。使用DNeasy Blood & Tissue Kit(Qiagen,德国)进行POCs基因组DNA提取,应用CNV文库构建试剂盒(北京贝瑞和康生物技术有限公司)进行DNA文库构建,使用Nextseq CN500(Illumina,美国)进行全基因组测序分析(单端测序,读长45 bp)。测序结果与人类基因组参考序列(GRCH37/hg19)进行对比分析。参考OMIM、DECIPHER、DGV、ClinGen等数据库进行分析,依据美国医学遗传学与基因组学学会(American College of Medical Genetics and Genomics,ACMG)指南[6]对结果进行致病性评估。采用荧光定量PCR对15个高度多态性短串联重复序列标记位点进行扩增,以判断母体细胞污染以及检测三倍体。
1.3 染色体核型分析 对POCs结果提示染色体末端重复和缺失,可疑染色体异常的夫妇进行高分辨外周血染色体核型检测。外周血染色体制备,G显带均依据天津医科大学总医院妇产科产前诊断中心实验室标准流程进行。使用全自动染色体显微图像扫描分析系统(蔡司,德国)进行扫描与分析,核型描述根据《人类细胞遗传学国际命名体制》(ISCN2016)完成。
1.4 统计学方法 应用SPSS 25.0软件进行数据分析。计数资料以例或例(%)表示,组间比较采用χ2检验;非正态分布的计量资料以M(P25,P75)表示,组间比较采用Mann-Whitney U检验,P<0.05为差异有统计学意义。
2 结果
2.1 2组一般情况 402例POCs中2例因母体细胞污染导致检测失败(均发生在RSA组),检测成功率99.5%,最终纳入400例,其中RSA组156例,年龄33(30,36)岁,流产孕周8(7,9)周,流产次数2~5次,流产2次106例(68%),流产3次35例(22.4%),流产≥4次15例(9.6%);SA组244例,年龄32(30,36)岁,流产孕周8(7,10)周。2组年龄和流产孕周差异均无统计学意义(Z分别为1.611和0.777,P>0.05)。
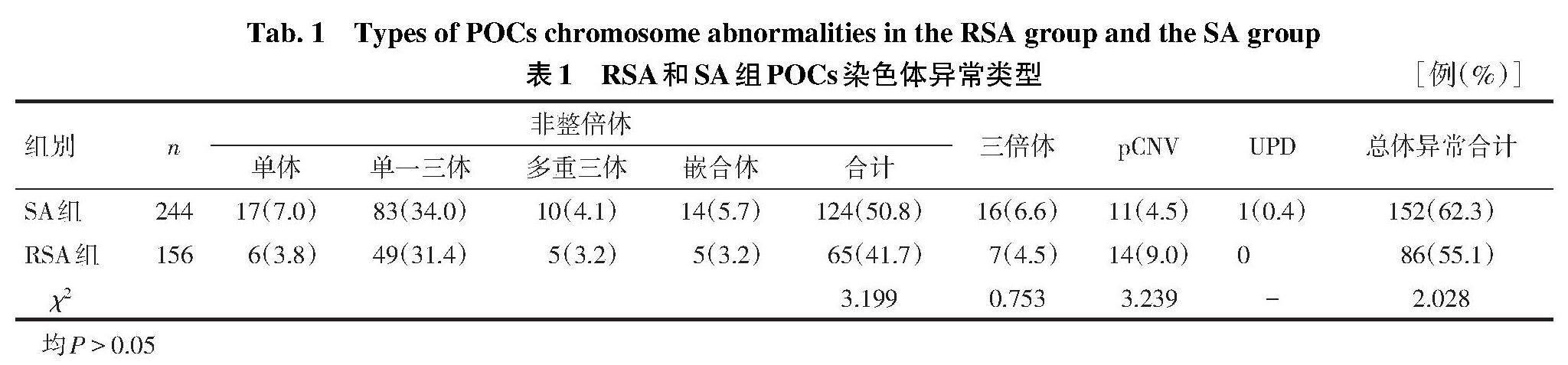
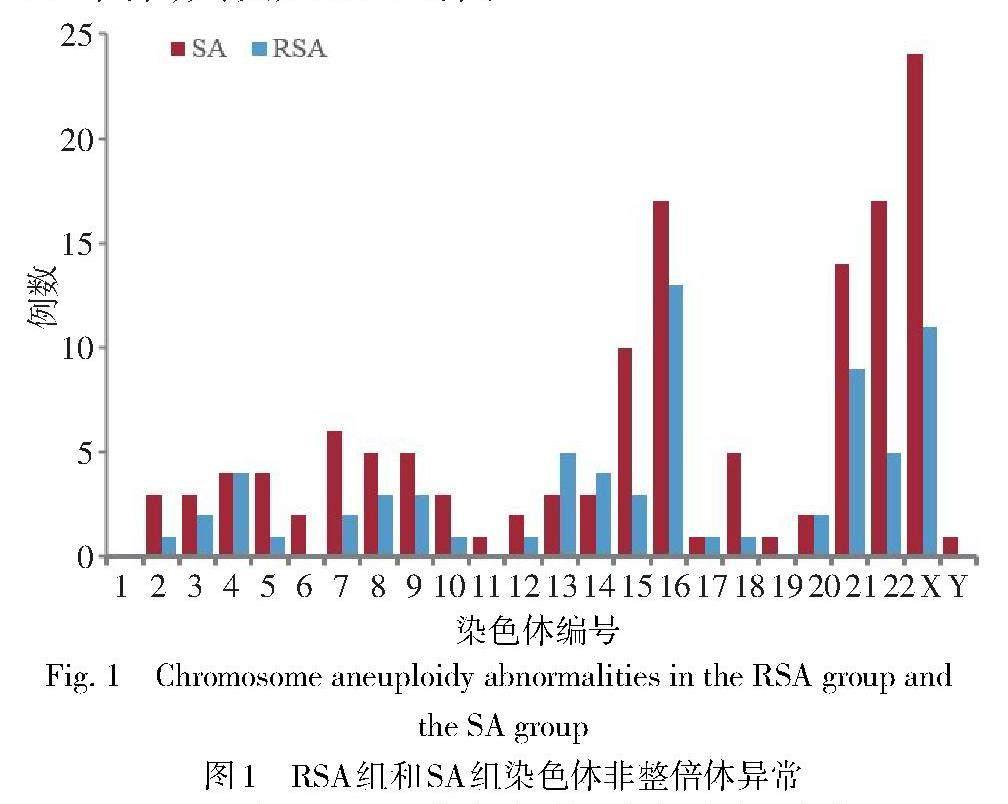
2.2 胚胎染色体总体异常分布情况 400例样本共检测出染色体异常238例(59.5%,238/400),包括染色體数目异常212例(89.1%,212/238)、致病性拷贝数变异(pathogenic copy number variation,pCNV)25例(10.5%,25/238)、单亲二倍体(uniparental disomy,UPD)1例(0.4%,1/238)。染色体数目异常中包括非整倍体189例(89.2%,189/212),三倍体23例(10.8%,23/212)。189例非整倍体异常中包括单体23例(12.2%,23/189),其中22例为X0;单一三体132例(69.8%,132/189),其中16-三体最常见,共25例(18.9%,25/132);多重三体15例(7.9%,15/189),包括2例涉及到3条染色体的多重三体以及13例涉及2条染色体的双三体;嵌合体19例(10.1%,19/189),其中9例X0嵌合体。RSA组和SA组POCs染色体异常情况分布见表1。非整倍体是最常见的染色体异常类型,在RSA组和SA组中POCs非整倍体在染色体异常中构成比分别为75.6%(65/86)和81.6%(124/152)。2组间总体染色体异常、非整倍体、三倍体以及pCNV发生率差异均无统计学意义。RSA组未发现涉及1、6、11以及19号染色体的非整倍体异常,SA组除1号染色体未发现非整倍体异常以外,其余染色体都检测出非整倍体异常。2组间染色非整倍体在1—22号染色体以及性染色体中的分布情况见图1。将嵌合体以及多重三体涉及到的染色体编号依次计算,RSA组非整倍体异常以16-三体构成比最高,SA组非整倍体异常中最常见的是X-单体,其次是22-三体。
2.3 pCNV在2组胚胎染色体异常中的分布 RSA组的14例pCNV含单个位点拷贝数重复2例,单个位点拷贝数缺失7例,多个位点拷贝数重复+缺失5例,片段大小0.26~79.72 Mb,包括6例致病性微缺失/微重复(<10 Mb)和8例致病性大片段缺失/重复(≥10 Mb)。SA组的11例pCNV含单个位点拷贝数重复2例,单个位点拷贝数缺失4例,多个位点拷贝数重复+缺失5例,片段大小位于0.86~99.42 Mb,包括1例致病性微缺失(<10 Mb)和10例致病性大片段缺失/重复(≥10 Mb)。RSA组pCNV在染色体异常中的构成比为16.3%(14/86),高于SA组的7.2%(11/152),差异有统计学意义(χ2=4.777,P<0.05)。高分辨外周血核型分析检测共发現4例平衡易位携带者,见表2。
2.4 年龄和胚胎染色体异常的相关性 将患者年龄按照≤29岁、30~34岁、35~39岁、≥40岁分为4段,SA组分别为56、109、60和19例;RSA组分别为29、69、45和13例。35~39岁年龄段中SA组胚胎染色体异常率高于RSA组(P<0.05),见表3。
2.5 流产孕周和胚胎染色体异常的相关性 将流产孕周以12周为分界,分成早期流产(流产孕周≤12周)和中期流产(流产孕周≥13周)。SA组早期流产和中期流产分别为208例和36例;RSA组为140例和16例。2组早期流产中胚胎染色体异常率均高于中期流产(P<0.05)。早期流产中,SA组的胚胎染色体异常率高于RSA组(P<0.05),见表4。
3 讨论
自然流产,尤其是RSA不仅是生育率下降的一个重要因素,也会对女性生育能力以及身心健康产生长期的危害,是生殖健康领域的重点问题。临床上可识别妊娠中有10%~15%以流产告终,其中RSA发病率为1%~5%[1,7]。RSA的病因和机制复杂,胚胎染色体异常是最常见的原因,对POCs进行遗传学检测对明确RSA病因有重要意义[8-9]。POCs遗传学检测方法经历了从染色体核型分析到荧光原位杂交(fluorescence in situ hybridization,FISH),多重连接依赖探针扩增技术(multiplex ligation dependent probe amplification,MLPA)到染色体微阵列分析(chromosomal microarray analysis,CMA)以及基于NGS的CNV-seq技术[5]。CNV-seq技术具有高灵敏度、高分辨率、全基因组覆盖等特点,目前在临床上可用于复杂POCs样本的遗传学检测。本研究采用CNV-seq技术对POCs进行检测,总体染色体异常检出率为59.5%,其中染色体数目异常占89.1%,pCNV占10.5%,UPD占0.4%,与以往报道基本相符[4,10]。
RSA和SA POCs遗传病因构成的差异仍存在争议。Sheng等[10]对1 556例POCs进行CMA和CNV-seq检测,发现RSA组和SA组POCs染色体异常率差异无统计学意义,但RSA组POCs有更高的CNV发生率并且涉及16q24.3和16p13.3的重复。2020年的一项荟萃研究发现,采用不同检测技术结合的情况下RSA和SA组染色体异常率相近,均为46%[11]。而2018年的一项系统评价发现SA POCs染色体异常率(49.7%,95% CI:34.9%~64.6%)高于RSA(40.4%,95% CI:25.2%~55.7%)[12]。2022年的一项荟萃分析共纳入8 320例POCs,发现SA POCs染色体异常率高于RSA(OR=1.20,95% CI:1.01~1.44,P=0.04)[13]。本研究中RSA组和SA组POCs染色体异常率差异无统计学意义,但当纳入年龄和流产孕周协变量分组研究后笔者发现,35~39岁SA组胚胎染色体异常率高于RSA组,而在母亲年龄小于35岁以及≥40岁时,2组差异无统计学意义。在早期流产中,SA组的POCs染色体异常率高于RSA组,而中期流产中,胚胎染色体总体异常率下降,但2组间胚胎染色体异常率差异无统计学意义。研究表明母亲年龄的增加是胚胎非整倍体发生的危险因素。胎儿时期卵母细胞会停滞在减数分裂Ⅰ期前期并持续相当长的时间,减数分裂长期停滞期间同源染色体间内聚力的退化是人类母亲年龄效应的基础[14-15]。本研究同样证实了随着年龄的增长,胚胎染色体异常率呈上升趋势,在35~39岁,SA总体POCs染色体异常率大于RSA,间接说明RSA中,虽然遗传因素占有一定权重,但尚有其他病因影响。对于40岁以上的RSA人群,胚胎染色体异常率急剧升高,这种趋势说明了超高龄带来的母亲年龄效应引起的胚胎染色体异常在RSA患者流产病因构成中的权重迅速增加。当然也不能除外纳入的≥40岁组的病例较少所导致。
本研究比较了RSA组和SA组间POCs染色体异常的构成。RSA组胚胎染色体异常病例中,非整倍体构成比高达75.6%,以16-三体最常见。SA组非整倍体在胚胎染色体异常中的构成比为81.6%,以X-单体最常见,其次是22-三体。2组均未涉及1号染色体的非整倍体。其原因可能为1号染色体基因组较大,1号染色体三体对胚胎发育是致死性的,往往着床前后就停止发育,很难获得可检测的POCs[16]。本研究发现RSA组pCNV在染色体异常中的构成比为16.3%,高于SA组的7.2%,与以往研究一致[10,17]。pCNV的变化涉及一段或者多段连续的基因,通过基因剂量改变、染色体断裂等方式影响基因表达,从而引起妊娠丢失,但到目前其与妊娠丢失关系尚不明确[18-19]。另外,亲本的结构异常,尤其是染色体平衡性结构重排是RSA的重要原因[20]。CNV-seq无法检测染色体交互易位、倒位等平衡性结构重排,但是当POCs CNV-seq结果提示同时具有缺失和重复异常,并且位于染色体末端位置时,要高度可疑亲本可能是平衡易位携带者。此时要应用核型分析的方法对夫妇双方的外周血染色体核型进行分析。本研究通过分析pCNV的大小和位置对可疑染色体结构异常的夫妇进行了进一步遗传学检测,共发现4例平衡易位携带者,并且易位片段均较小,常规的核型分析容易漏诊。因此,当POCs CNV-seq结果提示缺失或者重复位于染色体末端高度可疑亲本是平衡性结构重排携带者的情况下,建议完善夫妇双方核型分析,CNV-seq检测结果对于亲本染色体平衡易位断点的定位有很好的提示作用,可以增加核型分析的准确性,对于CNV片段较小、可疑隐匿性平衡易位的情况下,建议结合FISH、全基因组光学图谱等技术进行详细分析。
綜上,POCs染色体异常在RSA和SA人群中发生率相似,是妊娠丢失最主要的原因。高龄和孕早期流产是胚胎染色体异常的风险因素。相比SA患者,pCNV在RSA患者中具有更高的发生率,但其与RSA的关系尚需进一步研究。pCNV的检测为提示亲本可能存在平衡性染色体结构重排提供了重要证据。CNV-seq可以对胚胎染色体数目异常和染色体片段重复/缺失进行精准诊断,不仅能明确流产的病因,还可以提示亲本可能存在的染色体结构重排,在复发性流产和偶发流产的遗传学病因诊断中同样重要,为再生育指导提供依据。
参考文献
[1] 中华医学会妇产科学分会产科学组,复发性流产诊治专家共识编写组. 复发性流产诊治专家共识(2022)[J]. 中华妇产科杂志,2022,57(9):653-667. Obstetrics Subgroup,Chinese Society of Obstetrics and Gynecology Chinese Medical Association,Chinese Expert Consensus Group on Diagnosis and Management of Recurrent Spontaneous Abortion. Chinese expert consensus on diagnosis and management of recurrent spontaneous abortion(2022)[J]. Chinese Journal of Obstetrics and Gynecology,2022,57(9):653-667. doi:10.3760/cma.j.cn112141-20220421-00259.
[2] KOWALCZYK K,SMYK M,BARTNIK-G?ASKA M,et al. Application of array comparative genomic hybridization(aCGH)for identification of chromosomal aberrations in the recurrent pregnancy loss[J]. J Assist Reprod Genet,2022,39(2):357-367. doi:10.1007/s10815-022-02400-8.
[3] XIE C,TAMMI M T. CNV-seq,a new method to detect copy number variation using high-throughput sequencing[J]. BMC Bioinformatics,2009,10:80. doi:10.1186/1471-2105-10-80.
[4] WANG Y,LI Y,CHEN Y,et al. Systematic analysis of copy-number variations associated with early pregnancy loss[J]. Ultrasound Obstet Gynecol,2020,55(1):96-104. doi:10.1002/uog.20412.
[5] DAHDOUH E M,KUTTEH W H. Genetic testing of products of conception in recurrent pregnancy loss evaluation[J]. Reprod Biomed Online,2021,43(1):120-126. doi:10.1016/j.rbmo.2021.03.015.
[6] RIGGS E R,ANDERSEN E F,CHERRY A M,et al. Technical standards for the interpretation and reporting of constitutional copy-number variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics(ACMG)and the Clinical Genome Resource(ClinGen)[J]. Genet Med,2020,22(2):245-257. doi:10.1038/s41436-019-0686-8.
[7] RAI R,REGAN L. Recurrent miscarriage[J]. Lancet,2006,368(9535):601-611. doi:10.1016/S0140-6736(06)69204-0.
[8] FINLEY J,HAY S,OLDZEJ J,et al. The genomic basis of sporadic and recurrent pregnancy loss: a comprehensive in-depth analysis of 24,900 miscarriages[J]. Reprod Biomed Online,2022,45(1):125-134. doi:10.1016/j.rbmo.2022.03.014.
[9] TUR-TORRES M H,GARRIDO-GIMENEZ C,ALIJOTAS-REIG J. Genetics of recurrent miscarriage and fetal loss[J]. Best Pract Res Clin Obstet Gynaecol,2017,42:11-25. doi:10.1016/j.bpobgyn.2017.03.007.
[10] SHENG Y R,HOU S Y,HU W T,et al. Characterization of copy-number variations and possible candidate genes in recurrent pregnancy losses[J]. Genes (Basel),2021,12(2):141. doi:10.3390/genes12020141.
[11] SMITS M,VAN MAARLE M,HAMER G,et al. Cytogenetic testing of pregnancy loss tissue:a meta-analysis[J]. Reprod Biomed Online,2020,40(6):867-879. doi:10.1016/j.rbmo.2020.02.001.
[12] ZHANG T,SUN Y,CHEN Z,et al. Traditional and molecular chromosomal abnormality analysis of products of conception in spontaneous and recurrent miscarriage[J]. BJOG,2018,125(4):414-420. doi:10.1111/1471-0528.15052.
[13] LEI D,ZHANG X Y,ZHENG P S. Recurrent pregnancy loss:fewer chromosomal abnormalities in products of conception? a meta-analysis[J]. J Assist Reprod Genet,2022,39(3):559-572. doi:10.1007/s10815-022-02414-2.
[14] NAGAOKA S I,HASSOLD T J,HUNT P A. Human aneuploidy:mechanisms and new insights into an age-old problem[J]. Nat Rev Genet,2012,13(7):493-504. doi:10.1038/nrg3245.
[15] HASSOLD T,HUNT P. To err(meiotically) is human:the genesis of human aneuploidy[J]. Nat Rev Genet,2001,2(4):280-291. doi:10.1038/35066065.
[16] 陳俊坤,胡莉琴,杨晶珍,等. 用高通量测序技术检测流产物染色体数目和拷贝数变异的研究[J]. 中华医学遗传学杂志,2018,35(4):591-594. CHEN J K,HU L Q,YANG J Z,et al. Application of next generation sequencing for the detection of chromosomal aneuploidies and copy number variations in abortus tissues[J]. Chinese Journal of Medical Genetics,2018,35(4):591-594. doi:10.3760/cma.j.issn.1003-9406.2018.04.029.
[17] FAN L,WU J,WU Y,et al. Analysis of chromosomal copy number in first-trimester pregnancy loss using next-generation sequencing[J]. Front Genet,2020,11:545856. doi:10.3389/fgene.2020.545856.
[18] REDIN C,BRAND H,COLLINS R L,et al. The genomic landscape of balanced cytogenetic abnormalities associated with human congenital anomalies[J]. Nat Genet,2017,49(1):36-45. doi:10.1038/ng.3720.
[19] RAJCAN-SEPAROVIC E,DIEGO-ALVAREZ D,ROBINSON W P,et al. Identification of copy number variants in miscarriages from couples with idiopathic recurrent pregnancy loss[J]. Hum Reprod,2010,25(11):2913-2922. doi:10.1093/humrep/deq202.
[20] STEPHENSON M D,SIERRA S. Reproductive outcomes in recurrent pregnancy loss associated with a parental carrier of a structural chromosome rearrangement[J]. Hum Reprod,2006,21(4):1076-1082. doi:10.1093/humrep/dei417.
(2023-08-16收稿 2023-10-13修回)
(本文编辑 李志芸)

