藜麦CqAMSlike基因的克隆与功能分析
张玉敏 林春 刘正杰 林彤 董玉梅 毛自朝
摘 要 为探究藜麦(Chenopodium quinoa,Willd.)花粉发育的分子机制,基于基因组和转录组数据,经同源比对及RT-qPCR表达分析等,预测藜麦中AMS候选基因并克隆其编码序列,随后经病毒介导的基因沉默(VIGS)及双分子荧光互补(BiFC)法进行候选基因的功能验证。结果显示,藜麦中有 CqAMSlike1和 CqAMSlike2 两个基因,其中 CqAMSlike1的CDS长1 929 bp,而 CqAMSlike2的CDS长1 920 bp,两基因均在不同发育时期的圆锥花序中表达。与 CqAMSlike2相比, CqAMSlike1的表达更与AMS的表达模式相一致,而被进一步用于基因的功能验证。与对照相比,基于烟草脆裂病毒(TRV)沉默 CqAMSlike1的藜麦两性花出现部分花丝缩短、花药不裂,花粉败育,单株种子少,种子空瘪率增加的表型。BIFC结果显示CqAMSlike1自身,与 CqTDF1和CqMYB80均有互作,预示CqAMSlike1可形成同源或异源聚体调控藜麦绒毡层发育基因的表达。
关键词 藜麦;AMS;病毒介导的基因沉默;花粉发育;蛋白互作
藜麦(Chenopodium quinoa Willd.,2n= 4x=36)是苋科藜属1 a生双子叶植物,具有较高的营养价值[1],并对多种非生物逆境胁迫具有较强的抗性,近年来备受关注[2-5]。然而藜麦花器官结构复杂、尺寸小,同一植株的花由大小不同的雌花和两性花组成,比例受到遺传和环境的影响,导致人工去雄难,杂交育种困难[6]。目前在自然界中很难找到藜麦的自然突变的雄性不育材料,因而研究其花粉发育相关基因是创制人工雄性不育材料的基础。
花粉的败育与绒毡层的发育密切相关,绒毡层的缺陷会直接影响植物花粉的育性[7]。目前,花药发育的分子机制已在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)等模式植物中开展了大量的研究[8]。在拟南芥中AMS(aborted microspores)基因一个编码特殊bHLH家族的MYC2型的转录因子[9],其表达异常会造成绒毡层异常空泡化、小孢子退化[10]。AMS的表达受上游基因TDF1(defective in tapetal development and function 1)的调控[11-12]。TDF1基因编码一个R2R3 YMB转录因子[13],TDF1通过结合AACCT顺式元件调控AMS的表达,并与AMS发生互作形成复合物调节下游基因的表达[14];而AMS则可通过结合Ebox调节下游MYB80基因[15]。MYB80与TDF1均编码一个R2R3 YMB转录因子[16],YMB80也可以与AMS形成复合物而最终促进孢子发育[17]。在拟南芥中已阐明绒毡层发育后期的转录调控途径[DYT1-TDF1(MYB35)-AMS-MS188(MYB80)-MS1][18]。
2017公布了高质量参藜麦考基因组[19],为其基因发掘和利用创造了良好的基础,但由于藜麦中较难建立遗传转化体系,基因功能验证工作难以推进。目前病毒诱导基因沉默(Virusinduced gene silencing VIGS) 的方法已在茄科[20]和禾本科[21]的基因功能研究中得到广泛应用,但在藜麦的研究中应用甚少。本研究为挖掘藜麦中AMS基因及其表达作用来阐明CqAMSlike基因在藜麦花药发育过程中的作用,为藜麦雄性不育种质创制等提供依据。
1 材料与方法
1.1 材料、质粒、菌种
以白藜麦(BW25)为材料,种植于22 ℃,光照时间16 h/8 h(光照/黑暗)的温室环境。大肠杆菌 DH5α及农杆菌 GV3101宿主菌系均为云南农业大学特色小宗作物研究所保存;VIGS构建所用到的载体pTRV1、pTRV2 由云南农业大学植物保护学院李凡老师惠赠;蛋白质互作的双分子荧光表达载体pBRVC、pAgVN由云南农业大学特色小宗作物研究中心提供。
1.2 方法
1.2.1 藜麦RNA提取及cDNA第一链合成 参阅试剂盒说明书,利用京艾德莱生物科技有限公司的EASYspin Plus试剂盒提取RNA,然后用全式金公司的TransScript Onestep gDNA Removal and cDNA Synthesis SuperMix试剂盒完成cDNA第一链的合成。
1.2.2 CqAMSlike1/2、CqTDF1和CqMYB80like1/2的生物信息分析 从NCBI下载藜麦及其二倍体(Chenopodium pallidicaule、Chenopodium suecium)的全基因组、蛋白质序列(https://www.ncbi.nlm.nih.gov/datahub/assembly/GCF_001683475.1),以拟南芥bHLH和MYB家族为参照[22-23]构建藜麦以及藜麦二倍体的bHLH和MYB的系统进化树。拟南芥bHLH和MYB家族的信息来自于Tair(https://www.arabidopsis.org/),通过比对拟南芥AtAMS基因(AT2G16910.1)、 AtTDF1基因(AT3G28470.1)和AtMYB80基因(AT5G56110.1),预测藜麦AMS、TDF1和MYB80的同源基因。然后将藜麦以及二倍体藜麦同源基因与菠菜(Spinacia oleracea)、甜菜(Beta vulgaris)等物种已知确定功能的AMS、TDF1与MYB80用MEGA 10(https://www.megasoftware.net/)构建系统进化树。用ExPASy Proteomics ServerProtparam(http://www.expasy.ch/tools/ protparam.htm1) 预测候选蛋白的理化性质,SignalP(http:// http://www.cbs.dtu.dk/services/SignalP/)进行蛋白质的信号肽和跨膜结构预测,CellPLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/CellPLoc2/)进行蛋白亚细胞定位部位预测。
1.2.3 CqAMSlike、CqTDF1like和CqMYB80like基因的克隆 用SnapGene(https://www.snapgene.cn/)软件设计预测 CqAMSlike1/2、CqTDF1和CqMYB80like1/2的克隆引物(表1,用藜麦花序总RNA 反转录cDNA作为模板进行扩增,扩增片段与pUC57T Vector连接,转化至大肠杆菌DH5α感受态细胞,进行阳性克隆筛选后送测序公司进行鉴定,分别将所获质粒命名为pC57CqAMSlike1、pUC57CqAMSlike2、pUC57CqTDF1、pUC57CqMYB80like1 和 pUC57 CqMYB80like2。
1.2.4 CqAMSlike的表达模式分析 提取藜麦(显序期、开花初期、盛花期、开花末期)花序和不同藜麦器官部位(根、叶、花)的RNA并反转录为cDNA作为模板,以藜麦的actin基因作为内参基因[24]进行qRTPCR扩增后,采用2-△△Ct的方法[24]分析 CqAMSlike1和 CqAMSlike2基因在藜麦不同发育期和不同器官部位的表达情况。
1.2.5 pTRV2CqPDS和pTRV2CqAMSlike1载体的构建 以藜麦叶片RNA 反转录的cDNA为模板扩增,上下游引物分别添加BamHI和EcoRI酶切位点(表2),扩增长度为461 bp的CqPDS;以质粒pC57CqAMSlike1为模板扩增长度为498 bp的 CqAMSlike1的片段。将纯化的CqPDS、 CqAMSlike1片段与pTRV2载体同时用BamHI和EcoRI双酶切,连接后,转入大肠杆菌DH5α,提取阳性菌株质粒进行酶切验证后获得重组质粒TRV2CqPDS和TRV2CqAMS。采用电穿孔法(2 500 V)分别将重组质粒pTRV2CqPDS和TRV2CqAMS转染到农杆菌GV3101中进行选择培养,挑选单菌落进行PCR,挑选阳性菌株。
1.2.6 CqPDS和CqAMSlike1的VIGS沉默與表达量检测 将目的片段载体(pTRV2CqPDS和pTRV2CqAMSlike1)、空载体pTRV2和农杆菌接种至含50 mg·L-1卡纳霉素(Kan)和 50 mg·L-1 利福平霉素(Rif)的 YEB 液体培养基中,28 ℃振荡5 h,后用侵染液(1 mol·L-1 MgCl2、 1 mol·L-1 MES贮存液、50 mmol·L-1AS贮存液、pH 5.6)进行重新悬浮。将含有目的片段农杆菌与含pTRV1的农杆菌(pTRV1/GV3101)按照1∶1进行混合,分别注射到藜麦叶片作为试验组和阴性对照组;将没有注射任何菌株的藜麦作为正常对照组。黑暗条件下放置48 h,转至 22 ℃,光照时间16 h/8 h(光照/黑暗)条件下培养。设计CqPDS和CqAMSlike1的TR-qPCR检测引物(表1),用TR-qPCR检测目的基因在不同处理中的相对表达量。
1.2.7 花粉活力的鉴定 采用0.5% TTC(2,3,5三苯基氯化四氮唑)染色法测定藜麦花粉生活力。将同一时期正常、阳性分别置于2 mL 的离心管中,滴加 TTC摇晃均匀后,置于37 ℃恒温箱反应15 min后制片,在显微镜下观察并统计花粉活力,每组试验重复3 次,每次试验选取3 个视野。
花粉活力= 着红色花粉数 / 总花粉数×100%
1.2.8 BiFC载体构建与互作检测 分别设计引物下游引入不同酶切位的引物(表2), 以质粒pUC57 CqAMSlike1/2、pUC57 CqMS80like1/2和pUC57 CqTDF1为模板扩增相应目的全长CDS片段。将扩增的 CqAMSlike1/2片段分别用BamHI和SphI双酶切,分别回收连入pAgVN载体,获得表达CqAMSlike1、CqAMSlike2与venus蛋白N末端的融合蛋白;用SphI和EagI将需要连入的目的片段双酶切,然后分别回收连入pBRVC载体,获得CqAMSlike1、CqAMSlike2、CqMYB80like1和CqTDF1like与venus蛋白C末端融合表达载体。化学合成的IAAL蛋白中互作的结构域 E3和K3编码序列,分别插入pBRVC和pAgVN载体,获得融合表达载体pBRE3VC和pAgK3VN,用于BiFC阳性对照[25],以pAGVNvenus、pBRVCvenus的空白载体作为阴性对照。将构建的BiFC载体按照浓度 1∶1混合共同转入大肠杆菌DH5α感受态细胞中,均匀涂布于含有庆大霉素(Gen) 50 mg·L-1和卡那霉素(Kan) 50 mg·L-1的LB固体培养基上37 ℃过夜筛选培养。用菌落PCR检测单菌落,挑取阳性共转化菌株。共转化菌株于37 ℃, 200 r·min-1振荡培养12 h后, 换30 ℃,200 r·min-1振荡培养4~8 h。在载玻片上滴加少量甘油和菌液,混合后使用激光共焦聚显微镜(OLYMP FV 1000)观察黄色荧光 信号。
2 结果与分析
2.1 CqAMS、CqTDF1和CqMYB80基因的筛选、克隆及其生物信息分析
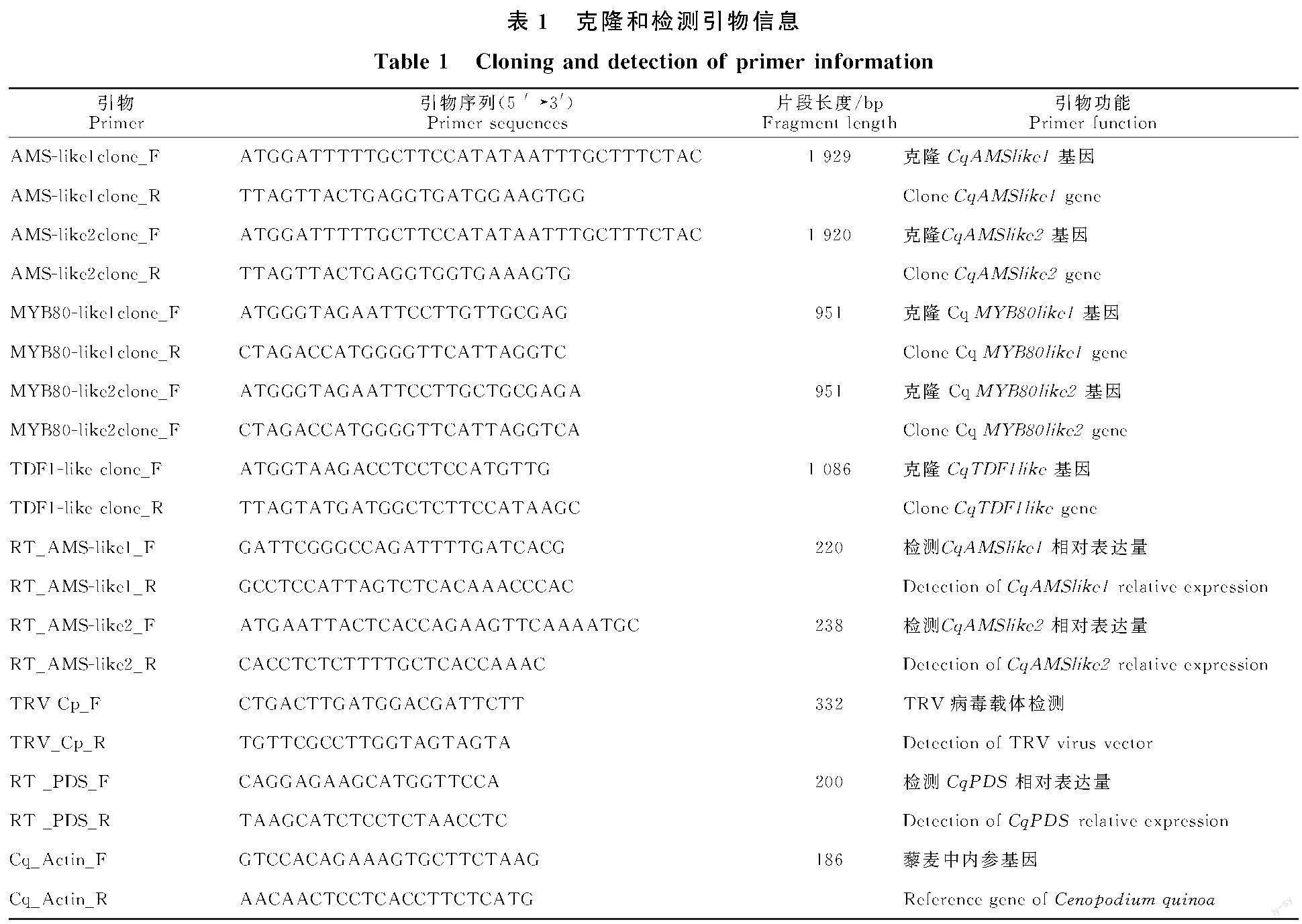
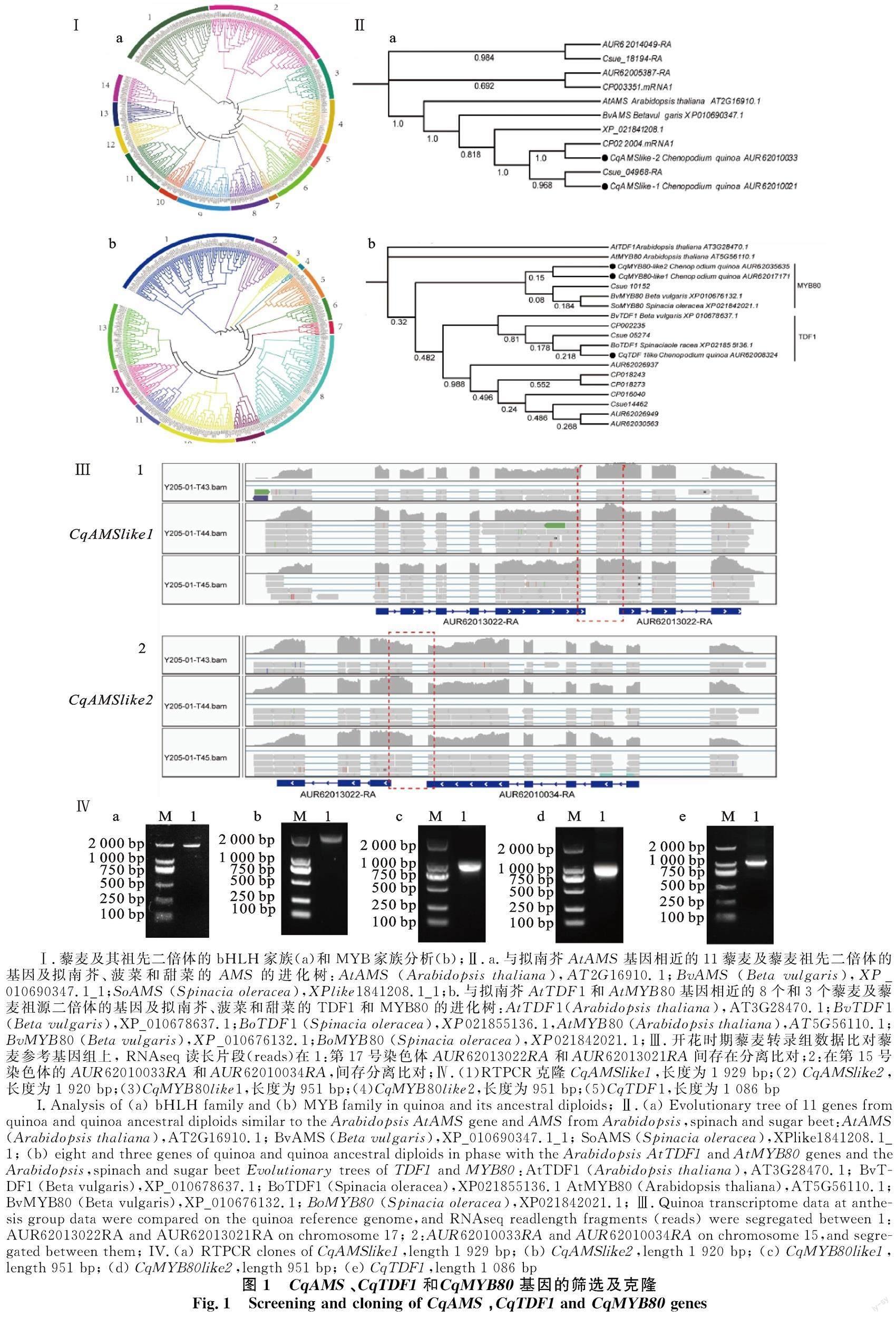
以拟南芥的bHLH和MYB家族的蛋白质序列为参照,筛选并构建藜麦及藜麦二倍体的相应基因家族系统进化树。藜麦的bHLH基因家族分为14个亚家族(图1-Ⅰa),而MYB基因家族分为13个亚家族(图1-Ⅰb)。其中与拟南芥AtAMS相近的基因有11个序列,包含6个藜麦基因(AUR62013022RA、AUR62013021RA、AUR62010033RA、AUR62010034RA、AUR620-05378RA、AUR62014049RA );与拟南芥AtTDF1相近藜麦基因有4个序列(AUR62-026937RA、AUR62030563RA、AUR62026949RA、AUR62018324RA);与拟南芥AtMYB80基因相近的藜麦基因有2个(AUR62017171RA和AUR62035635RA)。用Hisat2[26]对藜麦(BW25)不同发育时期花序的转录组数据与基因组序列进行比对,用IGV(Integrative Genomics Viewer)[27]可视化展示后发现, AUR62013022RA和AUR62013021RA、 AUR62010033RA和AUR62010034RA之间存在分离比对,确定AUR62013022RA、AUR62013021RA在一条CDS上(图1-Ⅲa),命名为 CqAMSlike1,全长CDS为1 929 bp;同理,AUR62010033RA和AUR62010034RA在另一条CDS上(图1-Ⅲb),命名为 CqAMSlike2,长度为1 920 bp。进一步将矫正的基因(CqAMSlike1/2)与菠菜和甜菜的AMS、TDF1和MYB80基因进行同源性比对,结果表明: CqAMSlike1和 CqAMSlike2与菠菜和甜菜的AMS基因有较高的同源性,并且两条基因编码的氨基酸序列具有93.77%的相似性,将其作为候选基因进行后续试验,其中 CqAMSlike1与 CP022004、 CqAMSlike2和Csue 04968RA具有较高同源性(图1-Ⅱa)。CqMYB 80like 1(AUR62017171RA)、CqMYB 80like 2(AUR62035635RA)与菠菜和甜菜的MYB80基因也具有较高的同源性,并且两条基因编码的氨基酸序列具有98.73%的相似性(图1-Ⅱb);进一步分析确定藜麦中仅有一条CqTDF1(AUR62008324RA)与甜菜和菠菜的TDF1具有相似的同源性(图1-Ⅱb)。
藜麦 CqAMSlike1基因位于17号染色体,包含有8个外显子 (图1-Ⅳa),编码642个氨基酸,蛋白质分子质量73.432 ku,等电点5.60; CqAMSlike2基因位于15号染色体,也包含有8个外显子,(图1-Ⅳb),编码639个氨基酸,其蛋白质分子质量73.139 ku,等电点5.72。CqMYB80like1、CqMYBlike2基因的CDS全长951 bp(图1-Ⅳc、d),CqMYB80like1位于2号染色体,CqMYB80like2位于1号染色,都含有3个外显子,编码316个氨基酸,其蛋白质分子质量35.160 ku,等电点6.76。CqTDF1基因位于16号染色体上,含有3个外显子,CDS全长1086bp(图1Ⅳ5),编码361个氨基酸,蛋白之分子质量为40.000 ku,等电点5.94。预测分析发现,这些基因都含有核定位信号,用CellPLoc 2.0预测,都定位在细胞核内。信号肽预测显示,这些蛋白没有信号肽,这些特征与他们作为转录因子的功能相一致。
2.2 CqAMSlike基因在藜麦植株不同生育时期及不同组织的表达特性
如图2所示,藜麦植株从显序期(花序刚出现)到末花期 CqAMSlike1/2基因的表达明显不同。其中 CqAMSlike1随着花发育进程的推进,基因的表达呈先升高后降低的趋势,在初花期阶段达到最高,末花期最低(图2-Ⅰ); CqAMSlike2基因的表达未发生变化,总体表达低于 CqAMSlike1。不同组织的表达分析表明, CqAMSlike1基因在藜麦花序中表达较高,在叶片和根中的表达较低,与拟南芥AMS表达模式相似,从而推测, CqAMSlike1可能在藜麦花粉发育中起主要调控作用,而 CqAMSlike2 可能作为冗余或功能或表达模式改变的重复基因,为此 CqAMSlike1被优选进行后续的功能分析。
2.3 藜麦VIGS系统的构建
含质粒pTRV2PDS和TRV1的根癌农杆菌侵染藜麦后,14 d的藜麦叶先从叶脉开始,随后至叶片出现白化现象,第20天时有明显叶片漂白现象(图3-Ⅲa、图3-Ⅲb),而空白对照(图3-Ⅲc)和阴性对照(图3-Ⅲd)叶片颜色表现正常。藜麦植株的外形,除叶片颜色外其他无明显改变。处理25 d后,观察50株藜麦叶片表型变化,共发现44株出现白化现象,其余6株叶片颜色无明显变化,侵染效率达到88%(n=50)。进一步观测发现空白对照未出现白化表型,仅接种空白TRV2载体的藜麦植株出现叶片轻微发黄现象,这可能与TRV接种后产生病症相一致,且随时间延长该病症逐渐减轻直至消失。
RT-qPCR檢测接种25 d的藜麦叶片基因表达发现,CqPDS表达量在接种pTRV2CqPDS藜麦中明显下降,而在接种TVR2空载体藜麦中未检测到变化(图3-Ⅱ),预示在藜麦中成功构建基于TRV的VIGS体系。
2.4 CqAMSlike1 的VIGS沉默
与空白对照相比,接种TRV2 CqAMSlike1和TRV1农杆菌的藜麦花序中 CqAMSlike1基因的表达量明显下降,而接种TRV2空载的表达量下降不明显(图4-Ⅱ、图4-Ⅳ)。在开花期藜麦顶端两性花中,接种TRV2CqAMSlike1的藜麦花相比正常植株(图4-Ⅲa、图4-Ⅲd)和接种TRV2空载体的藜麦(图4-Ⅲb、图4-Ⅲe)相比,出现了花丝缩短(图4-Ⅲc),花药不裂,花粉难释放的表型(图4-Ⅲf),因VIGS沉默的不稳定性,上述表型并非每一朵顶部两性花都会出现,特别是花药不裂,只发现3~4枚花药出现(图4-Ⅲe)。花粉活力检测结果显示,正常植株和接种TRV2空载体植株的花粉活力较高(图4-Ⅲg和图4- Ⅲh),而VIGS沉默 CqAMSlike1的藜麦中部分花粉未能染色(图4-Ⅲi),呈现花粉活力明显降低(图4-Ⅴ),藜麦部分雄性不育,从而降低藜麦的结实率并增加空瘪率(图4-Ⅴ)。
2.5 CqAMSlike1/2与CqTDF1和CqMYB80like1 蛋白的互作
通过BiFC对CqAMSlike1/2与CqTDF1和CqMYB80like1之间互作进行验证,结果表明:E3与Venus的C端、K3与Venus的N端的融合表达多肽能够互作产生明显黄色荧光(图5-Ⅱa); Venus的C端和N端之间不能产生互作,未产生黄色荧光(图5-Ⅱb)。CqAMSlike1与venues的N端和C端融合表蛋白能够互作,能产生较弱的黄色荧光(图5-Ⅱc),而CqAMSlike2与venues的N端和C端融合蛋白不能互作,未产生黄色荧光(图5-Ⅱd),预示着CqAMSlike1而非CqAMSlike2可形成二聚体或多聚体。进一步互作分析发现CqTDF1like与venues的C端融合表达蛋白能和CqAMSlike1与venues的N端融合表达蛋白形成二聚体产生较弱的荧光(图5-Ⅱe)但与CqAMSlike2与venues的N端融合表达蛋白未能检测到荧光(图5-Ⅱf)。CqMYB80like1与venues的C端融合表达蛋白能与CqAMSlike1的venus N端融合表达蛋白产生较强的荧光信号(图5-Ⅱg)而与CqAMSlike2的N端融合表达蛋白也能形成二聚体,但产生荧光信号较弱(图5-Ⅱh)。这些结果与此前报道在拟南芥中MYB80会与AMS互作调控下游基因表达相一致[15]。
3 讨 论
植物雄性不育性状受到遗传和环境的综合影响[28],目前雄性不育基因应调控功能不同可分为影响减数分裂至败育的PSS1[29]、AM1[30]等;影响胼胝质代谢的AtGsl5[31]等;影响绒毡层发育的AtAMS、OsTDR[7]、OsUDT[32]等;及影响花粉壁发育的CYP704B2[33]等调控基因。在这些基因中拟南芥AtDYT1、AtAMS和水稻OsTDR、OsUDT归属于bHLH超家族中的Ⅲ亚家族[34]。该bHLH亚家族中的成员与花药发育中绒毡层能发育相关,根据相似性比对,拟南芥AtAMS与水稻OsTDR、拟南芥AtDYT与水稻OsUDT均属直系同源基因[7]。在模式植物拟南芥中花粉绒毡层调控机制已被阐明,该发育过程涉及DYT1、TDF1、AMS、MYB80、MS等多个转录因子的协同调控网络。这些转录因子因功能的重要性其突变体常败育,在不同植物间高度保守[8],其中归属于bHLH超家族的AMS在绒毡层发育调控中起到关键作用[35]。
藜麦基因组的注释中将位于17、15号染色体的两个AMS同源基因基,均裂分为两个预测基因,本研究通过对比转录组数据将拆分的基因合并后重新分别命名为 CqAMSlike1和 CqAMSlike2,新预测基因的编码区长度,蛋白序列及在染色体上内含子数量与结构均与拟南芥AtAMS有较高的相似性[9]。AtAMS及其直系同源基因有著明显的时空表达特型,在花药发育的5~8期表达量最高,之后降低,该结论在番茄和辣椒中也获得了验证[9,21]。基因表达分析表明,藜麦 CqAMSlike1的表达模式与AtAMS 更相似,预示 CqAMSlike1比 CqAMSlike2更可能是主要发挥调控藜麦花粉绒毡层发育的AMS同源体。藜麦属于异源四倍体,由A和B亚基因组融合[19,36],藜麦基因组中染色体共线性分析表明定位于藜麦17号、15号染色体的分别与B基因组和A基因组二倍体物种有更多的共线性模块[27],推测, CqAMSlike1和 CqAMSlike2的冗余可能是在该物种形成中分别获于2倍祖先物种的AMS同源基因,但本研究中发现 CqAMSlike1和 CqAMSlike2表达模式和基因结构的变异,特别是功能上与CqTDFlike、CqMYB80like1互作的变化,可能源于A、B亚基因组中祖先基因的差异及进化中 CqAMSlike1和 CqAMSlike2结构和功能进一步异化,但具体演化机制有待进一步的研究。
VIGS沉默 CqAMSlike1的表型呈现与用VIGS降低辣椒CaAMS表达量的表型相似[21],初步确定了 CqAMSlike1是藜麦中有具有功能的AtAMS直系同源基因。AMS与上游的TDF1及与下游的MS188(MYB80)形成互作网络[7]。AMS基因的表达受上游转录因子的调控,并且还可以与上游基因编码的转录因子互作形成异源二聚体,扶艳艳等[25]在芦笋中验证AoTDF1与AoAMS之间存在互作,以异源二聚体的形式调节下游基因的表达。同样的,AMS也可以直接结合下游基因的启动子中Ebox区调控其表达[37],并与MYB80互作形成复合物,从而调控绒毡层和小孢子的发育。本研究用双分子荧光互补方法初步证实CqAMSlike1与CqTDF1和CqMYB80蛋白之间均有互作(图5-Ⅱe和5- Ⅱg)。同时CqAMSlike1自身也发生互作反应(图5-Ⅱc),结果预示CqAMSlike1可能与模式植物有相似的方式调控绒毡层发育[15,16,36]。藜麦中CqAMSlike1的功能发现,进一步预示不同物种的植物间调控绒毡层发育的模式保守的。本研究发现CqAMSlike2与CqAMSlike1,CqAMSlike2与CqTDF1均没检测到明显的互作荧光信号(图5),但能检测到CqAMSlike2与CqMYB80like1的互作荧光信号,预示CqAMSlike2可能也参与花粉发育通路的调控,但该假说的验证及具体的调控机制有待进一步研究。
综上所述,藜麦 CqAMSlike1是藜麦中有功能的AMS同源基因,其调控绒毡层的模式可能与模式植物中AMS相似,受CqTDF1like的调控,同时其编码产物又调控下游CqMYB80like1等靶基因的表达,且CqAMSlike1与CqTDF1like和CqMYB80like1可能形成复杂的转录调控复合物,调控藜麦绒毡层的发育。本研究将为阐明为藜麦花粉发育的分子机制及用于育种应用提供基础。
参考文献 Reference:
[1] VILCACUNDO R B H-L.Nutritional and biological value of quinoa (Chenopodium quinoa Willd.) [J]. Current Opinion in Food Science,2016,14:1-6.
[2] 陈阳郭,武 悦,张 春,等.藜麦CqSAP8基因克隆及其在非生物胁迫下的表达分析[J].西北植物学报,2021, 41(12):2014-2020.
CHEN Y G,WU Y,ZHANG CH,et al.Cloning and Expression Analysis of CqSAP8 in Chenopodium quinoa under Abiotic Stresses[J].Acta Botanica Boreali-Occidentalia Sinica,2021,41(12):2014-2020.
[3] 姚有华,白羿雄,吴昆仑.亏缺灌溉对藜麦光合特性、营养品质和产量的影响 [J].西北农业学报,2019,28(5):711-713.
YAO Y H,BAI Y X,WU K L.Effects of deficient irrigation on the photosyntheticcharacteristics,nutritional quality and yield of quinoa[J].Acta Agriculturae Boreali-occidentalis Sinica,2019,28(5):711-713.
[4] 魏玉明,杨发荣,刘文瑜,等.陇东旱塬区复种不同藜麦品种(系)的适应性初步评价 [J].西北农业学报,2020,30(5):675-686.
WEI Y M,YANG F R,LIU W Y,et al. adaptation evaluation of multiple cropping of 12 quinoa varieties on loess dryland in eastern gansu[J].Acta Agriculturae Boreali-occidentalis Sinica, 2020,30(5):675-686.
[5] GAMEZ A L,SOBA D,ZAMARRENO A M,et al.Effect of water stress during grain filling on yield,quality and physiological traits of illpa and rainbow quinoa (Chenopodium quinoa Willd.) cultivars [J].Plants (Basel),2019, 8(6):173-188.
[6] 阿圖尔·博汗格瓦,希皮尔·斯利瓦斯塔瓦.藜麦生产与应用 [M].北京:科学出版社,2014:90-102.
ATUL B,SHILPI S.Quinoa:Botany,Production and Uses[M].Beijing:Science Press,2014:90-102。
[7] PARISH R W,LI S.Death of a tapetum:aprogramme of developmental altruism [J].Plant Science,2010,178(2):73-89.
[8] GOMEZ J F,TALLE B,WILSON Z A.Anther and pollen development:a conserved developmental pathway [J].Journal Integrative Plant Biology, 2015,57(11):876-891.
[9] SORENSEN A M,KRBER S,UNTE U S,et al.The arabidopsis ABORTED MICROSPORES AMS gene encodes a MYC class transcription [J].The Plant Journal,2003,33:413-423.
[10]XU J,YANG C,YUAN Z,et al.The ABORTED MICROSPORES regulatory network is required for postmeiotic male reproductive development in Arabidopsis thaliana [J].Plant Cell,2010,22(1):91-107.
[11] GU J N,ZHU J,YU Y,et al.DYT1 directly regulates the expression of TDF1 for tapetum development and pollen wall formation in Arabidopsis[J].The Plant Journal,2014,80(6):1005-1013.
[12] FENG B,LU D,MA X,et al.Regulation of the arabidopsis anther transcriptome by DYT1 for pollen development [J].The Plant Journal,2012,72(4):612-624.
[13] YU J,ZHAO G,LI W,et al.A single nucleotide polymorphism in an R2R3 MYB transcription factor gene triggers the male sterility in soybean ms6 (Ames1) [J].Theoretical and Applied Genetics,2021,134(11):3661-3674.
[14] LOU Y,ZHOU H S,HAN Y,et al.Positive regulation of AMS by TDF1 and the formation of a TDF1-AMS complex are required for anther development in Arabidopsis thaliana[J].New Phytologist,2018,217(1):378-391.
[15] LOU Y,XU X F,ZHU J,et al.The tapetal AHL family protein TEK determines nexine formation in the pollen wall [J].Nature Communications,2014,5:3855.
[16] HUY A.PHAN S F L R W P.MYB80,a regulator of tapetal and pollen development,is functionally conserved in crops [J].Plant Molecular Biology,2011,78(1):171-183.
[17] ZHU J,LOU Y,XU X,et al.A genetic pathway for tapetum development and function in Arabidopsis [J].Journal of Integrative Plant Biology,2011,53(11):892-900.
[18] WANG K,GUO Z L,ZHOU W T,et al.The regulation of sporopollenin biosynthesis genes for rapid pollen wall formation [J].Plant Physiology,2018,178(1):283-294.
[19] JARVIS D E,HO Y S,LIGHTFOOT D J,et al.The genome ofChenopodium quinoa [J].Nature,2017,542(7641):307-312.
[20] Arce-Rodríguez M L,Ochoa-Alejo N.Virus-Induced gene silencing (VIGS) in Chili Pepper (Capsicum spp.)[J].Methods Molecular Biology,2020,2172:27-38.
[21] GUO J J,LIU C,WANG P,et al.The Aborted Microspores (AMS)-like gene is required for anther and microspore development in Pepper (Capsicum annuum L.) [J].International Journal of Molecular Sciences,2018,19(5):1341-1344.
[22] PIRES N,DOLAN L.Origin and diversification of basic-helix-loop-helix proteins in plants [J].Molecular Biology and Evolution,2010,27(4):862-874.
[23] ZHANG Z X Z R,WANG S C,ZHANG D,et al.Identification of Malus halliana R2R3-MYB gene family under iron deficiency stress and functional characteristics of MhR2R3-MYB4 in Arabidopsis thaliana [J].Plant Biology,2022,24(2):344-355.
[24] SONG C W L W,LI Y H,NIE P,et al.Alleviating the effect of quinoa and the underlying mechanism on hepatic steatosis in high-fat diet-fed rats [J].Nutrition & Metabolism,2021,18(1):106-122.
[25] 扶艳艳,刘正杰,林 春,等.芦笋性别决定基因 AoTDF1的表达及其编码蛋白与AoAMS间的互作分析 [J].植物生理学报,2021,2021(3):643-654.
FU Y Y,LIU ZH J,LIN CH,et al. Analysis of AoTDF1 expression in Asparagus officinalis and its coding protein interaction with AoAMS [J].Plant Physiology Journal,2021,57(3):643-654.
[26] KIM D P J,PARK C,BENNETT C,et al. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype.[J].Nature Biotechnology,2019,37:907-915.
[27] THORVALDSDOTTIR H R J,MESIROV J P.Integrative genomics viewer (IGV):high-performance genomics data visualization and exploration [J].Brief Bioinform,2013,14(2):178-192.
[28] 张高原,丁 谦,魏兵強.西瓜 PIP5K基因家族鉴定及其在雄性不育花蕾中的表达分析[J].西北农业学报,2021, 30(6):883-893.
ZHANG G Y,DING Q,WEI B Q.Identification of PIP5K gene family in watermelon (Citrullus lanatus) and its expression analysis in male sterile floral buds [J].Acta Agriculturae Boreali-occidentalis Sinica,2021,30(6):883-893.
[29] LI W C,JIANG L,ZHOU S R,et al.Fine mapping of pss1,a pollen semi-sterile gene in rice (Oryza sativa L.) [J].Theoretical and Applied Genetics,2007,114:939-946.
[30] PAWLOWKI W P,WANG C J R,GOLUBOVSKAYA I N,et al.Maize AMEIOTIC1 is essential for multiple early meiotic processes and likely required for the initiation of meiosis [J].Proceedings of the National Academy of Sciences,2009,106(9):3603-3608.
[31] L B B,SUN W W,ZHANG S P,et al.HrpN Ea-induced deterrent effect on phloem feeding of the green peach aphid Myzus persicae requires AtGSL5 and AtMYB44 genes in Arabidopsis thaliana [J].Journal of Biosciences,2011,36(1):123-137.
[32] JUNG K H,HAN M J,LEE Y S,et al. Rice Undeveloped Tapetum1 is a major regulator of early tapetum development [J].The Plant Cell Online,2005,17(10):2705-2722,
[33] LI H,PINOT F,SAUVEPLANE V,et al.Cytochrome P450 family member CYP704B2 Catalyzes the (omegahydroxylation of fatty acids and is required for anther cutin biosynthesis and pollen exine formation in rice [J].Plant Cell,2010,22(1):173-190.
[34] ZHENG X,HE L,LIU Y,et al.A study of male fertility control in medicago truncatula uncovers an evolutionarily conserved recruitment of two tapetal bhlh subfamilies in plant sexual reproduction [J].New Phytologist,2020,228(3):1115-1133.
[35] XUAN MA B F,HONG MA.AMS-dependent and independent regulation of anther transcriptome and comparison with those affected by other Arabidopsis anther genes [J].BMC Plant Biology,2012,12(1):23-43.
[36] HEITKAM T,WEBER B,WALTER I,et al.Satellite DNA landscapes after allotetraploidization of quinoa (Chenopodium quinoa) reveal unique A and B subgenomes [J].Plant Journal,2020,103(1):32-52.
[37] XU Y,LACUONE S,LI S F,et al.MYB80 homologues in Arabidopsis,cotton and Brassica:regulation and functional conservation in tapetal and pollen development [J].BMC Plant Biology,2014,14:278-292.
Cloning and Functional Analysis of CqAMSlike in Chenopodium quinoa
Abstract To investigate the molecular mechanism of pollen development of quinoa (Chenopodium quinoa,Willd.),CqAMS candidate genes were predicted and their coding sequences were cloned based on genomic and transcriptomic data,which included homologous alignment and RT-qPCR confirmation analysis of expression patter genes,followed by virus-indiated gene silencing (VIGS) and bimolecular fluorescence complementation (BiFC) methods to perform functional validation of the candidate genes.The results showed that quinoa had two copies of AMS homologs ,named as CqAMSlike1 and CqAMSlike2.The CDS of CqAMSlike1 has 1 929 bp,while that of CqAMSlike2 had 1 920 bp.Both genes were expressed in flowers at different developing stages of panicles,however,compared with the expression pattern of CqAMSlike2,the expression pattern of CqAMSlike1 was more consistent with the expression pattern of AMS gene in A.thaliana,thus the CqAMSlike1 was chosen for further function verification.Compared with the wild type,CqAMSlike1 is silenced in quinoa plants by VIGS based on tobacco rattle virus (TRV),showed phenotypes such as partial shortened filaments,shriveled,indehiscent stamens,abortive pollens,fewer seeds and increased seed shriveling rate.Further BiFC results showed that CqAMSlike1 could interact with itself,CqTDF1 and CqMYB80,indicating that CqAMS-like1 could form homo or heteropolymers to regulate the genes expression for controlling the development of quinoa anther tapetum.The study laid a basis for elucidating the molecular mechanism of pollen tapetum development,and creating male sterile quinoa germplasm.
Key words Chenopodiun quinoa; AMS; Virus-indiated gene silence(VIGS); Pollen devolpment; Protein interation

