空间转录组技术的研究进展及其在皮肤科的应用
林世颖,江建,陈宏翔,2
随着第二代高通量测序技术(next-generation sequencing technology, NGS)的兴起,引发了一场组学革命,转录组学、蛋白质组学、代谢组学等各种组学技术相继出现。尤其是单细胞转录组学(singlecell RNA-sequencing, scRNA-seq),作为一种革命性的技术,实现对单个细胞的转录水平进行测序,在肿瘤异质性、细胞类型的鉴定和谱系追踪上提供了新的见解[1]。然而细胞间相互作用具有时间和空间上的差异性,因此空间信息的维持对于理解发育生物学、神经生物学、肿瘤生物学等至关重要[2]。scRNA-seq在样本处理时需要对细胞进行消化分离,导致细胞空间位置信息的丢失,无法提供基因表达的空间模式。空间转录组技术(spatial transcriptome sequencing)的发展弥补了这一缺陷,它能直接从完整组织中量化RNA表达,达成转录本信息的空间映射[3]。目前,空间转录组在多种疾病研究中得到应用。本文将对空间转录组测序技术的发展、原理及其在皮肤科学研究的应用进行综述。
1 空间转录组技术的发展
空间分辨转录组学主要分为三大类[4]:①基于激光捕获显微切割技术(lasercapturemicrodissection, LCM)的方法;②基于成像的方法,包括基于原位杂交(insituhybridization, ISH)的方法和基于原位测序(insitusequencing, ISS)的方法;③基于原位捕获(insitucapturing, ISC)的方法。
1.1基于激光捕获显微切割的方法 LCM是在显微镜下,使用紫外线或红外线从组织切片中单独分离出单个细胞或感兴趣的区域,同时保留细胞的结构和空间信息,随后,可以通过直接RNA-seq或预先标记空间条形码的多路复用程序获取细胞信息[5-6](图1)。2016年,Kruse等[7]介绍了Tomo-seq(RNA tomography sequencing, Tomo-seq),一种利用冷冻切片机沿着感兴趣的轴对胚胎或组织进行切片,然后在每个切片上进行RNA-seq的技术。该研究团队先后在斑马鱼胚胎和受伤后的成年斑马鱼心脏组织上验证了Tomo-seq具有高空间分辨率和全基因组覆盖率。2017年,Chen等[8]将LCM与scRNA-seq结合展示了地域位置测序(geographical position sequencing, Geo-seq),Geo-seq的一个独特之处是将各个区域的标志基因作为编码,并通过编码检索单个细胞的位置。

图1 激光捕获显微切割原理
1.2基于成像的方法
1.2.1ISH ISH的基本原理是使用标记的探针与感兴趣的RNA互补杂交,这种方法使得研究人员能够在RNA原始位置进行定性、定量和可视化分析[9]。这种标记的思路可追溯到1969年,Gall等[10]早在那时就已利用放射性同位素标记的核酸探针可视化核糖体DNA。1982年,Singer等[11]尝试使用能够与核酸序列相结合的荧光核酸探针,实现了荧光原位杂交(fluorescenceinsituhybridization, FISH),该技术提高了检测分辨率、灵敏度和特异性。在FISH的基础上,Femino等[12]将FISH和数字成像显微镜结合以检测单个RNA分子,并称为单分子荧光原位杂交(single-molecule fluorescenceinsituhybridization, smFISH)。
然而,由于smFISH需要使用高分辨显微镜,受到显微镜下的荧光光谱重叠限制,检测的靶RNA数量有限。Lubeck等[13]在2014年开发了seqFISH,通过连续多轮杂交、成像和探针剥离,可以覆盖单个细胞的整个转录组。2019年,Eng等[14]在seqFISH的基础上进行改进,并将其命名为seqFISH+,与seqFISH 相比,该技术不仅将成像效率提高了8倍,还解决了光学拥挤的问题。尽管seqFISH可检测的基因数增多,但是其在应用过程中,存在价格昂贵、费时、检测误差高、显微成像时分子颜色堆叠等问题。为了克服误差,多重防错荧光原位杂交(multiplexed error-robust fluorescenceinsituhybridization, MERFISH)使用二进制条形码策略,提高了检测的质量和数量[15](图2)。但是,MERFISH仍存在杂交荧光背景高的问题,Goh等[16]提出split-FISH,采用分裂探针的设计,降低了脱靶背景荧光和假阳性。
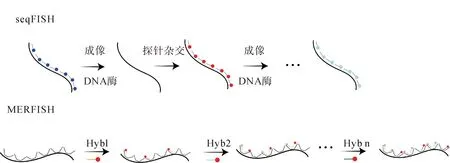
图2 顺序荧光原位杂交(seq-FISH)和多重抗错误荧光原位杂交(MERFISH)原理
1.2.2ISS ISS是另一种基于成像的空间转录测序技术。有研究[17-18]基于锁式探针、滚环扩增(rollingcircleamplification, RCA)原理开发了第一代ISS技术,该方法首先将mRNA在原位逆转录成cDNA,随后锁式探针可以通过缺口靶向或条形码靶向的方式与cDNA结合,探针末端被填补形成一个DNA环,经过RCA形成滚环产物(rolling-circleproduce, RCP),之后对RCP进行测序,重复进行连接、成像和洗涤步骤,经过多轮测序循环读取条形码序列,对这些序列进行解码最终能得到检测的mRNA种类(图3)。通过这种方法能检测到很小的序列变异,如剪切位点和突变,易于区分高度相似的序列[19]。

图3 原位测序原理
但ISS所用到的探针存在特异性差的问题,并且该技术难以扩展到全转录组,2015年,Lee等[20]开发了荧光原位测序(fluorescent in situ sequencing, FISSEQ),使用单链DNA环化酶将原位逆转录获取的cDNA进行环化,通过RCA信号放大后进行测序,由于其不需要设计靶基因,实现了非靶向RNA原位分析。
1.3基于原位捕获的方法 ISC是指通过在原位阵列上非靶向地捕获组织切片中的转录本来获取空间位置信息,然后在异位利用NGS测序[21]。2016年,StÅhl等[22]开发了一项新技术,并将其命名为空间转录组技术(spatialtranscriptomics,ST),在微阵列载玻片的每个微孔都固定上带有空间位置信息的条形码、唯一分子标识符(unique molecular identifier,UMI)、poly-dT的标签,微孔直径为100 μm,中心距为200 μm。然后将成年小鼠嗅球组织切片置于载玻片上,组织透化后,带有poly-A的mRNA被微孔内的寡核苷酸捕获,随后在原位进行逆转录得到cDNA,最后,使用基于NGS的RNA-seq获得基因序列,并通过解码位置信息标签,与组织图像联合,从而实现可视化分析。这项技术于2018年被10×Genomics收购,该公司发布的Visium就是ST的改进版本,其斑点的直径减小到了55 μm,中心距为100 μm,每个微孔能捕获超过5 000个转录本,实现了更高的分辨率和灵敏度(图4)。
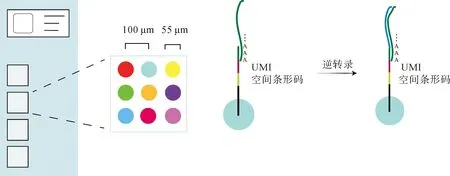
图4 10×Visium原位阵列捕获原理
然而,上述使用条形码寡核苷酸捕获阵列进行空间转录组测序的技术由于捕获点物理尺寸的限制,无法达到单细胞甚至亚细胞空间分辨率的要求。因此,科学家们开发了一种基于磁珠的、在高空间分辨率下测量全基因组表达的Slide-seq[23-24]。2021年,Stickels等[25]又在Slide-seq的基础上,报告了Slide-seq V2技术,对文库生成、磁珠合成方面进行了改进,大大提高了转录本的检测效率。除了磁珠,还有一种通过微流体通道生成空间阵列的技术,称为DBiT-seq,具有高空间分辨率(10 μm)、出色的数据质量和高基因组覆盖率[26]。
尽管基于磁珠和微流体的空间转录组技术的空间映射都接近单细胞级别,但它们仍不能直接解析单细胞。为了解决这个问题,Cho等[27]于2021年提出Seq-Scope,与之前的空间转录组技术相比,Seq-Scope不仅达到了0.5~0.8 μm的分辨率,还具有出色的mRNA捕获效率。近期,还报道了一种sci-Space技术,用于定位细胞核的mRNA[28]。
总之,空间转录组技术多种多样,并且仍在不断发展中,在具体的应用场景下应根据不同方法的利弊进行选择(表1)。
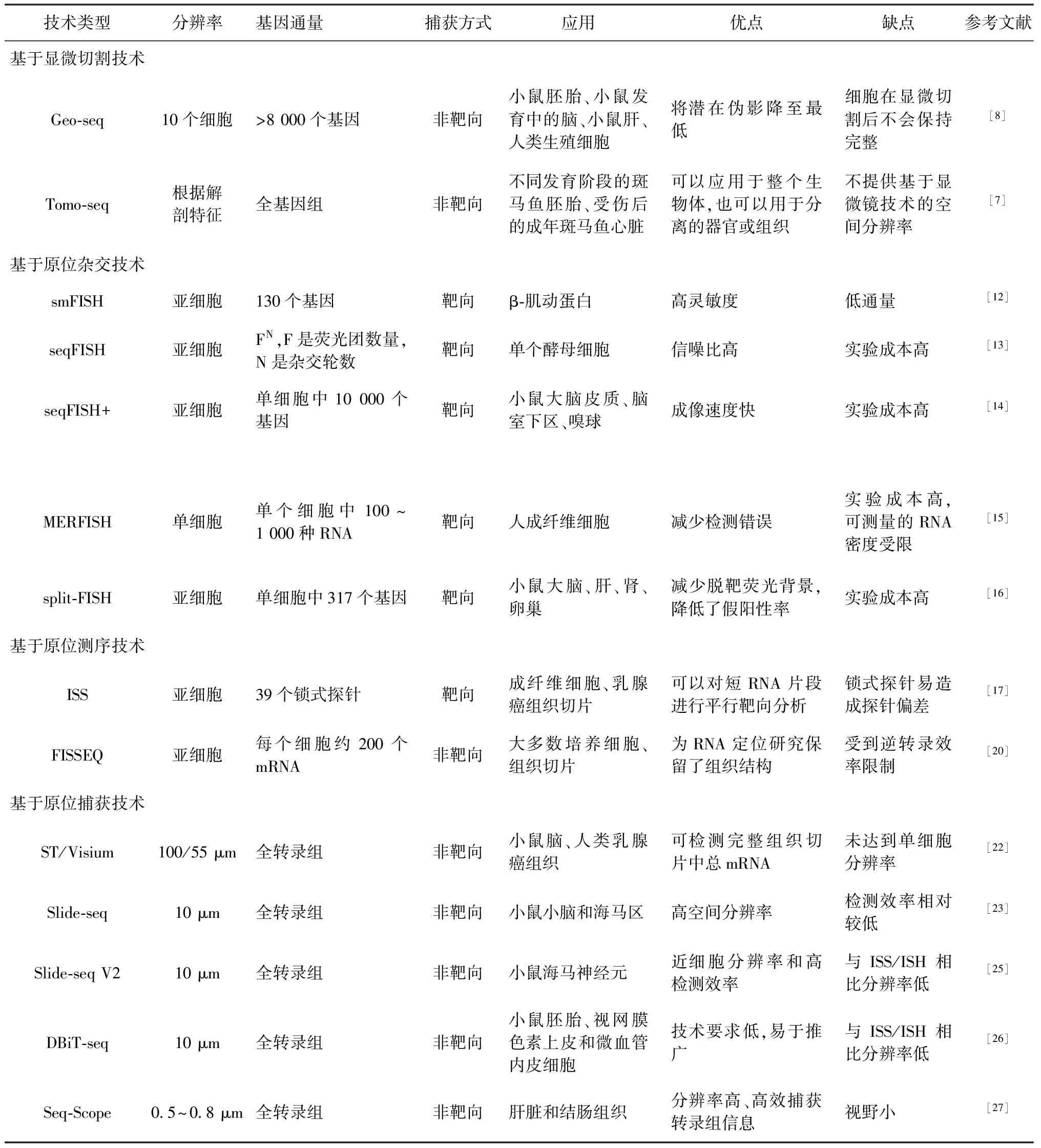
表1 空间转录组技术的对比
2 空间转录组技术与其他技术联合
在实际应用中,空间转录组技术通常与其他组学技术相结合,常见的如与scRNA-seq、蛋白质组学等结合。Lohoff等[29]在表征整个小鼠胚胎细胞类型时,将seqFISH和单细胞转录组图谱相结合,对未能通过seqFISH分析的基因的空间表达进行估算,并发现了在scRNA-seq数据中不明显的细胞分化轴。近年来,将蛋白质组学与空间转录组技术结合也得到发展和应用。2018年,Schulz等[30]提出了一种可以同时对蛋白质、蛋白质磷酸化和转录本进行空间测量的成像型质谱流式细胞术(imaging mass cytometry, IMC)。然而由于抗体标记数量有限,限制了IMC的应用,2020年,Piehowski等[31]对此提出了改进措施,采用了基于芯片的纳米微量液滴蛋白处理系统(Nanodroplet Processing in One potfor Trace Samples, nanoPOTS),与LCM结合,实现了以100 μm分辨率绘制大于2 000种蛋白质的定量图谱。除了与其他组学技术结合,现在还提出了将拟时序分析应用到空间转录组学中,从而对组织水平发展进行动态观察[23]。
3 空间转录组技术在皮肤病学的应用
目前空间转录组技术已在神经科学、胚胎发育、肿瘤微环境等方面得到广泛应用[32-34],在皮肤病学中,主要用于皮肤肿瘤与炎症方面的研究,如黑素瘤、基底细胞癌、鳞状细胞癌、瘢痕疙瘩、皮肤炎症等。
3.1黑素瘤 2018年,Thrane等[35]从4例Ⅲc期黑素瘤患者的淋巴结活检中获得了2 200个组织区域的空间转录组数据,并对4种转移瘤进行了主成分分析、因子分析和降维分析,显示了它们之间广泛的异质性,这是空间转录组技术首次应用到黑素瘤中。
2022年,Nirmal等[36]结合了高复杂成像(high-plex imaging)、3D高分辨率显微镜和空间转录组技术对原发性黑素瘤的免疫逃避和免疫编辑机制进行研究,发现随着癌前状态、原位黑素瘤和侵袭性肿瘤的演化过程,由肿瘤、免疫和基质细胞形成的免疫抑制环境发生着显著的变化,涉及到促进MHC-Ⅱ、代谢酶IDO1表达的局部细胞因子信号和PD1-PDL1介导的细胞间的相互作用。同年,Biermann等[37]开展的一项研究,通过应用单细胞转录组、SlideSeqV2技术,比较了黑素瘤脑转移和颅外转移的细胞形态、染色体形态、巨噬细胞的组成和表达差异等,发现黑素瘤脑转移的特征包括癌细胞染色体不稳定性增加、神经元样分化、CD8+T细胞功能障碍、髓系细胞比例增加、巨噬细胞表现出促肿瘤表型等,全面地描绘了黑素瘤脑转移的单细胞图谱,为进一步的机制研究奠定了基础。Plaschka等[38]对人黑素瘤免疫浸润物的空间和转录组联合分析,证明了黑素瘤细胞中的ZEB1转录因子直接抑制T细胞趋化因子,包括CXCL10、CCL3、CCL4的分泌,破坏CD8+T细胞募集,促进了黑素瘤的免疫逃逸。他们的研究成果为黑素瘤免疫治疗提供了新的可能的靶点。
3.2非黑素瘤皮肤癌 为了研究肿瘤-间质界面在基底细胞癌侵袭过程中的作用,Yerly等[39]首先对5例临床收集到的浸润性基底细胞癌的新鲜活检组织进行scRNA-seq,将cluster C0鉴定为肿瘤细胞,cluster C1、C2、C3分别代表基底、棘层/颗粒层和毛囊角质形成细胞。随后,使用数字空间分析技术划分了24个感兴趣区,将每个感兴趣区分为肿瘤感兴趣区和间质感兴趣区,通过比较浸润型和结节型基底细胞癌在肿瘤和间质中的差异表达基因(differentially expressed genes, DEG),利用DEG信号将构成肿瘤-间质交界面的细胞亚型鉴定为成纤维细胞。又使用了拟时序分析推断出驱动肿瘤细胞发展的各种基因,其中优先表达的基因INHBA主要产生ActivinA,通过旁分泌形式调节控制肿瘤细胞和侵袭部位中的肿瘤相关成纤维细胞的相互作用。
鳞状细胞癌也是一种常见的非黑素瘤皮肤癌。2020年,Ji等[40]整合了人鳞状细胞癌组织的单细胞和空间数据,在肿瘤组织中发现了肿瘤特有的角质形成细胞(tumor-specific keratinocyte,TSK),TSK细胞位于纤维血管壁龛,表达与上皮-间质转化(epithelial-mesenchymal transition, EMT)相关的基因,在细胞间通讯中发挥着枢纽作用。
3.3瘢痕疙瘩 Shim等[41]运用单细胞和空间转录的综合分析揭示瘢痕疙瘩的潜在病理机制。通过对比分析瘢痕疙瘩组织和正常组织的单细胞RNA测序数据,显示瘢痕疙瘩内有成纤维细胞、内皮细胞和肌成纤维细胞,空间转录组的结果显示与疾病相关的成纤维细胞集中在瘢痕疙瘩较深的区域,主要位于内皮周围,多重免疫荧光共定位表明瘢痕疙瘩内皮细胞发生间充质激活,ST对配体—受体定位的可视化也为成纤维细胞和内皮细胞之间互相串扰提供了证据支持。这些共同的结果表明纤维血管通讯和内皮间充质激活可能参与了瘢痕疙瘩的发病机制。
3.4皮肤损伤愈合 为了研究伤口再上皮化的分子机制,Konieczny等[42]运用了多模式单细胞和空间转录分析及功能研究,表明在受损上皮细胞中,RORγt+细胞衍生的IL-17A是激活HIF1的必要充分条件。首先,作者通过测序对转录本和表位进行细胞索引(cellular indexing of transcriptomes and epitopes by sequencing, CITE-seq)确定了皮肤驻留的RORγt+细胞直接促进伤口再上皮化。随后,作者绘制了伤口组织边缘的ST图谱,对ST结果进行多模态交叉分析,并经过实验验证RORγt+细胞分泌的IL-17A与上皮细胞IL-17RC结合,通过ERK 1/2-AKT-mTOR激活HIF1α,驱动伤口上皮细胞的糖酵解,从而参与伤口的愈合。
4 前景与展望
近年来,空间转录测序技术本身的不断改进以及与其他技术的结合,使它成为一个协助我们理解细胞表达和行为模式、识别生物标志物、发现新的治疗靶标的有力手段。尽管目前各种空间转录测序技术仍然存在需要改进的空间,如技术难度高、成本昂贵,主流的Visium技术还未达到单细胞水平等,但是基于它在阐明正常皮肤和疾病状态下的细胞类型以及细胞间相互作用等方面的优势,随着空间转录测序技术在皮肤疾病的广泛应用,其在皮肤病的早期诊断、治疗和预后上将取得更大的进展。

