纳米孔基因测序技术在HLA基因分型中的应用
朱孟琳,何燕琴,谢水莲,杨 影,万绍贵
(1. 赣南医科大学基础医学院;2. 赣州市中心血站检验科;3. 赣南医科大学基础医学院分子病理中心,江西 赣州 341000)
主要组织相容性复合物(Major histocompatibility complex,MHC)是一组能编码代表个体特异性抗原的基因群,其中编码人类基因的MHC又称为人类白细胞抗原(Human leukocyte antigen,HLA)复合体。人类白细胞抗原系统在免疫功能调节系统中,HLA分子与胸腺中T 细胞相互识别,来确定是否为自身分子[1]。因此准确的HLA分型在移植、输血、自身免疫疾病的临床诊断等方面均有重要作用[2]。
纳米孔基因测序技术是利用纳米级蛋白孔作为生物传感器,通过电流的变化识别DNA 或RNA的碱基序列[3]。该方法可以直接快速实时对DNA或者RNA碱基序列进行读取,为研究HLA分型提供了很大的助力。本文主要综述纳米孔基因测序技术在HLA基因分型中的应用。
1 HLA基因结构与功能
HLA 复合体位于第6 号染色体短臂2 区1 带3亚带(6p21.3)区域内,该区域是人类基因组中多态性最强的区域。截至2022年7月12日,世界卫生组织(World health organization,WHO)认证的HLA 序列数据储存库(IPD-IMGT/HLA 数据库)已经收录了35 220 个HLA 等位基因。按照HLA 复合体在染色体上距离着丝粒的远近,可将HLA分为3个区,即Ⅰ类基因区、Ⅱ类基因区、Ⅲ类基因区(图1)[4]。经典HLA 基因编码的产物直接参与机体免疫反应的抗原提呈过程。HLA Ⅰ类抗原分子由两条多肽链组成,即HLA 基因编码的α 链和非HLA 基因编码的β链,组成4 个结构区,其中α1 和α2 组成结构区形成HLA Ⅰ类分子的肽结合槽;同样,HLA Ⅱ类分子是由α1和β1形成肽结合槽,该区域是抗原识别区域,决定HLA 抗原特异性的部位,也是不同基因型HLA分子的结构差异[5]。故HLA 基因型的不同决定其HLA 抗原特异性,HLA 的抗原表位被认为是发生移植免疫反应的关键分子部位,HLA 抗原表位错配数越高,移植后发生免疫排斥反应的可能性越大[6]。因此,在进行器官或骨髓移植之前,对供体和受体的HLA 分子进行精准的配型极其重要,以防术后发生严重的移植免疫排斥。
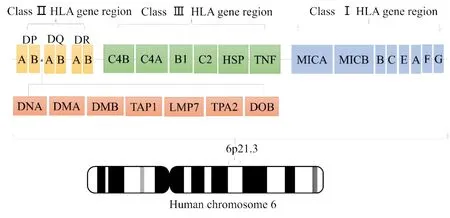
图1 HLA复合体结构示意图
2 HLA分型方法
随着测序技术的不断发展和进步,DNA 测序方法不仅可以带来更准确的HLA 基因分型,而且可以发现新的等位基因。为了更好分辨新的等位基因,国际上建立统一的命名规则(图2)。将不同HLA等位基因分为4 个数字区域和修饰后缀(N/S/L 等)进行区分,在进行HLA 分型中识别到第一个数字区域为低分辨率分型(Low resolution typing),识别到前两个数字区域以上为高分辨率分型(High resolution typing),识别到所有数字区域为等位基因水平分型(Allele level typing)[6-8]。2021 年8 月我国国家卫生健康委员会发布的《人类白细胞抗原基因分型检测系统技术标准》确定了聚合酶链反应——直接测序法(Polymerase chain reaction sequence-based typing,PCR-SBT)为HLA 分型方法的金标准。PCR-SBT是针对HLA Ⅰ类基因和HLA Ⅱ类基因的各外显子进行PCR 扩增,后用Sanger 测序方法(第一代测序方法)对扩增序列进行测序,该方法是直接在DNA水平对HLA 进行分型,相比于传统血清学方法对HLA 分型更为准确。但由于HLA 基因高度的序列相似性以及等位基因之间的大量多态性和镶嵌结构,PCR-SBT 方法在HLA 分型中会出现模棱两可等位基因组合等问题。同样下一代测序技术(Next generation sequencing, NGS)也在HLA 基因分型中研究广泛,但受限于测序读长较短和序列拼接困难的缺陷等,HLA 的精准分型仍存在挑战[9]。而作为第三代测序技术代表的纳米孔基因测序技术可能可以解决这些问题。
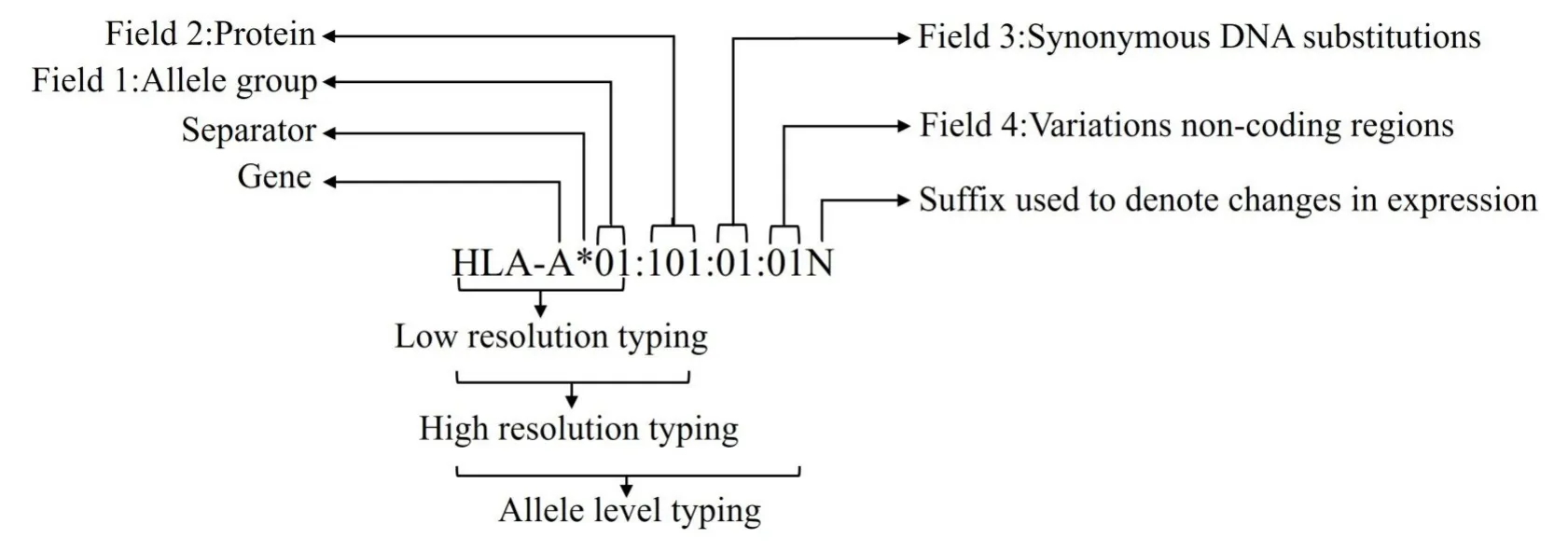
图2 HLA等位基因的命名
纳米孔基因测序技术核心是纳米级的蛋白质孔或“纳米孔”,设计来源于α-溶血素[10]。在充满离子的溶液中,给纳米孔两侧施加恒定电压,产生通过纳米孔的离子电流,单链DNA 或RNA 分子通过马达蛋白的驱动,穿过纳米孔,而其间各个碱基对离子电流的阻碍可形成不同的电流信号,后将不同的电流信号通过计算算法解码得出碱基序列[3]。纳米孔基因测序技术在对于HLA 应用方面,不仅有助于发现新等位基因,如WANG Y H 等[3]利用纳米孔基因测序技术发现一种新的HLA-A*11:335 等位基因、SHAW B E 等[11]在DPB1 测序中发现新突变(DPB1*02:01:02 + DPB1*04:01:01:13);而且可以对HLA 基因进行快速分型,极大提高器官移植效率。
3 纳米孔基因测序技术在HLA 基因分型的应用
除三代测序以外,其余测序均以短测序读长为主,而HLA 基因区域具有高度重复性,难以进行组装和易遗漏位置信息[12],纳米孔基因测序技术有着长读长的特点,在用于HLA 全长基因读取可以提供更为完整的基因信息。自2014 年牛津纳米孔技术公司(Oxford nanopore technologies, ONT)研发出的第一台纳米孔测序仪MinION 后,纳米孔测序技术就广泛应用于各种基础研究,AMMAR R 等[13]将纳米孔检测技术应用于HLA 基因分型,检测HLA-A、HLA-B 和CYP2D6 显示CYP2D6(单次最佳命中比对)平均覆盖率为1 236.4×,HLA-A(多次命中比对)为785.5×,HLA-B(多次命中比对)为1 416.3×,表明纳米孔基因测序在HLA 分型中具有很大的潜力。随着该测序技术的发展,2018 年LIU C 等[14]利用MinION 对HLA Ⅰ类基因进行测序并开发新的算法工具Athlon,HLA 分型准确率在低分辨率和高分辨率下皆为100%,促进了HLA 分型的发展和完善,提高HLA 分型的通量和准确性。到目前为止,2020年1月发布的R10.3测序芯片也被应用于HLA 基因分型检测,所测样本有102 个,样本检测有HLA-A、HLA-B、HLA-C、HLA-DPA1、HLA-DPB1、HLADOA1、HLA-DRB1、HLA-DRB3/4/5 位点共1 836 个等位基因(918 个基因型),原始读数准确率的中位数可达93.7%,相较于R9.4芯片其准确率提高6%,越来越多的研究表明,纳米孔基因测序技术可应用于HLA 分型[15]。实时纳米孔基因测序技术作为第三代测序中的代表,目前被广泛应用于各个生物学研究领域,如全基因组测序、靶向基因组测序、全长转录组测序、宏基因组测序、突变分析、表观遗传学研究等方面。目前主要利用纳米孔基因测序技术同靶向测序方法结合对HLA 基因进行分型,在准备基因组文库时主要采用2 种方法:一种是利用PCR富集目标基因区域;另一种是基于PCR 扩增cDNA样本。前者是对样本进行DNA 提取后直接测序分析,可以减少文库建立的繁琐;后者对样本进行RNA 提取后转录为cDNA 进行测序,该测序可同时对RNA进行表达分析,得到完整的转录本。
3.1 基于PCR 扩增基因组样本的纳米孔基因测序技术在HLA 基因分型中应用 利用PCR 对目标HLA 基因区域进行扩增,将得到的扩增子进行文库制备,将制备好的文库加载到纳米孔测序芯片(Flow cell)上进行测序,后对测序数据进行分析。靶向基因区域主要包括3部分:基因多态性区域、基因外显子区、全长基因区域。采用的靶向技术包括普通PCR、Long-range PCR和多重PCR等。DUKE J L等[16]通过Long-range PCR对HLA-DPB1基因座进行扩增;STOCKTON J D 等[17]通过Long-range PCR 对HLA Ⅰ类和HLA Ⅱ类基因进行扩增;MONTGOMERY M C等[18]通过Long-range PCR 对Ⅰ类HLA 基因中的HLA-A、HLA-B 和HLA-C 基因座进行全长扩增,通过MinION 进行测序皆可以得到高分辨率的HLA 基因分型。此外,HLA 系统在人体内基因组中呈现高度多态性,其遗传基础和主要表现为复等位基因,而纳米孔检测技术的长读长可以简化等位基因的定相分析,如STOCKTON J D 等[17]利用单样本测序获得HLA纯合子和重建来自父母本的单倍型。
3.2 基于PCR 扩增cDNA 样本的纳米孔基因测序技术在HLA 基因分型中的应用 PCR-cDNA 测序是ONT 基于cDNA 测序开发的一种测序方法,仅需1 ng RNA 作为起始材料,将RNA 转录为目标基因区域或全长cDNA 进行PCR 扩增,后将扩增的PCR产物进行测序文库制备(主要使用文库试剂盒为SQK-PCS108 试剂盒),将制备好的文库加载到纳米孔基因测序芯片进行测序,从而可得到基于cDNA的测序数据。MONTGOMERY M C 等[18]收集12 个健康供体外周血中的单核细胞(Peripheral blood mononuclear cell,PBMC),提取该细胞中的mRNA,后将mRNA转录为cDNA文库并加载到MinION芯片上进行测序,将其产生的数据与现有数据库中HLA-A、HLA-B、HLA-C的2号和3号外显子的参考序列和人类基因组hg38进行比对,结果表明HLA-B的相对表达量较HLA-A、HLA-C 高。JOHANSSON T 等[19]针对HLA 12 个基因座的cDNA 进行扩增,将扩增的基因文库进行MinION 测序。上述实验的HLA 基因分型均可达到高分辨率分型,表明基于PCR 扩增cDNA 样本的纳米孔基因测序技术可以同时进行HLA 的基因分型和HLA 表达。在实体器官移植中HLA 的表达会影响供受体的相容性[18],如果通过该项技术对供受体进行评估,可以更准确地评估患者接受该项治疗的风险,但目前移植中心未将此项纳入供受体评估。纳米孔基因测序技术相较于NGS突出的优点是长读长,可以检测出NGS 未检测到的剪接异构体位点。OKA M 等[20]利用该测序技术建立非小细胞肺癌细胞系的全长转录本,并从中鉴定出新型和潜在的新抗原。
4 纳米孔基因测序技术在临床上HLA 基因分型中的应用
4.1 纳米孔基因测序技术鉴定HLA分型在器官移植方面的应用 HLA 分布在所有有核细胞上,是免疫识别和免疫监视的重要效应分子,是器官移植成功率的关键影响因素。而供、受者间的HLA 基因型差异是引发急性移植排斥反应的主要因素,因此临床上需要检测供受体高分辨率的HLA-A、HLA-B、HLA-C、HLA-DRB1、HLA-DQB1、HLA-DPB1 是否匹配,特别是HLA-DBP 基因是否匹配对移植后器官存活率有重要影响[21]。器官移植时间越短对于器官在受体内运行的有效性越高,但目前DNA 测序对HLA 基因分型所用时间较长,未能在分配供体前实现基因分型。而纳米孔基因测序技术可实现快速、实时测序。STOCKTON J D 等[17]使用大约8 h进行建库测序,可得到测序深度为500 X 的数据。CORNABY C等[22]使用大约6~7 h便可提供高分辨率HLA 分型和完整的转录本表达。MOSBRUGER T L等[23]分别使用三种不同的PCR 方案进行样品基因扩增,总流程时长约6 h,均能实现高分辨率的HLA基因分型。DE SANTIS D 等[24]开发了一种快速高分辨HLA 分型方法,使用基于NGS的AllTypeTM产品对HLA 12 个基因座进行扩增,总流程时长大约4.5 h,同时利用此新型分型方法和SSO-PCR 分型方法同时进行,对10 例已故供体进行实时分型,证明该新型分型方法相对于SSO-PCR 方法产生数据分辨率更高、测序总流程时间更短。纳米孔测序技术对于HLA 分型时间介于NGS 和PCR-SSO 之间,但却可以获得较PCR-SSO 更多的额外信息和高分辨率分型,使得可在已故供体分配时间内实现分型,并提高分辨率、增加供受体相容性。
4.2 纳米孔基因测序技术鉴定HLA基因分型应用于临床诊断 随着检测技术的不断发展,人们可通过DNA 水平辅助疾病的治疗或探索疾病的发生发展。如痛风性关节炎患者治疗时会实时检测患者是否有携带HLA-B*5801 基因型,评估或检测患者疾病状态[25]。纳米孔基因测序技术可对HLA 基因进行高分辨基因分型,有助于临床了解相关疾病发生发展。MATERN B M 等[26]在研究中分两步进行实验:第一次实验数据可覆盖71个已知的Ⅰ类等位基因组中的70 个,包括除了HLA-B*83 以外的所有HLA Ⅰ类等位基因组;第二次实验,67 个诊断样本同时进行MinION测序和Sanger测序,实验结果证明两者测序结果的一致性达100%。Sanger 测序是目前认为技术成熟的测序技术并广泛应用于临床实践中,而目前纳米孔基因测序技术在HLA 系统中并未用于临床,但该实验证明纳米基因测序技术进行高准确度HLA 基因分型可用于临床常规诊断。不仅如此,ONT 的便携性测序仪MinION 可轻松用于基础实验室,无需使用测序核心设施,其基因组文库建立大多使用PCR 靶向技术,成本较低,可大范围应用于临床。STOCKTON J D 等[17]验证了纳米孔基因测序技术可在资源有限情况下进行HLA 分型诊断相应疾病。
5 总结与展望
纳米孔基因测序技术是一种新兴的技术,在HLA 基因研究中发挥着重要作用。尽管其准确率高达98.9%,但仍存在一些固有缺陷,如个别等位基因覆盖率低,文库制备导致的读长限制和测序深度低等。此外,数据量大、分析复杂以及分析软件缺乏等因素也限制了其应用。
纳米孔基因测序技术在HLA 分型中最常见的基因组文库准备方法是基于PCR 扩增出测序产物,该方法虽价格低廉,但针对HLA 目的基因的引物多和PCR 固有的问题,会导致结果中出现难以分辨的高度重复序列、高GC 难以扩增等问题。CRISPR/Cas9 是一项基因编辑工具,可通过设计特异性crRNA 探针对任意的靶向DNA 片段进行准确切割,而MinION 可直接针对该靶DNA 片段进行测序,基于Cas9的纳米孔基因测序技术无需PCR 扩增,不受CG 含量影响,具有测序深度高等优点,这将会是基于纳米孔基因测序技术的靶向测序在未来HLA分型方面的主攻方向。

