基于全基因组重测序的杏鲍菇菌株遗传多样性与性状分析
林俊彬,严俊杰,韩 星,赵 金,甘 颖,苗人云,冯仁才,李 斐,赖学飞,仝宗军,甘炳成
(1.中国农业科学院都市农业研究所,成都 610213;2.成都农业科技中心,成都 610213;3.金堂县现代农业装备产业园管理委员会,成都 610407;4.四川田岭涧生物科技发展有限公司,成都 610400)
【研究意义】杏鲍菇(Pleurotuseryngii)又名刺芹侧耳,分类学属于真菌界担子菌门伞菌纲伞菌目侧耳科侧耳属[1],是我国工厂化栽培的主要食用菌。2020年全国杏鲍菇工厂化栽培产量2.13×106t,占食用菌总产量的5.26%,略低于金针菇的产量2.28×106t,工厂化生产的食用菌产量排名第二[2]。实际生产中,杏鲍菇从栽培到采收需要大约2个月,菌种类型、菌种质量、培养料配方以及出菇过程中温度、光照、气体组成、空气湿度等环境因子均会影响子实体的性状[3],其中菌种本身的性质是最重要的影响因素之一[4]。通过建立菌株的分子标记与子实体性状之间的联系,可以方便快捷地在菌丝时期对杏鲍菇的性状进行预测,对遗传多样性的分析可为杂交育种的亲本选择提供参考。【前人研究进展】以往对食用菌菌株进行遗传多样性分析多采用RAPD、ISSR等第二代分子标记技术[5]。杏鲍菇在全国范围内栽培广泛,但不同地方的杏鲍菇存在差异,包括子实体的形状、产量、最适环境等,相同的品种在不同环境也会有一定差异,加之菌种没有统一的命名、保藏、管理,所以“同种异名、同名异种”现象较为严重,使杏鲍菇的遗传育种工作进展缓慢[6]。近年来,分子标记技术不断发展,被广泛应用于食用菌种质资源的遗传多样性分析。宿红艳等[7]利用RF-RAPD标记技术对5份杏鲍菇菌株进行遗传学差异分析,并将菌株分为2组,是国内采用分子标记技术对杏鲍菇进行遗传多样性分析最早的报道。杨和川等[6]利用ISSR标记技术对27份杏鲍菇菌株进行分析,将菌株分为4类。随后杨和川等[8]利用RAPD标记技术对10份杏鲍菇菌株进行分析,将菌株分为3个类群。刘晓红等[9]、尚晓冬等[10]、王波等[11]、许峰等[12]、李莹等[13]、陆娜等[14]、李小艳等[15]采用第二代标记技术也对杏鲍菇进行遗传多样性分析。通过PCR和凝胶电泳进行分析的第二代分子标记技术,存在工作量大、标记数量少等缺点,难以实现精度更高的遗传多样性分析。沈颖越等[16]利用SNP标记对20份金针菇菌株进行分析,将菌株分为5个亚群,并将自然栽培和工厂化栽培的菌株分为不同的亚支,证明以SNP为代表的第三代分子标记技术在食用菌资源遗传多样性鉴定上潜力巨大。【本研究切入点】基于测序技术的第三代分子标记技术应用于杏鲍菇种质资源分析的研究还未见报道。【拟解决的关键问题】本研究筛选出适宜工厂化栽培的高产、优质杏鲍菇菌株,为后期的杂交育种寻找合适的亲本。基于重测序技术对20份从全国收集到的杏鲍菇菌株进行全基因组重测序,通过SNP位点进行遗传距离分析、菌丝生长速度以及子实体性状的比较研究,为杏鲍菇种质资源的评价利用、品种鉴定以及杂交育种提供参考。
1 材料与方法
1.1 供试菌株和培养基制备
20份杏鲍菇菌株基本信息见表1。母种培养基(1 L):200 g马铃薯、20 g葡萄糖、20 g琼脂。栽培料配方:木屑38%、玉米芯30%、麸皮20%、豆粕5%、玉米粉5%、碳酸钙1%、石灰1%,含水量65%,pH 6~7。
1.2 试验方法
1.2.1 DNA提取及全基因组测序 收集20份杏鲍菇菌株的菌丝,采用CTAB法进行DNA提取,然后送武汉希望组生物科技有限公司进行文库制备与高通量测序。采用Illumina平台进行测序,对下机得到的二代 Raw data 使用fastq软件进行质控得到 Clean data[17]。
1.2.2 参考基因组序列比对 利用BWA软件将测序获得的过滤后数据比对到参考基因组上[18],比对结果采用经 GATK MarkDuplicates去除重复[19]。

表1 供试杏鲍菇菌株编号及来源
1.2.3 SNP/InDel检测及遗传多样性分析 采用 GATK HaplotypeCaller 进行SNP检测,过滤标准为测序深度过滤(--min-meanDP>5),MAF (Minor allele frequency)>0.05,Call rate>80%,并对检测到的SNP/InDel进行统计。使用MEGA 11软件对SNP数据采用邻位相接法构建系统发育树(Bootstrap method 1000次,其他参数默认),同时计算菌株之间的遗传距离[20]。
1.2.4 供试菌株生长速度及聚落形态试验 将活化好的菌种用5 mm打孔器取接种块接种到新的培养皿,待所有培养皿中接种块新萌发菌丝生长至培养基表面时画起始线,每隔24 h用记号笔在菌丝生长前端划线,连续划线5 d,测量每天的生长距离,计算平均生长速度(mm/d)。
1.2.5 栽培方法 栽培方法参照黄岳磊等[21]、王路朋等[22]的方法,出菇试验采用墙式袋栽的方式在成都汇菇源生物科技有限公司进行,疏蕾后每袋保留1个杏鲍菇子实体。
1.2.6 子实体数量性状测定 数量性状测试方法参照于海龙等[23]的方法,子实体不同部位名称及数量性状测定方法见图1和表2。
1.2.7 子实体质构分析 子实体成熟后,取菌柄中间位置进行质构分析。质构测试参数设定:测试前速度2.00 mm/s,测试速度1.00 mm/s,形变模式,目标30.0%,5.0 s,触发值5 gf。采用p2探头,在上述测定条件下对20份杏鲍菇菌株的子实体硬度、脆度、咀嚼性和胶着性进行测定分析,每个项目5个重复。

表2 子实体数量性状测定方法
1.3 数据分析
使用Execl进行数据统计整理,使用GraphPad Prism 8、TBtools[24]进行数据分析和图表的制作。
2 结果与分析
2.1 20份杏鲍菇资源的测序数据质量及与参考基因组比对结果
由表3可知,原始数据Raw reads数量为26 583 674~36 314 168,过滤后的Clean reads数量为26 428 896~36 108 006,质量值大于30的范围是91.51%~94.04%,说明测序数据质量比较高。将20份材料的重测序数据比对到参考基因组(PleurotuseryngiiJKXB130DA,NCBI Accession: QCWS0000 0000)后,比对率范围是83.27%~89.84%,覆盖度范围是92.43%~99.40%,覆盖深度范围为66.47~89.77,说明20份菌株的测序数据基本可以覆盖参考基因组,比对文件用于后续分析较为可靠。经过GATK检测,20份材料检测到InDel数量范围为98 544~112 652,SNP数量为182 952~492 504,具有较好的多态性。
2.2 遗传多样性分析
如图2~3所示,当遗传距离大于0.08时,20株杏鲍菇菌株被聚为6类,类群Ⅰ包括菌株Pe01,类群Ⅱ包括菌株Pe15、Pe22、Pe19,类群Ⅲ包括菌株Pe16,类群Ⅳ包括菌株Pe02,类群Ⅴ包括菌株Pe03、Pe05、Pe09、Pe10、Pe11、Pe13、Pe20、Pe23、Pe06、Pe08、Pe07、Pe24、Pe14,类群Ⅵ包括菌株Pe25。其中,类群Ⅳ、Ⅴ、Ⅵ的相似度比较高,结合20份菌株的来源,说明我国大部分地区和工厂栽培的杏鲍菇遗传背景比较接近,仅有少数遗传距离稍远的菌株分布在不同地区。
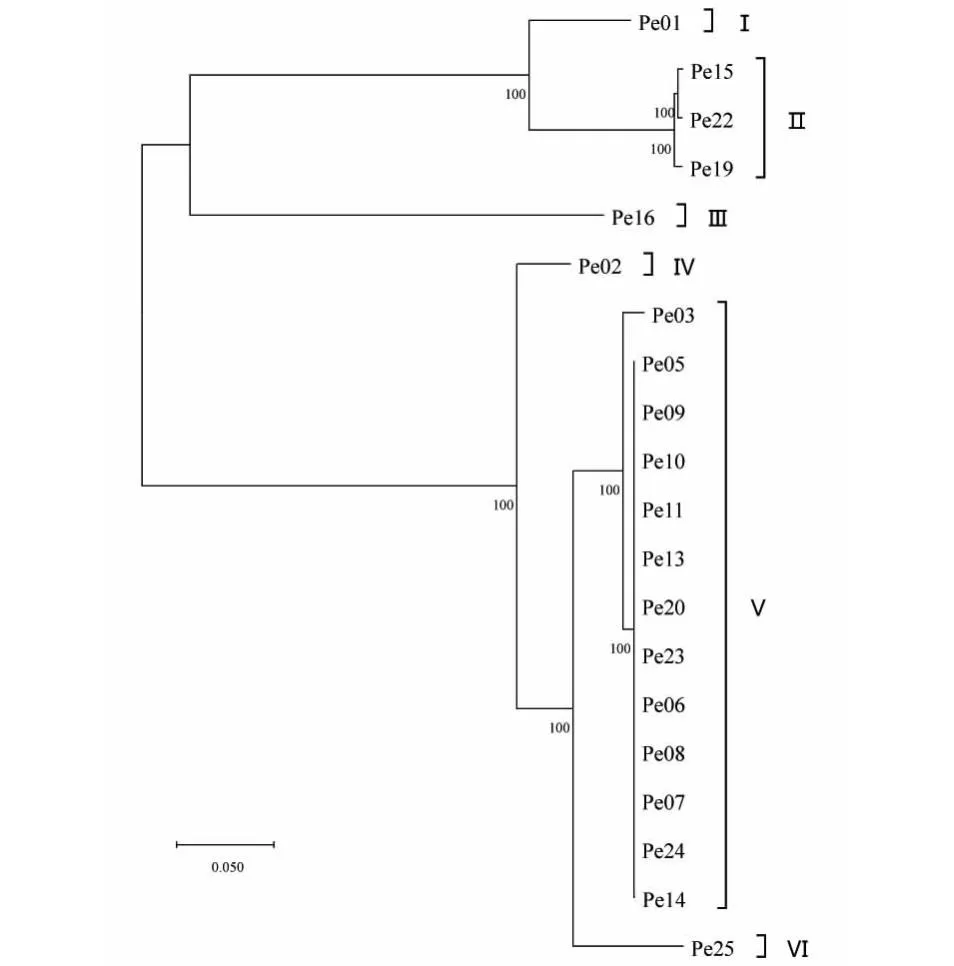
图2 20份杏鲍菇菌株的N-J进化树Fig.2 The neighbor-joining phylogenetic tree for the strains of 20 P.eryngii
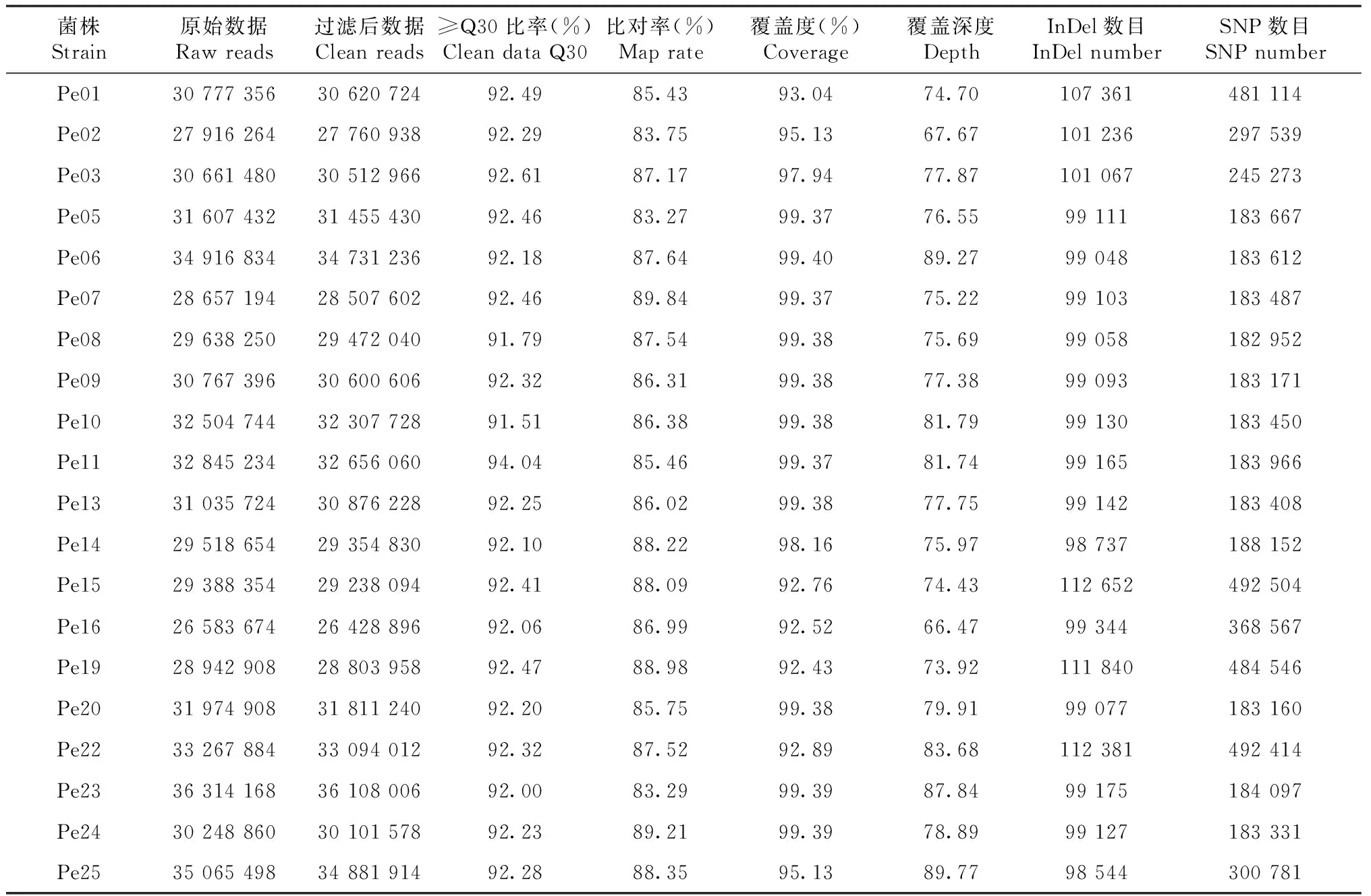
表3 20份杏鲍菇菌株测序数据、比对结果及SNP和InDel统计
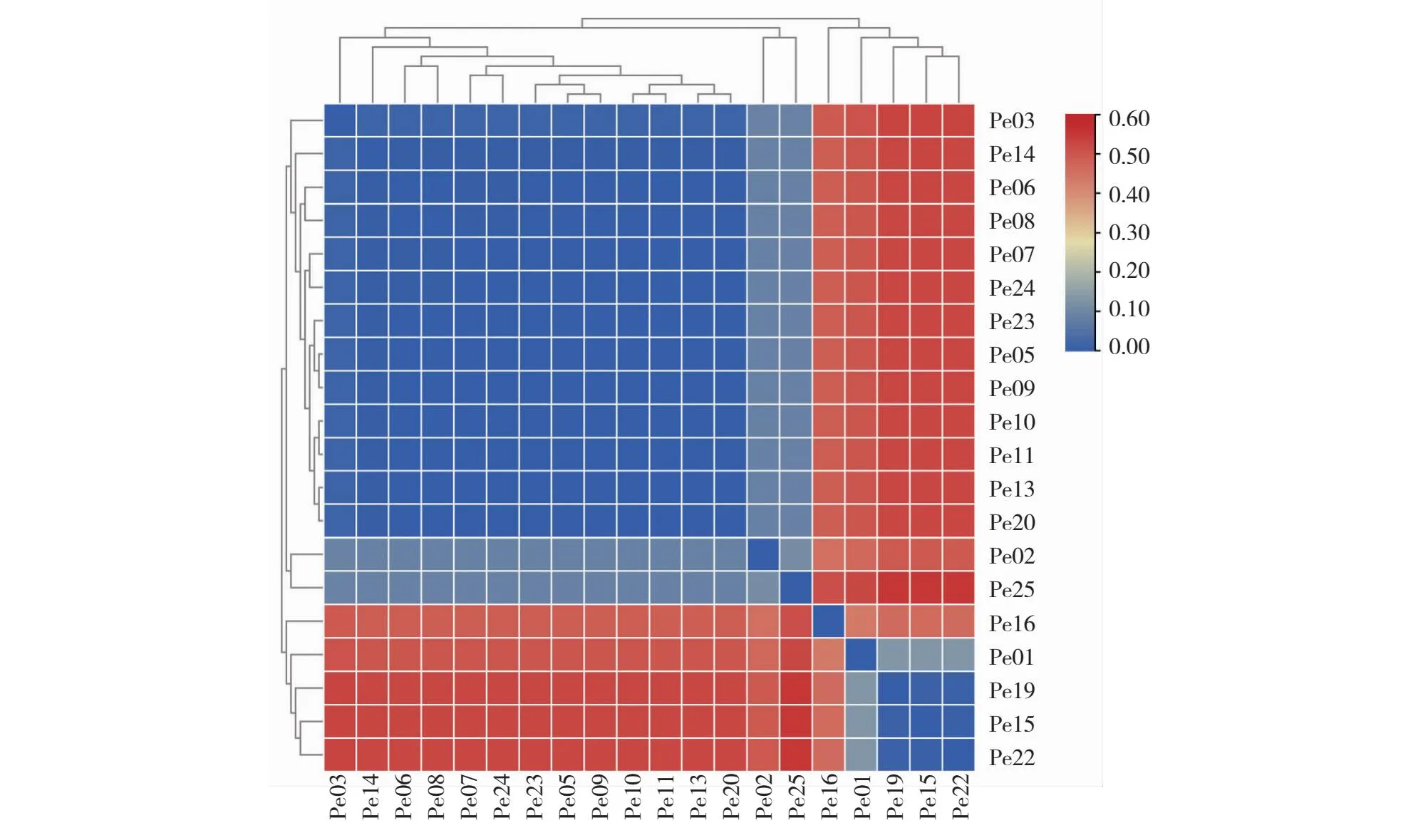
图3 20份杏鲍菇菌株的遗传距离热图Fig.3 The heatmap of genetic distance for the strains of 20 P.eryngii
2.3 菌丝生长速度比较
由图4可知,杏鲍菇菌株Pe01单独作为1个类群,菌丝生长速度明显慢于其他类群;类群Ⅱ3个菌株的菌丝生长速度最快,也相对接近;其他类群菌株的菌丝生长速度虽有差别但不大,与类群分布的关系不是特别密切。

图4 不同杏鲍菇菌株菌丝生长速度比较Fig.4 Comparison of mycelium growth rate among different strains of P.eryngii
2.4 杏鲍菇产量分析
由图5可知,菌株Pe16单独作为1个类群,产量显著低于其他类群;类群Ⅴ是包含菌株最多的类群,其包含的菌株整体产量普遍高于其他类群;其他几个类群包含的菌株产量比类群Ⅴ低,但高于类群Ⅲ。
2.5 不同子实体数量性状比较
由图6可知,结合子实体长度、菌盖直径、菌柄长度、菌柄直径4个指标对6个类群进行综合分析,类群Ⅴ的4个指标均比较突出且菌株间差异较小,只有菌株Pe14的子实体长度稍低;类群Ⅰ、Ⅳ最显著的特征为菌柄长度小、直径大,表现为子实体粗短;类群Ⅱ子实体长度较大,但菌盖大小、菌柄长度和直径均比较小;类群Ⅲ子实体长度较长,但菌柄直径最小,表现为子实体细长;类群Ⅵ几个指标均不突出。
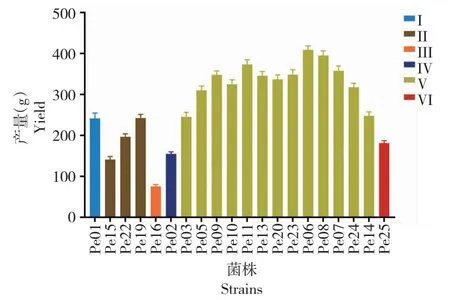
图5 不同杏鲍菇菌株产量比较Fig.5 Comparison of yield among different strains of P.eryngii
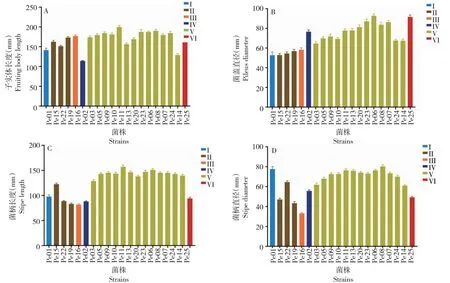
图6 不同杏鲍菇子实体数量性状比较Fig.6 The quantitative characteristics of different strains of P.eryngii
2.6 不同菌株子实体质构特性分析
由图7可知,结合杏鲍菇子实体硬度、脆度、咀嚼性、胶着性4个质构指标对6个类群进行综合分析,类群Ⅴ整体的质构特性虽有差异,但整体表现最为突出;类群Ⅰ的4个指标表现也比较均衡;除此之外,其他几个类群的菌株仅有个别指标表现稍好,大部分指标均较差。
2.7 主成分分析
由图8可知,不同类群的菌株距离均较近,其中,含有菌株最多的类群Ⅴ基本都聚集在一起,只有Pe14相隔较远,但也没有和其他类群聚到一起;类群Ⅱ的3个菌株距离也相对较近;其他几个只含有1个菌株的类群分布在不同位置,其中类群Ⅰ的菌株Pe01与其他所有菌株相隔最远,说明Pe01的性状表现与其他菌株相差较大。
3 讨 论
3.1 遗传多样性分析
相比传统分子标记技术,全基因组重测序技术可以准确、方便地产生大量的SNP和InDel标记,已经在许多动植物上被应用于进化、生物学性状的关联分析。沈颖越等[16]使用重测序技术对22份金针菇菌株进行遗传距离分析,不同类群的SNP和InDel标记的数量均比较接近。施肖堃等[25]对18个双孢蘑菇核心种质进行重测序以及SNP、SV检测,证明国内的As2796系列菌种与国外的U1系列菌种是两类区别明显的品系。沈秀芬等[26]利用InDel标记对国内44份香菇菌株进行分析,结果显示栽培菌株和野生菌株会聚为不同分支,并且将香菇划分为3个类群。基于重测序以及SNP位点对杏鲍菇进行遗传多样性分析的研究尚未见报道,而本研究对20份杏鲍菇菌株进行了全基因组重测序,平均数据高达3.1 Gb,Q30≥91.51%,GC含量正常,基因组覆盖率达92.43%以上,20份杏鲍菇菌株共获得182 952以上的SNP位点,具有丰富的多态性,说明重测序技术结合SNP位点的检测适用于杏鲍菇的遗传多样性分析。在遗传距离大于0.08的条件下,20份杏鲍菇菌株被划分为6个类群,综合其SNP数量进行分析,发现不同类群菌株的SNP存在着较大差异,且同一类群内SNP数量比较接近,说明SNP的多态性水平与遗传多样性紧密相关。本研究证明了采用SNP多样性分析进行菌株间遗传多样性分析的可行性,如果能对更多的菌株进行重测序并对数据进行更加深入的分析,可以对不同菌株的最初来源以及分化途径有更清晰的认识。

图7 不同杏鲍菇子实体质构分析Fig.7 Texture analysis of the fruiting bodies of different strains of P.eryngii

图8 20份杏鲍菇菌株性状的主成分分析Fig.8 Principal component analysis of characteristics of fruiting bodies of different strains of 20 P.eryngii
3.2 子实体性状分析
进行遗传多样性的研究过程中,除了采用SSR、RAPD、SNP等分子标记技术,表型性状也是非常重要的评价指标。物种的表型性状是进行资源收集、评价、利用的基础,其对资源的综合评价以及杂交育种亲本的选择具有重要的参考意义[27]。目前,表型性状研究在植物中的应用较为成熟,在食用菌中也有一定应用[28],在金针菇[29]、大球盖菇[30]、猪肚菇[31]等许多品种上均有报道。但在杏鲍菇中将表型性状、子实体质构指标联合共同作为参考指标对杏鲍菇菌株进行多样性分析的研究尚未见报道。本研究对20份杏鲍菇菌株的菌丝生长速度、产量、子实体长度、菌盖直径、菌柄长度、菌柄直径、硬度、脆度、咀嚼性、胶着性10个指标进行分析,不同类群在同一性状下的表现均有差异,且表现为同一类群内菌株性状接近。将所有指标的数据进行主成分分析后,同一类群的菌株比较明显的聚集在一起,且不会与其他类群混在一起,说明研究所选取的性状指标能很好地代表杏鲍菇子实体性状。如果能够借助表型分析设备采集到更多的性状指标,与分子标记联合进行分析,可以对品种鉴定、性状预测、杂交育种等提供基础的技术支撑。
4 结 论
通过全基因组重测序得到的SNP标记对20份杏鲍菇资源进行遗传多样性分析,并对菌丝、产量、子实体性状进行综合考量,发现通过SNP标记可以将20份杏鲍菇菌株划分为6个类群,并与产量、子实体数量性状、质构等方面有较好的一致性,对所有性状数据的主成分分析结果和SNP标记结果也有较好的一致性。在后续的工作中,可以在类群Ⅴ中选择1份菌株作为1个亲本,同时根据育种目的在其他类群中选择菌株进行杂交,以获得不同类群菌株的优良性状,适应不同的栽培条件或其他情况。

