非洲猪瘟病毒B646L基因序列分析及p72蛋白质结构与细胞抗原表位预测
罗世民 李中波

摘要:为探究非洲猪瘟病毒B646L基因序列的碱基组成特点,及预测该基因所编码的p72蛋白质结构与细胞抗原表位,利用PCR技术对非洲猪瘟病毒B646L基因ORF框的序列进行扩增、测序及碱基组成分析;运用软件EXPASY、PRABI与SWISS-MODEL对p72蛋白质进行理化性质分析及二、三级结构预测;运用在线软件ABCpred Prediction、Scratch、IEDB和NetCTL对p72蛋白在B、T细胞的抗原表位进行预测。结果表明,非洲猪瘟病毒B646L基因ORF框序列全长为1 941 bp,其中,碱基A含量为26.9%,T含量为28.9%,G含量为24.2%及C含量为20.0%,A+T的含量为55.8%,G+C的含量为44.2%,AT含量显著高于GC含量;该序列共编码646个氨基酸,p72蛋白大小为76 ku;在整个p72蛋白分子的二、三级结构中,α-螺旋占19.35%,β-转角占5.42%,无规卷曲占50.15%,扩展链为25.08%,以无规卷曲为主要结构;该蛋白存在细胞质的概率最大,其次是细胞核,存在于线粒体、高尔基体和内质网的概率较低;p72蛋白B淋巴细胞优势抗原表位分别为12~18 aa、27~37 aa、45~55 aa、77~88 aa、110~120 aa;T 淋巴细胞优势抗原表位分别为203~212 aa、298~307 aa、520~531 aa。说明非洲猪瘟病毒B646L基因ORF框序列呈明显AT偏好性;p72蛋白为一亲水性蛋白,其高级结构主要以无规卷曲为主,且具有B、T淋巴细胞优势抗原表位,是检测试剂和疫苗研发的理想靶标。
关键词:非洲猪瘟病毒;B646L基因;p72蛋白;序列分析;结构预测;抗原表位预测
中图分类号:S852.65+1文献标志码:A
文章编号:1002-1302(2023)16-0141-06
收稿日期:2022-09-04
基金项目:湖南省怀化市科学技术局项目(编号:2020R3107);湖南省教育厅项目(编号:21C1443);湖南省科技厅项目(编号:2022JJ50319)。
作者简介:罗世民(1980—),男,湖南新化人,硕士,副教授,主要从事病原分子生物学诊断。E-mail:124328975@qq.com。
通信作者:李中波,男,湖南新宁人,硕士,实验师,主要从事病原分子生物学诊断。E-mail:18390913065@163.com。
非洲猪瘟(African swine fever,ASF)是由非洲猪瘟病毒寄居于被感染猪的血液、组织液及内脏器官内所引起的且以高热不退、全身出血、脾脏肿大及呼吸与神经系统功能紊乱为病理特征的一种急性、高传染性、高致死性传染病[1-2]。1921年,該病首次于肯尼亚发现[3],随后蔓延、扩散至非洲、欧洲及南美洲等多个国家和地区[4],现已呈世界性分布。目前,全球共有37个国家和地区的养猪业已受非洲猪瘟的影响[5]。由于非洲猪瘟病毒在疾病扩散上具有高传染性及在致病上具有高致死性等特点[6-7],故而自其被发现以来一直对生猪的健康生产产生严重威胁,极大地阻碍了全球畜牧业的发展。现今,该病已被世界动物卫生组织(OIE)列为法定报告的动物疫病[8],在国内也被认定为一类动物疫病[9]。然而,迄今为止,市面上尚无该病的疫苗和治疗特效药[10]。因此,对那些能编码免疫原性蛋白的基因进行测序与分析,以及对具有疫原性蛋白进行结构预测,已成为研制非洲猪瘟疫苗的关键。
非洲猪瘟病毒为由媒介生物传播的双股线性DNA病毒。据资料显示,非洲猪瘟病毒的基因组全长为170~193 kb,共含150~167 个开放阅读框,编码150~200 种结构和非结构蛋白[7,11],其中,p12、p30、p35、p54、p72、pK205R与CD2v等蛋白具有较高的免疫源性[12],常用于非洲猪瘟的快速诊断和ELISA检测试剂盒的制备[13]。有研究表明,p72蛋白是由B646L基因所编码[14],大小约为 72 ku[15],为非洲猪瘟病毒粒子的主要衣壳蛋白,占比33%[7],具有构象中和表位且能诱导机体产生中和抗体[16],是检测试剂及疫苗开发的重要靶标[17]。如,Phillips M E等就利用p72蛋白制备了抗该蛋白的单克隆抗体[18];张文燕等在体外表达了全长和截短的p72蛋白,分析并比较了两者的免疫源性及反应源性[7]。由此可见,p72蛋白在非洲猪瘟的检测及疫苗研制等方面占有重要的一席之地,对其遗传信息及生物学功能研究已成为有效防控非洲猪瘟不可或缺的内容。因此,本研究扩增了非洲猪瘟病毒的B646L基因序列全长,并对其所编码p72蛋白的结构进行预测,以期为今后非洲猪瘟疫苗的研制提供一些有用的信息,为进一步研究p72蛋白生物学功能奠定坚实基础。
1 材料
1.1 样品来源
病料取于怀化市中方县某养猪场,经分子生物学鉴定,该病原体为非洲猪瘟病毒。
1.2 主要试剂
动物组织基因组DNA提取试剂盒,购自天根生化科技(北京)有限公司;2×EasyTaq PCR Super(Mix)保真酶,购自宝生物工程(大连)有限公司;蛋白酶K和DL-2 000 Marker,购自北京全式金生物技术有限公司。
1.3 引物设计与合成
试验于2021年9月怀化职业技术学院动物科技系分子实验室中进行,首先以ASFV中国分离株Pig/HLJ的B646L基因(登录号为MK333180)序列为参照序列,运用Primer Primer 5.0软件设计1对引物,序列为B646L-F:5′-TTAGGTACTNTAACGCAGCACAG-3′,B646L-R:5′-CATCAGGAGGAGCTTTTTGTC-3′,以扩增ASFV的B646L基因。再将设计好的引物序列发送至生工生物工程(上海)股份有限公司以进行生物合成。
2 方法
2.1 非洲猪瘟病毒的DNA提取
按照DNA提取试剂盒说明书,逐步提取非洲猪瘟病毒总DNA。再将DNA提取液于1.5%琼脂糖凝胶进行电泳,以检测DNA的提取是否成功。将阳性DNA提取液于-20 ℃保存、备用。
2.2 B646L基因的PCR扩增
以提取好的DNA为模板,利用引物、2×EasyTaq PCR Super(Mix)保真酶对非洲猪瘟病毒的B646L基因进行PCR扩增。PCR扩增体系(总体积为50 μL):2×EasyTaq PCR Super保真酶(Mix) 25 μL,ddH2O 22 μL,DNA模板1 μL,上、下游引物各1 μL。PCR擴增程序:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火45 s,72 ℃延伸2 min,共运行35个循环;最后,72 ℃再延伸5 min。吸取5 μL PCR产物在1.5%琼脂糖上进行电泳检测。将阳性PCR反应液发送至生工生物(上海)有限公司进行双向测序(每个样3个生物学重复)。
2.3 原始序列的校准、比对与分析
利用软件Chromas查看所有原始序列的测序峰图[19],去除峰图较差的序列;再将原始序列在NCBI中进行BLAST比对,确认其为目的基因序列;利用软件DNASTAR对所获序列进行碱基组成分析[20]。
2.4 B646L基因序列的翻译
利用在线软件EXPASY对B646L基因序列进行翻译[21-22],统计氨基酸数目,计算p72蛋白的分子量大小。
2.5 p72蛋白理化性质的分析
将上述所得的氨基酸序列于在线工具ProtParam(http://web.expasy.org/protparam/)进行分析[23],以获得p72蛋白分子质量、等电点、原子组成及稳定疏水性等理化性质。
2.6 p72蛋白的二、三级结构预测
利用在线软件SOMPA分析、计算p72蛋白所含的α-螺旋、β-折叠、β-转角、无规卷曲及扩展链的数目与百分比[23];利用在线软件PRABI与SWISS-MODEL对p72蛋白的二、三级结构进行预测[21]。
2.7 p72蛋白的定位分析
基于SVM算法和PSI-BLAST数据库对p72蛋白的定位进行预测和分析[21]。
2.8 p72蛋白B、T淋巴细胞抗原表位预测
利用在线工具ABCpred Prediction(http://crdd.osdd.net/raghava/abcpred/ABC_ submission. html)、Scratch(http://scratch.proteomics.ics.uci.edu/)、IEDB (http://www.Iedb.org/)对其优势 B淋巴细胞表位进行预测[23];利用在线软件IEDB、NetCTL Server(www.cbs.dtu.dk/services/NetCTL/)预测优势 T 淋巴细胞表位[23]。
3 结果与分析
3.1 B646L基因的PCR扩增
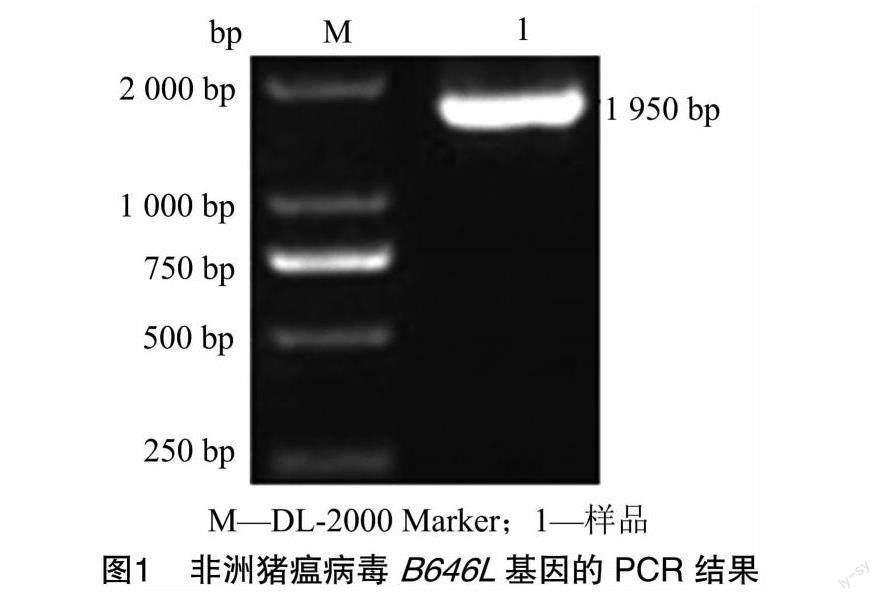
由图1可知,所扩增的PCR产物为阳性,泳道内仅含1个条带,清晰,无拖尾或含其他杂带,大小约为1 950 bp,与预期目的条带大小相一致。测序所得序列为1 941 bp。
3.2 目的基因序列的校准、比对与提交
所获原始序列经校准后,其长度为1 941 bp;再于NCBI中进行BLAST比对,发现所获序列与已公布B646L基因序列的相似度为99%~100%,确认该序列为目的基因序列。最后,将该基因序列提交到GenBank,获得其基因登录号为MK333180。
3.3 B646L基因序列碱基组成分析
对非洲猪瘟病毒B646L基因序列的碱基组成进行分析,结果显示,在非洲猪瘟病毒的B646L基因序列中,A、T、G、C等4种碱基含量分别为26.9%、28.9%、24.2%和20.0%;A+T含量为55.8%,G+C含量为44.2%。可见非洲猪瘟病毒的B646L基因序列的A+T含量高于G+C含量。
3.4 B646L基因序列的翻译
由图2可知,非洲猪瘟病毒B646L基因共编码646个氨基酸。
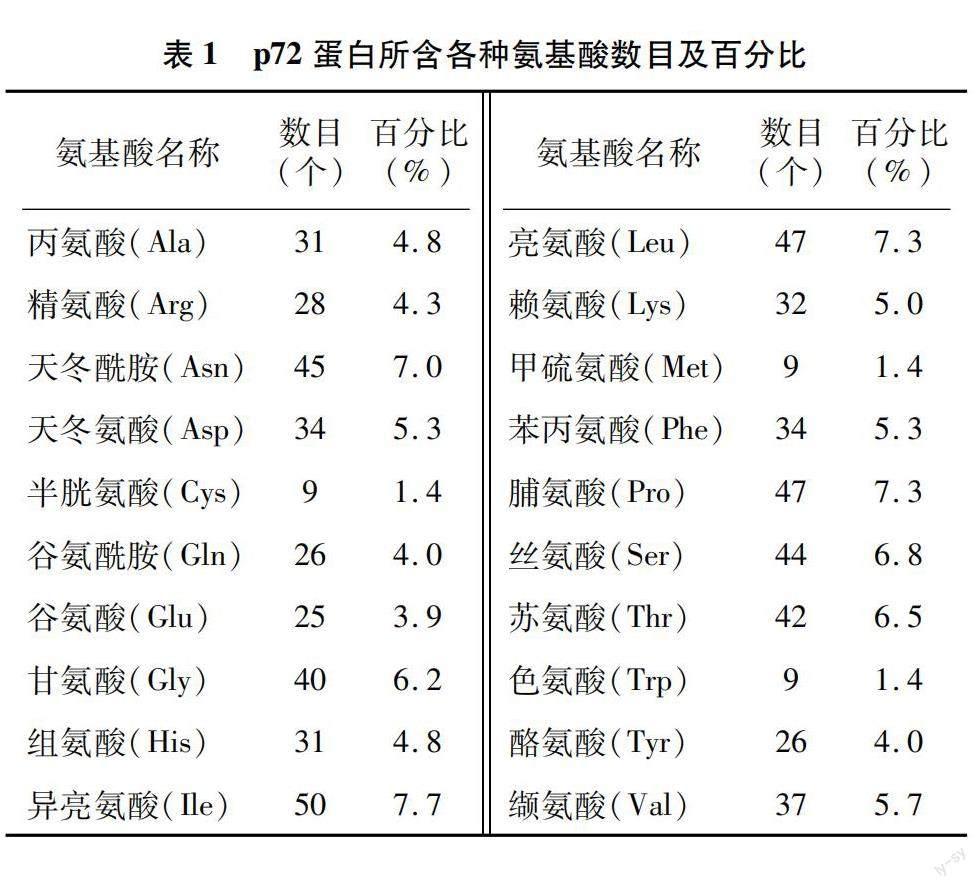
根据所获氨基酸序列,统计各种氨基酸在此氨基酸链的数目,并计算出其百分比。由表1可知,异亮氨酸占整个氨基酸链的百分比最高,其比例为7.7%,其后依次为亮氨酸和脯氨酸、天冬酰胺氨基酸、丝氨酸、苏氨酸、甘氨酸、缬氨酸、苯丙氨酸和天冬氨酸、赖氨酸、丙氨酸和组氨酸、精氨酸、酪氨酸和谷氨酰胺、谷氨酸、色氨酸、半胱氨酸和甲硫氨酸。
3.5 p72蛋白的理化性质分析
非洲猪瘟病毒p72蛋白的分子式为C3 303H5 036N904O948S18,大小为76 ku,共含646个氨基酸,其中,强碱性氨基酸个数为60,强酸性氨基酸个数为59,脂肪族氨基酸指数为79.97,等电点为7.71,平均亲水系数(GRAVY)为-0.392,不稳定系数为38.3。
3.6 p72蛋白的二、三级结构预测
利用在线软件SOMPA对p72蛋白的二级结构进行分析,结果显示,α-螺旋数目为125个,占19.35%;β-转角数目为35个,占5.42%,无规卷曲数目为324个,占50.15%,扩展链数目为162个,占25.08%。同时,也对p72蛋白的氨基酸位点进行了分析,由图3可知,p72蛋白所含的无规卷曲数目最多,其二级结构以无规卷曲为主。
此外,利用在线分析工具Swiss-Model(https://swissmodel.expasy.org/)对p72蛋白的三级结构进行预测。由图4可知,非洲猪瘟病毒p72蛋白的三级结构含有无规卷曲、α-螺旋、β-折叠及 β-转角等二级结构,其中以无规卷曲的数目最多,占比最高;p72蛋白三级结构的N端为一β-折叠结构,而其C端为一α-螺旋,其GMQE为0.82,QMEAN4为0.76±0.05。与模型c612tB、c6ku9B相比,其覆盖率均为100%,相似度卻分别为98%和100%。
3.7 p72蛋白的定位分析
基于SVM算法中的LOCSVMPSI预测方法及PSI-BLAST数据库的HSLpred预测方法对p72蛋白的定位进行了预测,结果显示,p72蛋白位于线粒体的可能性为4.3%,位于高尔基体、内质网及分泌系统囊泡的概率均为4.3%,位于真核细胞核内的概率为39.1%,位于真核细胞质内的概率为43.5%。
3.8 p72蛋白在B、T淋巴细胞抗原表位预测
运用软件ABCpred Prediction、Scratch及IEDB对p72蛋白B淋巴细胞抗原表位进行预测(表2)。
运用在线预测工具NetCTL、IEDB对p72蛋白T淋巴细胞抗原表位进行预测(表3)。
4 讨论
目前,关于ASFV的研究主要集中于2个方面,一是制备ASFV的抗体并将其应用于该病毒的检测中,以达到建立快速、精准检测方法的目的;二是研究ASFV抗体的保护性及抗体对病毒粒子复制的影响和作用机制[24]。一些研究结果表明,痊愈后和恢复期的病猪血清可保护健康猪,使其免受高毒力的ASFV攻击[25-26],这为研发非洲猪瘟疫苗提供了思路和线索。众所周知,非洲猪瘟病毒为一正二十面体粒子,其结构主要由DNA核心、衣壳及囊膜组成[27],含150~200 种结构和非结构蛋白[7,11],结构蛋白A104R、A151R、EP153R、B602L、K78R、p30、p54 和 p72的功能已被鉴定出[28],其中由p30、p54 和 p72等蛋白所制备的抗体可抑制病毒的复制[29]。此外,p72蛋白是ASFV衣壳的主要组成部分,并在病毒识别、结合及侵染宿主细胞等过程中起到十分关键的作用[30]。再者,也有研究表明p72蛋白在不同ASFV株之间高度保守[31-32],并具有良好的抗原性和免疫源性,不仅是ASFV 在宿主细胞内复制的指示器[7],也是检测试剂及疫苗开发的重要靶标。因此,本研究对ASFV的B646L基因序列进行分析,并预测其所编码p72蛋白的结构,以期为今后非洲猪瘟疫苗的研制提供一些有用的信息,为进一步研究p72蛋白生物学功能奠定坚实的基础。
从非洲猪瘟病毒B646L基因序列的碱基组成来看,AT含量(55.8%)显著高于GC含量(44.2%),与侯景等[21]、申超超等[33]研究的结果相一致,表明非洲猪瘟病毒B646L基因序列具有明显的AT偏好性。
从p72蛋白的氨基酸组成及理化性质来看,可知p72蛋白共含646个氨基酸,其中异亮氨酸(7.7%)、亮氨酸(7.3%)和脯氨酸(7.3%)为主要的氨基酸,其中强碱性氨基酸的数目为60个,所占百分比为9.29%,强酸性氨基酸数目为59个,所占百分比为9.13%。两者数目及所占比例几乎相等,故而推断该蛋白呈中性,与它的等电点为7.71相一致。此外,根据该蛋白的平均亲水系数为 -0.392,及不稳定系数为38.3(<40),可以推断非洲猪瘟病毒p72蛋白为一结构稳定的亲水性蛋白。
根据p72蛋白二级结构预测结果发现,该蛋白含3种二级结构,其中所含无规卷曲的数目最多(324,50.15%),其次为α-螺旋(125,19.35%),再次为β-转角(35,5.42%),与高瞻等研究非洲猪瘟病毒p72蛋白二级结构的结果[23]相一致,暗示该蛋白以无规卷曲为主要结构。然而,根据p72蛋白三级结构预测结果发现,该蛋白三级结构含4种(α-螺旋、β-转角、β-折叠及无规卷曲)二级结构,但以无规卷曲为主要结构,α-螺旋、β-折叠及β-转角的数目及占比较低,与高瞻等所预测非洲猪瘟病毒p72蛋白三级结构的结果[23]相符。此外,由图4可知,p72蛋白的三级结构比较紧密,与上述推断其为结构稳定的结果相符合。
基于p72蛋白定位分析结果,发现该蛋白存在细胞质的概率最大(43.5%),其次是细胞核(39.1%),这主要因为病毒的衣壳合成及病毒粒子的组装均在细胞质中进行,故而该蛋白存在细胞质的概率最高。然而,有学者推测非洲猪瘟病毒粒子的一些蛋白(包括p72蛋白)可能参与细胞核对核内IFN-β或NF-κB的抑制作用,从而导致宿主的天然免疫系统不能抵抗病毒[34],所以p72蛋白存在细胞核的概率为39.1%。
目前,市面上尚无用于防治非洲猪瘟的商品化疫苗和药物,但针对该病防控的核心是疫苗,而中和性抗原表位的研究却又是疫苗研制的基础[15]。抗原表位,俗称抗原决定簇,是病毒粒子结合淋巴细胞表面抗原受体的特定区段[23],能引发细胞、体液免疫反应。在细胞免疫反应过程中,T淋巴细胞受体(TCR)可在抗原的任何位置上识别线性表位,B淋巴细胞受体(BCR)能识别构象表位(多肽、多糖、脂多糖)和线性表位,且识别位置通常处于抗原分子的表面。一般而言,蛋白质的C 端因具有很好的亲水性、柔性和表面可及性等特点,往往是良好的优势表位区域,然而本试验所获得的p72蛋白的C 端为一α-螺旋结构(不易变形,且难以与抗体结合),很难形成表位。因此,p72蛋白C 端不作为抗原表位。然而,再根据p72蛋白的二、三级结构预测结果,发现无规卷曲为该蛋白的主体结构,在其中所占比例最高(50.15%),且该蛋白含有一定量的β转角(5.42%),两者均位于p72蛋白的表面,结构突出、可及性强、易与抗体结合,成为抗原表位概率大,筛选出5个p72蛋白B 淋巴细胞优势抗原表位,它们分别为12~18 aa、27~37 aa、45~55 aa、77~88 aa及110~120 aa;筛选出3个p72蛋白T 淋巴细胞优势抗原表位,即分别为203~212 aa、298~307 aa、520~531 aa,与高瞻等所筛选出B、T 淋巴细胞优势抗原表位[23]相同。
5 结论
本研究结果表明,非洲猪瘟病毒B646L基因序列呈明显的AT偏好特征,其所编码的p72蛋白以无规卷曲为主要结构,并具有B、T淋巴细胞优势抗原表位,是检测试剂和疫苗研发的理想靶标,不仅为非洲猪瘟病毒的感染机制研究提供了有用的信息,也为进一步研究p72基因生物学功能及疫苗的研制奠定坚实的基础。
参考文献:
[1]Wang T,Sun Y,Huang S J,et al. Multifaceted immune responses to African swine fever virus:implications for vaccine development[J]. Veterinary Microbiology,2020,249:108832.
[2]Sánchez-Cordón P J,Montoya M,Reis A L,et al. African swine fever:a re-emerging viral disease threatening the global pig industry[J]. Veterinary Journal,2018,233:41-48.
[3]Eustace M R. On A form of swine fever occurring in British East Africa (Kenya colony)[J]. Journal of Comparative Pathology and Therapeutics,1921,34:159-191.
[4]Mulumba-Mfumu L K,Saegerman C,Dixon L K,et al. African swine fever:update on eastern,central and southern Africa[J]. Transboundary and Emerging Diseases,2019:tbed.13187.
[5]劉学敏,张元峰,王永琦,等. 非洲猪瘟病毒E301R蛋白单克隆抗体的制备及其初步应用[J]. 中国预防兽医学报,2022,44(9):963-969.
[6]于学祥,陈晓雨,李栋凡,等. 非洲猪瘟病毒无标签p30-ELISA抗体检测方法的建立及应用[J]. 畜牧兽医学报,2022,53(5):1517-1526.
[7]张文燕,王亚文,冯亚文,等. 非洲猪瘟病毒全长与截短p72蛋白的抗原性比较[J]. 畜牧兽医学报,2022,53(4):1182-1191.
[8]戈胜强,吴晓东,李金明,等. 巴西非洲猪瘟根除计划的经验与借鉴[J]. 中国兽医学报,2017,37(5):961-964.
[9]徐玲玉,曹琛福,李中圣,等. 非洲猪瘟病毒p54蛋白单克隆抗体的制备及阻断ELISA方法的建立[J]. 畜牧兽医学报,2022,53(3):813-821.
[10]向志达,李长尧,张涛清,等. 非洲猪瘟病毒A137R蛋白单克隆抗体的制备与表位鉴定[J]. 中国预防兽医学报,2022,44(4):402-410.
[11]Arias M,Jurado C,Gallardo C,et al. Gaps in African swine fever:analysis and priorities[J]. Transboundary and Emerging Diseases,2018,65(Suppl 1):235-247.
[12]杨文兵,邹亚文,蒋一凡,等. 非洲猪瘟血清学诊断靶点的研究进展[J]. 畜牧兽医学报,2021,52(5):1208-1217.
[13]靳雯雯,楊俊兴,花群俊,等. 非洲猪瘟病毒抗体检测间接ELISA方法的建立[J]. 中国兽医学报,2014,34(7):1043-1046.
[14]李文傲. 非洲猪瘟病毒P72蛋白单克隆抗体的制备[D]. 秦皇岛:河北科技师范学院,2020.
[15]栗 宁,周景明,王爱萍. 非洲猪瘟病毒主要结构蛋白B细胞表位的研究现状[J]. 动物医学进展,2022,43(4):93-96.
[16]Neilan J G,Zsak L,Lu Z,et al. Neutralizing antibodies to African swine fever virus proteins p30,p54,and p72 are not sufficient for antibody-mediated protection[J]. Virology,2004,319(2):337-342.
[17]Petrovan V,Yuan F F,Li Y H,et al. Development and characterization of monoclonal antibodies against p30 protein of African swine fever virus[J]. Virus Research,2019,269:197632.
[18]Phillips M E. Epitope mapping of African swine fever virus p72 capsid protein using polyclonal swine sera and monoclonal antibodies[D]. Manhattan:Kansas State University,2016.
[19]Liu G H,Chen F,Chen Y Z,et al. Complete mitochondrial genome sequence data provides genetic evidence that the brown dog tick Rhipicephalus sanguineus (Acari:Ixodidae) represents a species complex[J]. International Journal of Biological Sciences,2013,9(4):361-369.
[20]Li Z B,Cheng T Y,Xu X L,et al. Genetic variation in mitochondrial genes of the tick Haemaphysalis flava collected from wild hedgehogs in China[J]. Experimental and Applied Acarology,2017,71(2):131-137.
[21]侯 景,申超超,张大俊,等. 非洲猪瘟病毒解旋酶D1133L基因序列分析、蛋白结构预测及亚细胞定位[J]. 畜牧兽医学报,2021,52(7):1953-1962.
[22]Gasteiger E,Hoogland C,Gattiker A,et al. Protein identification and analysis tools on the ExPASy server[M]//The proteomics protocols handbook. Totowa,NJ:Humana Press,2005:571-607.
[23]高 瞻,邵军军,常艳燕,等. 非洲猪瘟病毒p72蛋白抗原表位预测分析及多表位疫苗的构建[J]. 中国兽医杂志,2020,56(1):13-17,142.
[24]Tesfagaber W,Wang L L,Tsegay G,et al. Characterization of anti-p54 monoclonal antibodies and their potential use for African swine fever virus diagnosis[J]. Pathogens,2021,10(2):178.
[25]Arias M,de la Torre A,Dixon L,et al. Approaches and perspectives for development of African swine fever virus vaccines[J]. Vaccines,2017,5(4):35.
[26]Onisk D V,Borca M V,Kutish S,et al. Passively transferred African swine fever virus antibodies protect swine against lethal infection[J]. Virology,1994,198(1):350-354.
[27]Liu S,Luo Y Z,Wang Y J,et al. Cryo-EM structure of the African swine fever virus[J]. Cell Host & Microbe,2019,26(6):836-843.e3.
[28]Gallardo C,Reis A L,Kalema-Zikusoka G,et al. Recombinant antigen targets for serodiagnosis of African swine fever[J]. Clinical and Vaccine Immunology,2009,16(7):1012-1020.
[29]Gómez-Puertas P,Rodríguez F,Oviedo J M,et al. The African swine fever virus proteins p54 and p30 are involved in two distinct steps of virus attachment and both contribute to the antibody-mediated protective immune response[J]. Virology,1998,243(2):461-471.
[30]赵旭阳,樊 帅,靳家鑫,等. 非洲猪瘟病毒增殖过程中主要蛋白研究进展[J].中国预防兽医学报,2022.
[31]Jia N,Ou Y W,Pejsak Z,et al. Roles of African swine fever virus structural proteins in viral infection[J]. Journal of Veterinary Research,2017,61(2):135-143.
[32]Bao J Y,Wang Q H,Lin P,et al. Genome comparison of African swine fever virus China/2018/AnhuiXCGQ strain and related European p72 genotype Ⅱ strains[J]. Transboundary and Emerging Diseases,2019,66(3):1167-1176.
[33]申超超,李國丽,张大俊,等. 非洲猪瘟病毒 MGF 360-9L基因序列分析、蛋白结构预测及亚细胞定位[J]. 畜牧兽医学报,2020,51(6):1371-1381.
[34]Li J N,Song J,Kang L,et al. pMGF505-7R determines pathogenicity of African swine fever virus infection by inhibiting IL-1β and type Ⅰ IFN production[J]. PLoS Pathogens,2021,17(7):e1009733.

