辣椒MADS–box基因家族的鉴定及表达分析
杨光彬,王瑾,陈恺琳,单庆云,崔苏菲,熊程2,,4,5,邹学校2,,4,5,刘峰2,,4,5*
辣椒MADS–box基因家族的鉴定及表达分析
杨光彬1,王瑾3,6,陈恺琳3,单庆云3,崔苏菲3,熊程2,3,4,5,邹学校2,3,4,5,刘峰2,3,4,5*
(1.湖南大学隆平分院,湖南 长沙 410125;2.岭南现代农业实验室,广东 广州 510642;3.湖南农业大学园艺学院,湖南 长沙 410128;4.园艺作物种质创新与新品种选育教育部工程研究中心,湖南 长沙 410128;5.蔬菜生物学湖南省重点实验室,湖南 长沙 410128;6.南京农业大学园艺学院,江苏 南京 210095)
利用辣椒的全基因组数据鉴定到104个MADS–box基因,对它们的理化性质、染色体定位、系统进化关系、蛋白保守基序和组织表达水平进行分析。结果表明:辣椒MADS–box基因家族各成员在染色体上的分布不均,理化性质差异较大,104个家族成员编码的氨基酸长度为100~567 aa,蛋白相对分子质量为11 203.9~ 63 559.7,蛋白理论等电点(PI)为4.63~10.46,系统进化树分析结果表明,辣椒MADS–box基因家族可分为2大类,与拟南芥和番茄的进化关系类似;组织表达水平分析结果表明,主要在花、果实和种子中表达,在叶片中表达量相对较低,推测MADS–box基因可能参与调控果实的发育和成熟。
辣椒;MADS–box基因家族;基因表达
辣椒(L.)是一种重要的蔬菜和香料作物,由于辣椒素和抗坏血酸含量较高,越来越被关注[1–2]。研究表明,MADS–box基因在植物的发育过程中(尤其是在花器官形成和果实成熟过程中)发挥重要作用[3–8]。研究人员已经对多种植物进行了MADS–box基因的全基因组分析[9–11],但对辣椒MADS–box基因研究较少,仅有4个辣椒MADS–box基因被初步验证功能[12–15]。本研究中,根据辣椒全基因组序列[16–17]鉴定了104个辣椒MADS–box基因,分析了它们的基本信息,包括进化关系、基因结构、保守基序、染色体定位和种间共线性,同时预测了它们的亚细胞定位、顺式元件和表达模式,并通过qRT–PCR验证基因表达图谱,旨在了解辣椒的MADS–box家族成员及潜在功能,为研究MADS–box家族在辣椒中的作用提供依据。
1 材料与方法
1.1 材料
供试辣椒材料为野生型‘S8’,由湖南农业大学园艺学院提供。
1.2 方法
1.2.1辣椒MADS–box基因的鉴定
通过蛋白质保守结构域提取号PF00319 (SRF–TF结构域)和PF01486 (K–box结构域)从Pfam数据库(http://pfam.xfam.org/)[18]下载MADS结构域的隐马尔可夫模型(HMM)图谱,设定截断参数≤0.01,运用HMMER SEARCH 3.0程序在下载的HMM谱中识别含有MADS结构域的蛋白质[19]。利用SMART(simple modular architecture research tool)数据库预测辣椒中存在MADS–box保守结构域的候选蛋白[20],参考2个高质量的辣椒基因组数据CM334(http://peppergenome.snu.ac.kr/download.php)和Zunla–1(http://peppersequence.genomics.cn/),运用ExPaSy在线工具(https://web.expasy.org/compute_pi/)计算相对分子质量和理论等电点[21],并通过softberry online tool(http://www.softberry.com)预测基因的亚细胞定位。
1.2.2保守基序、基因结构和系统发育分析
运用在线工具Multiple EM(MEME,https:// meme–suite.org/meme/tools/meme)挖掘基序(motif),并用DNAMAN8.0.8(Lynnon Biosoft)进行比对;运用Gene Structure Display Server绘制CaMADSs的基因结构[22];利用MEGA–X程序[23]根据1000次步长检验(bootstrap test)的相邻连接法(NJ)构建CaMADSs的无根系统发育树,CaMADSs的完整蛋白质序列来源于Zunla–1参考基因组。
1.2.3染色体定位和共线性分析
利用Zunla–1参考基因组获取染色体位置,运用TBtools进行分析和绘图[24];利用MCScanX分析基因共线性和同线性;通过KaKs_Calculator1.2[25]计算同义替换率(s)和非同义替换率(a)。
1.2.4启动子区域的顺式元件分析
运用PlantCARE在线工具(http://bioinformatics. psb.ugent.be/webtools/plantcare/html/)预测CaMADSs启动子区顺式元件[26]。
1.2.5辣椒MADS–box基因家族表达分析
用于RNA–seq分析的原始数据均来源于PepperHub (http://PepperHub.hzau.edu.cn/)[27],通过Fastqc[28]和Trimmmatic–0.36[29]过滤低质量序列;利用HISAT2比对参考基因组Zunla–1序列,获得配对末端测序读数[30],并通过FeatureCounts计算读数之和[31];采用Rv3.6.1中的DESeq2包实现计数数据归一化[32],并构建热图。
1.2.6总RNA的提取和qRT–PCR分析
取‘S8’的花蕾、完全开放的花、未成熟绿色果、绿色成熟果、破色期果、成熟果、幼叶、成熟叶、绿色成熟果中的种子以及成熟果中的种子,采用TransZol Up(全式金生物技术有限公司,北京)提取样品总RNA,然后使用Evo M–MLV预混物(艾科瑞生物技术公司)合成用于qRT–PCR的cDNA。按照ChamQ Universal SYBR qPCR Master Mix(诺唯赞,南京)20 μL体系配制反应试剂,通过LightCycle® 96实时荧光定量PCR仪(Roche,瑞士)运行qRT–PCR程序,以辣椒基因Capana04g001698为内参[33],进行3次重复。采用2−∆∆Ct方法计算基因相对表达水平[34]。所有引物均通过GenScript网站(https://www.genscript.com/tools/real–time–pcr–taqman–primer–design–tool)设计,如表1所示。

表1 用于qRT–PCR 的基因和引物
1.2.7统计分析
使用SPSS软件进行数据分析。
2 结果与分析
2.1 辣椒MADS–box基因家族的鉴定
依据HMM MADS模型找到了104个包含在CM334和Zunla–1基因组数据库的辣椒MADS–box基因,根据其染色体位置,将其重命名为…。通过分析,其氨基酸序列的长度为100~567 aa,相对分子质量为11 203.9~63 559.7,基因的等电点(PI)为4.63~10.46。亚细胞定位的预测结果表明,除(Capana11g001822)存在于胞外结构中,其余均定位在细胞核中。
2.2 辣椒MADS–box基因的分类和系统发育分析
运用MEGA 5.02并采用NJ方法构建系统发育树(图1),结果表明,分类结果与拟南芥、番茄类似[10],104个基因可分为分支I和II,几乎所有只含MADS结构域的基因都属于分支I (Capana08g002103和Capana12g000929只含1个MADS结构域除外),其余小部分基因属于分支II。
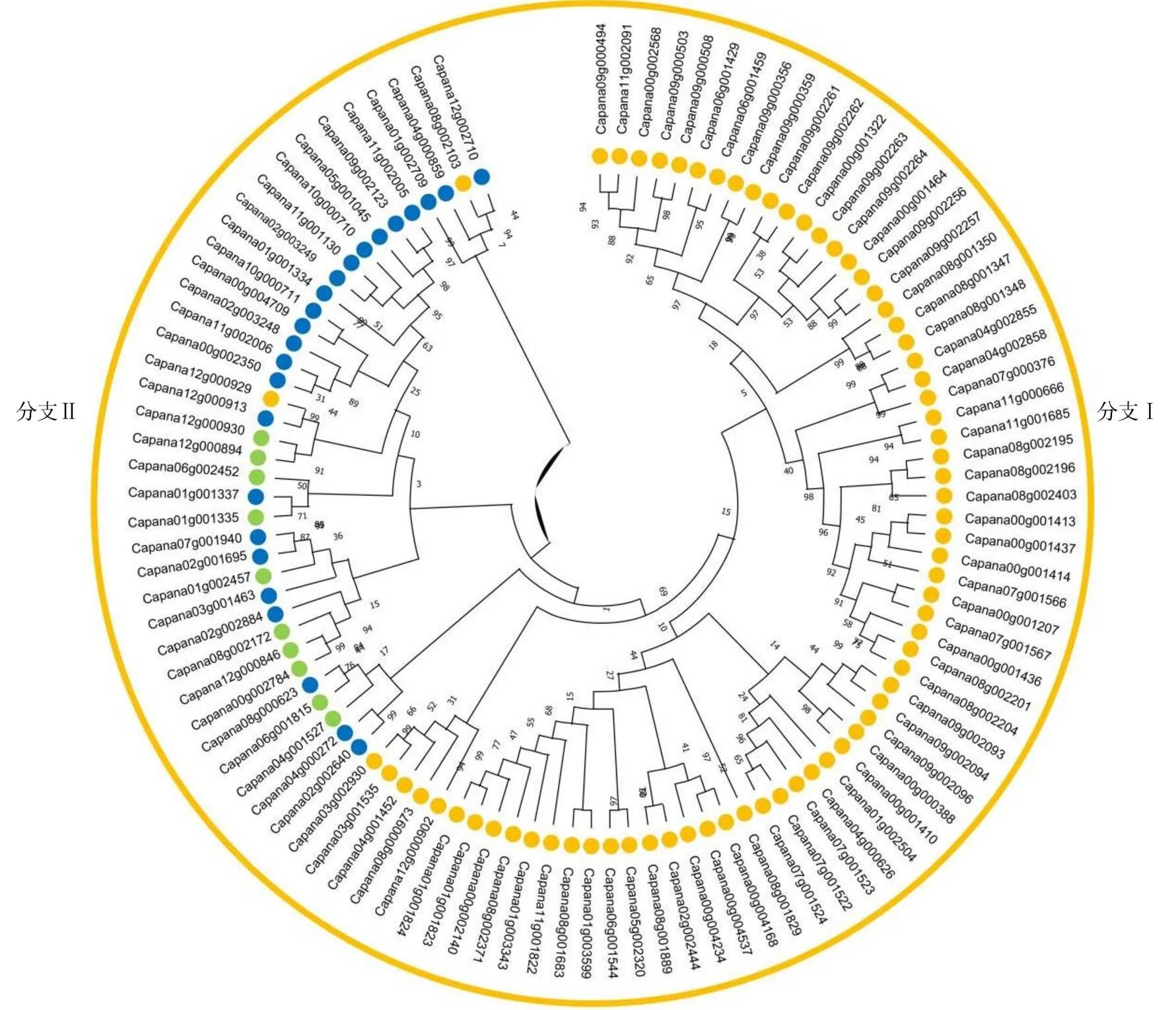
黄色圆点代表仅含MADS结构域的基因;蓝色圆点代表含有MADS结构域和K–box结构域的基因;绿色圆点代表仅含K–box 结构域的基因。
2.3 辣椒MADS–box基因的保守结构域和基因结构分析
为深入了解MADS–box基因的结构多样性、相似性及进化关系,分析了内含子–外显子的排列和保守基序,从结果(图2)可以看出,辣椒MADS–box基因包含0~9个内含子,50%以上的基因没有内含子,104个MADS–box基因中只有21个(20.2%)含有5个以上的内含子。另外,在辣椒MADS–box蛋白中鉴定到8个保守基序。

图2 辣椒MADS–box 基因的基序位置和基因结构
2.4 辣椒MADS–box基因的染色体定位和种间共线性分析
根据辣椒基因组注释,绘制了辣椒MADS–box基因的染色体定位及基因共线性图(图3)。结果表明,MADS–box基因不均匀地分布在12条染色体上。辣椒MADS–box基因多数集中在08至12染色体上,许多被定位在00染色体上,01至07染色体上辣椒MADS–box基因数量相对较少,05染色体上只有2个MADS–box基因,03染色体上只有3个MADS–box基因。红色曲线表示5组CaMADS重复基因对,每组重复基因对的a和s值及其比值(a/s)列于表2,重复基因对的a值为0.280~0.329,s值为0.997~2.145。a/s值为评价进化选择压力的指标,均小于1.00,说明辣椒在进化过程中存在纯合选择。
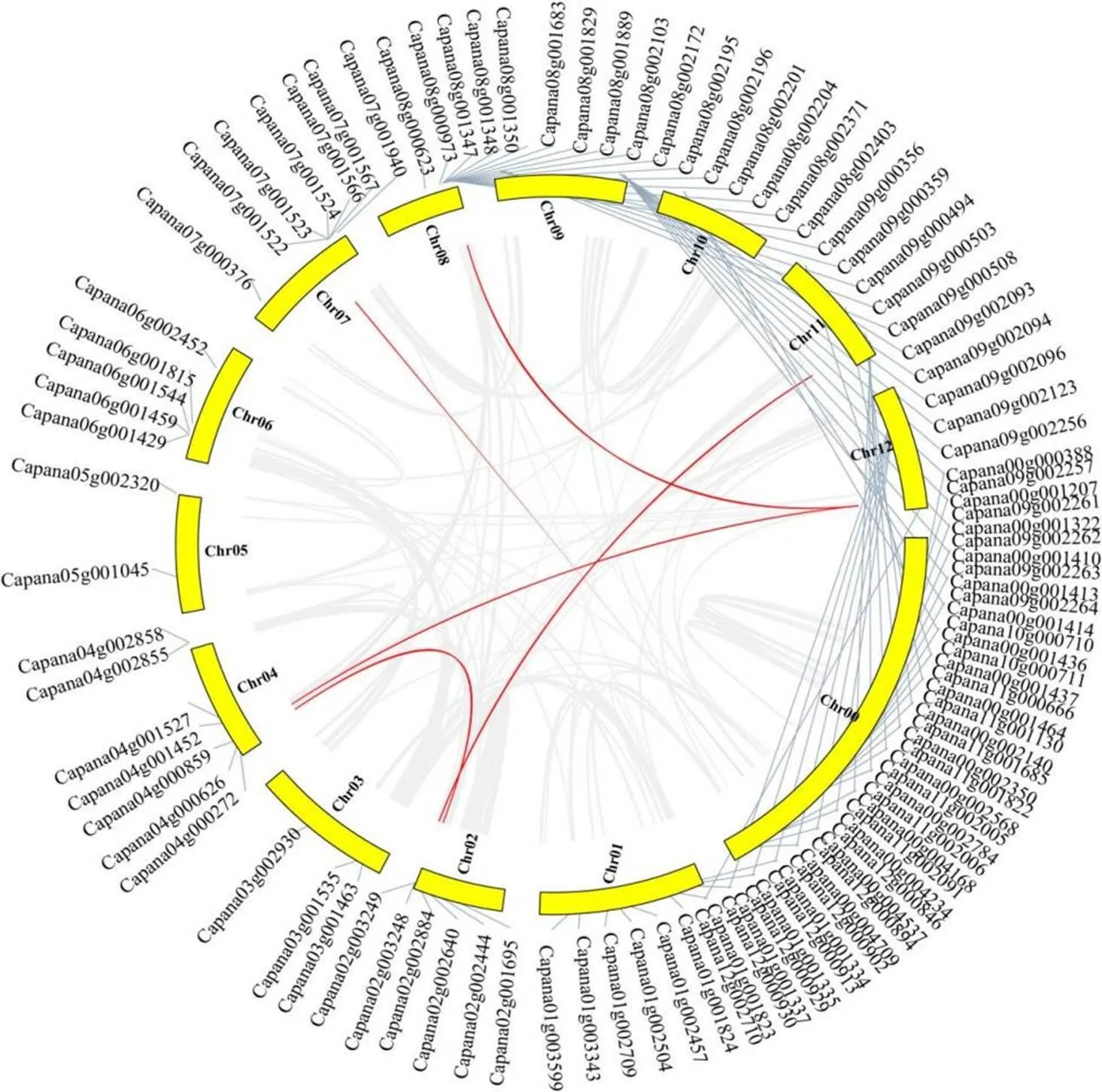
图3 辣椒MADS–box基因的染色体定位及基因共线性

表2 辣椒MADS–box基因的复制基因对
2.5 辣椒MADS–box基因启动子区顺式元件的预测
本研究鉴定到7个细胞发育顺式调控元件,负责胚乳、分生组织、栅栏叶肉细胞、类黄酮生物合成和细胞周期调控;预测了13个与激素相关的顺式元件,涉及乙烯、茉莉酸甲酯、赤霉素、水杨酸和生长素;还预测到33个与胁迫相关的顺式调控元件,包括非生物胁迫(光、低温、干旱反应和厌氧诱导)和生物胁迫(创伤响应等)。其中71个基因含有参与ABA反应的ABRE顺式作用元件,73个基因含有参与乙烯反应的ERE顺式作用元件。
2.6 辣椒MADS–box基因在不同器官中的表达谱预测
基于PepperHub数据集构建了分层聚类热图(图4),包含了花发育的9个时期(F1、F2、…、F9)、果实发育的11个时期(G1、G2、…、G11)、叶片发育的10个时期(L1、L2、…、L10)和种子发育的11个时期(S1、S2、…、S11)。基因表达谱显示,辣椒中大量基因在花、果实和种子中高表达,在叶中低表达。
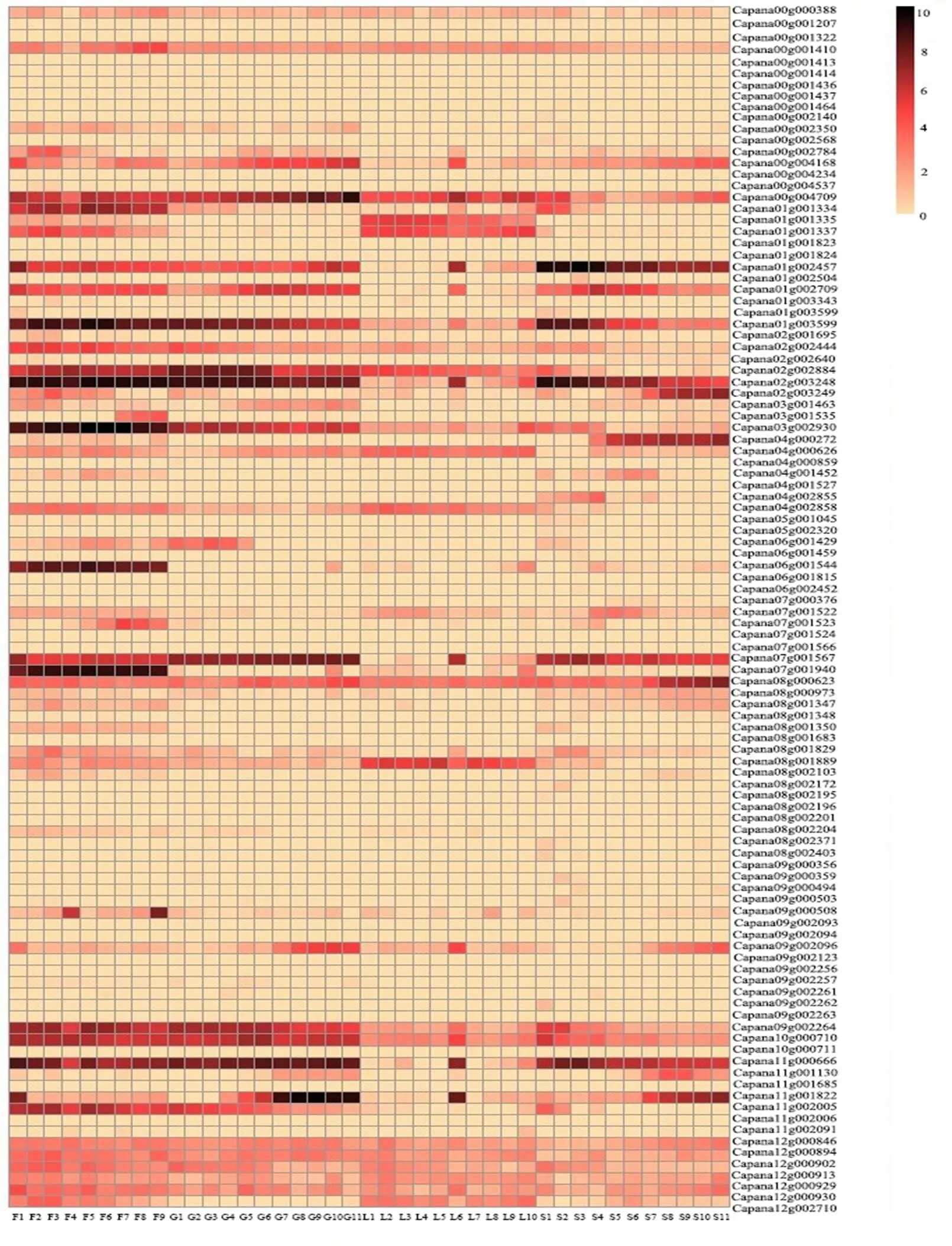
F1、F2、…、F9代表花的9个发育阶段;G1、G2、…、G11代表果实的11个发育阶段;L1、L2、…、L10代表叶片的10个发育阶段;S1、S2、…、S11代表种子的11个发育阶段。
2.7 辣椒MADS–box基因在果实发育阶段的差异表达分析
基于转录组数据集选择了6个呈现不同表达趋势的辣椒MADS–box基因,在整个发育时期均有表达,其中,在果实发育成熟阶段表达量较高;在花、果、叶中均有表达,在果实发育初期表达量较高;在花和幼果中的表达量相对较高;在果实和种子中表达量较高,且随着果实和种子逐渐成熟,表达量显著增加;在各阶段均有表达,在种子发育成熟时期表达水平尤为突出;在果实发育后期和成熟种子中的表达水平较高。通过qRT–PCR进行验证,结果(表3)发现表达量在花、果实和种子发育期间均呈现上升趋势,在果实成熟阶段达最高值。在果实发育初期表达量最高,约为花中的4倍,成熟果的2倍。在花器官中具有较高的表达水平,在其余组织中几乎无表达。在成熟果实中表达量最高,随果实逐渐成熟表达量显著增加,在叶中表达量接近0。在成熟种子中表达量最高(197.02),约是花中表达量的70倍,成熟果表达量的8倍。在果实初期几乎不表达,但随果实发育表达量迅速上升,果实成熟时达到最高值(216.59),在成熟种子中的表达量也较高,达133.70。qRT–PCR验证结果与表达谱预测趋势基本一致。
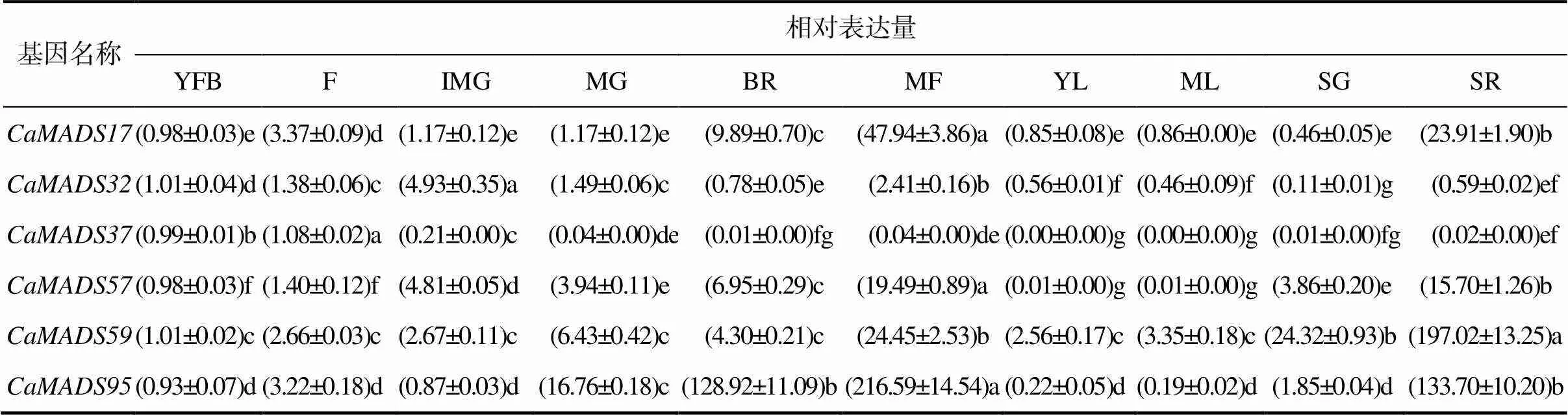
表3 辣椒中6个MADS–box基因4种组织不同发育阶段的相对表达量
YFB花蕾;F完全开放的花;IMG未成熟绿色果;MG绿色成熟果;BR破色期果;MF成熟果;YL幼叶;ML成熟叶;SG绿色成熟果中种子;SR成熟果中种子。不同小写字母示同行数据的差异有统计学意义(<0.05)。
3 结论与讨论
基因家族的全基因组分析方法是了解基因结构、进化和功能的有效分析方法。本研究结果显示,辣椒MADS家族中104个基因,与番茄中131个MADS家族成员[10]数量接近,推测该家族在进化中是保守的。对外显子和内含子数量和分布特征的分析结果表明,大多数基因具有相似的结构。表明这些基因在植物发育中的生物学功能相对保守。
鉴于基因的表达模式能反映其功能[35],对不同生育时期辣椒MADS–box基因家族在花、果实、叶和种子的表达模式进行分析。结果显示,该家族基因主要在生殖器官中出现高的表达量,在叶片中的表达量相对较低。qRT–PCR结果显示,6个基因的表达水平均随果实发育升高或降低,可能正向或反向调控果实发育。和随果实的成熟其相对表达量上升,表明它们可能促进果实成熟。此外,这4个基因在成熟种子中表达。根据这些结果推测,辣椒的MADS–box基因可能参与调控果实的发育和成熟,包括影响果实成熟时间、果实色泽和风味等[36–39],这在很多作物,如番茄、草莓等中已经被验证。在花器官中的表达水平较高,但在果实中显著降低,这可能意味着该基因主要参与花器官发育的调控。此外,和都属于分支II,推测分支II中的转录因子更可能参与调控辣椒果实的发育和成熟,这与吕山花等[40]的研究结果基本一致。但其潜在的功能与分子调控机制还需进一步研究。
[1] RAMCHIARY N,KOLE C.The Capsicum Genome[M]. Berlin:Springer,2019.
[2] 邹学校,马艳青,戴雄泽,等.辣椒在中国的传播与产业发展[J].园艺学报,2020,47(9):1715–1726.
[3] ZHAO D,CHEN Z,XU L,et al.Genome-wide analysis of the MADS-box gene family in maize:gene structure,evolution,and relationships[J].Genes,2021,12(12):1956.
[4] TAPIA-LÓPEZ R,GARCÍA-PONCE B,DUBROVSKY J G,et al.An-related MADS-box gene,(),regulates root meristem cell proliferation and flowering transition in[J].Plant Physiology,2008,146(3):1182–1192.
[5] ZHANG H,FORDE B G.AnMADS box gene that controls nutrient-induced changes in root architecture[J].Science,1998,279:407–409.
[6] DE BODT S,RAES J,VAN DE PEER Y,et al.And then there were many:MADS goes genomic[J].Trends in Plant Science,2003,8(10):475–483.
[7] HECK G R,PERRY S E,NICHOLS K W,et al.AGL15,a MADS domain protein expressed in developing embryos[J].The Plant Cell,1995,7(8):1271–1282.
[8] SLUGINA M A,EFREMOV G I,SHCHENNIKOVA A V,et al.Characterization ofisoforms and their expression in tomato fruit ripening[J].Cells,2021,10(7):1739.
[9] PARENICOVÁ L,DE FOLTER S,KIEFFER M,et al. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in:new openings to the MADS world[J].The Plant Cell,2003,15(7):1538–1551.
[10] WANG Y S,ZHANG J L,HU Z L,et al.Genome-wide analysis of the MADS-box transcription factor family in[J].International Journal of Molecular Sciences,2019,20(12):2961.
[11] SCHILLING S,KENNEDY A,PAN S R,et al. Genome-wide analysis of MIKC-type MADS-box genes in wheat:pervasive duplications,functional conservation and putative neofunctionalization[J].The New Phytologist,2020,225(1):511–529.
[12] DONG T T,CHEN G P,TIAN S B,et al.A non-climacteric fruit generegulates fruit ripening and ethylene biosynthesis in climacteric fruit[J].PLoS One,2014,9(4):e95559.
[13] CHEN R G,MA J H,LUO D,et al.CaMADS,a MADS-box transcription factor from pepper,plays an important role in the response to cold,salt,and osmotic stress[J].Plant Science,2019,280:164–174.
[14] BOROVSKY Y,RAZ A,DORON-FAIGENBOIM A,et al.Pepper fruit elongation is controlled by[J].Frontiers in Plant Science,2021,12:815589.
[15] WU S,CLEVENGER J P,SUN L,et al.The control of tomato fruit elongation orchestrated by,andin a wild relative of tomato[J].Plant Science,2015,238:95–104.
[16] QIN C,YU C S,SHEN Y O,et al.Whole-genome sequencing of cultivated and wild peppers provides insights intodomestication and specialization[J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111(14):5135–5140.
[17] KIM S,PARK M,YEOM S I,et al.Genome sequence of the hot pepper provides insights into the evolution of pungency inspecies[J].Nature Genetics,2014,46(3):270–278.
[18] EL-GEBALI S,MISTRY J,BATEMAN A,et al.The Pfam protein families database in 2019[J].Nucleic Acids Research,2019,47(D1):D427–D432.
[19] FINN R D,CLEMENTS J,EDDY S R.HMMER web server:interactive sequence similarity searching[J]. Nucleic Acids Research,2011,39(suppl_2):W29–W37.
[20] LETUNIC I,KHEDKAR S,BORK P.SMART:recent updates,new developments and status in 2020[J].Nucleic Acids Research,2021,49(D1):D458–D460.
[21] GASTEIGER E,GATTIKER A,HOOGLAND C,et al. ExPASy:the proteomics server for in-depth protein knowledge and analysis[J].Nucleic Acids Research,2003,31(13):3784–3788.
[22] HU B,JIN J P,GUO A Y,et al.GSDS 2.0:an upgraded gene feature visualization server[J].Bioinformatics,2015,31(8):1296–1297.
[23] KUMAR S,STECHER G,LI M,et al.MEGA X:molecular evolutionary genetics analysis across computing platforms[J].Molecular Biology and Evolution,2018,35(6):1547–1549.
[24] CHEN C J,CHEN H,ZHANG Y,et al.TBtools:an integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194–1202.
[25] ZHANG Z,LI J,ZHAO X Q,et al.KaKs_Calculator:calculating Ka and Ks through model selection and model averaging[J].Genomics,Proteomics & Bioinformatics,2006,4(4):259–263.
[26] LESCOT M,DÉHAIS P,THIJS G,et al.PlantCARE,a database of plant-acting regulatory elements and a portal to tools foranalysis of promoter sequences[J].Nucleic Acids Research,2002,30(1):325–327.
[27] LIU F,YU H Y,DENG Y T,et al.PepperHub,an informatics hub for the chili pepper research community[J].Molecular Plant,2017,10(8):1129–1132.
[28] BROWN J,PIRRUNG M,MCCUE L A.FQC dashboard:integrates FastQC results into a web-based,interactive,and extensible FASTQ quality control tool[J]. Bioinformatics,2017,33(19):3137–3139.
[29] BOLGER A M,LOHSE M,USADEL B.Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics,2014,30(15):2114–2120.
[30] KIM D,PAGGI J M,PARK C,et al.Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype[J].Nature Biotechnology,2019,37(8):907–915.
[31] LIAO Y,SMYTH G K,SHI W.FeatureCounts:an efficient general purpose program for assigning sequence reads to genomic features[J].Bioinformatics,2014,30(7):923–930.
[32] VARET H,BRILLET-GUÉGUEN L,COPPÉE J Y,et al. SARTools:a DESeq2- and edgeR-based R pipeline for comprehensive differential analysis of RNA-Seq data[J]. PLoS One,2016,11(6):e0157022.
[33] WANG J,YANG G,CHEN Y,et al. Genome-Wide characterization and anthocyanin-related expression analysis of the B-BOX gene family inL.[J].Frontiers in Genetics,2022,13:847328.
[34] SCHMITTGEN T D,LIVAK K J.Analyzing real-time PCR data by the comparativeTmethod[J].Nature Protocols,2008,3(6):1101–1108.
[35] 李宁,沈瑶,夏碧波,等.辣椒基因组中XTH基因家族的鉴定与表达特征分析[J].热带作物学报,2018,39(2):317–324.
[36] LI S,CHEN K S,GRIERSON D.Molecular and hormonal mechanisms regulating fleshy fruit ripening[J]. Cells,2021,10(5):1136.
[37] WANG S F,LU G,HOU Z,et al.Members of the tomatoMADS-box family regulate style abscission and fruit ripening[J].Journal of Experimental Botany,2014,65(12):3005–3014.
[38] LIU G Z,LI C X,YU H Y,et al.,encoding methylated TOMATO AGAMOUS-LIKE 1,regulates chloroplast development and Chl synthesis in fruit[J].The New Phytologist,2020,228(1):302–317.
[39] BARTLETT M E.Changing MADS-box transcription factor protein-protein interactions as a mechanism for generating floral morphological diversity[J].Integrative and Comparative Biology,2017,57(6):1312–1321.
[40] 吕山花,孟征.MADS-box基因家族基因重复及其功能的多样性[J].植物学通报,2007,42(1):60–70.
Identification and expression analysis of the MADS-box gene family in
YANG Guangbin1,WANG Jin3,6,CHEN Kailin3,SHAN Qingyun3,CUI Sufei3,XIONG Cheng2,3,4,5,ZOU Xuexiao2,3,4,5,LIU Feng2,3,4,5*
(1.Longping Branch, Hunan University, Changsha, Hunan 410125, China; 2.Laboratory of Lingnan Modern Agriculture, Guangzhou, Guangdong 510642, China; 3.College of Horticulture, Hunan Agricultural University, Changsha, Hunan 410128, China; 4.ERC for Germplasm Innovation and New Variety Breeding of Horticultural Crops, Changsha, Hunan 410128, China; 5.Key Laboratory for Vegetable Biology of Hunan Province, Changsha, Hunan 410128, China; 6.College of Horticulture, Nanjing Agricultural University, Nanjing, Jiangsu 210095, China)
In this study, 104 MADS-box genes were identified from pepper by using the whole genome data of pepper, and their physical and chemical properties, chromosome location, phylogenetic relationship, protein conserved motifs and tissue expression levels were analyzed. The results showed that the members of MADS-box gene family in pepper were uneven distribution on chromosomes and their physical and chemical properties were quite different. The length of amino acids encoded by 104 family members was 100-567 aa, the relative molecular masses of proteins were 11 203.9-63 559.7, and the theoretical isoelectric points(PI) of proteins were 4.63-10.46. Phylogenetic analysis showed that the MADS-box gene family of pepper could be divided into two categories, which were similar to the evolutionary relationship betweenand tomato. The analysis of tissue expression level showed that CaMADSs were mainly expressed in flowers, fruits and seeds, and was less expressed in leaves, speculating that MADS-box gene might be involved in regulating fruit development and ripening.
pepper; MADS-box gene family; gene expression
S641.3;Q786
A
1007–1032(2023)05–0558–09
杨光彬,王瑾,陈恺琳,单庆云,崔苏菲,熊程,邹学校,刘峰.辣椒MADS–box基因家族的鉴定及表达分析[J].湖南农业大学学报(自然科学版),2023,49(5):558–566.
YANG G B,WANG J,CHEN K L,SHAN Q Y,CUI S F,XIONG C,ZOU X X,LIU F.Identification and expression analysis of the MADS-box gene family in[J].Journal of Hunan Agricultural University(Natural Sciences),2023,49(5):558–566.
http://xb.hunau.edu.cn

2022–12–06
2023–10–18
国家自然科学基金项目(32002058、32130097);国家自然科学基金联合基金项目(U19A202);湖南省科技创新计划项目(2021JC 0007)
杨光彬(1999—),女,湖南常德人,硕士研究生,主要从事辣椒果实发育及分子育种研究,gbinyang@163.com;*通信作者,刘峰,博士,研究员,主要从事辣椒种质资源重要性状功能基因挖掘及新种质创制研究,liufengrich@126.com
10.13331/j.cnki.jhau.2023.05.009
责任编辑:毛友纯
英文编辑:柳正

