应用生物信息学筛选胃癌诊断和预后的生物标志物
董金凤 郑华川
承德医学院附属医院中心实验室,河北省承德市 067000
胃癌是全球最常见和癌症致死率最高的疾病之一。寻找胃癌生物标志物对早期胃癌患者的确诊、预后改善和生存率的提高具有重要意义[1]。 近年来,微阵列技术和生物信息学分析被广泛应用于寻找肿瘤诊断、治疗及预后的生物标志物[2]。本研究从GEO数据库下载GSE79973和GSE103236两数据集以获得胃癌组织和邻近非癌组织的差异表达基因(Differentially expressed genes,DEGs),并进行分析。报道如下。
1 材料与方法
1.1 下载数据集 从GEO数据库(http://www.ncbi.nlm.nih.gov/geo)下载胃癌的基因表达谱数据集GSE79973和GSE103236。GSE79973数据集包含10个胃癌组织样本和10个匹配的邻近非肿瘤组织样本;GSE103236数据集包含10个胃癌组织样本和9个匹配的邻近非肿瘤组织样本。
1.2 筛选差异基因 通过GEO2R在线工具分析两数据集中胃癌样本和邻近非癌组织样本的DEGs,adj.P<0.05和|logFC|>1被作为识别DEGs的标准,并将uDEGs定义为上调基因(up-regulated DEGs),dDEGs定义为下调基因(down-regulated DEGs),并使用仙桃在线数据平台(https://www.xiantao.love)绘制火山图。然后,通过维恩图在线工具(https://www.bioinformatics.com.cn)识别两个数据集之间重叠的DEGs,并绘制维恩图。
1.3 GO和KEGG富集分析 为更好地探索两数据集重叠DEGs的生物学意义,通过仙桃在线数据平台进行GO和KEGG富集分析。GO主要用于识别生物学功能, KEGG主要用于富集信号通路。
1.4 构建PPI网络和筛选hub基因 将两个数据集之间重叠的DEGs导入STRING在线工具(https://string-db.org)获得重叠 DEGs编码蛋白的PPI网络,“minimum required interaction score”为“medium confidence (0.4)”被作为限制条件。通过 Cytoscape 软件(3.9.1版)可视化DEGs基因的PPI网络,cytoHubba插件筛选PPI网络中连接度前十的hub基因。
1.5 hub基因的表达差异分析和生存分析,以及ROC曲线分析 使用GEPIA数据平台(http://gepia.cancer-pku.cn)的TCGA-GTEx数据库分析hub基因在胃癌样本和邻近非癌样本的表达差异。利用仙桃在线数据平台的TCGA在线数据库绘制hub基因的ROC曲线。最后,通过Kaplan-Meier Plotter在线数据平台(http://kmplot.com/analysis)绘制hub基因的总体生存率(Overall survival,OS)生存曲线。
2 结果
2.1 差异基因的识别 在GSE79973数据集中,487个基因被上调,919个基因被下调(见图1a)。在GSE103236数据集中,331个基因被上调,170个基因被下调(见图1b)。两数据集重叠的DEGs有156个,其中包括98个上调基因和58个下调基因(见图1c)。
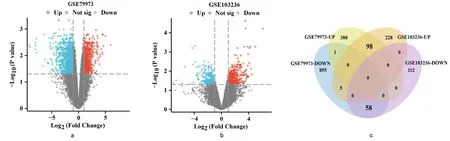
图1 两数据集DEGs的筛选与识别
2.2 重叠DEGs的GO和KEGG富集分析 为进一步探索重叠DEGs在胃癌中的生物学功能,我们通过仙桃在线数据平台对重叠DEGs进行GO和KEGG富集分析。uDEGs的GO分析主要富集在细胞外结构组织、细胞外基质组织、胶原蛋白纤维组织、含胶原蛋白的细胞外基质、内质网腔、胶原蛋白三聚体、细胞外基质结构成分、胶原蛋白结合、赋予拉伸强度细胞外基质结构成分(见图2a)。KEGG分析主要富集在蛋白质消化和吸收、细胞外基质受体相互作用(见图2a)。而dDEGs的GO分析主要富集在细胞二价无机阳离子稳定、肌肉系统过程、脂质定位、肌肉收缩、p型离子转运体活性、焦磷酸水解驱动的质子跨膜转运体活性、儿茶酚胺结合、胆汁酸结合(见图2b)。dDEGs的KEGG分析主要富集在钙信号通路、胃酸分泌、组氨酸代谢(见图2b)。
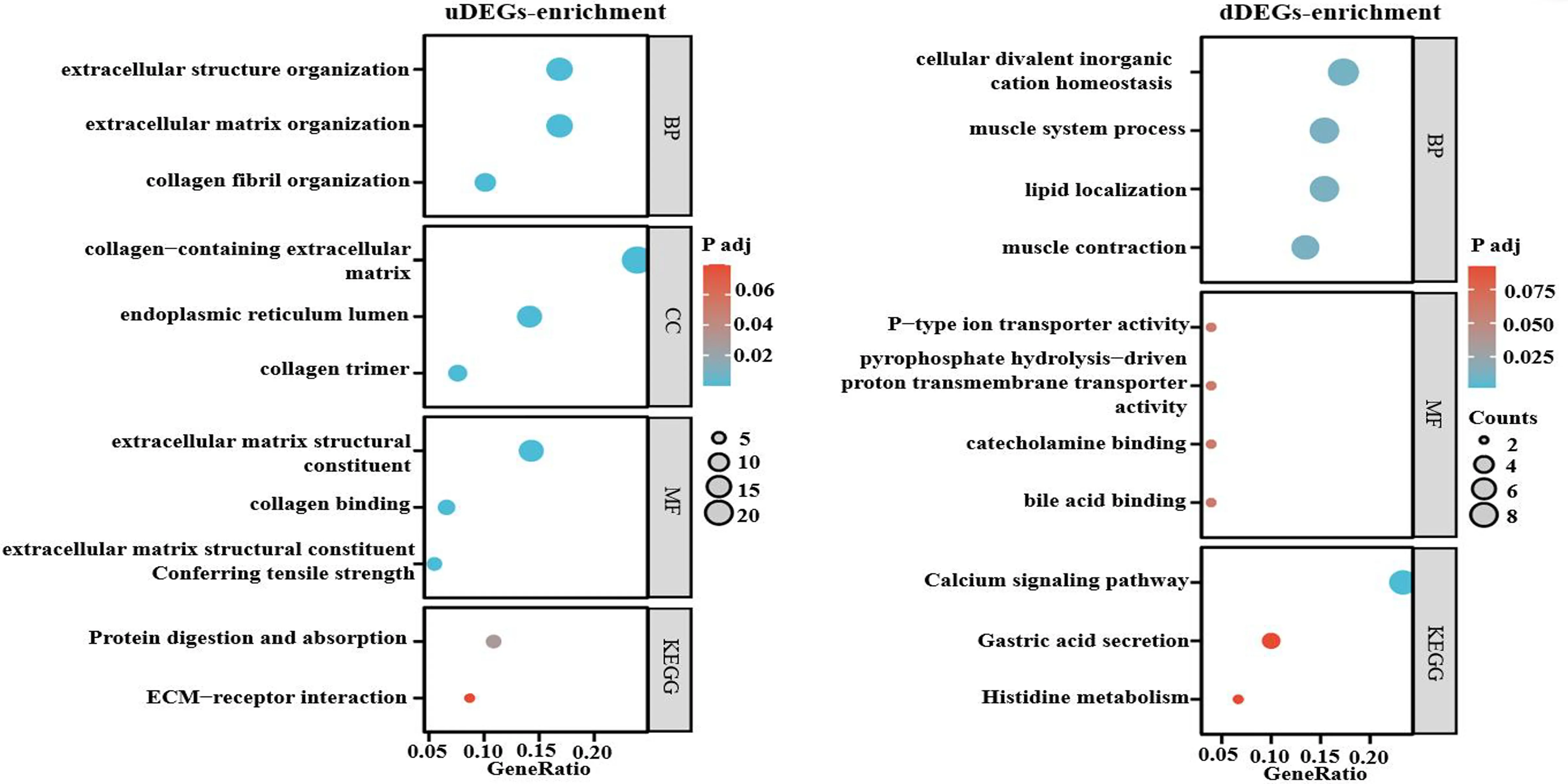
a b
2.3 构建PPI网络和筛选hub基因 为了解两数据集重叠DEGs编码蛋白之间的关系,利用STRING在线工具构建重叠DEGs的PPI网络,并通过Cytoscape软件可视化(见图3a)。使用Cytohubba插件识别PPI网络中连接度前十的hub基因,分别是COL1A1、BGN、SPARC、MMP14、LOX、THBS2、TIMP1、SPP1、VCAN、COL5A2(见图3b),且这10个hub基因均为uDEGs。
2.4 胃癌组织中hub基因的表达分析 我们使用GEPIA数据平台的TCGA-GTEx数据库进一步分析了胃癌组织中10个hub基因的mRNA表达水平。结果显示,这些基因在胃癌组织中的表达水平均显著高于胃正常组织(P<0.01),见图4。

a b

图4 hub基因在胃癌组织中高表达
2.5 评价hub基因的诊断价值 通过仙桃在线数据平台的在线TCGA数据库绘制ROC曲线评价hub基因对胃癌的诊断价值(见图5)。结果显示,10个hub基因的AUC 均>0.84,均具有一定诊断价值,其中COL1A1、BGN、TIMP1、SPP1、COL5A2>0.9,诊断价值较高。

图5 hub基因的ROC曲线
2.6 生存分析 为了评估hub基因在胃癌的预后价值,我们通过Kaplan-Meier Plotter在线数据平台进一步绘制了OS生存曲线(见图6)。表明10个hub基因均与较差的总体生存率显著相关 (P<0.01),提示这些基因可能是胃癌预后不良的生物标志物。
3 讨论
胃癌是全球肿瘤死亡的最常见原因之一。早期胃癌可以通过手术根治性切除,肿瘤生物标志物可为早期诊断提供诊断依据,并为分子治疗提供靶点[1,3]。
本研究通过生物信息学方法筛选胃癌的生物学标志物。我们分析了GSE79973和GSE103236两数据集重叠的DEGs,其中uDEGs的GO分析主要与细胞外基质及胶原蛋白相关。细胞外基质由胶原蛋白、纤连蛋白、弹性蛋白、层粘连蛋白、透明质酸、糖胺聚糖等多种成分组成[4]。在正常组织和器官中细胞外基质不仅可以发挥物理支架的作用和介导信号的转导,还可参与细胞形状、迁移、存活、增殖等多种细胞生物学行为的调节[5]。当细胞外基质的硬度和刚度发生病理变化时,可通过本身的物理性质和信号通路的激活促进恶性肿瘤细胞增殖、转移和血管生成的能力,并降低放化疗及靶向治疗的疗效[4]。胶原蛋白是细胞外基质的主要成分,与器官组织的纤维化、细胞外基质硬度增加、促进血管生成和引导肿瘤细胞的迁移和侵袭密切相关[6]。uDEGs的KEGG富集分析显示与细胞外基质受体相互作用有关。我们通过STRING在线工具和Cytoscape软件从两数据集重叠的DEGs中识别的10个hub基因均为uDEGs,提示这些基因可能与细胞外基质成分及其生物学行为密切相关。据报道,细胞外基质在正常胃上皮细胞—癌前病变—胃癌—胃癌进展整个过程均具有促进作用,是胃癌发生和发展的帮凶[7]。利用GEPIA、仙桃、Kaplan-Meier Plotter在线数据平台分析表明,hub基因在胃癌组织中均显著上调(P<0.01),具有一定诊断价值(AUC>0.84),并预示其预后不良 (P<0.01)。因此,这10个hub基因可能是诊断胃癌和判断预后的潜在生物标志物。
研究表明,COL1A1和COL5A2作为胶原蛋白的组成成分在胃癌组织中均高表达,对胃癌细胞的迁移和侵袭有促进作用[8-9]。BGN是一种富含亮氨酸的小蛋白,其高表达与胃癌的淋巴结转移、血管侵犯、复发率及较差的生存期密切相关[10]。SPARC是一种糖蛋白,在胃癌中高表达,与预后不良显著相关[11]。然而,有研究显示SPARC在胃癌中具有抑癌作用,抑制胃癌血管生成、增殖及迁移、侵袭及远处转移的能力[12]。基质金属蛋白酶是细胞外基质的蛋白水解酶,除了在侵袭和转移中起重要作用,还参与细胞的生长、增殖、凋亡及肿瘤血管生成和免疫逃逸的调节[13]。MMP14是基质金属蛋白酶家族成员,可通过调控细胞的增殖、侵袭、迁移和凋亡调节胃癌细胞的生长[14]。LOX是一种铜依赖性单胺氧化酶,在弥漫型胃癌细胞中高表达,与胃癌远处转移相关[15]。THBS2是血小板反应蛋白家族成员,在胃癌组织中表达上调,其表达下调对胃癌细胞的增殖、迁移和侵袭有抑制作用,对凋亡有促进作用[16]。TIMP-1是一种金属蛋白酶抑制剂,据报道TIMP-1阳性患者的复发率明显高于阴性患者,并且是不良预后的标志物[17]。SPP1(也称骨桥蛋白)是一种基质糖蛋白,在胃癌组织中显著高表达,其在血浆中的表达水平与胃癌向邻近组织的浸润和转移,以及预后不良密切相关[18]。VCAN是一种蛋白多糖,参与细胞外基质的组成,其高表达与晚期TNM分期、淋巴结转移、浸润深度和肿瘤分级相关,对胃癌细胞的增殖、迁移和侵袭具有促进作用[19]。因此,我们筛选出的10个hub基因均与胃癌的发生发展密切相关。
综上所述,本研究应用生物信息学方法从胃癌基因表达谱数据集中筛选的10个hub基因COL1A1、BGN、SPARC、MMP14、LOX、THBS2、TIMP1、SPP1、VCAN、COL5A2在胃癌中高表达,与胃癌的生物学行为及不良预后密切相关,可能是胃癌诊断和预后不良的潜在生物标志物。

