大豆抗旱遗传位点及候选基因发掘
田蕊, 张华, 黄玫红, 邵振启, 李喜焕, 张彩英
(河北农业大学农学院,教育部华北作物种质资源研究与利用重点实验室,河北 保定 071001)
大豆是重要的粮食和油料作物,是植物蛋白和食用油的主要来源[1]。干旱严重影响大豆生长发育,干旱条件下大豆植株生长缓慢,叶片萎蔫,生物量降低,以至减产甚至绝收。叶片是大豆植株重要光合器官,其叶绿素含量与光合速率及产量密切相关,易受干旱影响,是反映品种抗旱性的重要指标[2]。已有研究对作物干旱条件下的叶绿素含量进行定位分析,沈波等[3]利用水稻重组自交系(recombinant inbred lines, RIL)群体,在水分胁迫和正常条件下对叶绿素含量进行连锁分析,定位到13 个控制叶绿素含量的数量性状基因座(quantitative trait locus,QTL),其中水分胁迫条件下6 个,解释表型变异4.74%~19.86%;胡颂平等[4]以水稻F9重组自交系(珍汕97B×IRAT109)为材料,在正常与水分胁迫下对叶绿素含量进行定位,检测到13 个叶绿素含量QTLs,其中正常条件下检测到7个,联合贡献率为56.19%;干旱处理下检测到6 个,联合贡献率为47.39%。Kumar 等[5]利用C306×HUW206 构建的小麦RIL 群体在干旱条件下定位到1 个控制叶绿素含量QTL(QChl.ksu-3B),该QTL 解释表型变异的14.2%。Peleg 等[6]以硬粒小麦和野生二粒小麦构建的RIL 群体为材料,在干旱及正常水分条件下,对叶绿素含量进行QTL定位,获得8个叶绿素含量QTLs,解释表型变异的2%~10%。
崔世友等[7]以‘波高’ב南农94-156’构建RIL群体为材料,在4 个不同生育期对大豆叶绿素含量进行连锁分析,定位到与叶绿素含量相关的10 个位点,分别位于1、7、12、13、18 和19 号染色体,解释表型变异的6.9%~23.4%。Fang 等[8]利用809 份大豆自然群体重测序,对叶绿素含量进行关联分析,获得9 个大豆叶绿素含量相关位点,分别位于11、13、18、20 号染色体。Kato 等[9]利用‘Minsoy’בT225H’构建的F2:3家系在14 及17 号染色体定位到2 个叶绿素相关位点。由此可见,相较其他农作物,目前大豆多就正常水分条件下叶绿素含量进行研究。因此,本研究以199 份大豆品种资源构建的自然群体为材料,在干旱和正常浇水条件下,测定其叶绿素含量,并计算抗旱指数,结合20×重测序基因型数据进行关联分析,发掘基于叶绿素含量的抗旱遗传位点和相关基因,为大豆品种抗旱分子遗传改良提供依据。
1 材料与方法
1.1 供试材料
以本课题组前期构建的大豆自然群体为材料,包括152个育成品种和47个地方品种[10]。
1.2 大豆种植方法
将蛭石和土以体积比1∶1 进行混合,装入花盆(直径35 cm,高40 cm,花盆下铺塑料布),每盆装土20 kg,施复合肥5 g,浇水6 L;当基质含水量为20%时播种,每盆播种20 粒;待2 片真叶完全展开时定苗,每盆保留10株。试验分别于2021年6月(E1)、7月(E2)和8月(E3)在河北农业大学作物育种中心试验基地旱棚内进行,设置对照组和干旱胁迫处理组,2 次重复,其中干旱胁迫处理组于大豆V3 期开始进行干旱处理,对照组正常浇水,期间利用托普云农土壤水分含量测定仪对土壤水分含量进行测定,待干旱处理组的土壤含水量低于10%(植株受到干旱胁迫)时测定叶绿素含量[11-12]。
1.3 叶绿素含量测定
利用叶绿素仪SPAD-502Plus 分别测定干旱处理组和对照组的叶绿素含量。每盆随机选择3 个单株,分别测定其最上部完全展开三出复叶的叶绿素含量,其中每个叶片选择非叶脉处测定3次。
1.4 叶绿素含量抗旱指数计算
利用干旱处理及正常浇水条件下的叶绿素含量计算抗旱指数(drought resistance index,DRI),公式如下。
式中,CCD为干旱条件下叶绿素含量;CCW为正常条件下叶绿素含量;CCDM为干旱条件下供试大豆自然群体叶绿素含量平均值。
1.5 叶绿素抗旱指数遗传变异分析方法
利用SPSS 25.0 对基于叶绿素含量计算的抗旱指数进行描述统计及方差分析,利用公式计算其广义遗传力(broad-sense heritability,h2)[10]。
1.6 叶绿素抗旱指数关联分析方法
利用GEMMA(genome-wide efficient mixed model association algorithm)软件,采用混合线性模型(mixed linear model, MLM)对叶绿素抗旱指数进行全基因组关联分析(genome-wide association study, GWAS),其中供试大豆自然群体的基因型数据包含566万个单核苷酸多态性(single nucleotide polymorphism,SNP)标记[10];此外,参照已有报道根据LD(linkage disequilibrium)衰减距离,同一LD区域显著关联SNP视为1个位点[10,13]。
1.7 候选基因的筛选
在显著关联SNP 上、下游100 kb 筛选候选基因,结合大豆公共数据库Soybase(https://www.soybase.org/)注释基因功能,初步筛选候选基因;然后利用已有大豆干旱胁迫条件下的转录组数据[14],经标准化处理分析各基因表达量,根据基因差异表达结果确定候选基因。
2 结果与分析
2.1 大豆自然群体叶绿素含量抗旱指数遗传变异分析
对199 份供试大豆品种资源进行叶绿素含量抗旱指数方差分析(表1)发现,每个环境条件下的供试品种资源抗旱指数均具有极显著差异,说明干旱胁迫处理有效,品种资源抗旱能力存在较大差异。同时发现,不同环境下的叶绿素含量抗旱指数也存在差别,其平均值由大到小依次为E3、E2、E1,变化范围为1.35~1.48。进一步分析不同品种资源叶绿素抗旱指数遗传变异(表1)发现,变异系数为8.71%~11.64%,以E2 环境条件下的变异系数最大。分析叶绿素含量抗旱指数广义遗传力发现,其值为81.44%,表明主要由遗传因素决定。另外,分析叶绿素含量抗旱指数的偏度和峰度系数发现,绝对值均趋近于0,说明叶绿素抗旱指数属多基因控制数量性状(图1)。
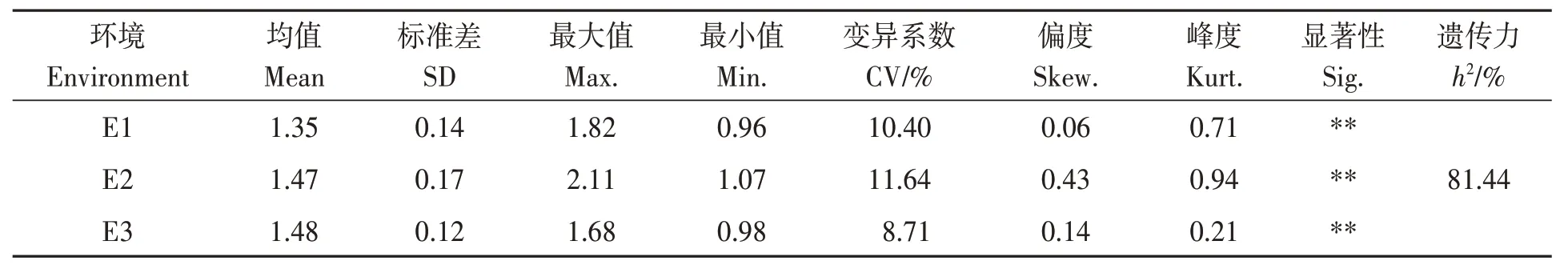
表1 大豆自然群体抗旱指数遗传变异分析Table 1 Genetic variation analysis of drought resistance index in soybean natural population
2.2 大豆叶绿素含量抗旱指数GWAS分析
对3 种环境条件下的叶绿素抗旱指数及其平均值、最佳线性无偏预测(best linear unbiased prediction,BLUP)值进行关联分析(图1)发现,关联到237 个SNPs,分别位于1、2、3、7、10、16、17 及18 号染色体,其中,11 个SNPs 位于基因外显子并引起非同义突变,28 个位于基因上游或下游,2 个位于基因3’UTR,这些SNPs位点可能影响基因功能或调控基因表达。此外,依据LD 衰减距离(100 kb)可将237 个SNPs 划分为18 个位点,其中位点1、5、6、11、12、13、14、15、17、18 在E1、E2、E3、平均值及BLUP 值中均被关联到,属多环境关联稳定位点(表2)。
进一步分析发现,在16号染色体关联到181个SNPs(分布于Locus 10~Locus 14),占总SNPs 的76.37%,其中4 个位点在上述3 种环境以及平均值和BLUP 值同时关联到,说明这4 个位点不仅是多环境稳定位点,而且是控制叶绿素抗旱指数热点区域;并且,上述181 个SNPs 中,49 个位于基因内,其中9 个在内含子,17 个在外显子(6 个属于非同义突变,9 个属于同义突变,2 个属于提前终止),11 个在基因上游,10 个在下游,2 个在3’UTR。此外,在10 号染色体关联到27 个SNPs(位于Locus 5~Locus 9),其中Locus 5 和Locus 6属多环境稳定关联位点,且在上述27 个SNPs 中,2 个位于基因上游或下游,2 个在内含子区域。
2.3 叶绿素抗旱指数显著关联SNP验证
为验证显著关联SNPs及其位点准确性,选取18 个位点的最显著P值关联SNPs,对参试品种含有的叶绿素抗旱指数优异等位SNPs 数目及其抗旱指数进行回归分析,结果(图2)表明,随优异等位变异数目增加,叶绿素抗旱指数逐渐增加(R2=0.76)。依据参试品种叶绿素抗旱指数,分别选取高、低极端品种各5个,对每个品种含有的18个位点中的11 个纯合SNPs 数目进行分析,结果发现(表3),5个抗旱品种中,3个具有所有的11个优异等位变异,另2 个分别含有9 和10 个优异等位变异,而5个敏感品种则不含任何优异等位变异。
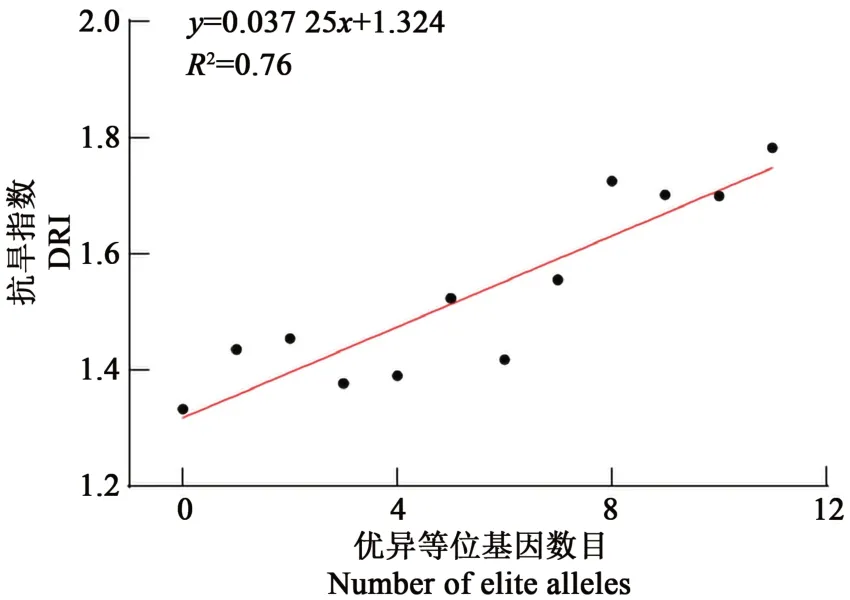
图2 大豆叶绿素抗旱指数与优异等位变异数量回归分析Fig. 2 Regression analysis of drought resistance index and elite allele numbers of soybean accessions

表3 大豆抗旱特异种质显著关联SNP分析Table 3 Analysis of associated SNPs in different soybean accessions with different drought resistance index
2.4 大豆抗旱相关基因发掘
在显著关联SNPs上、下游100 kb范围内寻找到57 个候选基因(表4),其中10 个基因外显子存在等位变异,10 个基因内含子存在等位变异,15 个基因上游或下游存在等位变异,2 个基因3’UTR存在等位变异。基于此,根据Soybase网站(https://www.soybase.org)Wm82 基因组注释信息,从57 个候选基因中初步筛选出16 个相关基因,分别参与植物激素信号转导、蛋白磷酸化、抗旱反应等;利用已有大豆转录组数据[14]分析候选基因表达量(图3),最终筛选出3 个抗旱相关基因,即Glyma.16G063600、Glyma.10G007000和Glyma.17G143900。

图3 大豆抗旱候选基因表达分析Fig. 3 Expressions of candidate genes for soybean drought tolerance

表4 大豆抗旱指数候选基因Table 4 Candidate genes of drought resistance index in soybean
Glyma.16G063600存在1 个非同义突变(C/A,天冬氨酸/谷氨酸)、3 个同义突变、2 个下游SNPs、17 个基因间SNPs(图4)。该基因编码F-box 蛋白,可与小热激蛋白结合,进而调控植物耐旱性[15]。依据已发表转录组数据[14]发现,该基因受干旱胁迫诱导显著上调表达。利用该基因的非同义突变分析供试群体发现,含有优异等位变异A与含有非优异等位变异C的品种间的叶绿素抗旱指数差异极显著。
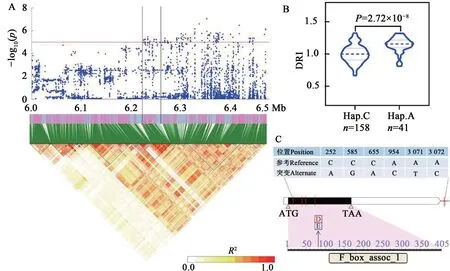
图4 候选基因Glyma.16G063600分析Fig. 4 Identification of candidate gene Glyma.16G063600
Glyma.10G007000编码AP2-EREBP 转录因子。AP2/EREBP 转录因子含有1 个或多个AP2/ERF 结构域,该类转录因子可响应植物盐、旱、冷等多种非生物胁迫[16]。转录组数据分析发现,干旱胁迫条件下,Glyma.10G007000表达显著上调;利用其上游SNP 分析群体发现,含有优异等位变异的品种其叶绿素抗旱指数显著高于含有非优异等位变异的品种(图5)。

图5 候选基因Glyma.10G007000分析Fig. 5 Identification of candidate gene Glyma.10G007000
Glyma.17G143900编码AP2/ERF 结构域蛋白。利用基因间最显著SNP 分析群体发现,含有优异等位变异T的品种其叶绿素抗旱指数显著高于含有非优异等位变异C的品种(图6)。

图6 候选基因Glyma.17G143900分析Fig. 6 Identification of candidate gene Glyma.17G143900
2.5 大豆抗旱优异基因型筛选
利用Glyma.10G007000、Glyma.16G063600及Glyma.17G143900等位变异,对供试品种资源进行分析,结果(表5)发现,可将其分为8种类型,其中基因型Ⅶ(AAT)含3 个优异等位变异,其叶绿素抗旱指数最高;基因型Ⅳ(CAT)、基因型Ⅵ(ACT)及基因型Ⅷ(AAC)含有2 个优异等位变异,叶绿素抗旱指数次之;而不含任何优异等位变异的基因型Ⅰ(CCC)叶绿素抗旱指数最低,说明3 个候选基因与干旱胁迫下的抗旱指数相关,为大豆抗旱育种提供了基础材料。

表5 供试大豆自然群体基于入选抗旱基因等位变异分析Table 5 Analysis of soybean natural population based on the allele variations of three selected causal genes
3 讨论
叶绿素是光能转化为化学能的关键色素[17]。对干旱条件下的叶绿素含量进行研究发现,随干旱胁迫增加,叶绿素含量呈现先增加后降低的趋势,说明叶绿素含量受干旱胁迫的影响[18-19]。抗旱指数是指性状抗旱系数与干旱条件下该性状指数的乘积[20]。刘桂茹等[21]对小麦抗旱系数、干旱敏感系数、单株产量降低指数分析发现,抗旱指数能反应品种的抗旱性,可作为小麦抗旱筛选的重要指标。杨玉敏等[22]以100 份小麦资源为材料,对抗旱系数、抗旱指数、耐旱指数进行分析发现,抗旱指数综合考虑了各品种在干旱条件下的敏感度和干旱胁迫下目标性状在整个群体中的地位,比较适合作为作物抗旱性评价的标准。基于此,本研究利用干旱处理和正常浇水处理下的叶绿素含量计算抗旱指数,用于大豆品种资源抗旱种质筛选以及遗传位点与候选基因挖掘;并依据群体供试品种资源的叶绿素抗旱指数,筛选出优异种质5 份,分别是‘冀10B5’‘沧0627’‘邯15-685’‘冀11B9’和‘HN1030’,其抗旱指数为1.70~1.83;进一步分析发现,其含有9~11 个优异等位变异,且均含有3 个入选相关基因的优异等位变异,为大豆抗旱育种奠定了重要物质基础。
目前,有关水稻、小麦等作物的干旱胁迫叶绿素含量遗传位点发掘研究已有报道,但大豆中开展相关研究较少[23-26]。本研究利用SPAD-502Plus测定干旱及正常条件下供试大豆自然群体叶绿素含量,计算抗旱指数,并依据本团队前期重测序数据对群体的叶绿素含量抗旱指数进行关联分析,结果获得18 个位点,通过与已有研究进行比较发现,Locus3 与李广军等[27]定位到的叶绿素含量QTLLeaflet chlorophyll 1-4重合,其加性效应值为0.97,解释表型变异5%。同时发现,本研究定位到的Locus15与Du等[28]定位到的干旱敏感指数相关QTLDrought index 1-9距离700 kb,该QTL 加性效应值为0.128 1,可解释表型变异6.42%;且QTLDrought index 1-5与本研究中的Locus10 相距4.2 Mb,该QTL 解释表型变异9.36%。另外,本研究获得的Locus3 与Carpentieri 等[29]定位到的耐旱相关QTLDrought tolerance 6-1及Shi 等[30]定位到的叶绿素相关QTLLeaflet chlorophyll 3-1相距2.2 Mb。上述结果证实了本研究结果的可靠性,为大豆抗旱遗传改良提供了选择标记。
为筛选大豆抗旱相关基因,依据关联区域内的基因SNP突变、基因注释及其表达量筛选出3个抗旱相关基因,其中候选基因Glyma.16G063600编码F-box 蛋白,该类蛋白既可调控植物生长发育,也可对植物多种生物及非生物胁迫作出响应[31-32]。Qu 等[33]发现,与拟南芥F-box 基因突变体fof2相比,过表达FOF2株系表现为ABA 含量增加及耐旱性提高;Lim 等[34]发现,辣椒F-box 类基因CaDIF1沉默植株表现出对干旱胁迫的敏感性,而过表达CaDIF1植株则表现为耐旱;Xu 等[15]发现,在干旱条件下,过表达大豆F-box 类基因GmFBL144可提高拟南芥过氧化氢及丙二醛含量。本研究筛选的F-box蛋白基因Glyma.16G063600与上述已报道的GmFBL144属同一亚族,说明其可能在大豆抵御干旱胁迫逆境中具有重要作用。
本研究还筛选出AP2/EREBP(APETALA2/ethylene-responsive element binding proteins)转录因子家族基因Glyma.10G007000,该类转录因子通常包含1 或2 个AP2/ERF 结构域,依据结构域数目,可将其分为2 个亚族,即EREBP 亚族(包含1 个AP2/ERF 结构域)和AP2 亚族(包含2 个AP2/ERF 结构域);AP2 亚族可调控花、胚珠及种子发育过程,EREBP 亚族(包括DREB 和ERF 类)参与植物激素调节,并响应非生物胁迫[16]。研究发现,过表达苹果AP2/EREBP 基因MdSHINE2可增加拟南芥植株茎和叶表面蜡质,并提高其抗旱性[35]。大豆DREB 同源基因GmDREB2在干旱、高盐及低温等非生物胁迫条件下被诱导表达,且过表达该基因会激活下游转录因子进而提高抗旱性[36]。因此,推断AP2/EREBP 转录因子基因Glyma.10G007000可能参与大豆抗旱反应。
另外,本研究还获得了诸如MYB 转录因子、Dof (DNA binding with one finger)结构域蛋白、CCR-4(carbon catabolite repressor 4)、LRR(Leucinerich repeat)候选基因,已有研究发现这些蛋白均可响应植物干旱胁迫,Cominelli 等[37]发现,AtMYB60通过调控叶片保卫细胞的开关影响植物抗旱;Ma 等[38]在干旱胁迫下对白菜9 个Dof 亚族基因表达模式进行分析发现,不同亚族Dof基因均可响应干旱胁迫;Li 等[39]发现,大豆LRR-RLK基因GmSARK可通过调控叶绿素降解基因Gmlls1及叶绿素合成基因Gmgtr1调控叶片衰老,说明该类基因位于Gmlls1和Gmgtr1上游,可间接调控叶绿素合成或降解。因此,本研究筛选出的这些候选基因可作为今后开展大豆抗旱分子育种研究的重要目标基因。

