渤海湾7种虾虎鱼科鱼类COI基因序列特征及分子分类研究
房恩军 郭彪 郑德斌 张 雪 王硕 王宇


摘 要:为获得渤海湾天津海域常见虾虎鱼线粒体条形码序列及系统分类信息,采集渤海湾7种虾虎鱼样本,通过形态初步鉴定物种,并提取DNA进行线粒体COI基因部分序列扩增测序,用系统进化分析及分子鉴定软件MEGA对渤海湾常见12种虾虎鱼COI基因部分片断进行序列特征分析及分子分类分析。扩增得到7种虾虎鱼COI基因片断,分析得到其平均GC含量低于AT含量,碱基在密码子位置分布中具有偏向性,且GC含量最高为第2位碱基,与多种鱼类COI基因碱基特点相似。基因密码子碱基变异率为19.4%,转换颠换比为1.3。遗传距离分析及系统进化树分析理清了渤海湾天津海域12种主要虾虎鱼的系统分类及进化关系。遗传距离显示,矛尾刺虾虎鱼(Amblychaeturichthys hexanema)和斑尾复虾虎鱼(Synechogobius ommaturus)为同种鱼。虾虎鱼COI编码基因密码子高变异率可能与其良好的环境适应性有关。
关键词:渤海湾虾虎鱼;线粒体细胞色素c氧化酶亚基(COI);系统进化
中图分类号:Q917.4 文献标识码:A DOI 编码:10.3969/j.issn.1006-6500.2023.09.006
Sequence and Molecular Classification of COI Gene of Seven Species of Goby from the Bohai Bay
FANG Enjun,GUO Biao,ZHENG Debin,ZHANG Xue,WANG Shuo,WANG Yu
(Tianjin Fisheries Research Institute, Center for Marine Ranching Engineering Science Research of Tianjin,Tianjin 300221, China)
Abstract: In order to clarify the mitochondrial barcode sequence and taxonomic information of common goby fish in Tianjin area of Bohai Bay, seven species of goby fish were collected from Bohai Bay, identified by morphology, and DNA was extracted for amplification and sequencing of mitochondrial COI gene fragments. Phylogenetic analysis and molecular identification software MEGA were used to analyze the sequence characteristics and molecular classification of COI gene fragments of 12 common goby species in Bohai Bay. Seven COI gene fragments of Goby were amplified and sequenced, the average GC content was lower than AT content, the base had a bias in the codon position distribution, and the highest GC content was the second base, which was similar to the COI gene base of many fishes. The codon base variation rate was 19.4%, and the ratio of transitions to transversions was 1.3. Phylogenetic distance analysis and phylogenetic tree analysis clarified the phylotaxonomic and evolutionary relationships of 12 major goby species in Tianjin area of Bohai Bay. Genetic distance indicated that Amblychaeturichthys hexanema and Synechogobius ommaturus were the same species. The high codon variation rate of codon encoding gene in Goby may be related to its good environmental adaptability.
Key words: Goby of the Bohai Bay; cytochrome oxidase subunit I (COI) gene; phylogenetic analysis
渤海灣是一个浅水淤泥质半封闭海湾,历史上饵料丰富,基础条件优越,是渤海鱼类的产卵场。近年来,由于围填海工程、水域污染和过度捕捞等因素的影响,渔业资源的生存环境恶化,影响了其生态系统结构和功能,渔业资源衰退,小型化、低值化问题突出,种类和数量也在减少。年度调查表明,天津海域产卵场逐渐退化,与历史调查数据相比,渤海湾鱼类浮游生物的丰度和多样性显著减少,经济鱼类渔获和产卵量也逐渐减少[1-2]。而虾虎鱼由于环境适应力强,繁殖力强和生命周期短等特点[3],其资源量稳定增加,已逐渐代替其他经济鱼类,成为渤海湾天津海域春季休渔期前鱼类群落及仔稚鱼的优势种,在海洋食物网中发挥着重要的作用。但是虾虎鱼种类繁多,且有关仔稚鱼的形态相近,相关资料较少,鉴定难度大,准确性难以保证,虾虎鱼仔稚鱼鉴定也是世界难点和是鱼类早期资源研究的重要内容。
20世纪90年代以来,国内外科学家发现线粒体一些短的基因(如细胞色素氧化酶第一亚基(COI)基因、12s rRNA、细胞色素B(Cty B)等)进化速率快且相对保守,其中COI基因5'端650 bp片断可有效区分种间物种,可作为物种分类的DNA条形码(DNA Barcoding),随后与之配套的数据库也日趋完善,广泛应用于生命科学、法医学、食品质量控制等领域,也为鱼类浮游生物种类的分子鉴定奠定了良好的基础[4-9]。
虾虎鱼亚目(Gobioidei)有两千余种虾虎鱼,腹鳍合成一个吸盘,是鲈形目中最大的一个类群。蝦虎鱼多为小型底栖鱼类,主要以小型底栖甲壳动物为食,在渤海湾生态系统中能量流动和物质循环中有重要作用,是底层食物链的关键物种[10]。根据渤海鱼类生物学记载, 渤海湾天津海域历史上能采集到的虾虎鱼有12种左右[11],其中拉氏狼牙虾虎鱼(Odontamblyopus rubicundus)、斑尾腹虾虎鱼(Synechogobius ommaturus)具有一定的经济价值。除了小头栉孔虾虎鱼。常见的虾虎鱼的线粒体(COI)基因片断均能在数据库中获取。但是虾虎鱼形态多变、各地名称不统一等原因,需要通过扩增进一步明确序列信息。本研究选择渤海湾常见7种虾虎鱼类作为研究对象,通过获取线粒体(COI)基因序列分析比较其序列变异情况,进一步进行系统发育及遗传分析,从而为鱼类DNA条形码库及鱼类浮游生物的鉴定积累基础数据,为渤海海洋食物网的研究和稳定,为渤海湾虾虎鱼资源保护和合理开发利用提供科学依据。
1 材料与方法
1.1 样品采集
样品来源于2018年春季和夏季渤海湾水域地笼或拖网采样,参照《渤海鱼类生物学》共鉴定出矛尾刺虾虎鱼(Amblychaeturichthys hexanema)、裸颈缟虾虎鱼(Tridentiger Nudicervicus)、矛尾虾虎鱼(Chaeturichthys stigmatias)、红狼牙虾虎鱼(Odontamblyopus rubicundus)、弹涂鱼(Periophthalmus modestus)、钟馗虾虎鱼(Triaenopogon barbatus)、小头栉孔虾虎鱼(Ctenotrypauchen microcephalus)7种7个虾虎鱼[11]。样本用95%的乙醇混合保存后取鱼鳍。
1.2 DNA提取
采用酚-氯仿法提取DNA,取鱼尾鳍0.5 g放入1.5 mL的离心管中,将鱼鳍剪碎后加入裂解液和蛋白酶涡旋混匀,离心后于56 ℃消化3 h,酚-氯仿抽提后2倍无水乙醇沉淀,75%乙醇洗涤吹干后以ddH2O溶解,4 ℃冰箱保存。
1.3 引物设计及PCR扩增
在上海生工合成Ward[12]文献中鱼类COI通用引物:FishF1(5'-TCAACCAACCACAAAGACATTGG
CAC-3'), FishR1(5'-TAGACTTCTGGGTGGCCAAAG
AATCA-3')。
组建一个25 μL PCR反应体系:
模板DNA 2.0 μ ddHO 6.5 μlPremix Taq 12.5 μL引物 各2.0 μL
PCR扩增的程序为:
95 ℃ 2 min
95 ℃变性40 s92 ℃退火1 min72 ℃延伸1 min 35个循环
72 ℃延伸5 min
PCR扩增产物通过琼脂糖凝胶(1.5%)电泳15~20 min,凝胶电泳成像仪检测后送至上海欧易生物技术有限公司进行双向测序,测序引物为扩增引物。
1.4 数据分析
从BOLD数据库中获取鲈形目的石首鱼科黄姑鱼(Nibea albiflora)、马鲅科的四指马鲅(Eleutheronema tetradactylum)、鲽形目的舌鳎科短吻红舌鳎(Cynoglossus joyneri)、半滑舌鳎(Cynoglossus semilaevis)共4种鱼类的同源序列作为外群,同时从NCBI和BOLD数据库下载天津可能存在的虾虎鱼序列,结合测序序列进行系统进化分析(表1)。对所获得的序列利用MEGA进行多序列比对,人工核查后,用“Statistics”程序计算序列碱基组成、遗传颠换值、变异位点、密码子偏向性等。不同个体间的遗传距离用“Distance”程序下的Kimura双参数模型计算。用Modeltest检测出最适合的核苷酸序列分析模型后采用邻位连接法(Neighbor-Joining,NJ)构建进化树Bootstrap置信值重复抽样1 000次。
2 结果与分析
2.1 渤海湾7种虾虎鱼的COI基因序列特征
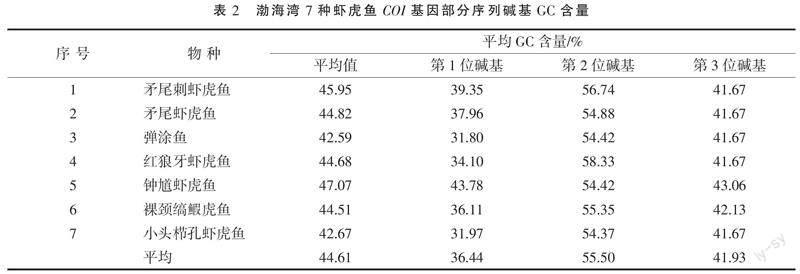
将扩增测序得到的COI序列进行blast比对,相似度大于98%的即为同一种,比对结果支持形态鉴定结果。多序列比对得到652 bp共有序列,证实COI可以用作物种鉴定条形码[8]。
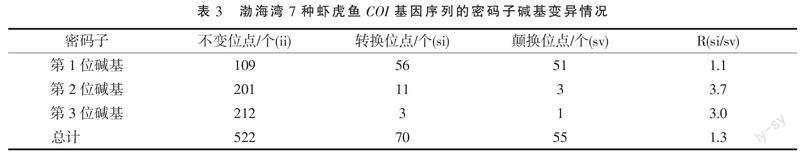
碱基特征分析得到7种虾虎鱼的COI基因部分序列符合硬骨鱼类G+C含量(44.6%)小于A+T含量(55.4%)的特点[14]。密码子的碱基使用频率存在一定的偏向性,平均GC含量最高为第2位碱基(55.50%),4种碱基在第1、2、3位密码子分布含量都不平均。本研究中COI基因密码子变异情况表明,共有不变位点(522个)多于变异位点(126个),转换位点(71个)多于颠换位点(55个),转换与颠换比为1.3,其中第1位碱基变异位点最多,第3位最少。
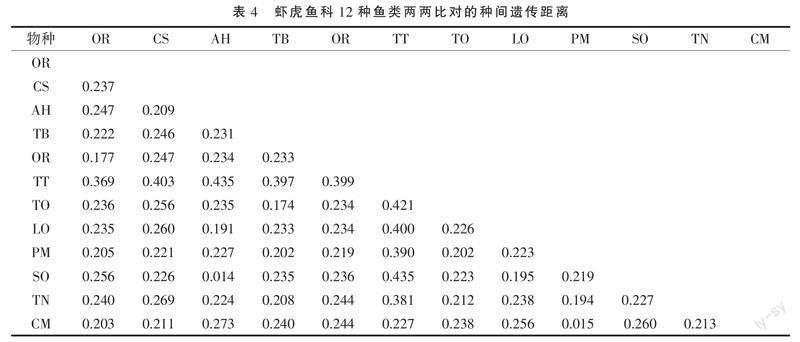
2.2 虾虎鱼科12种鱼类的种间遗传距离
12种虾虎鱼的种间遗传距离平均值为0.21。矛尾刺虾虎鱼和斑尾复虾虎鱼的种间遗传距离最小(0.014),小于0.02的临界值[13],应为同种虾虎鱼,斑尾复虾虎鱼和纹缟虾虎鱼种间遗传距离最大(0.435)。
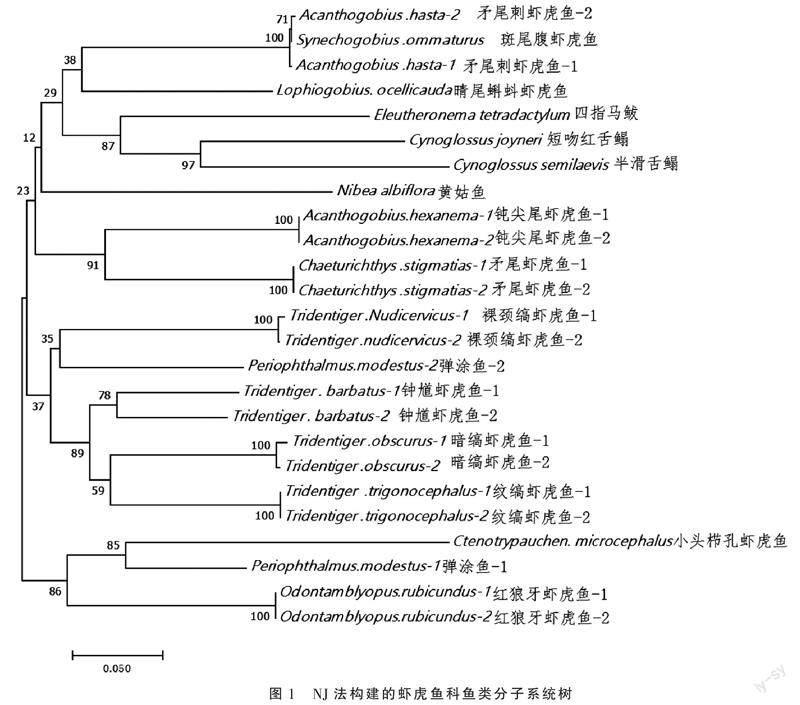
2.3 虾虎鱼科鱼类的分子系统进化树
系统树显示,7种虾虎鱼中,红狼牙虾虎鱼与矛尾刺虾虎鱼距离较远,小头栉孔虾虎鱼和弹涂鱼聚为一支,矛尾刺虾虎鱼和斑尾复虾虎鱼聚为一支,纹缟虾虎鱼和暗缟虾虎鱼遗传距离较近。
3 讨论与结论
3.1 7种渤海湾虾虎鱼类的COI基因序列特点
COI基因部分序列第2位碱基的GC含量最高,与石首鱼科COI基因碱基特征一致[5],与于亚男长江口虾虎鱼科鱼类COI基因碱基特征不一致等[15]。第3位碱基的GC含量比较恒定,而第1位碱基则因种类不同而呈现含量差异,第1位碱基能否作为不同种类区分的特征也需要进一步扩大样本量做深入的研究。本研究中7种虾虎鱼的密码子碱基使用存在一定偏向性,但与李明晖等[16]研究结果不一致。由于其与基因长度、碱基组分、翻译起始效应、基因的表达水平等多种因素有关[16-17],需要系统地对虾虎鱼亚目不同地理群体的该基因密码子碱基使用情况进行分析再下结论,或者精确到不同科属的具体情况来进行探讨。7种虾虎鱼的COI基因颠换大于转换,转换/颠换值(R)值为1.3小于2,且第1位碱基承担着主要选择压力,表明虾虎鱼可能通过高度基因突变实现快速进化,从而增强其抗逆性和适应性[9,18]。
3.2 COI基因在渤海湾天津海域虾虎鱼科鱼类分类中的应用
虾虎鱼的COI基因的种间和种内遗传距离相差20倍以上,可作为天津海域虾虎鱼科鱼类分子分类的条形码和仔稚鱼分子鉴定的依据[13]。
在系统进化树上,12种虾虎鱼节点支持率较高,除了刺虾虎鱼属、晴尾蝌蚪虾虎鱼与其他虾虎鱼遗传距离较远,其他虾虎鱼均聚为一大类。于亚男等[14]研究成果也出现了有的虾虎鱼先与外群聚在一起,之后再与其他虾虎鱼相聚的现象。该基因条形码可以很好地区分不同种,但对于不同科或更大的分类界元的效果不太明显。由于有的虾虎鱼比较稀有、难以采集,本研究采集样品有限,需要加大样本量进一步优化进化树。形态学上,孔虾虎鱼和拉氏狼牙虾虎鱼具有相似的体型、体色、眼睛高度退化等特点,有些科学家将它们归为近盲虾虎鱼亚科,这与进化树分类结果比较一致。但是它们遗传距离大于0.2,确定是不同的种。本研究中,背眼虾虎鱼科的弹涂鱼与近盲虾虎鱼亚科的小头栉孔虾虎鱼聚为一支,相关研究也证实他们亲缘关系较近,具体还需加大样本进行确认。
本研究得出,矛尾刺虾虎鱼和斑尾复虾虎鱼为同一物种,种间遗传距离仅为0.014,小于 Hebert等[13]提出的临界值0.020;进化树上,节点支持率达到100%聚为一支。
缟虾虎鱼是虾虎鱼科的一个属,渤海常见的有钟馗虾虎鱼(髭缟虾虎鱼)、纹缟虾虎鱼、裸颈缟虾虎鱼等。本研究中所有的缟虾虎鱼遗传距离较近,与已知分类结果一致。晴尾蝌蚪虾虎鱼由于头部形态特征与其他虾虎鱼相差较大,从形态上很好区分,与其他虾虎鱼科属遗传距离较远。
由于虾虎鱼种类众多,变异较大,其鉴定除了分子鉴定还需结合形态学和生态地理分布等因素,也可以结合其他条形码基因综合分析其进化、地理分布及系统分类等。
参考文献:
[1] 李宝华, 张素青, 王秀芹, 等. 2004-2009年渤海湾(天津区域)鱼虾贝产卵场水质环境综合评价[J]. 水产科学, 2010, 29(11): 661-665.
[2] 徐海龙, 谷德贤, 乔秀亭, 等. 渤海湾主要渔业资源长度与体质量关系分析[J]. 南方水产科学, 2014, 10(1): 57-63.
[3] 刘静, 陈咏霞, 马琳. 黄渤海鱼类图志[M]. 北京: 科学出版社, 2015.
[4] SACCONE C, DE GIORGI C, GISSI C, et al. Evolutionary genomics in Metazoa: the mitochondrial DNA as a model system[J]. Gene, 1999, 238(1): 195-209.
[5] 柳淑芳, 陈亮亮, 戴芳群, 等. 基于线粒体CO1基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J]. 海洋与湖沼, 2010, 41(2): 223-232.
[6] 高天翔, 毕潇潇, 赵林林, 等. 基于线粒体Cytb基因全序列的松江鲈群体遗传结构分析[J]. 水生生物学报, 2013, 37(2): 199-207.
[7] 彭居俐, 王绪祯, 王丁, 等. 基于线粒体CO1基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用[J]. 水生生物学报, 2009, 33(2): 271-276.
[8] 彭士明, 施兆鸿, 侯俊利, 等. 银鲳3个野生群体线粒体COI基因的序列差异分析[J]. 上海海洋大学学报, 2009, 18(4): 398-402.
[9] 毕潇潇, 高天翔, 肖永双, 等. 4种鳕鱼线粒体16S rRNA、COI和Cyt b基因片段序列的比较研究[J]. 南方水产, 2009, 5(3): 46-52.
[10] 杨纪明. 渤海鱼类的食性和营养级研究[J]. 现代渔业信息, 2001, 16(10): 10-19.
[11] 李明德, 张洪杰, 等. 渤海鱼类生物学[M]. 北京: 中国科学技术出版社, 1991: 62-68.
[12] WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia’s fish species[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360(1462): 1847-1857.
[13] HEBERT P D N, STOECKLE M Y, ZEMLAK T S, et al. Identification of birds through DNA barcodes[J]. PLoS Biology, 2004, 2(10): e312.
[14] AQUINO L M G, TANGO J M, CANOY R J C, et al. DNA barcoding of fishes of Laguna de Bay, Philippines[J]. Mitochondrial DNA, 2011, 22(4): 143-153.
[15] 于亚男, 宋超, 侯俊利, 等. 基于线粒体COI基因部分序列的长江口虾虎鱼科鱼类系统分类[J]. 淡水渔业, 2014, 44(5): 3-8.
[16] 李明晖, 安长廷, 李昂, 等. 黄渤海虾虎鱼DNA条形码分类体系构建[J]. 中国水产科学, 2022, 29(8): 1179-1188.
[17] FEDOROV A, SAXONOV S, GILBERT W. Regularities of context-dependent codon bias in eukaryotic genes[J]. Nucleic Acids Research, 2002, 30(5): 1192-1197.
[18] 郭新紅, 刘少军, 刘巧, 等. 鱼类线粒体DNA研究新进展[J]. 遗传学报, 2004, 31(9): 983-1000.

