京津冀地区猪源沙门氏菌耐药性及耐药基因分析
谢 飞,刘 滢,郭致君,范祥伟,魏 博,张 乐,刘雪连*
( 1. 北京大北农科技集团股份有限公司,北京 100192 ;2. 北京市饲料安全生物调控工程技术研究中心,北京 100192 )
随着我国经济以及食品行业发展,猪肉等各类肉类食品的需求与日俱增,而在猪养殖、屠宰和零售过程中极易受到沙门氏菌(Salmonella)的污染。沙门氏菌是主要的人畜共患病的病原之一,目前国际上已发现约2 500 个血清型,中国有200多种,是许多国家发生食源性细菌肠炎的主要原因之一[1]。其中鼠伤寒沙门氏菌于免疫功能正常的人体内可引起自限性胃肠炎,在免疫功能低下的人体内引发败血症,而伤寒沙门氏菌则可引起伤寒[2-3]。
为维护我国动物源性食品安全和公共卫生安全,本研究在京津冀地区猪饲料厂、猪养殖场、猪屠宰场、超市、农贸市场等采集饲料、猪肛拭子、水、猪肉等样品5 024份,采用国标GB 4789.4—2016[4]的方法分离筛选得到沙门氏菌菌株484株,通过抗菌药物浓度梯度法测定了沙门氏菌对12类22种抗菌药物的敏感性,得到对7类以上药物的多重耐药菌株50株;通过分子生物学、全基因组测序技术等对50株多重耐药菌株的耐药基因进行了分析和预测,构建系统进化树,建立了包含50株具有多重耐药特点的沙门氏菌全基因组数据库,以期为沙门氏菌引起的疾病控制和指导用药提供一定参考。
1 材料与方法
1.1 样品来源
本试验采集京津冀地区猪饲料、猪养殖场、猪屠宰场和猪肉零售等4个环节的样本,合计5 024份。其中饲料环节分别采集蛋白原料和配合饲料样本(521 份);养殖环节采集猪的肛拭子(2 703 份,1 头猪1 份),水样分别采集水井、储水箱和饮水头样本(25 份);屠宰环节采集预冷后分割前的猪胴体样本(322份,1头猪1份,颈部);零售环节则以超市和农贸市场的生鲜肉和猪肉馅作为样本(1 453份)。样品放入无菌PBS 缓冲液中保存,以装有冰袋泡沫箱封闭,24 h内低温送至实验室进行预增菌处理。
1.2 试验菌株
大肠杆菌标准菌株ATCC25922、肠炎沙门氏菌标准菌株CVCC3377 和鼠伤寒沙门氏菌标准菌株ATCC700408均来自饲用微生物工程国家重点实验室。
1.3 仪器与试剂
主要仪器:SPX-250B培养箱(上海博迅实业医疗设备厂)、CF16RXⅡ低温冷冻离心机(日立公司)。
主要试剂:缓冲蛋白胨水(buffered peptone water,BPW)、四硫磺酸钠煌绿(tetrathionate broth base,TTB)、亚硒酸盐胱氨酸(selenite cystine,SC)、亚硫酸铋(bismuth sulphite,BS)、木糖赖氨酸脱氧胆盐(xylose lysine desoxycholate,XLD)均购自北京奥博星生物科技有限公司;基因组抽提试剂盒购自宝生物工程公司。
1.4 试验方法
1.4.1 样品沙门氏菌分离
采用国标GB 4789.4—2016 的方法分离沙门氏菌:将采集的样品在超净工作台加入含无菌BPW培养基的试管中,37 ℃ 180 r/min 振荡培养8~18 h,次日分别吸取1 mL BPW 增菌液至10 mL 的TTB 和10 mL 的SC 增菌液中,分别置于42 ℃培养18~24 h和37 ℃培养18~24 h。培养后的TTB增菌液分别划线接种于BS琼脂平板,SC增菌液划线接种于XLD琼脂平板,于37 ℃分别培养40~48 h(BS琼脂平板)或18~24 h(XLD琼脂平板)。
1.4.2 耐药性分析
根据美国临床和实验室标准协会(CLSI)[5]标准,选取头孢曲松(CRO)、头孢噻呋(CEF)、头孢喹肟(CEQ)、氨曲南(ATM)、庆大霉素(GEN)、阿米卡星(AMK)、阿莫西林(AMX)、碳青霉烯(IMP)、亚胺培南(IPM)、四环素(TET)、多西环素(DOX)、环丙沙星(CIP)、恩诺沙星(ENO)、左氧氟沙星(LEV)、萘啶酸(NAL)、氯霉素(CHL)、氟苯尼考(FFC)、磷霉素(FOS)、多黏菌素(BPB)、阿奇霉素(AZM)、喹乙醇(OLA)、乙酰甲喹(MEQ)等22种常用抗菌药物,以大肠杆菌标准菌株ATCC 25922作为质控菌株。抗菌药物通过微量肉汤稀释法进行稀释,将筛出的沙门氏菌液态培养和接种,采用纸片扩散法进行抗菌药物敏感性试验,37 ℃孵育18~24 h,记录菌落生长情况。
1.4.3 基因组提取与检测
按照基因组抽提试剂盒试验步骤,将耐7 种以上抗菌药物的50株样品提取好的基因组放在冰上融化,吹打混匀离心待检测。采用琼脂糖凝胶电泳(胶浓度1%,电压150 V)检测样品的完整性,用于构建DNA文库[6-7]。
1.4.4 全基因组测序、组装及预测
测序:样本按照全基因组测序要求,送至深圳华大基因科技有限公司进行全基因组测序;细菌基因组用PacBio Sequel Ⅱ平台进行de novo 测序,测序得到的原始数据(raw data),对illumina测序平台的数据进行污染和低质量过滤,过滤掉500 bp以下的片段,得到50株沙门氏菌的有效数据(clean data)。
组装:基于测序平台获得有效数据组装使用SPAdes软件,经过多次调整获得最优组装结果。
基因组分析组装完成后,分析样品基因组的成分,针对编码基因序列进行CARD 数据库的功能注释。将测到的50 株猪源沙门氏菌全基因组数据上传至NCBI,编号为PRJNA673054。
1.4.5 耐药基因分析
基于耐7 种以上抗菌药物的50 株沙门氏菌的全基因组测序结果,耐药基因分析使用抗性基因数据库(comprehensive antibiotic research database,CARD),共检测到43 种耐药相关基因,包括β-内酰胺类:blaTEM-1、blaCTX、blaOXA-1、blaOXA-10、pbp1A、blaIMP、blaDHA-1;氨基糖苷类:aac(3)-Iva、aac(6')-Iaa、aadA1、aadA8b、aph(3')-Ia、aph(3'')-Ib、aph(3')-Iia、aac(3)-Iid;大环内酯类:erm(B)、erm(T);多黏菌素类:mcr-1、mcr-3、mcr-5;环素类:tet(A)、tet(B);磺胺类:sul1、sul2、sul3;喹诺酮类:oqxB、oqxA、gyrA(S83F)、parC(S80R)、qnrD1、qnrS1、gyrA(D87Y);磷霉素类:Fos3、FosA;氯霉素类:catA1、catB4、catB8、cmlA1、floR;三甲氧苄二氨嘧啶类:dfrA12;利福平类:arr-3;氨基糖苷类和喹诺酮类:aac(6')-Ib-cr。
1.4.6 进化树分析
基于全基因组序列构建系统进化树。系统进化树(phylogenetic tree)是表明被认为具有共同祖先的各物种相互间演化关系的树。wgMLST 称为全基因组多位点序列分型(whole genome multilocus sequence typing),是多位点序列分型(MLST)的一个拓展方法。由于wgMLST包含更多的基因位点(1 500~4 000),因此与传统的基于7个基因位点的MLST分型手段相比具有更高的精度及分辨率。菌株高精度分型wgMLST以其高精度、高可靠性的分型结果,成为流行病学菌株分型的新标准。本试验依据wgMLST对沙门氏菌进行了亲缘关系分析和分型。
2 结果与分析
2.1 京津冀地区不同来源样本沙门氏菌检出率(见表1)
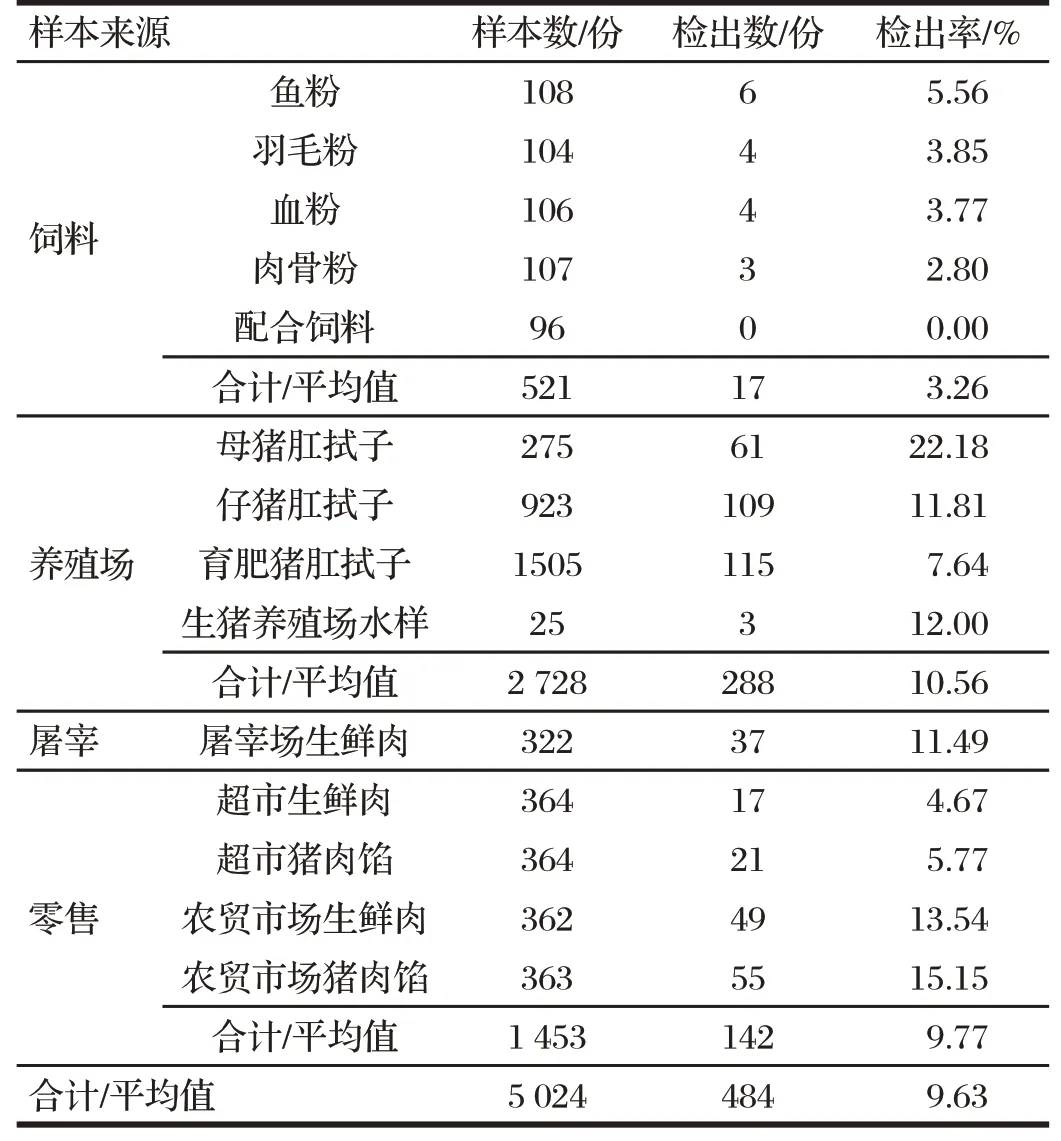
表1 京津冀地区不同来源样本沙门氏菌检出率Tab.1 Detection rate of Salmonella in samples from different sources in Beijing-Tianjin-Hebei
由表1 可知,按照国标GB 4789.4—2016 对采集自饲料厂、养殖场、屠宰场、超市、农贸市场等来源的饲料、猪肛拭子、水样、猪肉、猪肉馅等5 024份样品进行检测,经过分离鉴定,共检出阳性样品484份,平均检出率为9.63%。在养殖环节,母猪肛拭子中沙门氏菌检出率为22.18%,高于断奶仔猪(菌株数/样本数=109/923,11.81%)和育肥猪(菌株数/样本数=115/1 505,7.64%)。在零售环节,农贸市场猪肉的沙门氏菌检出率(菌株数/样本数=49/362,13.54%)高于超市的猪肉(菌株数/样本数=17/364,4.67%)。
在检出484 株沙门氏菌中,鼠伤寒沙门氏菌占25.41%、伊鲁木沙门氏菌占15.91%、猪伤寒沙门氏菌占11.78%、德尔卑沙门氏菌占10.33%、汤卜逊沙门氏菌占5.99%、塔克松尼沙门氏菌占5.79%、山夫登堡沙门氏菌占4.96%、猪霍乱沙门氏菌占2.48%、纽兰沙门氏菌占1.45%。
2.2 484 株沙门氏菌对22 种抗菌药物的耐药率分析(见表2、图1)
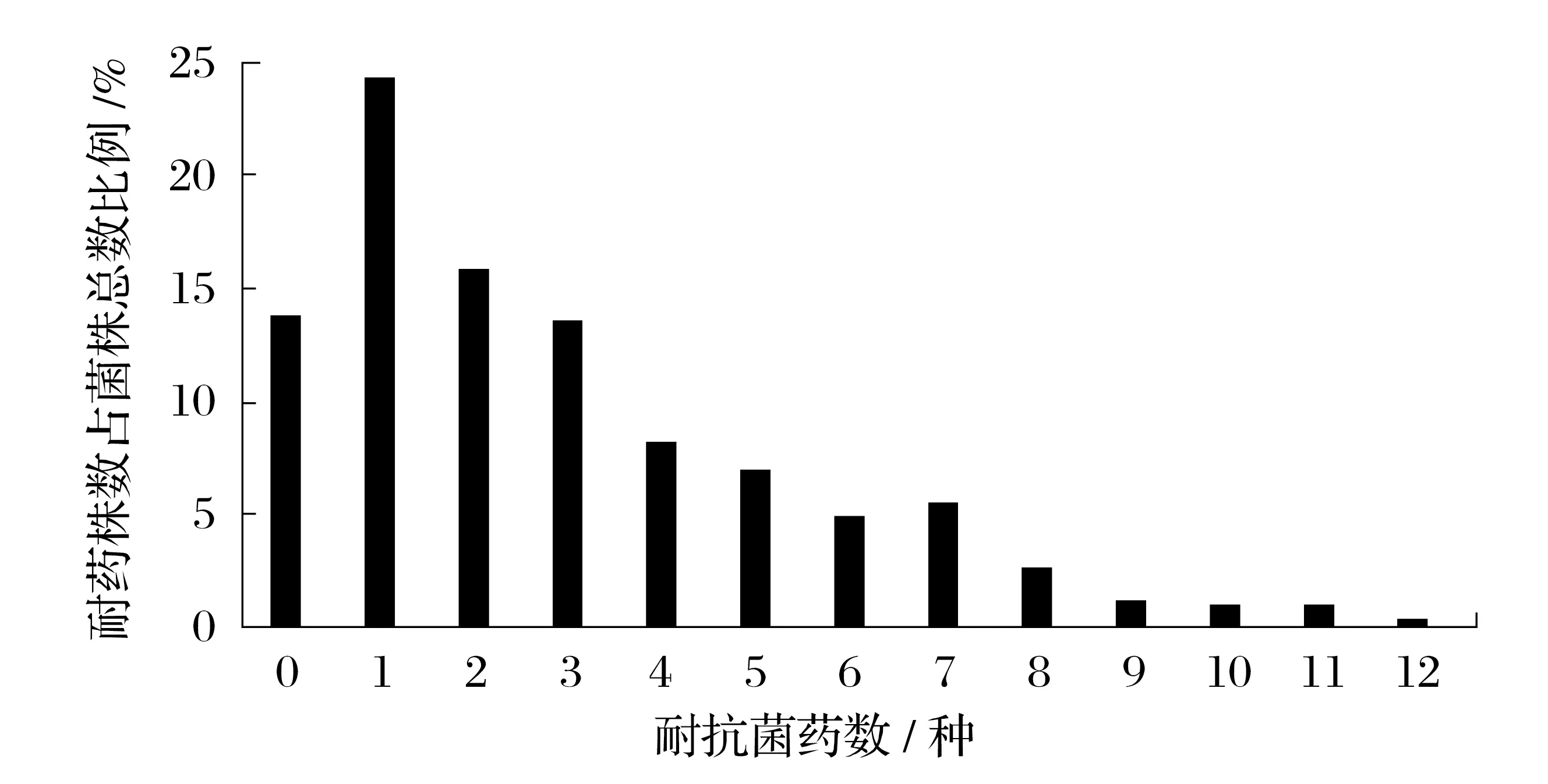
图1 484株沙门氏菌对22种抗菌药物的多重耐药性Fig.1 Salmonella 484 strains with multiple resistance to 22 antimicrobial agents

表2 484株沙门氏菌对22种抗菌药物的耐药性分析Tab.2 Resistance analysis of 484 Salmonella strains to 22 antibiotics
控制菌株孔内菌落数为1×105CFU/mL 左右,生长表现为浑浊或微孔底部沉淀,记录肉眼可见抑制细菌生长药物的最小浓度。
由表2 可知,对484 株沙门氏菌的耐药率大于30%的抗菌药物有GEN 30.8%、AMX 31.6%、环素类(TET 36.8%、DOX 33.5%)、NAL 31.8%。对沙门氏菌耐药率小于30%大于10%的抗菌药物有氟喹诺酮类(CIP 19.2%、ENO 26.7%、LEV 16.9%)、氯霉素类(CHL 21.5%、FFC 20.7%)、FOS 13.0%、BPB 13.0%、AZM 11.4%、喹恶啉类(OLA 26.4%、MEQ 25.2%)。
484 株沙门氏菌对22 种抗菌药物耐药性具体分析结果见图1。由图1 可知,对22 种抗菌药物敏感的菌株数占总检测出沙门氏菌总数的比例是13.84%,对1种抗菌药物耐受菌株数比例最高为24.38%;随着检测的抗菌药物越多,沙门氏菌耐药菌株数逐渐减少,耐7种以上抗菌药物的沙门氏菌比例为11.98%,对12 种抗菌药物耐受菌株数仅占沙门氏菌总数的比例0.41%。
2.3 耐7 种以上抗菌药物的50 株沙门氏菌耐药基因分析(见图2、图3)
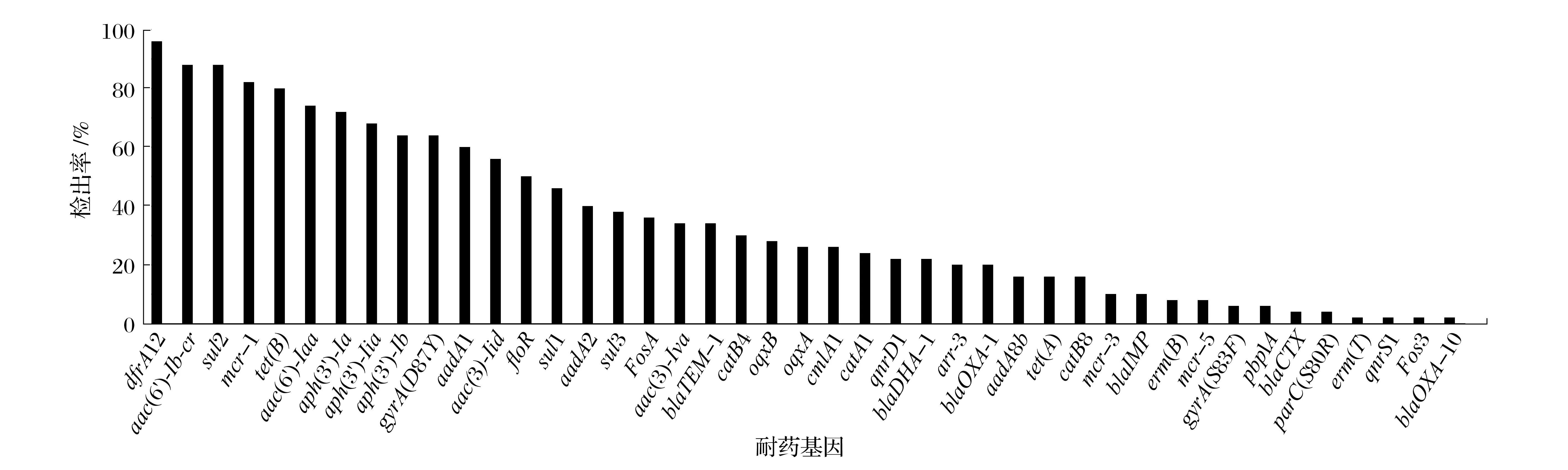
图2 耐7种以上抗菌药物的50株沙门氏菌耐药基因检出率Fig.2 Detection rate of drug resistance genes in 50 Salmonella strains resistant to more than seven antibiotics
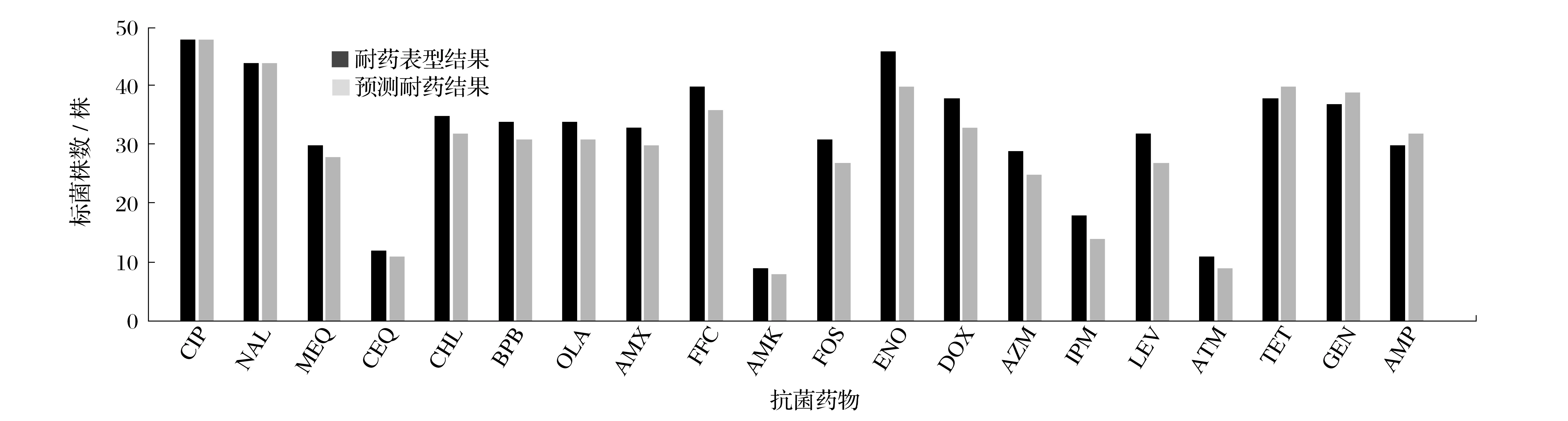
图3 耐7种以上抗菌药物的50株沙门氏菌耐药表型与耐药基因检测符合性Fig.3 Antibiotic resistance phenotypes of 50 Salmonella strains resistant to more than seven antibiotics were consistent with drug resistance gene detection results
由图2可知,利用CARD数据库对50株沙门氏菌的耐药基因进行比对分析,共得到43种耐药基因检出率,包括β-内酰胺类:blaTEM-1(35%)、blaCTX(4%)、blaOXA-1(20%)、blaOXA-10(2%)、pbp1A(6%)、blaIMP(10%)、blaDHA-1(22%);氨基糖苷类:aac(3)-Iva(34%)、aac(6')-Iaa(74%)、aadA1(60%)、aadA8b(16%)、aph(3')-Ia(72%)、aph(3'')-Ib(64%)、aph(3')-Iia(68%)、aac(3)-Iid(56%);大环内酯类:erm(B)(8%)、erm(T)(2%);多黏菌素类:mcr-1(82%)、mcr-3(10%)、mcr-5(8%);环素类:tet(A)(16%)、tet(B)(80%);磺胺类:sul1(46%)、sul2(88%)、sul3(38%);喹诺酮类:dfrA12(96%)、oqxB(28%)、oqxA(26%)、gyrA(S83F)(6%)、parC(S80R)(4%)、qnrD1(22%)、qnrS1(2%)、gyrA(D87Y)(64%);磷霉素类:Fos3(2%)、FosA(36%);氯霉素类:catA1(24%)、catB4(30%)、catB8(16%)、cmlA1(26%)、floR(50%);利福平类:arr-3(20%);氨基糖苷类和氟喹诺酮类:aac(6')-Ib-cr(88%)。
由图3 可知,对比耐药表型和通过全基因组数据预测耐药结果,CIP和NAL的对应率达到100%,44株沙门氏菌对氟喹诺酮类CIP 有耐药性,测序具有aac(6')-Ib-cr耐药基因;48 株沙门氏菌对喹诺酮类NAL 有耐药性,测序有dfrA12 耐药基因。抗菌药物平均对应率>90%,说明全基因组测序结果可较好地预测菌株的耐药性;IPM对应率仅为77.78%,可能与本试验分离得到的耐受该抗菌药物的菌株较少有关,也可能仍有未知的耐药机制在发挥作用。
2.4 基于全基因组测序结果的亲缘关系分析
对全基因组测序得到的50 株沙门氏菌菌株进行wgMLST的分型和聚类,结果见图4。

图4 耐7种以上抗菌药物的50株沙门氏菌wgMLST聚类分析Fig.4 WgMLST cluster analysis of 50 strains of Salmonella resistant to more than seven antibiotics
由图4 可知,除7 株为未知型外,分离的沙门氏菌有27株为ST19,10株为ST34,4株为ST40,2株为ST64。
3 讨论
本文调查了京津冀地区的饲料、养殖、屠宰和零售等4 个环节的猪源沙门氏菌污染分布情况,平均污染率达9.63%。在养殖环节,母猪肛拭子的沙门氏菌检出率为22.18%,高于断奶仔猪(11.81%)和育肥猪(7.64%),原因可能是母猪经历多次繁殖周期和产仔应激,肠道内沙门氏菌侵入和增殖概率更高,母猪抗菌药物治疗次数比仔猪和育肥猪多,导致沙门氏菌耐药概率也随之增加。
农贸市场猪肉的沙门氏菌检出率(13.54%)比超市的猪肉(4.67%)高,原因可能是超市的冷藏环境比农贸市场的环境好,管理更规范。猪场水样的沙门氏菌检出率是12.00%,沙门氏菌污染率比较高。因此,检测猪场的水源安全是比较重要的工作。
近年来,国内外由沙门氏菌感染人体和猪群并暴发疫情数逐年上升,进一步加强监控饲料和肉类食品尤为重要。本研究检出的484株沙门氏菌中,不同来源的沙门氏菌中优势菌株为鼠伤寒沙门氏菌、伊鲁木沙门氏菌、猪伤寒沙门氏菌和德尔卑沙门氏菌等。484 株沙门氏菌对22种抗菌药物均出现了不同程度的耐受性,耐药菌株所占比例86.5%。根据药敏试验分析,对TET的耐受性最强,耐药率高达36.8%;其他抗菌药物如DOX 耐药率为33.5%,GEN 为30.8%,AMX 为31.6%,NAL 为31.8%,提示应降低或避免耐药率较高的上述几种药物如TET、DOX、GEN、NAL、阿莫西林、恩诺沙星、喹乙醇、乙酰甲喹等在畜禽临床中的使用情况。
对耐受7 种以上抗菌药物的50 株沙门氏菌全基因组进行测序发现,有5 种常见耐药基因的检出率在80%以上,分别为三甲氧苄二氨嘧啶类dfrA12、磺胺类sul2、氟喹诺酮类aac(6')-Ib-cr、多黏菌素类mcr-1 和环素类tet(B);有3种耐药基因的检出率在50%以上,分别为氨基糖苷类aac(6')-Iaa、喹诺酮类gyrA和氯霉素类floR;有3种耐药基因的检出率在20%以上,分别为磷霉素类FosA、β-内酰胺类BlaTEM-1和利福平类arr-3;沙门氏菌的耐药基因的携带率与该菌株对药物的耐药性情况对应。
本研究中,沙门氏菌对BPB 的耐药率为13.0%,属于较低水平,可能与我国2017 年禁止BPB 作为饲料添加剂使用等措施相关[8]。BPB 长期应用于感染性疾病治疗[9],如大肠杆菌、肺炎杆菌、绿脓杆菌等所致的泌尿系统感染、脑膜炎、肺部感染、败血症等。目前国内外已有许多报道耐BPB的沙门氏菌的研究[10-11],如猪伤寒沙门氏菌和鼠伤寒沙门氏菌等携带耐药基因mcr-1、mcr-3和mcr-5[12]。因此,生猪养殖过程中使用BPB 应引起足够重视,防止有害菌如沙门氏菌对BPB的耐药性增强[13]。
研究表明,动物体分离的沙门氏菌中,最常见的β-内酰胺酶基因是BlaTEM-1和BlaPSE-1编码对氨苄青霉素的抗性[14-15]。本研究表明,沙门氏菌的耐药表型、致病性与毒力基因相关。全基因组测序已鉴定出多个毒力基因,包括菌毛黏附类因子、调节因子、毒力岛、质粒上的毒性基因、毒素等[16-17]。多重耐药菌株的产生机制复杂,控制难度大,且极易通过食物链水平传播给人类,对人体健康造成巨大威胁[18]。
我国农业农村部第194 号公告提出,自2020 年7 月1 日起,饲料生产企业停止生产含有促生长类药物饲料添加剂(中药类除外)的商品饲料[19]。307 号公告提出,养殖者日常生产自配料时不得添加允许在商品饲料中使用的抗球虫和中药类药物以外的兽药。上述两个公告将极大减少耐药性沙门氏菌污染。
4 结论
本研究通过调查京津冀地区饲料、养殖、屠宰和零售等环节的猪源沙门氏菌污染分布,检测分离的猪源沙门氏菌对抗菌药物耐药率,并进行耐7种以上抗菌药物的沙门氏菌全基因组测序分析。结果发现,京津冀地区沙门氏菌检出率、耐药率均较高,且得到的菌株具有多重耐药性,耐药基因分析结果与耐药表型相关性较高,沙门氏菌的耐药基因可决定耐药表型。
本研究结果为京津冀地区沙门氏菌耐药基因的监控提供一定参考,提示猪场临床中应限制某些抗菌药物使用,以降低沙门氏菌的耐药性。

