绿豆GR 基因生物信息学分析及其CRISPR/Cas9重组载体的构建与转化
李晓驹,方淑梅,王庆燕,梁喜龙,郑殿峰,3
(1.黑龙江八一农垦大学农学院,大庆 163319;2.黑龙江八一农垦大学生命科学技术学院;3 广东海洋大学)
绿豆(Vigna radiataL.)是蛋白质、膳食纤维、矿物质、维生素和大量生物活性化合物(包括多酚、多糖和肽)的极好来源[1-4],因其生育期短、适应性广泛和固氮性能[5],在种植产业结构调整和优质高效农业生产及经济发展中具有重要作用[6]。近年来,由于其产量低且价格不稳定,我国绿豆种植面积及其在国际市场中的竞争力受到严重影响[7]。此外,由于其特殊的花结构,严格闭花授粉,杂交不易成功。加之我国绿豆的品种改良工作起步较晚,种植面积相对较小,所以在一定程度上限制了绿豆转基因育种的发展。为此绿豆经常被农业生产者种植在贫瘠的土地上,产量远远没有到达自给自足,进口量逐年增加,且由于多年连续种植,常存在着连作障碍现象[7-9]。现有的绿豆品种已不能满足当下种植需求,绿豆的育种方式也亟待拓展。基因编辑育种因其育种效率高、育种周期短、且能定向改良某一个或多个目标性状,能解决多种自交不亲和性及不易杂交等问题,被广泛用于作物育种,但因绿豆遗传转化体系尚不完善,直至目前为止,国内外对绿豆基因编辑育种的研究鲜见报道。
所在团队经甲基化测序研究发现,大豆谷胱甘肽还原酶基因(GR)在大豆抗(耐)连作方面具有积极作用。谷胱甘肽还原酶(GR)是抗坏血酸—谷胱甘肽(AsA-GSH)循环中的关键抗氧化酶,其主要的生理功能是将氧化型谷胱甘肽(GSSG)还原为还原型谷胱甘肽(GSH),从而为活性氧(ROS)的清除提供还原力,保护植物免受伤害[10]。再加之前人研究发现GR基因可在多种环境胁迫中发挥作用,所以对GR基因的功能展开研究具有重要意义。因此,论文将在生物信息分析的基础上,利用绿豆GR基因构建CRISPR/Cas9 重组载体,初步揭示遗传转化体系在绿豆中的应用潜力。
CRISPR/Cas 系统是一些细菌和古生菌用于抵抗外来质粒或者噬菌体入侵的免疫系统[11]。一般来说,CRISPR/Cas9 系统对外来DNA 的作用可分为三步:一是免疫获得;二是免疫表达;三是免疫反应。具体表现为其通过碱基配对方式与sgRNA(single guide RNA)形成Cas9 蛋白复合物,参与靶双链DNA 的切割。经过人工改造后,CRISPR/Cas 技术现已成为一项能对基因组完成精确修饰的基因编辑技术,具有操作简单、效率高、脱靶效率低等特点。随着CRISPR/Cas 系统的发展,其在越来越多的领域中得到广泛应用,包括人[12]、小鼠[13]、拟南芥[14]、玉米[15]、大豆[16],以及传染病的诊断和治疗等[17-18]。尤其是在“十四五”期间作物育种工作中,CRISPR/Cas9 早已成为主流的基因编辑技术和手段[16]。
基于上述分析,以期通过生物信息学分析了解GR基因及其相应蛋白产物特点,构建CRISPR/Cas9-GR-gRNA 敲除载体,同时利用发根农杆菌转化绿豆子叶节,获取阳性毛状根,从而为构建绿豆CRISPR/Cas9 基因编辑体系及获取优质的突变体遗传材料奠定前期基础。
1 材料与方法
1.1 植物材料、菌株、载体、试剂及仪器
试验中用于基因遗传转化的绿豆品种“绿丰2号”,由实验室保存。大肠杆菌(Escherichia coli)菌株DH5α 和DNA 回收试剂盒购于北京全式金生物技术有限公司。发根农杆菌K599、CRISPR/Cas9 载体pYLCRISPR/Cas9 -DB 及gRNA 载体pYLgRNA -AtU3d、pYLgRNA-AtU3b、pYLgRNA-AtU6 -1、pYLgRNA-AtU6-29 由广州大学孔凡江教授馈赠。质粒提取试剂盒购自康为公司;2×Taq Master Mix、T4 连接酶购自TaKaRa 公司;限制性内切酶AscI、BsaI 购自NEB 公司;PCR 试剂KOD plus 购自Toyopo 公司;蔗糖、琼脂、各种抗生素、用于培养基配制的试剂以及其他化学试剂购自Solarbio 公司。
1.2 绿豆GR 生物信息学分析
利用NCBI 网站获取大豆GR基因(Glyma.02G141800)的蛋白质序列,再使用BLASTp 工具对大豆GR基因的蛋白质序列在绿豆基因数据库进行BLAST 比对到绿豆的GR基因(LOC106772498)序列,接着使用ExPASy ProtParam 在线软件对绿豆GR基因编码的氨基酸序列进行理化性质(分子量、等电点、氨基酸组成、不稳定系数等)分析,采用Netphos3.1Server 在线软件对绿豆GR 蛋白进行磷酸化位点预测,利用Protscale 在线软件进行疏水性或亲水性预测与分析,运用TMHMM Server v.2.0 预测蛋白的跨膜结构域。通过SignalP5.0 在线软件预测GR蛋白的信号肽,根据ProtComp9.0 在线软件进行亚细胞定位预测,通过SMART 在线软件分析GR 的保守结构域,利用Phyre2 在线软件预测该蛋白的三级结构模型。
1.3 同源比对及进化树分析
通过NCBI 网站中的blastp (protein-protein BLAST)与CD- Search 在线比对工具对绿豆GR基因编码氨基酸序列进行同源比对,在其中选取相似性较高的物种, 包括绿豆(Vigna radiata,XP_014514412.1)、赤豆(Vigna angularis,XP_017415 276.1)、菜豆(Phaseolus vulgaris,XP_007144694.1)、大豆(Glycine max,KAG5002780.1)、豌豆(Pisum sativum,P27456.1)、粳稻(Oryza sativa subsp.JaponicaGRoup,XP_015627860.1)、大麦(Hordeum vulgare,BAF80308.1)、小麦(Triticum aestivum,KAF7041206.1)、玉米(Zea mays,NP_001292747.1)、高粱(Sor-ghum bicolor,XP_002468457.1)、西红柿(Solanum lycopersicum,NP_001234243.2)、马铃薯(Solanum tu-berosum,KAH0636848.1)、 辣椒(Capsicum chinense,PHU22703.1)、烟草(Nicotiana tabacum,P80461.1)、山荆子(Malus baccata,TQE13711.1)、苹果(Malus domestica,XP_028958518.1)、桃(Prunus perosica,XP_0 07199772.1)、扁桃(Prunus dulcis,XP_034228289.1)、樱桃(Prunus avium,XP_021821920.1)、拟南芥(Arabidopsis thaliana,NP_191026.1)、甘蓝(Brassica oleracea,VDD57363.1)、芜菁(Brassica rapa,RID47097.1)、芥菜(Brassica juncea,AAD28177.1)。
(1)利用DNAMAN 软件做氨基酸多重序列比对;
(2)利用MEGA_X 软件对作物中的氨基酸序列进行同源比对;
(3)利用Jalview 软件对氨基酸序列进行同源比对和可视化分析;
(4)利用iTOL(https://itol.embl.de/tree/)在线分析软件基于邻接法构建绿豆GR 蛋白与其他22 个物种GR 蛋白的系统进化树,分析其亲缘关系。
1.4 靶点选择及sgRNA 引物设计
利用CRISPOR 网站(http://crispor.tefor.net/)在线工具对绿豆GR基因序列进行gRNA 靶位点设计。选择4 个长度为20 bp 的靶位点序列(图1),4 个靶位点,能同时敲除绿豆GR基因,分别为GR-AtU3d-T1-F1/GR-AtU3d-T1-R1、GR-AtU3b-T2-F2/GRAtU3b-T2-R2、GR-AtU6-1-T3-F3/GR-AtU6-1-T3-R3、GR-AtU6-29-T4-F4/GR-AtU6-29-T4-R4,同时设计CRISPR 鉴定引物GR-CasF-Test/GR-Cas R-Test(表1)。

表1 引物序列Table 1 Primer sequence
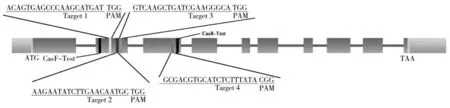
图1 靶点在GR 基因内的位置Fig.1 Location of the target in the GR gene
1.5 四靶点CRISPR/Cas9-GR-gRNA 载体的构建
构建CRISPR /Cas9 载体步骤如下:(1)靶点接头制备,将靶点引物放置PCR 仪中90 ℃退火30 s。(2)gRNA 载体的酶切、连接与扩增反应,使用T4 DNA Ligase 酶和BsaⅠ酶将靶点引物与对应的gRNA 连接起来,进行两轮巢式PCR 反应后使用胶回收试剂盒回收产物,按此方法分别获得四个靶点的gRNA表达盒,各取3 μL 电泳检查产物长度是否符合。(3)四靶点pYLCRIS PR/Cas9-GR-gRNA 载体(图2)的酶切、连接及测序,将四个靶点gRNA 表达盒按一定浓度比例混合,加入未切割的pYLCRISPR/Cas9 质粒,BsaⅠ37 ℃酶切10 min,加入T4 DNA ligase,设置两个循环温度实现先酶切后连接。将连接产物转化大肠杆菌DH5α 感受态细胞后,挑取单克隆接种培养,并使用Cas9 载体上的特异引物SP1/3 进行菌落PCR,成功筛选出单克隆后抽提质粒后将酶切鉴定正确的进行测序,测序正确的菌株甘油保存备用。
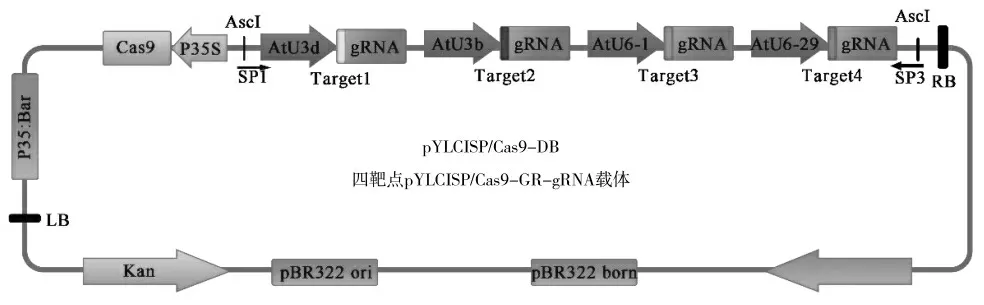
图2 载体构建信息Fig.2 Vector construction information
1.6 绿豆子叶外植体的遗传转化
(1)侵染液的制备。取出备用质粒并转化到农杆菌K599 感受态细胞中,挑单克隆到YEP 液体培养基,摇至OD600=0.6 左右,吸取5 mL 菌液,离心弃上清,加入1 mL 10 mM MgCl2溶液重悬,做侵染液。
(2)外植体的获得。选取籽粒均匀、饱满的绿豆种子,消毒灭菌后用无菌水清洗4~5 次,均匀放置在绿豆培养基中,5 d 后选取子叶肥厚无病斑的外植体以及粗壮的下胚轴备用。
(3)外植体的侵染与培养。将外植体放置灭菌纸上,用无菌刀水平切割顶端(未切离),用无菌毛笔或棉签沾取含有重组体的K599 菌液,均匀涂抹在切面,放置在含有特美丁、头孢、苄基霉素的发根培养基中,放到培养室,暗光培养,直到伤口长出毛状根。
1.7 毛状根检测
收集绿豆毛状根,提取绿豆毛状根DNA,利用SP1/3 引物进行PCR 扩增,检测阳性植株。根据侵染后统计PCR 扩增阳性毛状根数量计算得出毛状根转化率。
计算方法:毛状根转化率=转化阳性毛状根外植体数量/染总外植体数量×100%。
2 结果与分析
2.1 绿豆GR 基因及相关蛋白的一般特性分析
(1)理化性质分析
使用ExPASy ProtParam 在线软件对绿豆GR基因进行分析,结果显示绿豆GR基因编码蛋白的相对分子量为58.38 kDa,其分子式为C2612H4132N716O775S13,原子总数为8 248,氨基酸数为544,由19 种氨基酸组成,其中高含量氨基酸为丙氨酸(Ala)、甘氨酸(Gly)和丝氨酸(Ser),其比例分别为9.7%、9.6%和8.1%;有59 个带正电荷的氨基酸(Arg 和Lys),有54个带负电荷的氨基酸(Asp 和Glu),其理论等电点为8.69,属于弱碱性蛋白;预测半衰期为30 h,属于一种较为稳定的蛋白质。
(2)磷酸化位点的预测
通过Netphos 3.1 Server 在线软件对GR蛋白进行磷酸化位点预测。GR 蛋白存在47 个磷酸化位点,其中丝氨酸(Ser)位点28 个,苏氨酸(Thr)位点25个,络氨酸(Tyr)位点4 个,该蛋白Ser 磷酸化位点最多,推测其活性可能受到磷酸化的调控。
(3)亲水性预测和分析
利用Protscale 在线软件对绿豆GR 蛋白预测与分析。结果表明:GR 最低疏水值为-2.678,最高亲水值为2.278,亲水值大于疏水值,由此推测该蛋白为亲水性蛋白。这一结果与ExPASy ProtParam 的预测结果一致。
(4)跨膜结构域预测
使用TMHMM 2.0 Server 在线软件对绿豆GR蛋白的跨膜结构域进行预测,未发现GR 蛋白有明显的跨膜结构域。通过SignalP3.0 软件预测GR 蛋白的信号肽,也未发现GR 蛋白有信号肽,表明GR 蛋白是一种非分泌蛋白。接着根据ProtComp9.0 在线软件对绿豆GR 蛋白进行亚细胞定位预测,发现其在细胞质积分值(inteGRal)最高,说明该蛋白主要在细胞质中行使功能。
(5)蛋白质二级结构预测
为了更好地了解GR 蛋白的结构与功能,采用SOPMA 对绿豆GR 蛋白二级结构进行预测。发现GR蛋白中α-螺旋有163 处,占29.96%;延伸链有120处,占22.06%;β-转角有31 处,占5.07%;无规则卷曲有230 处,占42.28%。
(6)保守结构域
通过SMART 在线软件分析GR 的保守结构域,发现该蛋白在第67~390 个氨基酸处有Pyr_redox_2结构域,在第410~519 个氨基酸处有Pyr redox dim结构域(图3)。PROSITE 在线程序也同样显示GR 蛋白是含有Pyr redox 2、Pyr redox dim 结构域的ATP结合蛋白,属于PLN025465 超家族(图3)。

图3 GR 蛋白结构域预测Fig.3 Domain prediction of GR protein
(7)蛋白质三级结构模型预测
在预测GR蛋白二级结构的基础上,利用Phyre2在线软件预测该蛋白的三级结构模型。该模型是以C5w1jA 为模板进行构建,二者的序列一致性达到83%,预测可信度为100%。由图4 可知,GR蛋白主要由α-螺旋、无规则卷曲和延伸链组成,β-转角比较少,这也与GR 蛋白二级结构预测的结果一致。并且在预测的GR 蛋白三维结构中,其具有GR 蛋白与GSSG 和NADPHet al 作用的结合位点,C 端具有黄素腺嘌呤二核苷酸(FAD)的结合域。

图4 GR 蛋白三级结构预测Fig.4 Prediction of the tertiary structure of GR protein
2.2 同源比对及进化树分析
通过NCBI 网站中的blastp(protein-protein BLAST)与CD- Search 在线比对工具对绿豆GR基因编码氨基酸序列进行同源比对,在其中选取相似性较高的物种,用DNAMAN 软件做氨基酸多重序列比对。结果表明,绿豆GR基因与其他物种的GR基因编码氨基酸序列高度保守(图5)。

图5 不同物种GR 同源比对Fig.5 GR homology comparison of different species
利用MEGA7 软件基于邻接法绘制绿豆GR 蛋白和其他22 条来自物种GR 蛋白的系统发育进化树。如图6 所示,进化树聚为2 个大组,其中豆科、蔷薇科、茄科和十字花科属于同一大组,禾本科单独成组。其中豆科和蔷薇科属于同一四级分支,前者与茄科属于同一三级分支,前者与十字花科属于同一二级分支。分级数越大,说明各个植物科目情缘关系越密切。
(1)绿豆GR基因编码蛋白与赤豆处在同一个末级分支,说明与赤豆的亲缘关系最近。其次是菜豆,在豆科植物中与豌豆亲缘关系最远。
(2)豆科植物GR基因与蔷薇科植物GR基因属于同一大类分支,说明其与苹果、桃等蔷薇科植物的亲缘关系较为接近,而与粳稻、大麦、玉米、高粱等禾本科植物亲缘关系最远。
(3)绿豆和赤豆同处于豆科最末级分支,说明其GR基因可能是豆科中最后演化(图6)。

图6 不同植物的GR 进化树分析Fig.6 GR phylogenetic tree analysis of different plants
2.3 四靶点CRISPR/Cas9-GR-gRNA 载体的构建与转化
(1)CRISPR/Cas9-GR-gRNA 载体的构建
首先构建四个分别由AtU3d、AtU3b、AtU6-1、AtU6-29 启动子启动的靶位点。通过巢式PCR 扩增的方法分别进行四个靶位点sgRNA 表达盒的连接,扩增产物的片段都约为500 bp(图7A)。再将4 个sgRNA 表达盒通过一步法组装到pYLCRISPR/Cas9载体上,构建pYLCRISPR/Cas9-GR-gRNA 表达载体,组装完成的重组体转化大肠杆菌DH5α,随后挑取阳性单克隆,摇菌培养,随后进行质粒的提取。经Asc Ⅰ酶切鉴定,切出目的片段大小分别约为15.5 kbp 和2 000 bp(图7B),表明四个靶位点gRNA表达载体盒已经成功插入到编辑载体上。最后将酶切验证正确的质粒进行测序,测序正确的载体保存用于后续试验。

图7 pYLCRISPR/Cas9-GR-gRNA 表达载体的构建及验证Fig.7 Construction and verification of pYLCRISPR/Cas9-GR-gRNA expression vector
(2)发根农杆菌的转化
将构建好的pYLC RISPR/Cas9-GR-gRNA 表达载体转化至农杆菌K599,利用通用引物SP1/3 进行PCR,为阳性克隆,则能扩增出2 000 bp 左右的目的条带。(图8)说明pYLCRISPR/Cas9-GR-gRNA 重组质粒已转到农杆菌K599 中,挑取阳性单克隆制备好农杆菌K599 菌液,待菌液OD600=0.6 时侵染萌发4~5 d 的绿豆子叶,暗条件下培养,直至长出毛状根(图9)。

图8 重组质粒pYLCRISPR/Cas9-GR-gRNA PCR 鉴定Fig.8 PCR identification of recombinant plasmid pYLCRISPR/Cas9-GR-gRNA

图9 发根农杆菌K599 转化绿豆子叶节和下胚轴Fig.9 Agrobacterium K599 transformation of mung bean cotyledon nodes and hypocotyls
2.4 毛状根检测
利用CRISPR/Cas9 载体上通用引物SP1/3,PCR扩增产物约为2 000 bp。本次共检测20 株毛状根,共检测出12 株阳性转基因毛状根,其毛状根转化率约为60%(图10)。

图10 阳性转基因根毛检测Fig.10 Detection of positive transgenic roots
3 讨论
3.1 GR 基因
GR(谷胱甘肽还原酶)是维持还原型谷胱甘肽含量的关键酶,还原型谷胱甘肽直接参与应激抵抗和适应机制,从而确保细胞免受氧化损伤,在抵抗干旱、低温、高盐、高温、强光、重金属等环境胁迫中具有重要作用[10,19-20]。丁舜华等[21]研究表明,拟南芥谷胱甘肽还原酶的减少导致拟南芥中年龄依赖性和H2O2诱导的提前衰老。Tang G 等[22]构建了苜蓿根瘤菌GR基因缺失突变体,使得苜蓿延迟结瘤,总固氮容量减少75%,还发现通过GR途径GSSG 回收谷胱甘肽有助于苜蓿根瘤的发育和竞争能力,在固氮共生中起着关键作用。Bashir K 等[23]研究表明,GR可能在禾本科植物的缺铁应答中发挥作用。Wu TM 等[24]研究表明,水稻GR3 基因敲除导致水稻对盐胁迫的敏感性增加。同时有研究表明,GR在抵抗生物胁迫方面也具有一定的作用。研究通过生物信息学分析发现绿豆GR基因编码一个58.38 kDa,含有544 个氨基酸的蛋白质,并显示其与赤豆、菜豆、豌豆、苹果、桃、大麦等,具有高度序列同源性,都具有保守的GSSG 和NADPH 序列,含有Pyr_redox_2 和Pyr_redox_dim 两个结构域,这与Madhu[25]的研究结果相一致。
3.2 CRISPR/Cas 基因编辑及育种
CRISPR/Cas 系统作为一项主流的基因编辑技术,由于其操作简单、效率高,广泛应用于哺乳动物和植物的基因突变和转录调控[26]。与传统育种方法相比,CRISPR/Cas 系统能快在改良作物产量、品质、抗病性、抗除草剂和缩短育种周期均起到重要作用[27]。Jiang B[28]研究表明,利用CRISPR/Cas9 介导大豆雄性不育基因MS1 的诱变,花粉活性测试表明,虽然野生型植物的花药充满了有活力的花粉,但两个编辑品系的花药都是空心的,并且有活力的花粉粒数量显著减少。Do[29]利用CRISPR/Cas9 编辑GmFAD2-1A和GmFAD2-1B,使大豆油酸含量超过80%的显著增加,而亚油酸降低到1.3%~1.7%。蔡宇鹏等[30]利用CRISPR/Cas9 介导的GmFT2a 靶向诱变延迟大豆开花时间。
随着国内外转基因植物新品种的越来越多,但转基因食品的安全问题引起了人们的思考。利用CRISPR/Cas9 技术,可以很简单、方便且在理论上相对安全地获得具有稳定遗传的目标基因突变植物材料,但方法也存在许多技术上的难题。技术在不同的物种其遗传效率不同,豆科作物就是遗传转化效率极低的一类物种,很难将基因编辑载体质粒转运到细胞内并发挥基因编辑功能;其次,不同物种编辑基因的效率有所不同,在相同物种不同基因型的生物编辑效率也有所差异。最后,CRISPR/Cas9 成功进入细胞内并发挥基因编辑功能,使目标靶点断裂,但由于生物同源修复能力不同,导致被切割的目的基因难以被修复重组等。
我国绿豆的基因编辑育种尚无报道,目前国内外还没有CRISPR/Cas9 技术在绿豆中的应用。研究成功构建了分别由AtU3d、AtU3b、AtU6-1、AtU6-29启动子启动四靶点CRISPR/Cas9 编辑载体,接着利用靶点筛选引物GR-CasF/R-Test 对阳性毛状根进行PCR 扩增后测序。通过测序发现,AtU3d、AtU3b、AtU6-1、AtU6-29 启动子启动的Target 1、2、3、4 靶点均未出现有效的基因编辑(图11)。导致上述结果的可能原因如下:(1)由于试验样本量不足,绿豆基因编辑效率低,导致难以获得突变体;(2)CRISP/Cas9技术中也存在脱靶效应,Target 2、3、4 各靶点序列大小仅有20 bp,全基因组中这段序列可能存在相同或类似的序列,因此不知道靶点设计是否合理有效;(3)由于未见有AtU3d、AtU3b、AtU6-1、AtU6-29 启动子在绿豆中的报道,潜在的因素尚未确定,暂时无法确定这些启动子能否在绿豆中正常启动。
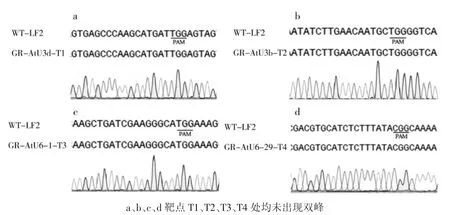
图11 编辑靶点测序峰图Fig.11 Detailed sequence of the targets site
4 结论
研究通过生物信息学手段明确了绿豆GR基因及其所编码蛋白的理化特性、结构特征、亚细胞定位及生物进化关系。同时以此为基础,成功构建了CRISPR/Cas9-GR-gRNA 基因编辑载体,同时通过K599 农杆菌介导绿豆转化并产生毛状根,最后利用基因编辑重组载体通用引物进行PCR 鉴定,成功获取阳性毛状根12 个,研究可为今后深入分析GR基因在绿豆中的功能提供有效信息和理论依据,同时为CRISPR/Cas9 技术在绿豆中的应用提供前期基础。

