利用双向导入系和重组自交系定位和挖掘水稻苗期耐冷QTL和有利等位基因
曹慧敏 杨贤莉 王立志 李苹苹 翟来圆 姜树坤 郑天清 邱先进,* 徐建龙,4,5,*
利用双向导入系和重组自交系定位和挖掘水稻苗期耐冷QTL和有利等位基因
曹慧敏1,2,**杨贤莉3,**王立志3李苹苹1翟来圆2姜树坤3郑天清2邱先进1,*徐建龙2,4,5,*
1农业农村部长江中游作物绿色高效生产重点实验室(省部共建), 长江大学农学院, 湖北荆州 434025;2中国农业科学院作物科学研究所, 北京 100081;3黑龙江省农业科学院耕作栽培研究所, 黑龙江哈尔滨 150086;4岭南现代农业科学与技术广东省实验室深圳分中心, 中国农业科学院农业基因组研究所, 广东深圳 518210;5海南省崖州湾种子实验室, 海南三亚 572024
苗期低温冷害常造成水稻生长发育受阻, 影响形态建成最终导致减产。鉴定和挖掘优良的耐冷基因, 选育耐冷水稻品种, 是减少冷害造成水稻产量损失的有效措施。本研究以籼稻明恢63和粳稻02428为亲本构建的双向导入系及重组自交系群体为研究材料, 通过人工气候室进行苗期耐冷性鉴定, 考查枯萎度和冷害恢复生长后的存活率, 结合重测序构建的Bin基因型数据, 进行苗期耐冷性QTL定位, 3个群体在1号、4号、5号、7号和12号染色体上共检测到13个与水稻苗期耐冷性相关的QTL, 解释0.36%~13.63%的表型变异。未定位到不同遗传背景下稳定表达的QTL, 但有5个同时影响枯萎度和存活率的QTL被检出, 分别位于1号、5号、7号和12号染色体, 其中位于1号染色体12,732,139~13,202,097 bp和14,445,778~14,585,009 bp及5号染色体14,658,891~15,684,510 bp的3个QTL的LOD值都高于25, 被认为是可靠的主效QTL。结合基因注释、表达谱数据和3K种质资源的苗期耐冷表型数据分析, 推测位于5号染色体区间内影响枯萎度和存活率的基因很可能是已克隆的耐冷基因, 而1号染色体2个区间的2个基因是新基因位点, 其候选基因分别是和。单倍型分析结果表明,的Hap2、Hap4和Hap9,的Hap1、Hap8和Hap10为有利单倍型。研究结果将为水稻品种耐冷分子改良提供宝贵的耐冷资源和有利基因。
水稻; 苗期耐冷; 枯萎度; QTL定位; 单倍型
水稻(L.)是全球约半数人口的主食, 总产量居全球粮食作物产量第2位。作为喜温作物, 水稻容易受到冷害的影响。冷害对水稻的伤害程度一般取决于其生长时期、冷害的严重程度及低温持续的时间长短[1]。我国水稻种植区域分布范围广, 东北地区、长江流域、珠江流域及部分高海拔地区均会遭受到不同程度的低温冷害。苗期是水稻最易受到冷害的时期, 幼苗叶片受冷害后会变黄、卷曲或枯萎, 生长缓慢, 严重时还会死苗烂秧, 最终导致水稻成活率降低和水稻减产。苗期耐冷性与芽期、孕穗开花期的耐冷性相关, 其鉴定结果也可为后期耐冷性评价提供重要的参考[2]。因此, 挖掘优异的耐冷基因、培育耐冷品种既能保证低温地区的水稻产量, 又能扩大水稻的种植范围。
水稻苗期耐冷性是复杂的数量性状, 前人采用不同的遗传群体及耐冷鉴定方法定位和克隆到了多个耐冷QTL/基因, 分布于水稻12条染色体上。Lou等[3]以耐冷品种AAV002863和低温敏感品种珍汕97B为亲本构建的DH群体共定位到5个苗期耐冷QTL, 分别位于1号、2号和8号染色体上。Zhao等[4]在不同低温处理条件下, 利用耐冷品种LTH (丽江新团黑谷)和低温敏感品种SHZ-2 (三黄占2号)为亲本构建的重组自交系群体定位到1个苗期耐冷QTL, 并鉴定到一个提高苗期耐冷的新基因。Kim等[5]利用粳稻品种Jinbu和籼稻品种BR29为亲本构建的重组自交系群体, 在1号、2号、4号、10号和 11号染色体上共定位到6个苗期耐冷性相关的QTL, 其中位于1号和11号染色体上的QTL被精细定位。是已克隆的苗期耐冷性基因, 编码一个G蛋白信号调节因子, 位于细胞膜和内质网上, 与G蛋白α亚基RGA1互作感受低温, 激活钙离子通道, 增强G蛋白GTP酶活性, 最终增强水稻抗寒的能力[6]。的2个等位基因和均负调控水稻对低温胁迫的反应[7]。Chen等[8]证明转录因子和互作并调控靶基因和的表达, 以此来调节水稻耐冷性。此外, 与和对水杨酸和茉莉酸处理均有响应, 这种共调控模式在拟南芥中也有报道, 而且被认为是水杨酸和茉莉酸介导的植物防御信号的汇聚节点[9-10]。MicroRNA319 (miR319)是miRNA家族之一, 水稻中的基因家族包括和, 在RNA干涉植株中, 下调2个miR319靶标基因和中任意一个的表达, 会导致耐冷性的增强[11]。
虽然目前已经定位和克隆了一些水稻耐冷的QTL/基因, 但这些耐冷QTL/基因表达的遗传背景效应不清楚, 尤其具有育种利用价值的耐冷基因还很匮乏, 远远不能满足水稻耐冷遗传改良的需求。
本研究以籼稻品种明恢63和粳稻品种02428为亲本构建的双向导入系和重组自交系为研究材料, 对水稻进行苗期耐冷性鉴定, 结合已有基因型数据进行QTL定位, 并针对重要耐冷位点, 利用3K种质资源进行候选基因的单倍型分析, 挖掘有利等位基因。研究结果将为精细定位和克隆苗期耐冷基因提供理论基础, 为分子育种改良水稻耐冷性提供有利基因。
1 材料与方法
1.1 试验材料
本研究以籼稻品种明恢63 (MH63)和粳稻品种02428为亲本构建的双向导入系和重组自交系为研究材料[12], 其中明恢63背景的导入系(MH63-ILs)共226份, 02428背景的导入系(02428-ILs)共198份, 重组自交系(RILs)共245份。将明恢63和02428进行全基因组重测序后获得的5,336,108,154和5,562,905,674个碱基[12]与作为参考基因组的日本晴序列(IRGSP 1.0)进行对比, 明恢63和02428分别比对上5,062,106,567和5,278,080 725个碱基, 占全基因组的96.57%和94.03%。根据序列比对, 利用双亲测序中3倍、4倍和5倍的测序片段分别检测到48,498、42,124和36,140个SNP, 进行归并后双亲共鉴定到58,936个SNP。利用3套群体采用Xie等[13]的方法构建了含有4568个Bin标记的遗传连锁图谱。
为对定位到的重要苗期耐冷QTL进行单倍型分析挖掘有利等位基因, 本研究还从3K种质资源中随机挑选了1548个资源材料。这些种质资源来源于全球68个国家, 包括印度(227份)、中国(199份)、印度尼西亚(142份)、菲律宾(119份)、孟加拉国(108份)、老挝(103份)、泰国(75份)、缅甸(46份)、柬埔寨(42份)、马达加斯加(42份)、尼泊尔(32份)、越南(32份)、马来西亚(31份)、巴基斯坦(28份)、意大利(25份)、美国(22份)、科特迪瓦(21份), 其他地区合计233份。这些种质资源共分为5个亚群, 其中籼稻845份、粳稻475份、Aus稻135份、Basmati稻50份和中间型43份。
1.2 苗期耐冷性评价
2022年在黑龙江省农业科学院耕作栽培研究所人工气候室进行苗期耐冷试验。选取饱满的水稻种子在50℃的烘箱中放置2~3 d打破休眠, 用2%的过氧化氢消毒液对种子进行消毒30 min并用自来水冲洗干净, 在30℃黑暗条件下浸种48 h, 28℃的条件下催芽12 h, 选取发芽一致的种子播种在装满壤土的育秧盘中, 每个材料播种约50粒, 2次重复。在28℃环境中将幼苗培养至三叶一心期后低温(9℃)处理5 d, 然后在正常条件下恢复生长7 d后调查枯萎度(wilting degree, WD)以及存活率(survival rate, SR)[14-15]。枯萎度的评价标准参考韩龙植等[14]的方法。
1.3 数据分析及QTL定位
利用Microsoft Excel 2016进行耐冷性的统计描述分析, 利用ICI Mapping 4.2软件的完备区间作图法(ICIM)在BIP模块中定位水稻苗期耐冷QTL[16], 并估算每个QTL对表型的加性效应和贡献率。将02428基因型定义为A, 明恢63基因型定义为B, 杂合型定义为H, QTL定位阈值设置为LOD≥2.5。
1.4 有利等位基因挖掘
针对不同群体同时定位到影响同一性状或相同遗传背景定位到影响不同性状的重要QTL, 在RGAP数据库(http://rice.uga.edu/)查找QTL区间内的候选基因, 根据基因注释、表达谱数据和相关文献报道预测QTL区间内可能的候选基因, 结合3K种质资源进行单倍型分析。以启动子区1 kb以内和基因编码区的SNP构建单倍型, 并根据Zhang等[17]的方法将同义SNP合并为一个单倍型。每个单倍型至少有30份种质, 使用R中的“agricolae”包完成显著性检验, 比较单倍型之间的表型差异, 挖掘苗期耐冷候选基因的有利单倍型。
2 结果与分析
2.1 不同群体的供体导入频率
4568个Bin组成的遗传连锁图谱总长度为总长为373.24 Mb, 相邻标记间的平均距离为81.71 kb。明恢63背景导入系的供体基因组平均导入比例为7.16% (0~86.51%), 02428背景导入系的供体基因组平均导入比例19.84% (0~100%), 重组自交系中明恢63基因组所占比例为46.90% (图1)。显然, 粳稻供体导入籼稻背景的比例要明显低于籼稻供体导入粳稻背景的比例; 重组自交系群体中, 粳稻亲本基因组所占的比例略高于籼稻亲本。
2.2 亲本与群体苗期的耐冷表现
双亲苗期的耐冷性差异很大(表1), 02428的枯萎度为1, 存活率为81.9%, 耐冷性强; 明恢63的枯萎度为8, 存活率为15.69%, 耐冷性较差, 表明在低温胁迫下, 02428更易存活。明恢63背景导入系、02428背景导入系和重组自交系3个群体的枯萎度和存活率均呈偏态分布, 且均出现超亲分离, 表明苗期耐冷性是受多基因控制的数量遗传性状(图2)。
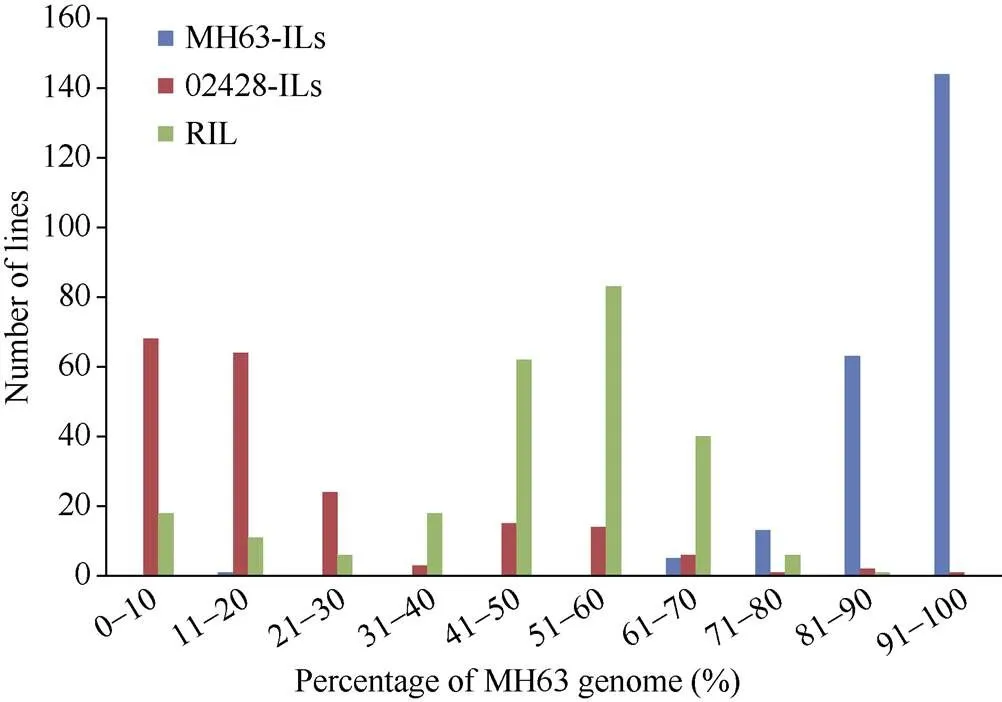
图1 双向导入系和重组自交系群体的明恢63基因组成成分的频数分布
2.3 苗期耐冷性QTL定位
明恢63背景导入系共定位到1个影响枯萎度和2个影响存活率的QTL, 分别位于水稻5号和12号染色体, 对表型的贡献率分别为8.17%、5.86%和3.28% (表2和图3); 02428等位基因在和均提高苗期耐冷性。02428背景导入系共定位到4个苗期耐冷QTL, 分别位于水稻4号和12号染色体上, 对表型的平均贡献率为9.13%; 其中, 明恢63等位基因在和增强苗期耐冷性, 在和则降低苗期耐冷性。重组自交系群体共鉴定到6个苗期耐冷QTL, 分别位于1号和7号染色体, 解释0.36%~10.67%的表型变异; 其中明恢63等位基因在和处提高苗期耐冷性, 02428等位基因则在、、和处提高苗期耐冷性。
在不同遗传背景下没有定位到影响同一性状的相同主效QTL, 但在5个染色体区间同时检测到影响幼苗枯萎度和存活率的主效QTL (表2), 其中明恢63背景导入系检测到5号染色体14,658,891~ 15,684,510 bp区间, 02428背景导入系检测到12号染色体26,529,940~26,660,385 bp区间, 重组自交系检测到1号染色体14,445,778~14,585,009 bp和7号染色体16,793,414~17,068,785 bp区间, 提高水稻苗期耐冷性的等位基因均来自02428; 重组自交系检测到1号染色体12,732,139~13,202,097 bp区间, 提高苗期耐冷的等位基因来源于明恢63。
2.4 候选基因预测和有利等位基因挖掘
在定位到的13个耐冷QTL中, 明恢63背景导入系群体位于5号染色体14,658,891~15,684,510 bp区间和重组自交系群体位于1号染色体12,732,139~ 13,202,097 bp和14,445,778~14,585,009 bp区间的QTL同时影响枯萎度和存活率, 其LOD值均大于25 (表2), 可以认为是比较可靠的重要主效QTL。前人在5号染色体的14,658,891~15,684,510 bp区间内已克隆了一个耐冷相关基因[7]; 1号染色体的12,732,139~ 13,202,097 bp和14,445,778~14,585,009 bp区间是新定位到的苗期耐冷QTL所在区间, 共有47个候选基因,根据基因注释和表达差异数据(图4-C, F), 最终预测区间内对苗期耐冷有影响的2个可能候选基因, 分别为和。
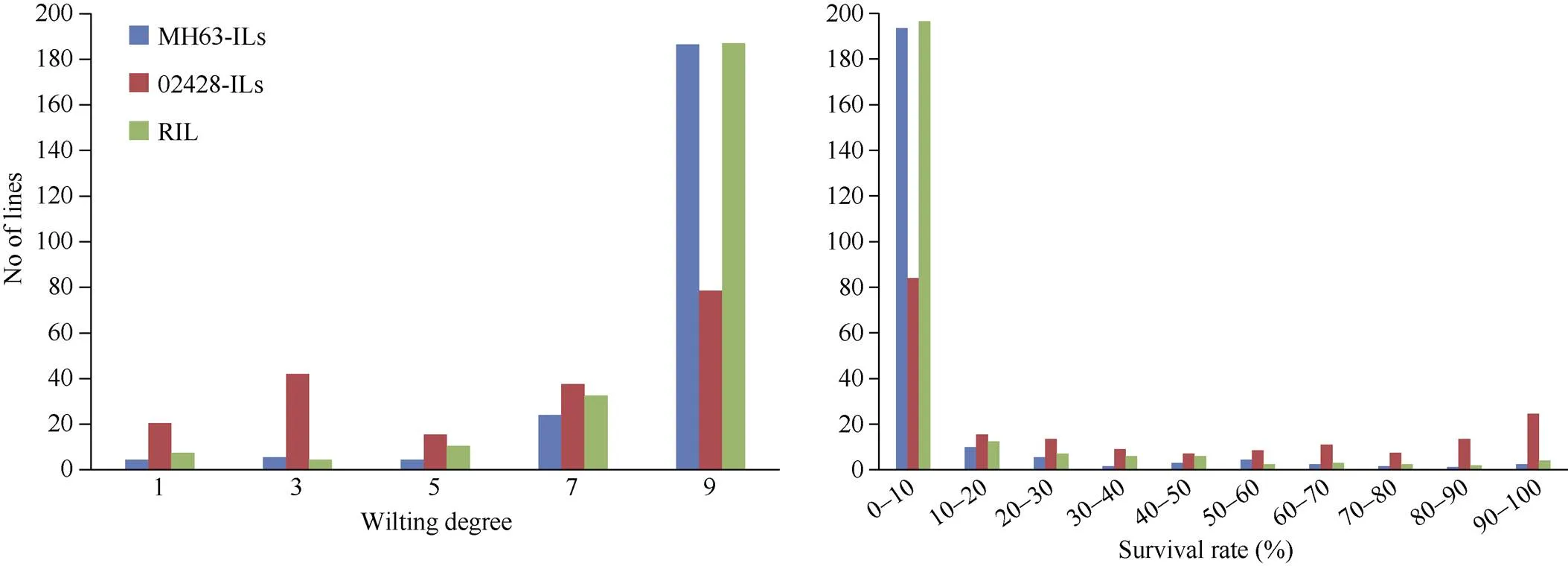
图2 明恢63和02428衍生群体的苗期耐冷分布

表1 亲本、双向导入系和重组自交系群体苗期耐冷表现
**代表亲本在<0.01水平差异显著。
**represents significantly difference at the 0.01 probability level between two parents.

表2 明恢63和02428衍生群体定位到的苗期耐冷QTL
WD: 枯萎度; SR: 存活率。加性效应正值表示增效等位基因来源于02428, 负值表示增效等位基因来源于明恢63。
WD: wilting degree; SR: survival rate. The positive value of additive effect means the favorable allele from 02428, while the negative value of additive effect means favorable allele from MH63.

图3 明恢63和02428衍生群体定位到的水稻苗期耐冷QTL在染色体上的分布
WD: 枯萎度; SR: 存活率。WD: wilting degree; SR: survival rate.
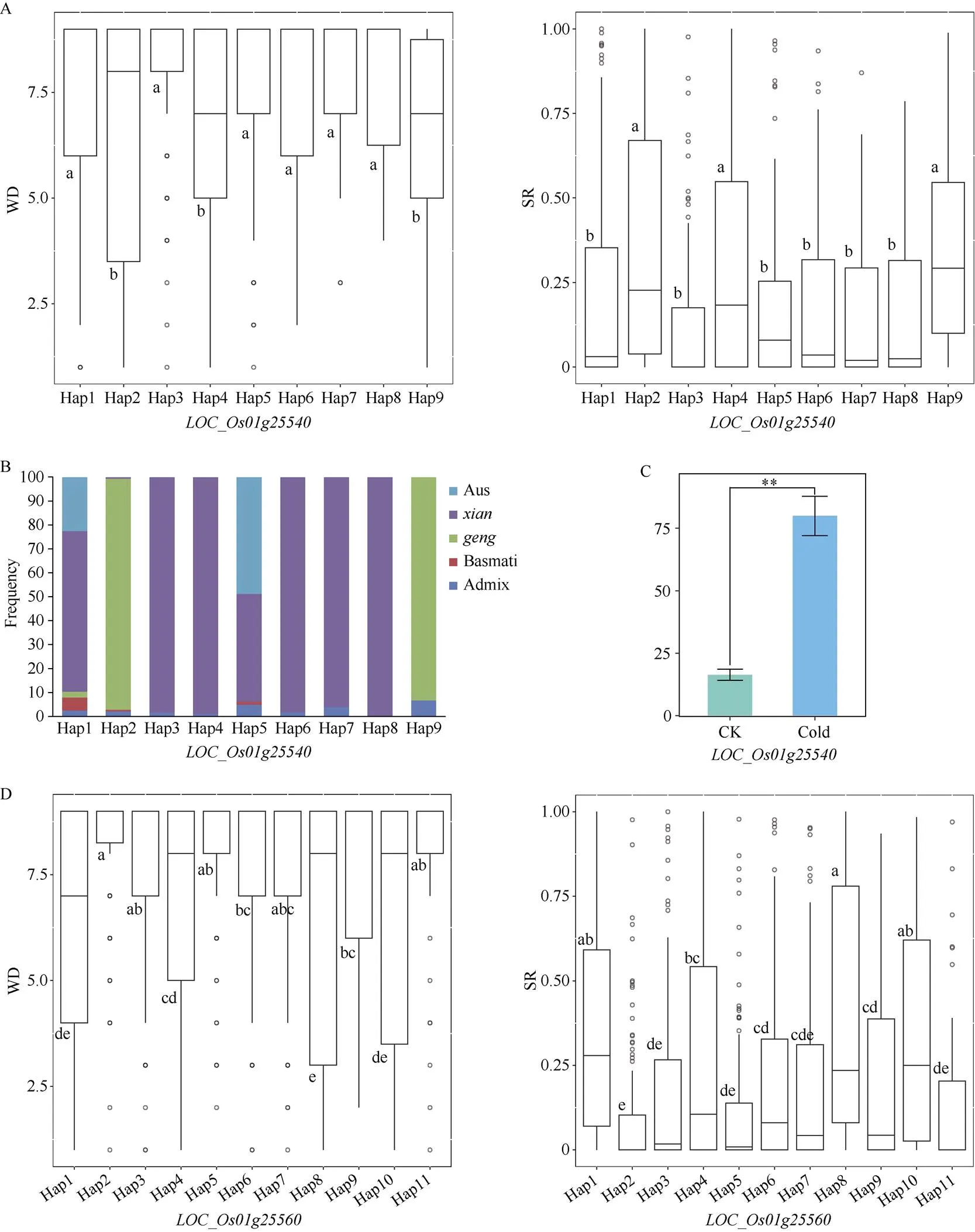
(图4)
A、D: 2个耐冷性状不同单倍型间的表型差异, 不同字母代表不同单倍型之间表型在< 0.01水平差异显著; B、E: 不同亚群在不同单倍型内的频率分布; C: 低温和常温条件下空育131苗期叶片的表达量(数据来源于Yu等[18]的报道); F: 低温和常温条件下日本晴苗期根部的表达量(数据来源于Yu等[18]的报道); **和***分别代表表达量在< 0.01和< 0.001水平差异显著。
A, D: phenotypic difference between different haplotypes of the two cold tolerance traits, the different lowercase letters indicate significant differences among different haplotypes at the 0.01 probability level; B, E: frequency distribution of five subpopulations in the different haplotypes; C: the relative expression level ofof leaf of Kongyu 131 at seedling stage under cold and normal conditions (data reported by Yu et al.[18]); F: the relative expression level ofof root of Nipponbare at seedling stage under cold and normal conditions (data reported by Yu et al.[18]); ** and *** represent significant difference at the 0.01 and 0.001 probability levels, respectively.
在3K种质资源中共有9种单倍型(图4-A和附表1), Hap2、Hap4和Hap9的枯萎度显著低于其他几种单倍型, 存活率显著高于其他几种单倍型, 为有利单倍型(图4-A)。其中Hap2和Hap9主要分布于粳稻中(图4-B), 耐冷品种空育131在冷处理后的表达量上升[18](图4-C), 表明该基因可能正调控水稻苗期耐冷性。在种质资源共11种单倍型(图4-D和附表1), 其中Hap1、Hap8和Hap10的枯萎度显著低于Hap2、Hap3、Hap5、Hap6、Hap7、Hap9和Hap11, 存活率显著高于Hap2、Hap3、Hap5、Hap6、Hap7、Hap9和Hap11, 为有利单倍型(图4-D)。这3种单倍型也主要存在于粳稻种质中(图4-E), 日本晴在冷处理后该基因的表达量下降[18](图4-F), 表明该基因可能负调控水稻苗期耐冷性。
3 讨论
本研究利用明恢63和02428为亲本构建的双向导入系和重组自交系群体, 共定位到13个水稻苗期耐冷QTL, 其中有6个QTL与前人克隆的耐冷基因处于相同或相近区间。例如, 明恢63背景导入系5号染色体上影响枯萎度的和影响存活率的与处于相同位置[7], 12号染色体上影响存活率的与已克隆的耐冷基因和的位置相近[8]; 02428背景导入系4号染色体上影响枯萎度的与已克隆的孕穗期耐冷基因处于相近位置[19], 12号染色体上影响枯萎度的和影响存活率的与耐寒相关基因和位置相近[11]。后续要通过精细定位和转基因验证确定这些QTL与已克隆基因是相同基因还是紧密连锁的基因。
QTL检测受遗传背景影响较大。王韵等利用粳稻Lemont和籼稻特青构建的双向回交导入系定位影响抽穗期和株高的QTL, 只有5个主效QTL在2种背景下同时被检测到, 占15.2%[20]; Liu等[12]在明恢63和02428构建的双向导入系中分别定位到9个耐铁QTL和10个耐锌QTL, 其中仅2个QTL在不同背景中被共同检测到, 占10.5%; Wang等[21]利用特青背景导入系和Lemont背景导入系分别定位到29个和23个抗旱QTL, 只有5个在不同背景中同时检测到。这些研究表明, 不同遗传背景能同时定位到的QTL数量都较少, 遗传背景对QTL的检测影响较大。本研究定位到的13个苗期耐冷QTL中, 未鉴定到在不同遗传背景稳定表达的QTL, 再次表明遗传背景对水稻苗期耐冷性QTL的检测影响大。因此, 利用分子育种进行水稻耐冷品种改良时, 应特别注意QTL定位群体与育种群体的异同, 在进行分子标记辅助选择前要确认QTL在育种群体中是否具有明显的表型效应。只有在育种群体中有显著表型效应的QTL, 才能借助标记辅助选择用于育种群体的目标性的改良。
一般来说, 籼稻的耐冷性劣于粳稻, 利用籼粳稻进行回交育种时, 虽然籼稻供体亲本苗期表现为冷敏感, 但以粳稻为背景的回交后代中仍出现了耐冷性强于轮回亲本的个体, 表明有些耐冷有利基因会以“隐蔽”的方式存在于水稻种质资源中[22]。枯萎度和存活率的鉴定结果显示(表1), 02428为耐冷粳稻品种, 明恢63为冷敏感籼稻品种。在02428导入系群体中仍筛选到27份存活率高于轮回亲本02428的耐冷导入系, 其中5份材料拥有43%以上明恢63导入片段, 分别是DQ355、DQ357、DQ424、DQ456、DQ464, 说明耐冷有利基因以一种“隐蔽”的形式存在于亲本明恢63中。因此, 在耐冷性改良育种工作中应重视这些“隐蔽”的有利基因, 培育回交导入系结合分子标记辅助选择技术挖掘存在于供体亲本中的“隐蔽”耐冷基因, 进而通过不同耐冷基因的聚合来进一步提高粳稻品种的耐冷性。
4 结论
本研究共定位到13个影响苗期耐冷性的QTL, 其中5个同时影响枯萎度和存活率的QTL被检出, 分别位于1号、5号、7号和12号染色体, 位于1号染色体12,732,139~13,202,097 bp和14,445,778~ 14,585,009 bp及5号染色体14,658,891~15,684,510 bp的3个QTL属效应较大的主效QTL。苗期耐冷QTL定位受遗传背景影响较大, 不同遗传背景下没有检测到影响同一性状稳定表达的耐冷QTL, 但影响枯萎度和存活率的QTL具有较大的遗传重叠。对位于1号染色体上的2个新的耐冷区间(12,732,139~ 13,202,097 bp和14,445,778~14,585,009 bp), 鉴定到和2个候选基因,利用3K种质资源分别挖掘到的Hap2、Hap4和Hap9、的Hap1、Hap8和Hap10 等6个有利单倍型。
[1] Li T G, Visperas R M, Vergara B S. Correlation of cold tolerance at different growth stages in rice., 1981, 23: 203–207.
[2] 李霞, 戴传超, 程睿, 陈婷, 焦德茂. 不同生育期水稻耐冷性的鉴定及耐冷性差异的生理机制. 作物学报, 2006, 32: 76–83. Li X, Dai C C, Cheng R, Chen T, Jiao D M. Identification for cold tolerance at different growth stages in rice (L.) and physiological mechanism of differential cold tolerance., 2006, 32: 76–83 (in Chinese with English abstract).
[3] Lou Q J, Chen L, Sun Z X, Xing Y Z, Li J, Xu X Y, Mei H W, Luo L J. A major QTL associated with cold tolerance at seedling stage in rice (L.)., 2007, 158: 87–94.
[4] Zhao J L, Zhang S H, Dong J F, Yang T F, Mao X X, Liu Q, Wang X F, Liu B. A novel functional gene associated with cold tolerance at the seedling stage in rice., 2017, 15: 1141–1148.
[5] Kim S, Suh J, Lee C, Lee J, Kim Y, Jena K K. QTL mapping and development of candidate gene-derived DNA markers associated with seedling cold tolerance in rice (L.)., 2014, 289: 333–343.
[6] Ma Y, Dai X Y, Xu Y Y, Luo W, Zheng X M, Zeng D L, Pan Y J, Lin X L, Liu H H, Zhang D J, Xiao J, Guo X Y, Xu S J, Niu Y D, Jin J B, Zhang H, Xu X, Li L G, Wang W, Qian Q, Ge S, Chong K. COLD1 confers chilling tolerance in rice., 2015, 160: 1209–1221.
[7] Tao Z, Kou Y J, Liu H B, Li X H, Xiao J H, Wang S P.alleles play different roles in abscisic acid signalling and salt stress tolerance but similar roles in drought and cold tolerance in rice., 2011, 62: 4863–4874.
[8] Chen L P, Zhao Y, Xu S J, Zhang Z Y, Xu Y Y, Zhang J Y, Chong K. OsMADS57 together with OsTB1 coordinates transcription of its targetandto switch its organogenesis to defense for cold adaptation in rice., 2018, 218: 219–231.
[9] Ryu H S, Han M, Lee S K, Cho J I, Ryoo N, Heu S, Lee Y H, Bhoo S H, Wang G L, Hahn T R, Jeon J S. A comprehensive expression analysis of thegene superfamily in rice plants during defense response., 2006, 25: 836–847.
[10] Li J, Brader G, Palva E T. The WRKY70 transcription factor: a node of convergence for jasmonate-mediated and salicylate-mediated signals in plant defense., 2004, 16: 319–331.
[11] Yang C C, Li D Y, Mao D H, Liu X, Ji C J, Li X B, Zhao X F, Cheng Z K, Chen C Y, Zhu L H. Overexpression of microRNA319 impacts leaf morphogenesis and leads to enhanced cold tolerance in rice (L.)., 2013, 36: 2207–2218.
[12] Liu H, Soomro A, Zhu Y J, Qiu X J, Chen K, Zheng T Q, Yang L W, Xing D Y, Xu J L. QTL underlying iron and zinc toxicity tolerances at seedling stage revealed by two sets of reciprocal introgression populations of rice (L.)., 2016, 4: 280–289.
[13] Xie W B, Feng Q, Yu H H, Huang X H, Zhao Q, Xing Y Z, Yu S B, Han B, Zhang Q. Parent-independent genotyping for constructing an ultrahigh-density linkage map based on population sequencing., 2010, 107: 10578–10583.
[14] 韩龙植, 张三元. 水稻耐冷性鉴定评价方法. 植物遗传资源学报, 2004, 5: 75–80. Han L Z, Zhang S Y. Methods of characterization and evaluation of cold tolerance in rice., 2004, 5: 75–80.
[15] Morsy M R, Jouve L, Hausman J, Hoffmann L, Stewart J M. Alteration of oxidative and carbohydrate metabolism under abiotic stress in two rice (L.) genotypes contrasting in chilling tolerance., 2007, 164: 157–167.
[16] Meng L, Li H H, Zhang L Y, Wang J K. QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations., 2015, 3: 269–283.
[17] Zhang F, Wang C C, Li M, Cui Y R, Shi Y Y, Wu Z C, Hu Z Q, Wang W S, Xu J L, Li Z K. The landscape of gene- CDS-haplotype diversity in rice: Properties, population organization, footprints of domestication and breeding, and implications for genetic improvement., 2021, 14: 787–804.
[18] Yu Y M, Zhang H, Long Y P, Shu Y, Zhai J X. Plant public RNA-seq database: a comprehensice online database for expression analysis of ~45000 plant public RNA-seq libraries., 2022, 20: 806–808.
[19] Li J L, Zeng Y W, Pan Y H, Zhou L, Zhang Z Y, Guo H F, Lou Q J, Shui G H, Huang H G, Tian H, Guo Y M, Yuan P R, Yang H, Pan G J, Wang R Y, Zhang H L, Yang S H, Guo Y, Ge S, Li J J, Li Z C. Stepwise selection of natural variations atandimproves cold adaptation during domestication ofrice., 2021, 231: 1056–1072.
[20] 王韵, 程立锐, 孙勇, 周政, 朱苓华, 徐正进, 徐建龙, 黎志康. 利用双向导入系解析水稻抽穗期和株高QTL及其与环境互作表达的遗传背景效应. 作物学报, 2009, 35: 1386–1394. Wang Y, Cheng L R, Sun Y, Zhou Z, Zhu L H, Xu Z J, Xu J L, Li Z K. Genetic background effect on QTL expression of heading date and plant height and their interaction with environment in reciprocal introgression lines of rice., 2009, 35: 1386–1394 (in Chinese with English abstract).
[21] Wang Y, Zhang Q, Zheng T Q, Cui Y R, Zhang W Z, Xu J L, Li Z K. Drought-tolerance QTLs commonly detected in two sets of reciprocal introgression lines in rice., 2014, 65: 171.
[22] 张帆, 郝宪彬, 高用明, 华泽田, 马秀芳, 陈温福, 徐正进, 朱苓华, 黎志康. 利用籼稻资源中的“隐蔽有利基因”提高粳稻苗期耐冷性. 作物学报, 2007, 33: 1618–1624. Zhang F, Hao X B, Gao Y M, Hua Z T, Ma X F, Chen W F, Xu Z J, Zhu L H, Li Z K. Improving seedling cold tolerance ofrice by using the “Hidden Diversity” inrice germplasm in backcross breeding program., 2007, 33: 1618–1624 (in Chinese with English abstract).
附表1 两个候选基因的单倍型信息
Table S1 Haplotype information of the two candidate genes
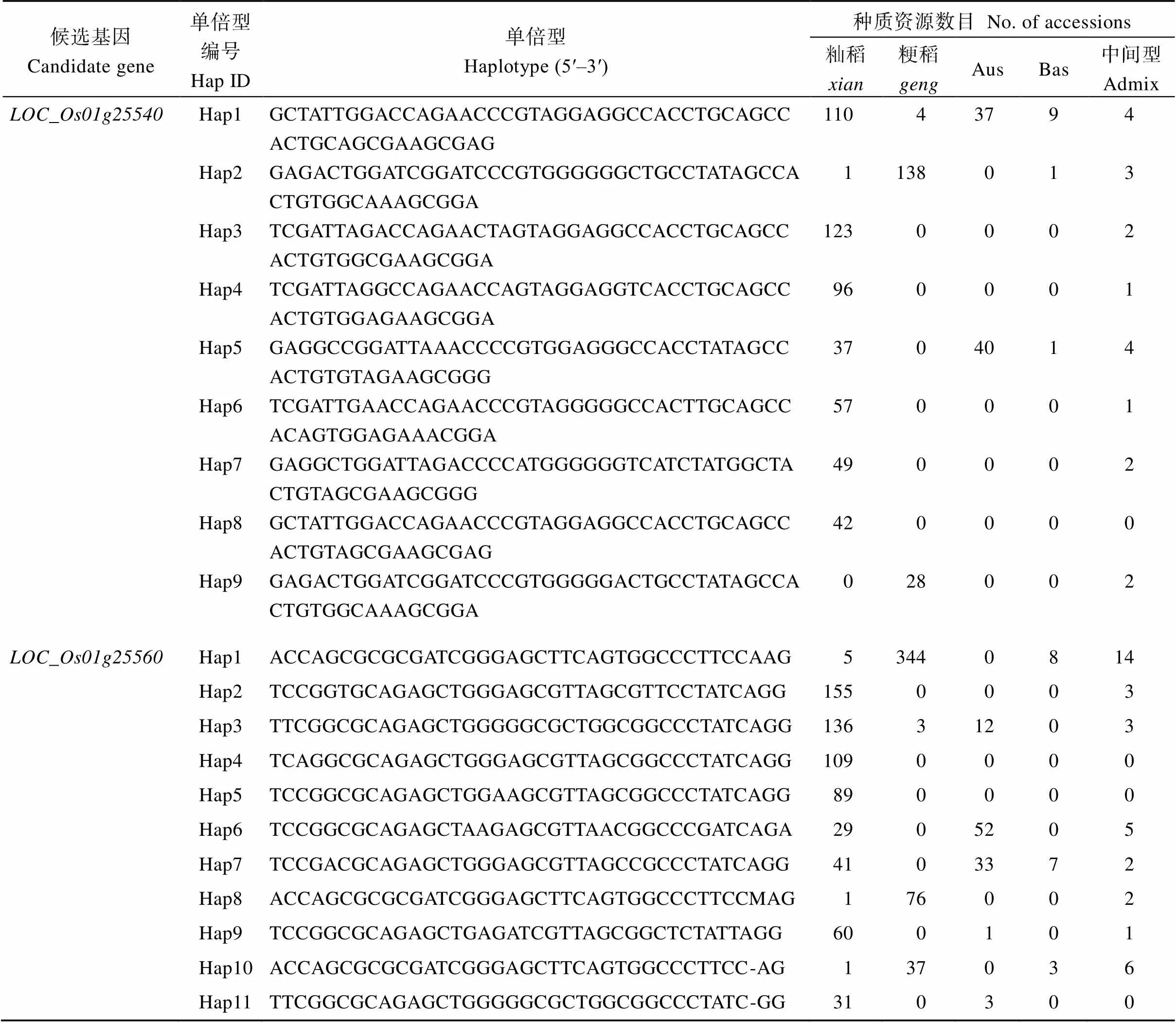
候选基因Candidate gene单倍型编号Hap ID单倍型Haplotype (5′–3′)种质资源数目 No. of accessions 籼稻xian粳稻gengAusBas中间型Admix LOC_Os01g25540Hap1GCTATTGGACCAGAACCCGTAGGAGGCCACCTGCAGCCACTGCAGCGAAGCGAG11043794 Hap2GAGACTGGATCGGATCCCGTGGGGGGCTGCCTATAGCCACTGTGGCAAAGCGGA1138013 Hap3TCGATTAGACCAGAACTAGTAGGAGGCCACCTGCAGCCACTGTGGCGAAGCGGA1230002 Hap4TCGATTAGGCCAGAACCAGTAGGAGGTCACCTGCAGCCACTGTGGAGAAGCGGA960001 Hap5GAGGCCGGATTAAACCCCGTGGAGGGCCACCTATAGCCACTGTGTAGAAGCGGG3704014 Hap6TCGATTGAACCAGAACCCGTAGGGGGCCACTTGCAGCCACAGTGGAGAAACGGA570001 Hap7GAGGCTGGATTAGACCCCATGGGGGGTCATCTATGGCTACTGTAGCGAAGCGGG490002 Hap8GCTATTGGACCAGAACCCGTAGGAGGCCACCTGCAGCCACTGTAGCGAAGCGAG420000 Hap9GAGACTGGATCGGATCCCGTGGGGGACTGCCTATAGCCACTGTGGCAAAGCGGA028002 LOC_Os01g25560Hap1ACCAGCGCGCGATCGGGAGCTTCAGTGGCCCTTCCAAG53440814 Hap2TCCGGTGCAGAGCTGGGAGCGTTAGCGTTCCTATCAGG1550003 Hap3TTCGGCGCAGAGCTGGGGGCGCTGGCGGCCCTATCAGG13631203 Hap4TCAGGCGCAGAGCTGGGAGCGTTAGCGGCCCTATCAGG1090000 Hap5TCCGGCGCAGAGCTGGAAGCGTTAGCGGCCCTATCAGG890000 Hap6TCCGGCGCAGAGCTAAGAGCGTTAACGGCCCGATCAGA2905205 Hap7TCCGACGCAGAGCTGGGAGCGTTAGCCGCCCTATCAGG4103372 Hap8ACCAGCGCGCGATCGGGAGCTTCAGTGGCCCTTCCMAG176002 Hap9TCCGGCGCAGAGCTGAGATCGTTAGCGGCTCTATTAGG600101 Hap10ACCAGCGCGCGATCGGGAGCTTCAGTGGCCCTTCC-AG137036 Hap11TTCGGCGCAGAGCTGGGGGCGCTGGCGGCCCTATC-GG310300
QTL identification and favorable allele mining of cold tolerance at seedling stage by reciprocal introgression and recombinant inbred line populations in rice
CAO Hui-Min1,2,**, YANG Xian-Li3,**, WANG Li-Zhi3, LI Ping-Ping1, ZHAI Lai-Yuan2, JIANG Shu-Kun3, ZHENG Tian-Qing2, QIU Xian-Jin1,*, and XU Jian-Long2,4,5,*
1Key Laboratory of Sustainable Crop Production in the Middle Researches of the Yangtze River,Ministry of Agriculture and Rural Affairs (Co-construction by Ministry and Province), College of Agriculture, Yangtze University, Jingzhou 434025, Hubei, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3Institute of Crop Cultivation and Tillage, Heilongjiang Academy of Agricultural Sciences, Harbin 150086, Heilongjiang, China;4Shenzhen Branch, Guangdong Laboratory for Lingnan Modern Agriculture, Agricultural Genomics Institute at Shenzhen, Chinese Academy of Agricultural Sciences, Shenzhen 518120, Guangdong, China;5Hainan Yazhou Bay Seed Laboratory, Sanya 572024, Hainan, China
Cold damage often hinders rice growth, affects its morphogenesis and leads finally to serious yield loss. Identifying favorable genes tolerant to cold, and breeding elite rice varieties with cold tolerance is an effective way to solve this problem. In this study, two sets of reciprocal introgression lines and a set of recombinant inbred line derived fromvariety Minghui 63 andvariety 02428 were evaluated for cold tolerance in climate chamber using cold tolerance-related traits such as wilting degree and survival rate after recovered growth. Combined with the available Bin genotype data generated by previous re-sequencing, 13 QTLs for cold tolerance at seedling stage were identified on chromosomes 1, 4, 5, 7, and 12, which explained 0.36% to 13.63% of phenotypic variation. No QTL was stably detected in different genetic backgrounds, whereas five QTLs were simultaneously detected for both wilting degree and survival rate on chromosomes 1, 5, 7, and 12. Among them, QTLs in the regions of 12,732,139–13,202,097 bp and 14,445,778–14,585,009 bp on chromosome 1, and 14,658,891–15,684,510 bp on chromosome 5 had high LOD values above 25 and were considered as reliable main-effect QTLs. Combined with gene annotation, gene expression profile and phenotyping data of cold tolerance of 3K germplasms, the previously cloned genefor cold tolerance was regarded as the responsible gene for the QTL in the region of 14,658,891–15,684,510 bp on chromosome 5, while the two candidate genes,andin the two regions on chromosome 1, were the new candidate genes for both wilting degree and survival rate. Haplotype analysis indicated that Hap2, Hap4, and Hap9 ofand Hap1, Hap8, and Hap10 ofwere favorable haplotypes for cold tolerance. The results provide the novel germplasms and favorable genes for molecular improvement of cold tolerance in rice.
rice; cold tolerance at seedling stage; wilting degree; QTL mapping; haplotype
10.3724/SP.J.1006.2023.32007
本研究由深圳市基础研究专项(JCYJ20200109150713553)和海南省崖州湾种子实验室揭榜挂帅项目(B21HJ0216)资助。
This study was supported by the Shenzhen Basic Research Special Project (JCYJ20200109150713553) and the Hainan Yazhou Bay Seed Lab Project (B21HJ0216).
邱先进, E-mail: xjqiu216@yangtzeu.edu.cn; 徐建龙, E-mail: xujianlong@caas.cn
**同等贡献(Contributed equally to this work)
曹慧敏, E-mail: chm98316@163.com; 杨贤莉, E-mail: aiwei.ni@163.com
2023-02-16;
2023-04-17;
2023-04-24.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20230424.0833.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

