基于转录组测序的姜黄侧根发育分析及相关基因筛选
李永宁,尹彦棚,周罗静,蓝 鑫,侯飞侠,彭 成,高继海
• 药材与资源 •
基于转录组测序的姜黄侧根发育分析及相关基因筛选
李永宁,尹彦棚,周罗静,蓝 鑫,侯飞侠,彭 成,高继海*
成都中医药大学 西南特色中药资源国家重点实验室,四川 成都 611137
姜黄主根形成中药姜黄,侧根形成药材郁金。为探知姜黄主根与侧根形成过程的差异和侧根生长膨大的分子机制,对姜黄主根与侧根进行转录组测序,挖掘促使侧根生长膨大的关键基因。以姜黄主根和侧根为材料,采用Illumina HiSeq高通量测序平台进行转录组测序,组装与注释后进行差异表达基因筛选。转录组测序共获得42.69 Gb clean data,共得到42 748条Unigene,其中35 456条被注释。主根和侧根中的差异表达基因(differentially expressed genes,DEGs)有1551个,(gene ontology,GO)富集结果表明,姜黄主根与侧根的DEGs主要富集于代谢过程、细胞过程、催化过程、转运过程、结合等功能,COG富集结果表明,主根与侧根中DEGs主要富集于碳水化合物代谢和信号转导机制。KEGG富集分析显示,根中DEGs主要富集在淀粉和糖代谢、植物激素信号传递等代谢通路中。通过注释信息筛选出姜黄侧根生长膨大相关的差异表达基因,关键差异表达基因与通路分析表明侧根中蔗糖合成酶基因、生长素响应基因(、、)的表达上调可能是姜黄侧根生长膨大的影响因素。通过对姜黄主根与侧根高通量转录组测序,揭示了姜黄侧根生长膨大的关键基因,可以为姜黄根系生长的生物学机制提供参考。
姜黄;转录组;主根;侧根;生长;代谢过程;细胞过程;催化过程;转运过程
姜黄L.为姜科(Zingiberaceae)姜黄属多年生草本植物,主根炮制后作药材“姜黄”,侧根炮制后作药材“郁金”,姜黄主根呈不规则倒卵圆形,表面深黄色,有明显环节;侧根呈纺锤形,表面棕灰色[1]。姜黄具破血行气、通经止痛的功效,郁金具活血止痛、行气解郁、清心凉血的功效。姜黄主根与侧根主要化学成分为姜黄素类和挥发油类成分,此外,主根中包含姜黄二酮、樟脑,侧根中包含郁金二酮、龙脑及樟脑等成分。现代药理表明姜黄主根及侧根有抗炎、抗肿瘤、抗氧化作用,侧根还可治疗抑郁,对心血管病有较好的治疗作用[2]。
姜黄根系生长膨大是多因素作用的复杂过程,受内源激素、蔗糖及淀粉代谢等相关因素的影响[3]。其中生长素是侧根生长的主要调控因子,在侧根发生的建成细胞选择、侧根原基的发育和侧根分生组织的激活3个阶段均起调控作用[4],可以促进侧根的生长发育[5-6]。侧根膨大需要物质和能量,在光合产物从源到库后,侧根作为库器官,蔗糖在库器官转化为干物质,促使侧根膨大。姜黄的侧根在10月下旬进入膨大期,部分侧根末端开始发育膨大,光合产物向侧根转移,侧根干物质量增加,于12月侧根质量达到最大值。而姜黄主根在8月开始膨大,11月中旬至次年1月为主根干物质量达到最大值[7]。姜黄主根与侧根的发育及膨大时间均不相同,乃至种植方式亦有区别,因此在实际生产中,受内外环境影响,形成犍为、沐川的姜黄道地产区和崇州、双流的郁金道地产区[8]。姜黄侧根相对主根的生长过程差异较多,其受生长素、蔗糖及淀粉代谢等其他内环境因素影响的分子机制不明,利用RNA-Seq技术,选择发育成熟的姜黄植株根系进行转录组测序研究,对姜黄侧根发育相关的差异表达基因进行筛选,从转录组水平探究姜黄侧根发育的基因表达特征,可为实际生产中姜黄主根与侧根的栽培和育种提供数据基础。
1 材料
姜黄于2021年12月采收于四川省犍为县(坐标:104°07′22″E,029°03′48″N),经成都中医药大学高继海副教授鉴定为姜科植物姜黄L.。挖取新鲜姜黄主根和侧根共6个样本,立即投入液氮保存,之后置于−80 ℃保存备用。主根编号为JH-1、JH-2、JH-3;侧根编号为YJ-1、YJ-2、YJ-3。
2 方法
2.1 总RNA的提取与文库构建
采用Trizol Kit (Burlington Co.,加拿大)提取各样品总RNA, Oligo (dT)磁珠分离纯化,将mRNA分解成短基因片段后反转录成cDNA链,委托北京百迈客生物科技有限公司构建测序文库。
2.2 转录组测序、组装及功能注释
采用Illumina Hiseq2500高通量测序平台对姜黄转录组文库进行测序。截取测序引物,过滤原始序列的低质量数据,得到clean reads。使用Trinity软件将测序Reads打断为较短是片段(K-mer),然后将这些片段延伸成较长的片段(Contig),并得到长片段集合Comonent(Unigene)。利用BLAST软件将Unigene序列与NCBI的非冗余蛋白序列(NR)、Swiss-Prot蛋白序列(Swiss-Prot)、(gene ontology,GO)、直源同源群集数据库(clusters of orthologous group,COG)、真核同源群数据库(eukaryotic orthologous groups,KOG)、eggNOG4.5、京都基因与基因组百科全书Kyoto encyclopedia of genes and genomes(KEGG)等数据库进行比对。使用KOBAS2.0得到了Unigene在KEGG中的KEGG Orthology结果,并得到了Unigene的注释信息。
2.3 基因表达与差异富集分析
利用TransDecoder软件预测Unigene的编码区域及其对应的氨基酸序列。用MISA软件对大于1 kb的Unigene进行SSR分析。利用STAR软件对每个样本的reads和Unigene序列进行比较,并通过GATK针对RNA-Seq的单核苷酸多态性(single nucleotide polymorphisms,SNP)识别(SNP Calling)流程,识别SNP位点。在FPKM表达量分析的基础上,设置错误率(false discovery rate,FDR≤0.5)、绝对值差异倍数(Fold Change,FC)≥2,采用DEGseq R进行姜黄转录组样品差异表达分析,使用Goseq R、KOBAS软件对差异表达基因(differentially expressed genes,DEGs)进行GO、COG、KEGG、KOG、Pfam、eggNOG等的富集分析。
2.4 实时荧光定量PCR(qRT-RCR)验证
随机选取5个参与生长素途径相关差异表达基因进行qRT-RCR验证,5个差异基因为生长素响应基因16(auxin response factor 16)、生长素运输蛋白BIG(auxin transport protein BIG)、生长素响应基因12(auxin response factor 12)、吲哚-3-乙酸-酰胺合成酶GH3.1(indole-3-acetic acid-amido synthetase GH3.1)、生长素响应基因7(Auxin transport protein 7),以测序实验同批次样品的cDNA为模板,使用为Actin内参基因,利用CDS序列和Primer5软件设计qRT-PCR特异性引物(表1)。采用SsoFastTMEva Green Su-peremix (Bio-Rad,美国)进行定量表达检测,同2−∆∆Ct方法计算基因的相对表达量。
表1 qRT-RCR扩增特异引物
Table 1 Specific primers of qRT-PCR amplification
基因ID正向引物(5’-3’)反向引物(5’-3’) actinAGGAACCACCGATCCAGACAGGTGCCCTGAGGTCCTGTT c410320.graph_c0CCTGCGGCTTGTGGATTAGTTTCGCTGTGACCTTGGGGA c409034.graph_c0CATGAGGATGACGCAGCAAACCGCAAACTGTCGGAGAACG c397769.graph_c0GGCAACCCCGTAGACATCTGATTCCCAACCTCAACACCCC c405834.graph_c0CTTTCGCTGGTTTGTACCGTTATCCACCTCGTCCGTCTTGTC c404082.graph_c0TACAGGGAGCCAGGCAAGATGCGCTAGGGGGACAGGAAACT
3 结果与分析
3.1 转录组测序与注释
对姜黄主根和侧根6个样品转录组文库进行测序,共获得42.69 Gb clean data,各样品clean data均达到6.48 Gb,Q30碱基百分比在93.32%及以上,其中主根70 279 161条clean reads,侧根72 305 640条Clean Reads,经过Trinity组装共得到42 748 条Unigene,50为1768,表明本实验姜黄转录组数据库质量较高,可用于后续数据分析。
通过选择BLAST参数E-value不大于1×10−5和HMMER参数E-value不大于1×10−10,最终获得35 456个有注释信息的Unigene,统计结果见表2。
3.2 差异表达分析
设置筛选差异条件为FDR<0.01且FC≥2作为筛选标准,共获得1551个差异基因,其中侧根表达量上调基因为675个,下调基因876个(图1)。
表2 Unigene注释统计
Table 2 Statistics of annotated Unigene
数据库注释数量长度≥300长度≥1000 COG10 362 4 297 6 065 GO25 66711 90713 760 KEGG20 082 8 93011 152 KOG17 341 7 766 9 575 Pfam24 48010 79413 686 Swissprot18 887 7 96810 919 TrEMBL29 16513 54115 624 eggNOG23 51710 31513 202 nr34 98718 04316 944 All35 45618 45517 001

图1 姜黄主根侧根DEGs统计
3.3 主根、侧根DEGs分析
将姜黄和郁金之间的DEGs注释到GO蛋白质数据库,按照功能被注释到生物过程(biological process)、细胞组分(cellular component)、分子功能(molecular function)3个大类58个功能小类,富集最多的是细胞组分亚类。与细胞组分相关的功能主要富集于细胞485个、细胞部分485个。与分子功能相关的富集功能主要有结合共622个,有254个DEGs上调和368个DEGs下调。与生物过程有关的功能主要富集在代谢过程489个(图2)。476条差异基因获得COG注释(图3),在注释到的26个功能分类中,其中碳水化合物运输和代谢包含的Unigene数量最多(G,84),其次是信号传导机制(T,53),一般功能预测项目(R,50)和次生代谢物的生物合成、运输和分解代谢(C,40)。

图2 姜黄主根与侧根中DEGs的GO功能分类

图3 姜黄主根与侧根转录组序列DEGs的COG分类
3.4 DEGs KEGG代谢通路分析
本研究将姜黄与郁金的差异基因在KEGG数据库对比,结果表明,有909条差异基因被注释到5个大类中,包括细胞过程、环境信息处理、遗传信息处理、新陈代谢和生物系统,富集最多的为新陈代谢类。差异基因被富集到115个代谢通路,按照差异基因注释量大小依次排序,选取前20个代谢通路(表3),主要注释到植物激素信号转导(plant hormone signal transduction)、碳代谢(carbon metabolism)、植物-病原互作(plant-pathogen interaction)、内质网蛋白加工(protein processing in endoplasmic reticulum)、淀粉和蔗糖代谢(starch and sucrose metabolism)、MAPK信号通路-植物(MAPK signaling pathway-plant)、氨基酸生物合成(biosynthesis of amino acids)等通路,富集程度最高的代谢通路为植物激素信号转导,有46个差异基因。在姜黄生长素信号通路途径上发现了10个生长素受体,相对姜黄主根,上调表达的生长素相关基因有6个,下调表达的4个,生长素IPA合成途径基因有4个,上调表达3个,下调表达1个(表4),表明相对主根,姜黄侧根生长素信号通路途径更活跃。其次为碳代谢和植物病原菌互作,分别有44、33个差异基因。此外淀粉和蔗糖代谢(29个)、苯丙素生物合成(22个)也有大量差异基因富集。生长素合成和响应关键差异表达基因热图和生长素促进植物根系生长通路图见图4。
表3 姜黄转录组DEGs KEGG通路分析统计
Table 3 KEGG pathway classifications of DEGs in C. longa transcriptome
编号通路通路IDDEGs数量 1植物激素信号转导(plant hormone signal transduction)ko0407546 2碳代谢(carbon metabolism)ko0120044 3植物-病原互作(plant-pathogen interaction)ko0462633 4内质网蛋白加工(protein processing in endoplasmic reticulum)ko0414129 5淀粉和蔗糖代谢(starch and sucrose metabolism)ko0050029 6MAPK信号通路-植物(MAPK signaling pathway-plant)ko0401629 7氨基酸生物合成(biosynthesis of amino acids)ko0123026 8糖酵解/糖异生(glycolysis/gluconeogenesis)ko0001022 9苯丙烷类化合物生物合成(phenylpropanoid biosynthesis)ko0094022 10剪接体(spliceosome)ko0304019 11氨基糖和核苷酸糖的代谢(amino sugar and nucleotide sugar metabolism)ko0052018 12乙醛酸和二羧酸代谢(glyoxylate and dicarboxylate metabolism)ko0063015 13半胱氨酸和蛋氨酸代谢(cysteine and methionine metabolism)ko0027014 14光合生物中的碳固定(carbon fixation in photosynthetic organisms)ko0071013 15内吞作用(endocytosis)ko0414412 16半乳糖代谢(galactose metabolism)ko0005212 17RNA运输(RNA transport)ko0301312 18核糖体(ribosome)ko0301011 19丙氨酸、天冬氨酸和谷氨酸代谢(alanine, aspartate and glutamate metabolism)ko0025011 20甘氨酸、丝氨酸和苏氨酸代谢(glycine, serine and threonine metabolism)ko0026011
表4 姜黄根中生长素途径基因
Table 4 Genes of auxin pathway regulated in roots of C. longa
基因编号表达变化基因注释 c397769.graph_c0上调生长素响应因子(auxin response factor) c391677.graph_c0上调生长素响应因子(auxin response factor SAUR32) c389801.graph_c0上调生长素响应因子(auxin response factor SAUR76) c409034.graph_c0上调生长素响应因子(auxin response factor) c407275.graph_c0上调生长素响应因子(auxin response factor AUX/IAA) c395155.graph_c0上调生长素响应因子(auxin response factor SAUR50) c410320.graph_c0下调生长素响应因子(auxin response factor AUX/IAA) c404082.graph_c0下调生长素响应因子(auxin response factor GH3) c405834.graph_c0下调生长素响应因子(auxin response factor) c407971.graph_c0下调生长素响应因子(auxin response factor AUX/IAA) c396355.graph_c0上调生长素合成酶(auxin response factor YUCCA2) c401872.graph_c0上调生长素合成酶(auxin response factor TAA4) c409165.graph_c0上调生长素合成酶(auxin response factor TAA2) c387677.graph_c0下调生长素合成酶(auxin response factor YUCCA10)

JH1~JH3-主根 YJ1~YJ3-侧根
3.5 转录因子分析
将所注释的Unigene与Plant TFDB(Plant transcription factor database)和Animal TFDB (Animal transcription factor database)数据库进行比较,在姜黄主根与侧根差异基因转录组数据中共有2510条Unigene被注释为转录因子,最多的转录因子类型有 C2H2、bZIP、bHLH、AP2/ERF等,其中数目最多的转录因子是C2H2(图5)。AP2/ERF家族转录因子中91个差异基因主要在侧根中高表达,反映该家族转录因子通过转录调控姜黄侧根发育中起重要作用。
3.6 qRT-PCR验证
本研究为验证转录组数据的可靠性,随机选取5个生长素相关差异表达基因进行qRT-PCR验证,5个差异基因的相对表达量与转录组数据的FPKM值变化趋势基本上一致(图6),结果表明生长素响应基因2、生长素响应基因16在侧根中表达上调,与转录组测序结果趋势基本一致,说明转录组数据结果可靠。

图5 姜黄转录因子家族统计
4 讨论
植物侧根生长膨大需要能量补充和生物量累积,在淀粉和糖代谢、碳代谢等基础代谢的共同作用下,侧根开始生长发育。本研究通过富集分析发现,姜黄主根和侧根在碳代谢、淀粉和糖代谢差异基因富集较多,侧根中淀粉和蔗糖代谢相关的基因表达上调,侧根膨大和蔗糖和淀粉代谢相关,姜黄侧根郁金是作为块根,淀粉是其生长膨大的原因之一,本研究中淀粉和蔗糖代谢通路富集差异基因较多,蔗糖合成酶通路找到3个基因sucrose synthase 1()、、上调,基因在植物中可以促进淀粉合成和生长发育,在百合鳞茎[9]、石蒜[10]、铁棍山药[11]、白及[12]等植物中得到证实,占雷雷[13]发现促进甘薯块根膨大,表明蔗糖合成酶可能是促进姜黄侧根膨大的重要因素(图4)。
植物激素是植物体内天然存在的一系列有机化合物,对植物根系发育起着重要的调节作用[14]。主要包括生长素(auxin,IAA)、脱落酸(abscisic acid,ABA)、赤霉素(gibberellin,GA)、细胞分裂素(cytokinin,CTK)、乙烯(ethylene)等。本实验通过对姜黄主根和侧根的转录组学差异分析发现,与姜黄根系生长发育相关调控激素的信号转导通路显著富集。
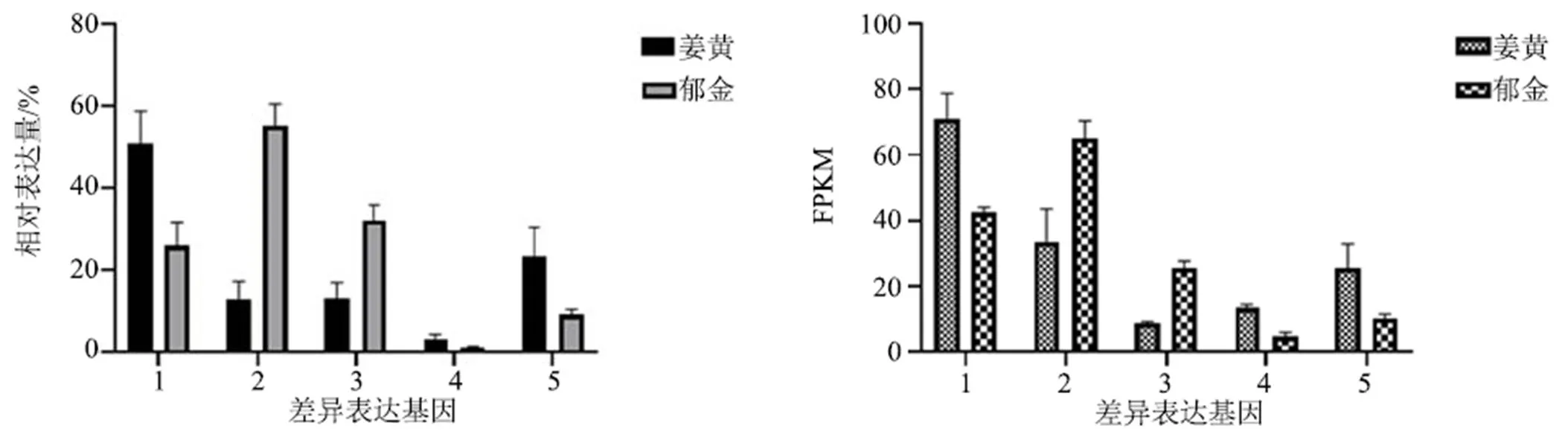
1-生长素响应基因16(c410320.graph_c0) 2-生长素运输蛋白BIG(c409034.graph_c0) 3-生长素响应基因2(c397769.graph_c0) 4-吲哚-3-乙酸-酰胺合成酶GH3.1(c405834.graph_c0) 5-生长素响应基因7(c404082.graph_c0)
生长素是侧根形成的主要激素调控因子[15],和是生长素吲哚-3-丙酮酸(indole-3-pyruvic acid,IPA)合成途径的关键酶[16],通过转氨基将色氨酸转变为IPA,IPA被进而催化为IAA[17],本研究中生长素合成途径的3个关键基因、和在姜黄侧根中表达上调,在侧根中促进生长素合成和积累。是生长素早期响应基因家族,在生长素调控下基因通过酸生长理论正向调控植物细胞膨胀[18],本研究中姜黄的生长素响应基因、和在姜黄侧根中表达上调,且上调倍数最高。酸生长理论提出生长素可以激活细胞质膜上的H+-ATP酶,降低细胞原生质体的PH,进而激活细胞壁上相关蛋白促使细胞膨胀[19]。研究发现基因可以在生长素诱导下激活H+-ATP酶,如通过促进磷酸化来激活质膜H+-ATP酶的活性,进而加速拟南芥细胞膨大伸长[20]。可以促进拟南芥侧根生长,是将生长素信号整合到乙烯信号中调控拟南芥根生长的,Li等[21]在研究中表明可以促进细胞扩张,可以和乙烯受体ETR2和EIN4相互作用,作为乙烯受体信号转导下游降低乙烯反应,促进植物生长。Markakis等[22]在拟南芥过表达显示,拟南芥植株根比敲除拟南芥植株根长20%,用IAA处理拟南芥幼苗后,拟南芥侧根原基的的表达量高于对照,证明了在侧根发育中的正向调控作用。此外有研究发现在拟南芥中亦可生长素刺激下通过诱导细胞壁酸化来促进细胞伸长[23],因此推测姜黄侧根膨大过程可能和侧根中生长素响应基因、和的表达上调有关。
基因家族是不同转录因子控制植物发育和生长的共同靶点,其启动子中有多种转录因子结合位点,如AP2/ERF类转录因子。AP2/ERF类转录因子是植物最大的转录因子家族之一,AP2/ERF转录因子可响应生长素的调节从而影响植物发育过程,如在杨树、菊花、拟南芥等植物中可以促进侧根生长[24],在姜黄主根与侧根间的DEGs中,筛选出含91个差异基因的AP2/ERF类转录因子,此外,在生长素诱导的液体培养基培养后,表达显著增加[23-24],故姜黄侧根发育可能和AP2/ERF类转录因子调控基因家族,特别是SAUR50有关。
本研究通过高通量转录组测序,初步揭示了促进姜黄侧根生长膨大的基因调控机制,对其中一些关键基因进行深入研究,可为姜黄侧根发育的生物学机制提供科学依据。
利益冲突 所有作者均声明不存在利益冲突
[1] 中国药典[S]. 一部. 2020: 263.
[2] 何雪娇, 余智城, 林金水, 等. 姜科姜黄属植物药用价值及其研究进展 [J]. 福建热作科技, 2020, 45(2): 52-55.
[3] 王柳萍, 覃坤坚, 赵立春, 等. 植物根膨大的研究进展 [J]. 湖北农业科学, 2020, 59(11): 5-9.
[4] 刘大同, 荆彦平, 李栋梁, 等. 植物侧根发育的研究进展 [J]. 植物生理学报, 2013, 49(11): 1127-1137.
[5] Cavallari N, Artner C, Benkova E. Auxin-regulated lateral root organogenesis [J]., 2021, 13(7): a039941.
[6] 邹锋康, 王秋红, 周建朝, 等. 生长素调节植物生长发育的研究进展 [J]. 中国农学通报, 2018, 34(24): 34-40.
[7] 刘峰群, 肖小河, 夏文娟, 等. 姜黄地下部分的形态发育动态与药材品质形成的初步研究 [J]. 解放军药学学报, 2000, 16(3): 131-133.
[8] 李青苗. 姜黄、郁金道地性差异及其形成机制研究 [D]. 雅安: 四川农业大学, 2014.
[9] 朱迎迎. 百合鳞茎发育过程中蔗糖合成酶基因的功能研究 [D]. 沈阳: 沈阳农业大学, 2020.
[10] 许俊旭, 李青竹, 李叶, 等. 石蒜鳞茎膨大过程中碳水化合物代谢相关基因的差异表达研究[J]. 核农学报, 2021, 35(10): 2250-2257.
[11] 刘苏伟, 文艺, 张骆琪, 等. 铁棍山药SUS基因CDS区克隆与生物信息学分析 [J]. 中国实验方剂学杂志, 2019, 25(9): 136-143.
[12] 蒋素华, 牛苏燕, 周一冉, 等. 白及蔗糖合成酶基因的克隆及表达分析 [J]. 广西植物, 2020, 40(2): 192-199.
[13] 占雷雷. 甘薯块根膨大和品质形成中蔗糖分解酶的初步探究 [D]. 海口: 海南大学, 2019.
[14] 黎家, 李传友. 新中国成立70年来植物激素研究进展 [J]. 中国科学: 生命科学, 2019, 49(10): 1227-1281.
[15] Casanova-Sáez R, Mateo-Bonmatí E, Ljung K. Auxin metabolism in plants [J]., 2021, 13(3): a039867.
[16] Cao X, Yang H L, Shang C Q,. The roles of auxin biosynthesisgene family in plants [J]., 2019, 20(24): 6343.
[17] 王海霞, 崔大勇. 陆生植物生长素合成的主要途径及调控 [J]. 生物学教学, 2015, 40(9): 2-5.
[18] Wong J H, Klejchová M, Snipes S A,. SAUR proteins and PP2C.D phosphatases regulate H+-ATPases and K+channels to control stomatal movements [J]., 2021, 185(1): 256-273.
[19] Ren H, Park M Y, Spartz A K,. A subset of plasma membrane-localized PP2C.D phosphatases negatively regulate SAUR-mediated cell expansion in[J]., 2018, 14(6): e1007455.
[20] Stortenbeker N, Bemer M. The SAUR gene family: The plant’s toolbox for adaptation of growth and development [J]., 2019, 70(1): 17-27.
[21] Li Z G, Chen H W, Li Q T,. Retraction Note: Three SAUR proteins SAUR76, SAUR77 and SAUR78 promote plant growth in[J]., 2022, 12(1): 1994.
[22] Markakis M N, Boron A K, van Loock B,. Characterization of a small auxin-up RNA (SAUR)-like gene involved indevelopment [J]., 2013, 8(11): e82596.
[23] van Mourik H, van Dijk A D J, Stortenbeker N,. Divergent regulation ofSAUR genes: A focus on the SAUR10-clade [J]., 2017, 17(1): 245.
[24] 悦曼芳, 张春, 吴忠义. 植物转录因子AP2/ERF家族蛋白结构和功能的研究进展 [J]. 生物技术通报, 2022(12): 11-26.
Screening and analysis of genes related to lateral root development inbased on transcriptome sequencing
LI Yong-ning, YIN Yan-peng, ZHOU Luo-jing, LAN Xin, HOU Fei-xia, PENG Cheng, GAO Ji-hai
State Key Laboratory of Southwestern Chinese Medicine Resources, Chengdu University of Traditional Chinese Medicine, Chengdu 611137, China
The main roots ofform Chinese medicine Jianghuang (), and the lateral roots form Yujin (). Transcriptome sequencing was conducted to explore the differences in the formation process of main roots and lateral roots ofand the molecular mechanism of growth and expansion of ateral roots, so as to explore the key genes promoting lateral root growth and expansion.The main roots and lateral roots ofwere used as materials, and transcriptome sequencing was performed by Illumina HiSeq high-throughput sequencing platform. After assembly and annotation, differentially expressed genes were screened.A total of 42.69 Gb Clean Data and 42 748 Unigenes were obtained by transcriptome sequencing, of which 35 456 were annotated. There were 1551 differentially expressed genes (DEGs) in the main roots and lateral roots. The GO enrichment results showed that the DEGs in the main and lateral roots ofwere mainly enriched in metabolic processes, cellular processes, catalytic processes, transport processes, binding and other functions, and the COG enrichment results showed that the DEGs in the main and lateral roots were mainly enriched in carbohydrate metabolism and signal transduction mechanisms. KEGG enrichment analysis showed that DEGs were mainly enriched in starch and sugar metabolism and plant hormone signaling pathways. The differentially expressed genes related to lateral root development were screened from the annotated information. The key differentially expressed genes and pathway analysis indicated that the up-regulation of sucrose synthase gene and auxin response genes (,,) in the lateral roots ofmay be the influencing factors of the lateral root growth expansion of
L.; transcriptome; main root; lateral root; growth; metabolic processes; cellular processes; catalytic processes; transport processes
R286.12
A
0253 - 2670(2023)14 - 4623 - 08
10.7501/j.issn.0253-2670.2023.14.023
2022-11-06
西南特色中药资源多维评价多学科交叉创新团队(ZYYCXTD-D-202209);中药品质性形成分子机制研究四川省青年科技创新研究团队(22CXTD0009);川产地道药材大品种精深加工关键技术及产品开发的研究与示范(2020YFN0152);杏林学者(QNXZ2018017,QNXZ2019001)
李永宁(1998—),男,研究生,研究方向为中药资源学。E-mail: liyongning2021@163.com
高继海(1983—),男,副教授,研究方向为中药资源与开发、种质资源研究。E-mail: gaojihai@cdutcm.edu.cn
[责任编辑 时圣明]

