混合线性非线性网络的多源miRNA-疾病关联预测方法
赵 静,李浩琳,王会青,王 彬
(太原理工大学信息与计算机学院, 太原 030600)
miRNA 是一类长度约为20 个核苷酸的非编码RNA[1],广泛存在于真核生物中,并在许多生物过程中发挥着至关重要的作用,如早期细胞生长、发育、增殖、分化、肿瘤侵袭和细胞凋亡等[1-3]。突变或生物功能障碍的miRNA 可能会导致疾病的发生。Calin等[4]通过研究miR15 和miR16 的频繁缺失和下调证明了miRNA 水平的降低与慢性淋巴细胞白血病之间存在显著相关性,Yang 等[5]研究发现肿瘤的发生或凋亡依赖于miRNA 的调控。随着 miRNA 研究的发展,miRNA 与疾病的关联已扩展到多种疾病,包括白血病[6]、肺癌[7]和乳腺癌[8]等。因此,研究miRNA与疾病之间的关联有助于从分子水平了解疾病的发病机制,对于研究疾病的预后、诊断、评估和治疗具有重要意义[9]。
在miRNA-疾病关联预测方面,大多数研究方法采用miRNA 功能相似性和疾病语义相似性作为输入,进行miRNA-疾病关联预测。Zhao 等[10]基于miRNA 功能相似性、疾病语义相似性,采用最近邻与SVM 对miRNA-疾病对加权投票,以预测miRNA-疾病关联。Chen 等[11]基于miRNA 功能相似性、疾病语义相似性构建决策树,并级联多个决策树进行miRNA-疾病关联预测。然而上述方法仅利用了miRNA 功能相似性和疾病语义相似性,未利用miRNA 序列相似性、疾病功能相似性和汉明相似性,忽略了miRNA 和疾病不同相似性信息间的潜在关联,影响了miRNA-疾病关联预测性能。
早期miRNA-疾病关联预测依赖于传统生物实验,其过程复杂、昂贵且耗时,因此基于计算的方法被提出,用于miRNA-疾病关联预测。目前基于计算的方法可分为基于矩阵分解的方法和基于机器学习的方法。基于矩阵分解的方法通过将miRNA 和疾病相似矩阵映射到底层子空间来获得线性特征,从而挖掘miRNA 和疾病间深层的关联信息。Gao 等[12]使用图正则化L2,1非负矩阵分解方法来推断潜在的 miRNA-疾病关联。Chen 等[13]基于已知关联以及miRNA 和疾病的相似性矩阵,提出归纳矩阵补全模型预测缺失的miRNA-疾病关联。基于机器学习的方法可以利用非线性函数自动学习miRNA 和疾病相似性信息中的非线性特征,实现miRNA-疾病关联的高效预测。Peng 等[14]基于异构网络提出MDACNN 方法,使用自动编码器提取miRNA 和疾病的非线性特征并采用CNN 预测miRNA-疾病关联。Li 等[15]使用图卷积网络自动学习miRNA 和疾病的非线性特征进行miRNA-疾病关联预测。然而,基于矩阵分解的方法仅采用miRNA 和疾病的线性特征,基于机器学习的方法仅关注了miRNA 和疾病的非线性特征,两者忽略了miRNA 和疾病的线性特征与非线性特征间的信息互补性,降低了miRNA-疾病关联预测性能。
近年来,相关研究提出将矩阵分解方法与机器学习相结合,融合线性特征和非线性特征,并已成功应用于生物信息领域。Zeng 等[16]使用SVD 和深度网络融合lncRNA 和疾病的线性和非线性特征,在关联矩阵的基础上预测 lncRNA-疾病关联,取得了较好的结果。Xie 等[17]基于多源相似性,使用SVD 以及深度矩阵分解融合cirRNA 和疾病的线性特征和非线性特征,提高了cirRNA-疾病关联预测精度。因此将矩阵分解方法与机器学习相结合,可以实现线性特征和非线性特征互补,有助于潜在miRNA-疾病关联预测。
基于上述问题,本文提出了一种miRNA-疾病关联预测模型GCNMSF。GCNMSF 基于miRNA 功能相似性和疾病语义相似性,引入了miRNA 序列相似性、疾病功能相似性和汉明相似性,采用相似性核融合方法(SKF)分别融合miRNA 功能和疾病语义等多源相似性信息,捕获了miRNA 和疾病不同相似性信息间的潜在关联,构建了更丰富的初始特征空间;然后采用两路图卷积(GCN)分别学习miRNA 和疾病的非线性拓扑特征,嵌入卷积注意块(CBAM)从通道和空间两个层面自适应优化非线性特征空间,并引入非负矩阵分解方法(NMF)学习miRNA 和疾病的线性特征;最后,融合miRNA、疾病的线性特征和非线性特征重构miRNA-疾病关联矩阵,实现miRNA-疾病关联预测。
1 数据集及预处理
1.1 miRNA-疾病关联数据
本文从miRNA 疾病数据库HMDDv2.0[18-19]中下载了5 430 个miRNA-疾病关联对,包含495 种miRNA和383 种疾病。然后将495 种miRNA 和383 种疾病构成二维miRNA-疾病关联矩阵Y∈{0,1}m×n。如果miRNAmi与疾病dj之间存在关联,那么Y(i,j)=1 ,否则Y(i,j)=0 。
1.2 miRNA 和疾病多源相似性数据
本文采用miRNA 功能相似性、miRNA 序列相似性、疾病语义相似性、疾病功能相似性以及汉明相似性作为多源相似性数据,miRNA 功能相似性、miRNA 序列相似性、疾病语义相似性、疾病功能相似性均从公开数据库获得,miRNA、疾病汉明相似性基于miRNA-疾病关联相似性计算其汉明距离获得。miRNA 和疾病多源相似性数据如表1 所示。
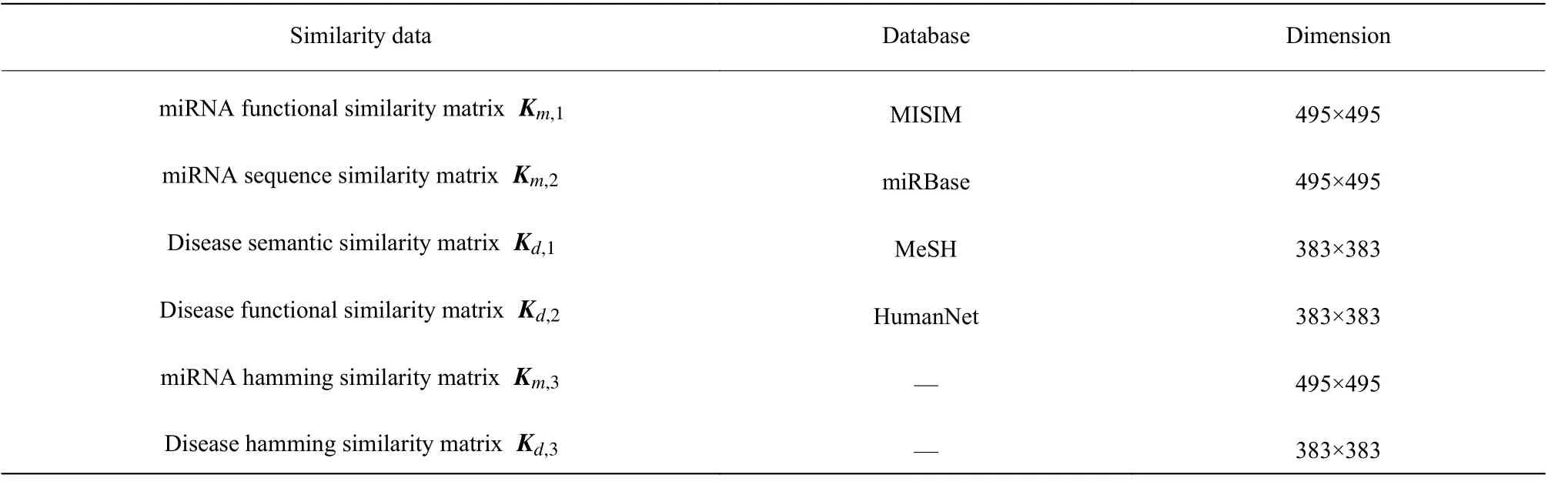
表1 miRNA 和疾病多源相似性数据Table 1 Multi-source similarity data of miRNA and disease
2 miRNA-疾病关联预测模型
基于相似性较高的miRNA 趋向于与相似的疾病相关联这一假设[20],本文提出了miRNA-疾病关联预测模型GCNMSF,模型架构如图1 所示。
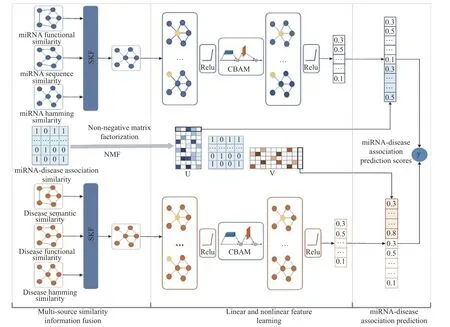
图1 GCNMSF 模型架构Fig.1 Model framework of the GCNMSF
2.1 多源相似性信息融合
融合miRNA 和疾病的多源相似性,可以实现不同miRNA 和疾病相似性间的信息互补,丰富初始特征空间。本文采用SKF 方法[20]融合miRNA 功能相似性、序列相似性、汉明相似性为miRNA 融合相似性信息,融合疾病语义相似性、功能相似性、汉明相似性为疾病融合相似性信息,从而捕获miRNA 和疾病不同相似性信息间的潜在关联,提高miRNA-疾病关联预测精度。
在SKF 过程中,Km,l和Kd,l分别表示miRNA 相似性矩阵和疾病相似性矩阵,其中l=1,2,3 。对于miRNA 每个原始相似性矩阵Km,l,SKF 首先进行归一化处理,得到归一化矩阵Pm,l。然后,基于Km,l中每个节点mj的邻接节点集Ni构建稀疏矩阵Sm,l,miRNA 稀疏矩阵构建过程如式(1)所示。
基于归一化矩阵和稀疏矩阵,每个miRNA 原始相似性矩阵Km,l经过t次迭代获得优化后的相似性矩阵,l=1,2,3 ,然后融合不同miRNA 相似性矩阵的得到miRNA 多源融合相似性矩阵Km,具体过程如式(2)所示。
式中:α1、α2均为SKF 方法中的偏置量。
此外,为了消除多源相似性矩阵融合过程中存在的噪声,SKF 在miRNA 多源融合相似性矩阵Km中添加了权重矩阵Wm,其构建如式(3)所示。
2.2 NMF 学习线性特征
矩阵分解能够通过隐语义学习数据的深层特征关联,被广泛应用于生物数据分析和计算机领域[21-22]。由于NMF 的非负性和高效性,本文采取NMF 方法提取miRNA 和疾病的线性特征。以miRNA-疾病关联矩阵Y∈Rm×n作为输入,使用NMF将其分解为两个非负低秩矩阵Um∈ Rm×r和Vd∈Rn×r,使满足Y≈,其中r≤min (m,n) 。因此miRNA的线性特征矩阵Um和疾病的线性特征矩阵Vd可通过NMF 分解得到,目标函数如式(4)所示,其中,||*||F表示矩阵 * 的 Frobenius 范数。
2.3 GCN 学习非线性拓扑特征
2.3.1 GCN GCN 利用卷积核学习miRNA 和疾病的拓扑结构信息,可以补充miRNA 和疾病的非线性特征中的拓扑结构信息[23-24]。本文将经过相似性核融合得到的miRNA 多源融合相似性矩阵和疾病多源融合相似性矩阵输入到GCN 中学习非线性拓扑特征。
GCN 使用图的拉普拉斯矩阵的特征值和特征向量来提取数据的拓扑信息。经过拉普拉斯矩阵转换和特征分解,第t-1 步的miRNA 多源融合相似性矩阵可以转化为,如公式(5)所示。其中,Λm=diag(λ1,λ2,λ3,···,λm)表示特征值矩阵。
相似地,疾病的非线性拓扑特征可表示为:
2.3.2 卷积注意块优化特征空间 不同节点具有不同信息,而GCN 在特征提取过程中不同节点间共享权重,难以提取重要节点特征。因此本文在GCN 中添加CBAM[26],CBAM 包含通道注意力模块和空间注意力模块,可以通过通道和空间两个层面研究不同特征的重要性差异,赋予拓扑相似的邻域更大的权值,以此实现特征的自适应优化,提升网络节点的表示能力。
输入特征Fx首先通过通道注意力模块生成一维通道注意力图Mc。通道注意力的具体架构如图2 所示。
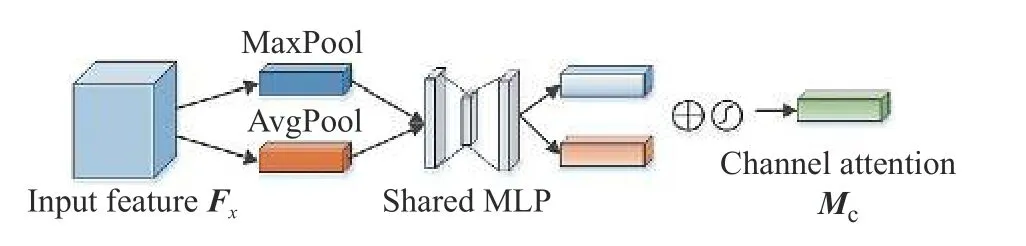
图2 CBAM 通道注意模块Fig.2 Channel attention module in the CBAM
在通道注意力模块中,Fx首先通过平均池化和最大池化分别生成和,然后将和输入到共享网络MLP 中,生成通道注意图Mc∈RC×1×1,具体计算过程见公式(9):
通道注意图Mc与原始特征矩阵Fx点乘 ⊗ 得到,然后通过空间注意力模块生成二维空间注意力图Ms。空间注意力模块具体架构如图3 所示。
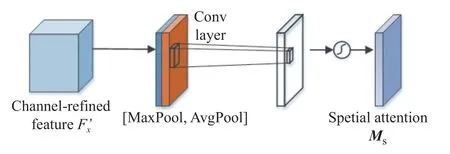
图3 CBAM 空间注意模块Fig.3 Spatial attention module in the CBAM
最后,将空间注意力图Ms与进行点乘操作,得到经过卷积注意块优化的特征。
2.4 miRNA-疾病关联预测
在miRNA-疾病关联预测阶段,本文首先将miRNA 和疾病的线性特征和非线性特征进行拼接,得到miRNA 特征和疾病特征Dnew=然后,采用矩阵补全的方法补全miRNA-疾病关联矩阵,得到重构miRNA-疾病关联矩阵Z,如式(11)所示。
其中:Wm∈Rfm×r和Wd∈Rfd×r分别为将miRNA 和disease 特征映射到原始特征空间的低秩特征映射矩阵。
在误差回溯过程中本文采用均方误差损失函数。为了防止模型训练过程中GCN 的过平滑问题,本文在损失函数中添加偏置项 α 调整阳性样本与阴性样本的损失比,以更好地训练模型。偏置损失公式如式(12)所示。
其中:Φ(·) 表示均方误差损失;Y+和Y-分别表示miRNA-疾病关联矩阵中的已知关联集和未知关联集。
在模型训练优化过程中,对于非线性拓扑特征学习,本文设置GCN 层数为2,各层节点数为[256,256],并将卷积注意块CBAM 嵌入到两层神经网络之间;对于线性特征学习,本文设置NMF 迭代次数为1,输出维度256,以保证线性和非线性特征的均衡;对于特征优化,本文选择Adam 优化器对均方误差损失函数进行优化,以获取GCN 的最优特征,并设置学习率为0.000 1。
3 实验结果与分析
3.1 模型评估指标
在miRNA-疾病关联预测和参数选择过程中,本文基于miRNA-疾病关联数据集,将其中5 430 个已知miRNA-疾病关联作为阳性数据集,184 155 个未知miRNA-疾病关联作为阴性数据集,采用五折交叉验证评估本文模型GCNMSF 的性能。在每折交叉验证中,将5 430 个阳性样本划分为5 个子集,选取其中1 个子集作为测试集,其余作为训练集;在184 155个阴性样本中,选取与阳性训练样本相同数量的阴性样本作为测试集,其余阴性样本作为训练集。
在模型预测性能的评估过程中,本文采用AUC、AUPR 和F1_score 统计度量指标。AUPR 表示PR 曲线下面积,横坐标为召回率Recall,纵坐标为精确率Precision。AUC 表示ROC 曲线下面积,横坐标是伪阳性率(FPR),纵坐标是真阳性率(TPR)。TPR、FPR、Recall、Precision 和F1_score 计算公式如下所示。
其中:TP 为正样本被正确识别的数量;FP 为负样本被预测为正样本的数量;TN 为负样本被正确识别的数量,TP 为正样本被预测为负样本的数量。
3.2 模型参数选择
在本文模型GCNMSF 性能评估之前,需要对损失函数中的偏置项 α 以及GCN 层数进行确定。本文基于miRNA-疾病关联数据集,分别对偏置项 α 以及GCN 层数进行五折交叉验证。偏置项 α 是损失函数中阳性样本和阴性样本的损失比,其取值区间为(0,1)。取步长0.1 对不同 α 值进行验证,结果如图4(a)所示,α=0 表示损失函数未添加偏置 α 。当偏置项 α 为0.4 时,AUC 值为0.945 2,达到最优,表明适当调整阳性样本和阴性样本的损失比有利于模型的预测。然后,保持偏置项 α=0.4 不变,分别取GCN 层数为1、2、3,依次对GCN 的不同层数进行验证,结果如图4(b)所示。由图可见,当GCN 层数为2 时,模型性能最优,表明图卷积层数的加深可能会导致过平滑,降低模型预测能力。因此,本文选择偏置项 α 为0.4,GCN 层数为2。
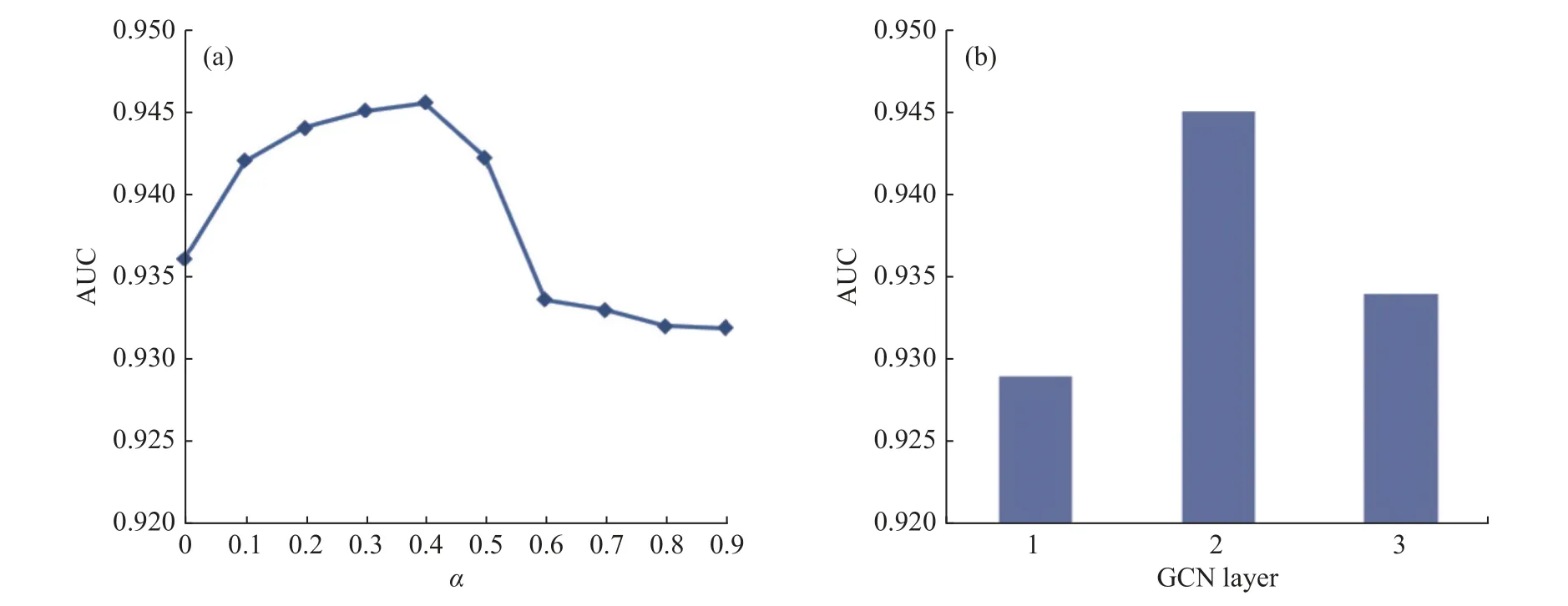
图4 GCNMSF 模型的参数选择实验结果Fig.4 Experimental results of the GCNMSF model with different parameters
3.3 消融实验
本文模型GCNMSF 引入了多源相似性信息,并融合了miRNA 和疾病的线性特征和非线性特征预测miRNA-疾病关联。为了验证多源相似性的有效性以及线性特征和非线性特征的必要性,引入了消融实验。
在miRNA 和疾病的多源相似性消融实验中,以本文模型GCNMSF 作为基线模型,对不同miRNA 和疾病相似性组合进行五折交叉验证,结果如表2 所示。表2 中第一列表示不同相似性组合,其中MFS表示miRNA 功能相似性,MSS 表示miRNA 序列相似性,DSS 表示疾病语义相似性,DFS 表示疾病功能相似性,HMS 表示汉明相似性。
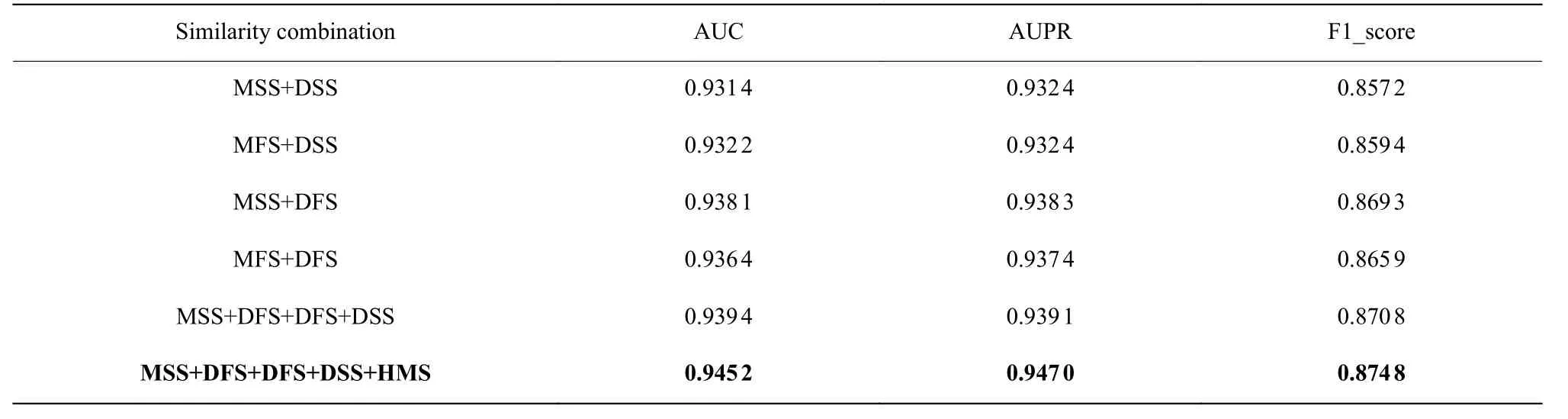
表2 不同相似性组合消融实验数据表Table 2 Ablation experiments with different similarity combinations
从表2 可以看出,采用miRNA 和疾病的单源相似性时的各项指标均低于采用多源相似性时的指标,这是因为高相似性的 miRNA 往往与相似的疾病相关,而多源相似性融合可以学习多种不同相似性信息间的潜在关联,而且汉明相似性融入了拓扑结构信息,从而丰富了初始特征空间,提高了miRNA-疾病关联预测精度。在线性特征和非线性特征消融性实验中,分别对线性特征学习模块GCN、非线性特征学习模块NMF 以及本文模型GCNMSF 进行五折交叉验证。结果如图5 所示。
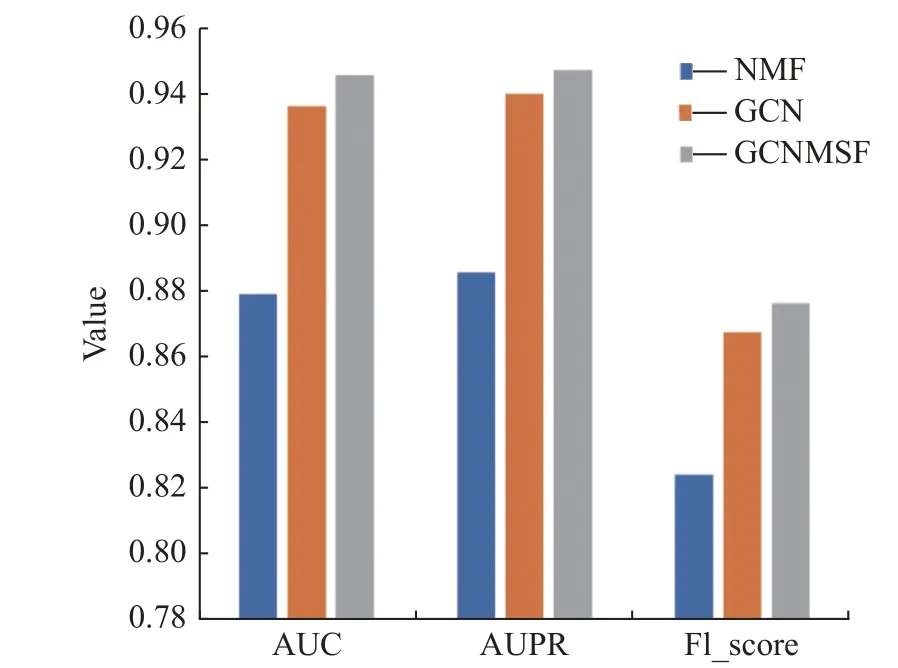
图5 线性和非线性特征学习模块消融实验结果Fig.5 Results of linear and nonlinear feature learning module ablation experiments
由图5 可得,本文模型GCNMSF 融合miRNA和疾病的线性特征和非线性特征,性能优于仅使用线性特征的NMF 模块和仅使用非线性特征的GCN 模块,这表明线性特征和非线性特征的融合可以实现特征信息互补,为miRNA-疾病关联预测提供更丰富的特征表示。
3.4 模型性能评估
为了评估本文模型GCNMSF 的有效性,本文选取了 ABMDA[10]、EDTMDA[11]、IMCMDA[13]、NIMCGCN[15]、DMA[27]、TCRWMDA[28]作为对比方法,并在miRNA-疾病数据集上进行五折交叉验证,,结果如图6 所示。其中,IMCMDA、TCRWMDA 基于矩阵分解和随机游走,ABMDA、EDTMDA 基于传统机器学习,DMA、NIMCGCN 基于深度学习。
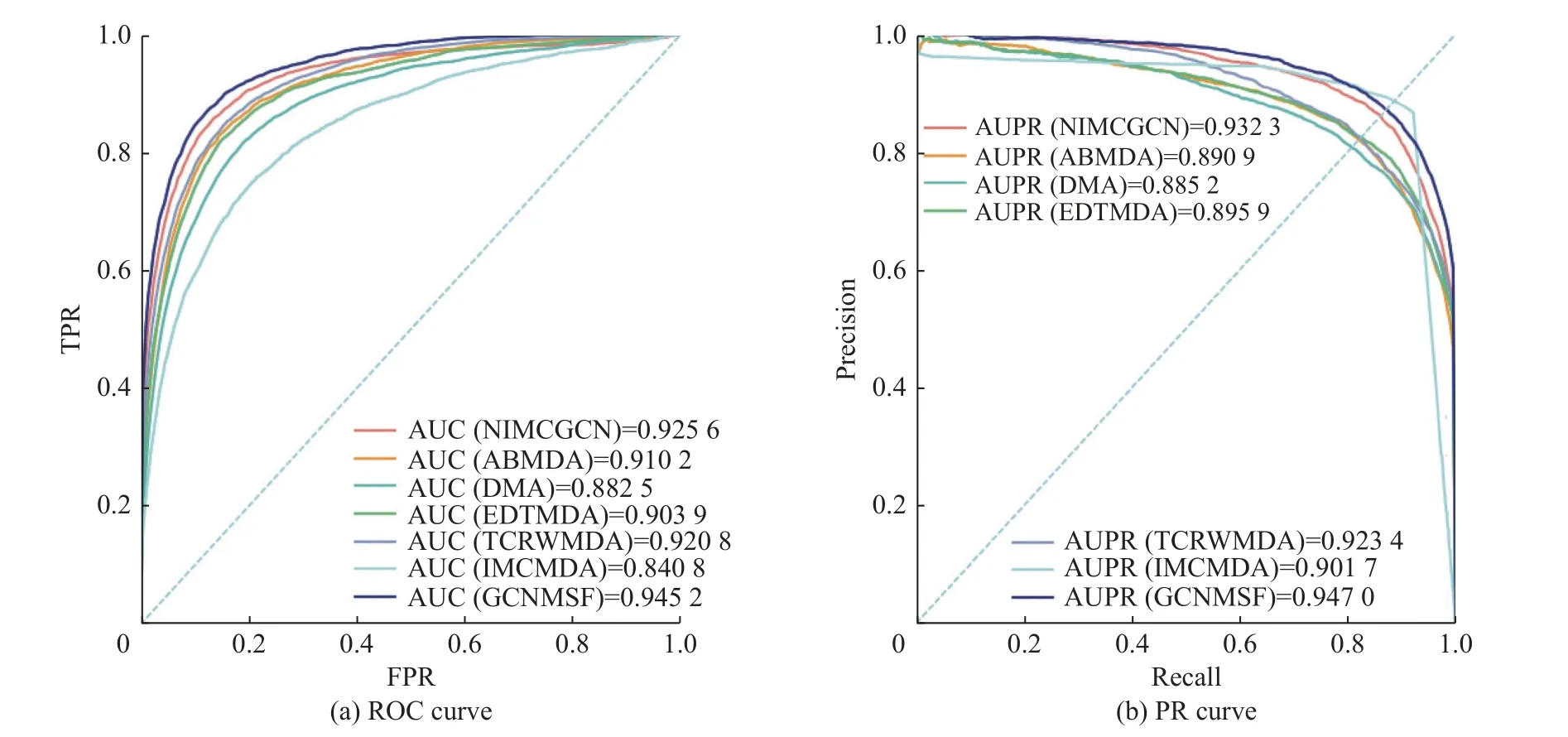
图6 不同方法五折交叉实验结果Fig.6 Five-fold-cross experiments of different methods
由图6 可得,本文模型GCNMSF 在五折交叉实验中的AUC 和AUPR 值为0.945 2 和0.947 0,均优于对比方法。DMA、ABMDA、EDTMDA、TCRWMDA、IMCMDA、NIMCGCN 均采用miRNA 功能相似性和疾病语义相似性,未融合miRNA 序列和疾病功能等多源相似性信息,这表明融合多源相似性信息可以为miRNA-疾病关联预测提供更丰富的初始特征信息,有利于miRNA-疾病关联预测。而且上述对比方法仅采用miRNA 和疾病的线性特征或非线性特征进行预测,未融合线性特征和非线性特征,这表明融合线性特征和非线性特征可以实现信息互补,丰富miRNA 和疾病的特征表示,提高miRNA-疾病预测性能。综上可知,在miRNA-疾病关联预测中,本文模型通过融合多源相似性信息以及线性特征和非线性特征取得了更高的结果,能够更有效地预测miRNA-疾病关联。
为了进一步验证本文模型GCNMSF 对特定疾病的识别和预测能力,本文以乳腺癌、肝癌、结肠癌3 种常见疾病为例,选取ABMDA[10]、EDTMDA[11]、IMCMDA[13]、NIMCGCN[15]、DMA[27]、TCRWMDA[28]作为对比方法进行验证,结果如图7 所示。由图7可知,本文模型GCNMSF 在3 种疾病上获得的AUC 值均高于其他模型,进一步表明本文模型在特定疾病的小样本集上,能够依据相似性数据的特征信息实现特定疾病相关miRNA 的预测,具有较好的稳健性。
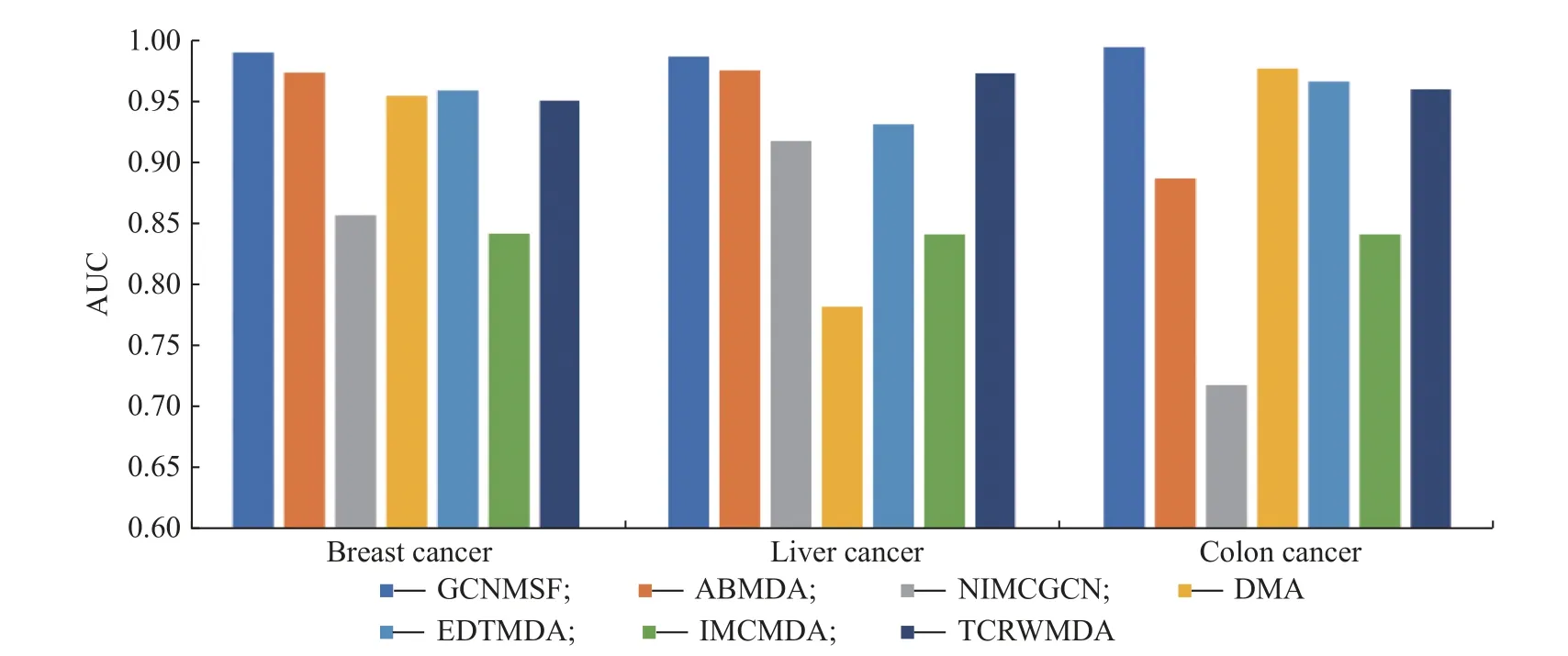
图7 不同方法预测特定疾病的实验结果Fig.7 Experimental results of different methods on specific diseases prediction
3.5 案例研究
为了进一步验证本文模型GCNMSF 在真实案例中的准确性,本文模型选取两种常见疾病肺癌和乳腺癌进行案例研究,并使用dbDEMCv3.0[29]和miR2Disease[30]数据库进行验证。
为了验证本文模型GCNMSF 在特定疾病未知miRNA-疾病关联预测中的有效性,将GCNMSF 用于预测与肺癌相关的潜在miRNA,并将排名前50 的miRNAs 在dbDEMCv3.0 和miR2Disease 数据库进行验证。如果dbDEMCv3.0 和miR2Disease 数据库中存在miRNA-肺癌关联,则表示预测成功,具体结果如表3 所示。表3 所示证明了肺癌前50 个相关miRNA中有46 个相关miRNA 在dbDEMCv3.0 和miR2Disease数据库中得到了验证,hsa-mir-378a、hsa-mir-296、hsa-mir-151a 和 hsa-mir-520c 未在 dbDEMCv3.0 和miR2Disease 数据库中得到相关文献支持。肺癌的验证结果说明了本文模型GCNMSF 可以有效地预测特定疾病潜在miRNA-疾病关联。
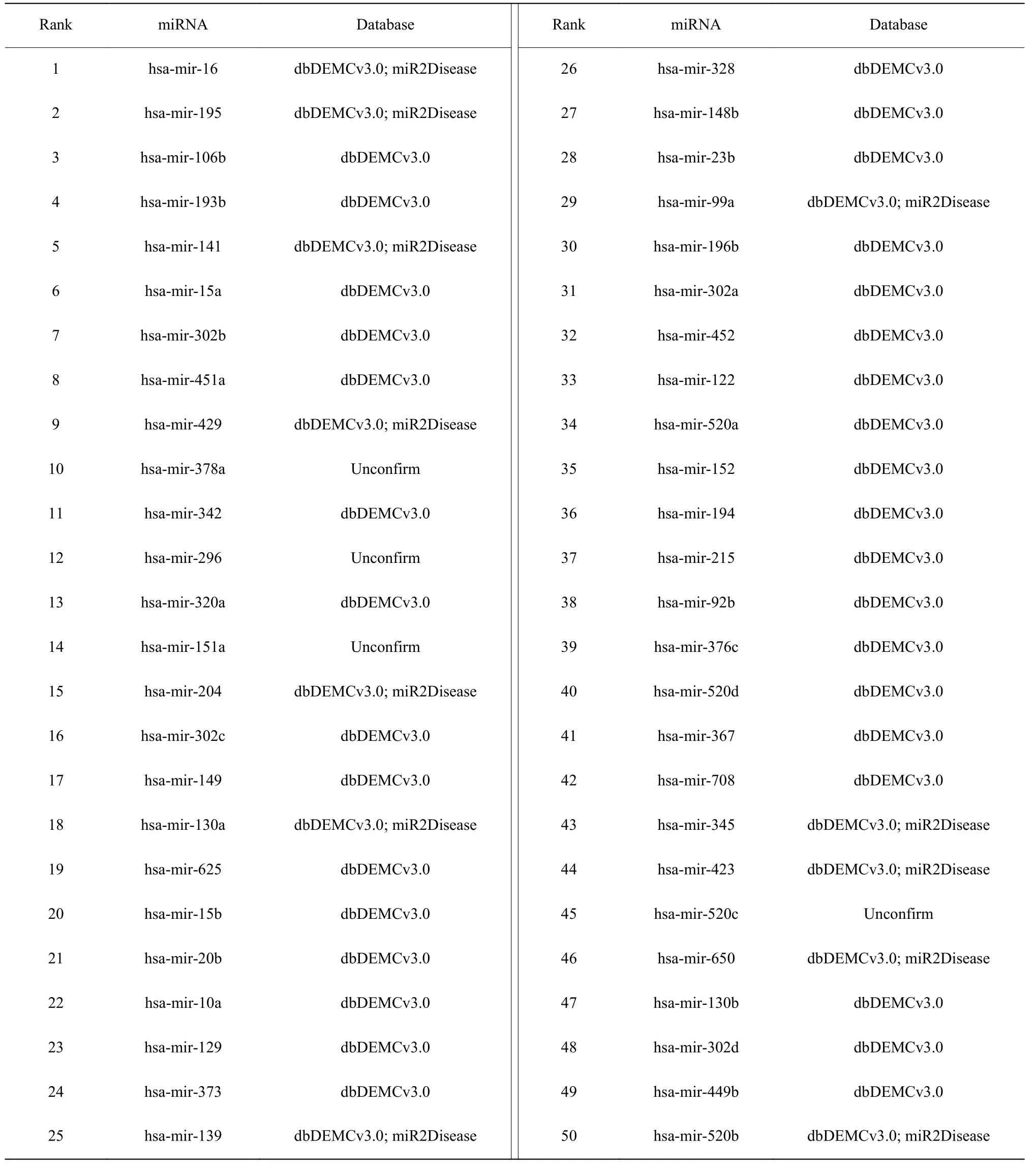
表3 肺癌相关miRNA 预测实验数据表Table 3 Predicted data of miRNAs associated with lung cancer
为进一步验证本文模型GCNMSF 预测未知疾病miRNA-疾病关联的性能,本文首先去除miRNA-疾病关联矩阵中已知miRNA-乳腺癌关联,重新训练模型GCNMSF 并预测乳腺癌相关的miRNA。然后将排名前50 的miRNA 在dbDEMCv3.0 和miR2Disease数据库进行验证,前50 个相关miRNA 的预测和验证结果见表4。表4 表明乳腺癌前50 个相关miRNA均在dbDEMCv3.0 和miR2Disease 数据库中得到了验证,预测精度为100%,表明本文模型GCNMSF 可在没有任何已知miRNA 关联的情况下,预测未知疾病与miRNA 的潜在关联。
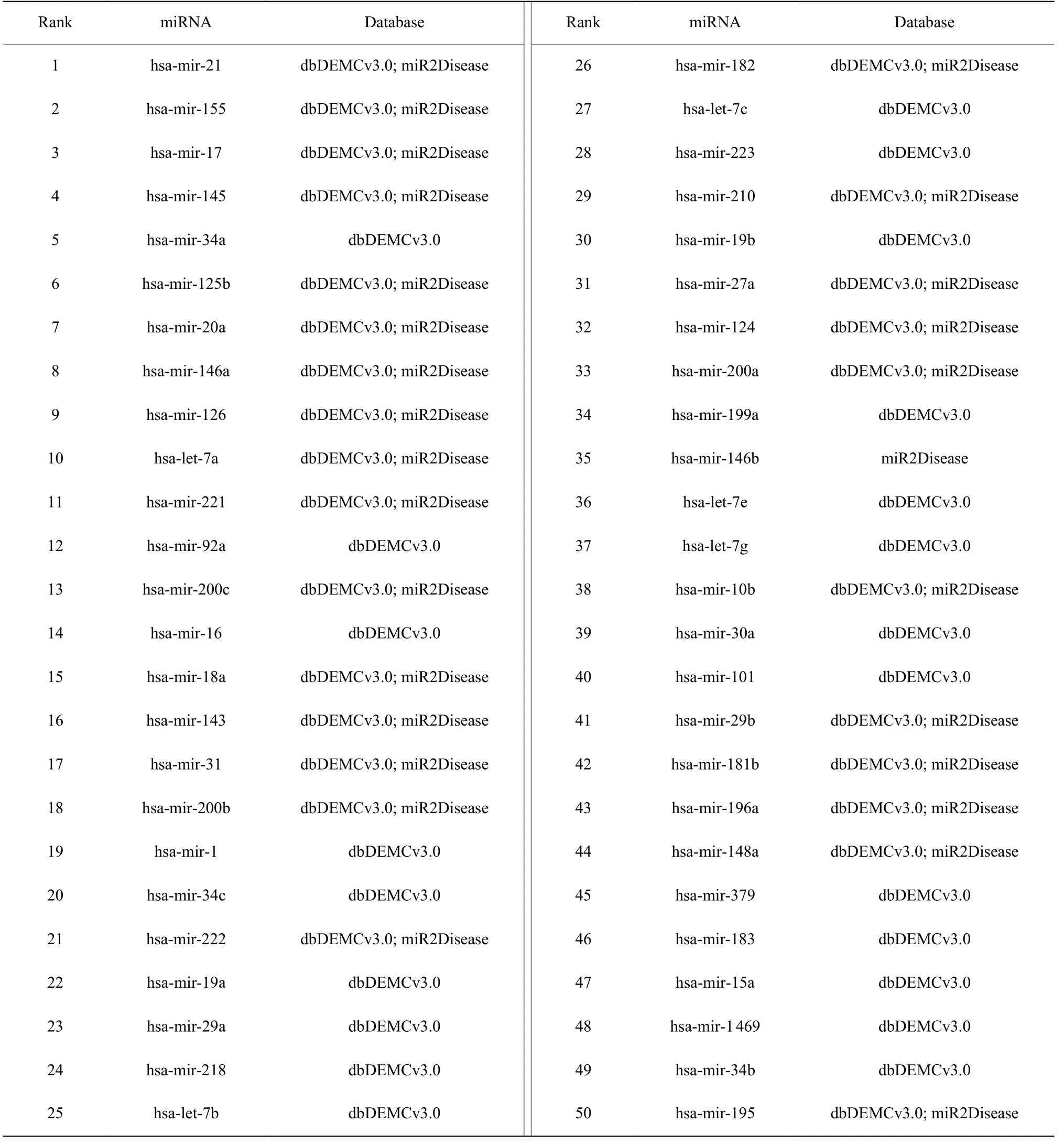
表4 乳腺癌相关miRNA 预测实验数据表Table 4 Predicted miRNAs associated with breast cancer
4 结 论
本文针对现有miRNA-疾病关联研究考虑信息不全面以及特征提取方式单一的问题,提出了一种miRNA-疾病关联预测模型GCNMSF。该模型引入miRNA 序列相似性、疾病功能相似性和汉明相似性,采用SKF 融合miRNA 和疾病多源相似性数据,丰富初始特征空间;采用嵌入CBAM 的两路图卷积分别学习miRNA 和疾病的非线性特征,并引入NMF方法学习miRNA 和疾病的线性特征,融合miRNA和疾病的线性特征和非线性特征,实现信息互补,最后利用融合的线性特征和非线性特征预测miRNA-疾病关联。消融实验结果表明,多源相似性信息有助于miRNA、疾病信息互补,丰富特征信息,而且相较单一线性特征或非线性特征,二者的结合更有利于miRNA-疾病关联的预测。肺癌和乳腺癌的案例研究进一步证实了本文模型的实际有效性。尽管本文模型GCNMSF 在miRNA-疾病预测中表现出较好的性能,但仍需要进一步改进。由于图卷积特征提取时存在深度拓展局限性,因此本文将继续探索其他深度学习技术在miRNA-疾病中的可应用性。

