蛋白质芯片检测技术结合多维统计方法推断死亡时间
张旭东,姜垚如,梁芯瑞,田甜,靳茜茜,张小红,曹洁,杜秋香,孙俊红
1.山西医科大学法医学院,山西 晋中 030600;2.禹州市公安局,河南 禹州 452570;3.四川大学华西基础医学与法医学院,四川 成都 610041;4.邹平市公安局,山东 邹平 256200
准确推断死亡时间(postmortem interval,PMI)是法医学实践中的一个关键问题,死后相关生物技术分析可为其提供重要、可靠的数据。法医昆虫学[1]或形态学[2]分析可为死亡时间推断提供基础依据,但因易受外界复杂环境的影响,存在一定的局限性和不稳定性。
随着生物技术的发展,研究者们愈发关注生物大分子如RNA[3-4]、DNA[5-6]或蛋白质[7-8]的死后变化。近年来,机体死后骨骼肌蛋白质降解的相关研究取得了重大进展。在标准化动物模型中,可通过十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfonate-polyacrylamide gel electrophoresis,SDS-PAGE)和Western 印迹分析,发现如蛋白磷酸酶2A、结蛋白、肌钙蛋白[7-8]等特定蛋白质的降解模式。此外,死后变化如天然蛋白条带的丢失或特定降解产物的出现,与死亡时间显著相关[9]。
利用蛋白质芯片毛细管电泳进行蛋白质分离,具有快速、准确、灵敏等优势[10]。2100 生物分析仪(美国Agilent 公司)集成多个实验程序,包括样品处理、分离染色、脱色、检测和分析,在其基础上结合蛋白质芯片,可分析多种蛋白质样品。目前,蛋白质表达谱的应用主要集中在临床疾病检验、药物研制及物种鉴定等领域[11]。
本研究收集死后不同时间点大鼠骨骼肌及人体骨骼肌样本,采用蛋白质芯片检测技术获取死后蛋白质表达谱,通过多维统计方法,探索蛋白质含量变化与死亡时间的关系,建立死亡时间预测模型,为死亡时间推断提供新的思路和技术支持。
1 材料与方法
1.1 样本制备
健康成年雄性SD 大鼠8 只,10~12 周龄,体质量200~230 g,由山西医科大学实验动物中心提供。温室饲养2 d,腹腔注射20%乌拉坦(1 g/kg,北京索莱宝科技有限公司)麻醉后颈椎脱臼处死,死后放入16 ℃人工气候箱。于死后0 d、1 d、2 d、3 d、4 d、5 d、6 d、7 d、8 d 和9 d 分别取每只大鼠右后肢腓肠肌(200±2)mg,重复收集大鼠的肌肉组织样本,每次收集后保持皮肤覆盖,共收集80份样本。样本于液氮速冻后放入-80 ℃冰箱待检。
收集2021 年山西医科大学司法鉴定中心9 例不同死亡时间人体骨骼肌样本,按序编为1~9 号,其中3 例经冷藏(计算死亡时间时除去尸体冷藏时间),样本信息详见表1。样本均为无损伤、无出血的胸大肌,每份肌肉组织为(200±2)mg,于液氮速冻后放入-80 ℃冰箱待检。

表1 9 例人体样本的详细信息Tab.1 Details of 9 human cadaver samples
本研究已获得山西医科大学科学研究伦理审查委员会批准(审批文号:2020LL149)。
1.2 蛋白质提取
将大鼠和人体的骨骼肌在液氮中研磨至粉末状后置入微量离心管,同时将蛋白酶抑制剂苯甲基磺酰氟(phenylmethylsulfonyl fluoride,PMSF;武汉博士德生物工程有限公司)与纯水按1∶3.5的质量浓度比(kg/L)加入离心管,冰上孵育1 h,以12 000×g离心20 min,吸取上清液400 μL 分装备用。
从每只大鼠10 个时间点的蛋白上清液中各取50 μL,混匀制成400 μL 的质量控制(quality control,QC)样本,共8 份。
1.3 蛋白质芯片样本制备及检测
根据蛋白质230 试剂盒(美国Agilent 公司)说明书在4 μL 样本中加入2 μL 变性剂,将样本溶液和Ladder 置入100 ℃水浴中加热5 min,再加入84 μL 去离子水进一步稀释,从稀释液中取6 μL 加载到蛋白质芯片相应的孔道中。
每个芯片加载10 个样本,然后将芯片置于2100生物分析仪中进行分析,20~30 min 后,获取每个样本相对分子质量为14 000~230 000 的水溶性蛋白质表达谱数据。
1.4 数据预处理
采用2100 Expert 软件(美国Agilent 公司)获取大鼠和人体骨骼肌蛋白质凝胶电泳图和电泳色谱图。根据内标“lower marker”和“upper marker”对电泳色谱进行定标识别,并对峰位进行校正调整。为消除杂质干扰,去除荧光强度在10 FU 以下的峰[12]。将迁移时间(相对分子质量大小)基本一致的峰记为相同编号,对峰含量进行归一化处理。
1.5 统计分析
使用SPSS 22.0 软件(美国IBM 公司)对8 份QC样本进行单因素方差分析,检验组间是否存在差异;再行多重比较最小显著性差异(least significant difference,LSD)检验,分析QC 样本是否存在差异。采用SIMCA 14.1 软件(瑞典Umetrics 公司)对死后各时间点大鼠骨骼肌样本的峰高数据进行主成分分析(principal component analysis,PCA)降维,结合正交偏最小二乘(orthogonal partial least squares,OPLS)判别分析死后各时间点之间的差异,其中R2、Q2表示模型的解释能力和预测能力,R2、Q2终点越接近,同时Q2回归直线与Y轴有负截距,说明模型有效,无过度拟合现象。R2X、R2Y分别为模型对自变量X、因变量Y的解释能力。当Q2>0.5 且与R2Y差值不超过±0.3 时,认为模型有效、数据可靠[13],用基于交互验证的方差分析(cross validation-analysis of variance,CV-ANOVA)检验死后不同时间点样本间的差异。采用SPSS Clementine 12.0 软件(美国IBM 公司)分别建立Fisher 判别模型和反向传播(back propagation,BP)神经网络模型,对死后不同时间点大鼠骨骼肌样本进行分类预测,随机选取70%的数据作为训练集、30%的数据作为测试集,分别获取两个模型的内部交叉验证准确率及外部验证准确率,同时以混淆矩阵总结BP模型的预测能力。使用Morpheus分析软件(https://clue.io/morpheus/)对人体骨骼肌蛋白质中各峰的含量进行热图及聚类分析。检验水准α=0.05。
2 结果
2.1 大鼠骨骼肌蛋白质表达谱峰分析及模型建立
2.1.1 大鼠骨骼肌蛋白质谱峰特征及差异性
QC 样本的单因素方差分析及多重比较结果表明,组间差异无统计学意义(P>0.05)。
死后不同时间点蛋白质的谱峰形状相近,多数峰位置一致但峰高不同(图1A~B);凝胶电泳图中各条带随死亡时间的延长有所变化(图1C),在保留时间24 s 时,死后0 d 与9 d 蛋白条带存在明显差异。在死后0~9 d 的所有蛋白质样本中共识别出25 个峰(按序编号为1~25),各峰含量(蛋白质相对分子质量)在死后不同时间点具有差异性,峰10 在死后0~3 d 含量较高,而在死后4~9 d 含量逐渐减少,同时某些峰(如峰14)在死后0~9 d 的含量始终维持较高的水平(图1D)。
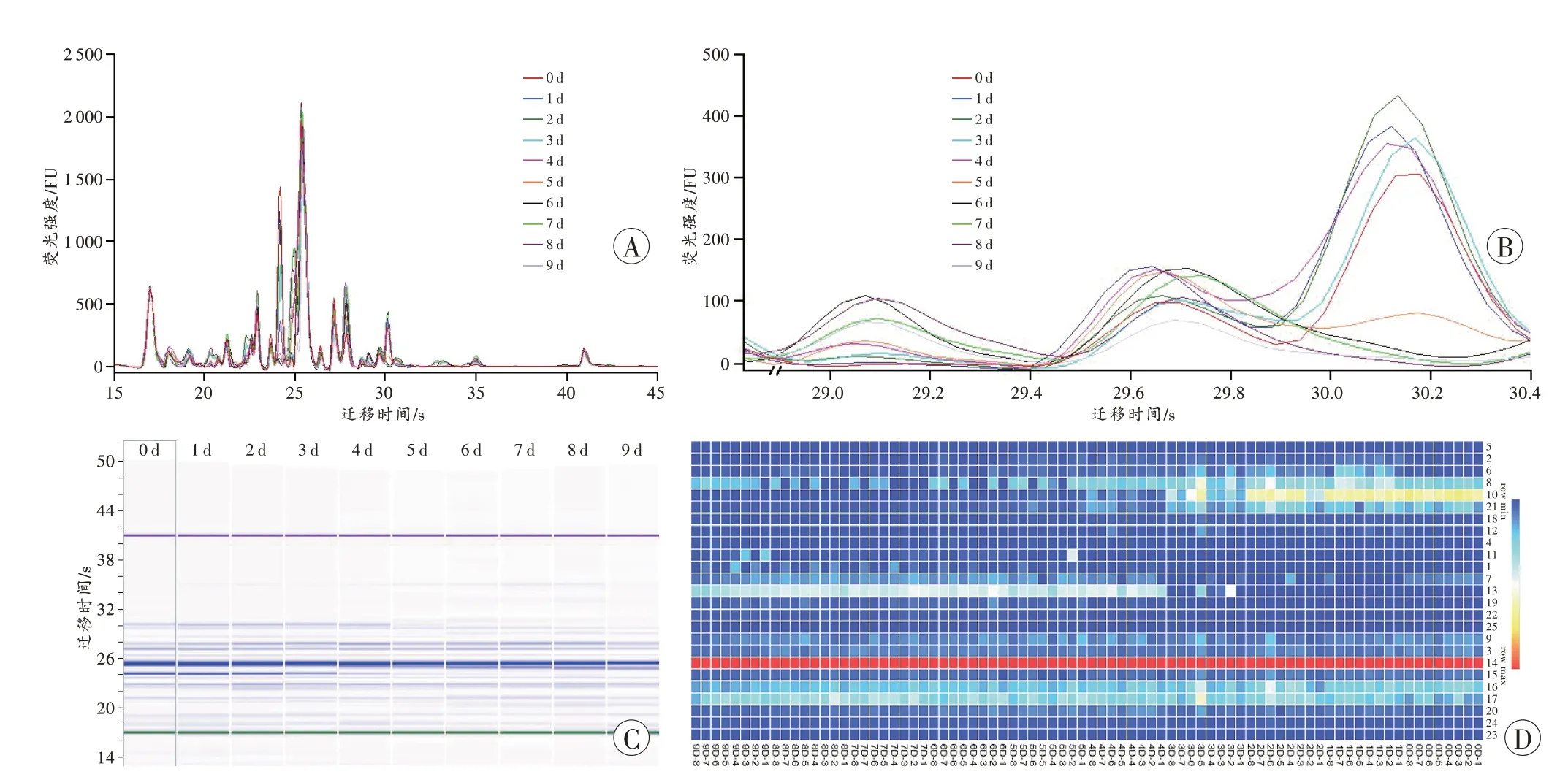
图1 死后不同时间点大鼠骨骼肌蛋白质的表达Fig.1 Protein expression of rat skeletal muscles at different time points after death
PCA 模型指标良好(R2=0.866,Q2=0.654),可解释大部分的数据变异(图2)。死后不同时间点大鼠骨骼肌蛋白质谱峰随死亡时间的延长呈一定的聚集趋势,但模型为无监督学习,部分死亡时间段内差异不能完全显现,需进一步分析。

图2 死后不同时间点大鼠骨骼肌蛋白质谱峰的PCA 模型Fig.2 PCA model of rat skeletal muscle protein peaks at different time points after death
组间OPLS 判别分析发现,死后0 d、1 d、2 d、3 d、4 d、5 d 和9 d 两两时间点间均可分离,模型R2、Q2值均大于0.5,且CV-ANOVA 结果显示各组间差异均有统计学意义(P<0.05);6 d、7 d、8 d 3 个时间点间较难分离且CV-ANOVA 中P>0.05。
2.1.2 死亡时间预测模型
对80 份大鼠骨骼肌蛋白质样本进行Fisher 判别分析,典型判别函数图显示死后10 个时间点分离效果尚可(图3A),但部分样本存在重叠交叉现象,模型内部交叉验证和外部验证的准确率分别为71.4%和66.7%,准确率较低。

图3 大鼠骨骼肌蛋白质样本的死亡时间预测模型Fig.3 Prediction model of postmortem interval in protein samples from rat skeletal muscles
以蛋白质样本为数据节点、死亡时间为输出节点、25 个峰为输入节点的BP 神经网络模型中,内部交叉验证和外部验证的准确率分别为98.2%和95.8%,以混淆矩阵结合散点图判断BP 神经网络模型分类预测结果中,行代表预测时间点,列代表实际时间点,发现在5 d、7 d 各有1 例错判(图3B)。
经两种模型的比较,BP 神经网络模型具有良好的稳定性和对未知样本的预测能力。
2.2 人体骨骼肌蛋白质表达谱峰分析
将人体骨骼肌蛋白质样本中识别出的16 个峰按序标记为a~p,发现各峰形状、位置基本一致(图4A)。案例1(死后4 d)样本的个别峰与其余8 例略有差异,其峰识别个数最少(10 个),案例6(死后22 h)样本的峰识别个数最多(13 个)。

图4 死后人体骨骼肌蛋白质的表达Fig.4 Protein expression of human skeletal muscles after death
进一步探究人体蛋白质谱峰含量在不同死亡时间点的分布状态和规律,对样本数据进行热图分析,发现不同谱峰含量随死亡时间的延长发生变化,如峰a 含量随时间的延长而增加,峰e 含量呈现先减少后增加的趋势,峰i 与峰e 趋势相反。根据峰含量对死亡时间进行聚类,发现死后4 d 的蛋白质样本与死后25 h 内的蛋白质样本明显分离,各峰含量随死亡时间的延长呈一定的时序性变化(图4B)。
3 讨论
死后机体蛋白质随死亡时间延长而降解,其降解过程为基于蛋白质降解的死亡时间推断提供了可靠的证据[9,14]。蛋白质的测定和分析多采用Western 印迹、质谱和免疫组织化学等方法[15-17];肾、肺、肝,尤其是骨骼肌,常作为蛋白质降解的研究对象[16]。考虑到死后机体内环境的相互作用对蛋白质的影响,多指标联合研究可能更全面、精准。LI 等[15,18]利用质谱技术发现死后蛋白质表达与死亡时间具有相关性,为死亡时间推断提供了新思路。然而质谱分析成本昂贵,操作流程复杂且严格,在一定程度上限制了其实际应用。
2100 生物分析仪基于微流控毛细管电泳技术,可对蛋白质进行高通量的自动化分析。蛋白质230试剂盒可用于分离和分析相对分子质量为14 000~230 000 的蛋白质,分辨率为10%[12],与传统的SDSPAGE 相比具有易于处理、可重复性及环保等优点[11]。李文晋等[12]利用蛋白质芯片检测技术高通量、准确、便捷等特性,获取死后大鼠肝组织蛋白质表达谱,推测其与死亡时间的关系。
本研究使用蛋白质芯片检测技术对大鼠骨骼肌蛋白质进行检测,结果显示,不同死亡时间的蛋白质芯片谱峰及峰含量具有差异,其变化趋势和程度不同。PCA 是一种数据降维方法,可利用较少的综合指标来解释原始变量信息[19-20]。本研究中PCA 结果显示,死后不同时间点大鼠骨骼肌蛋白质谱峰分布有一定趋势,但不能完全体现组间差异。OPLS 判别分析是将连续变量数据分为预测信息和不相关信息,使结果更易解释和可视化[21]。本研究对死后不同时间点的蛋白质表达进行OPLS 判别分析发现,除死后6 d、7 d 和8 d 外,其余7 个时间点间差异均有统计学意义(P<0.05),可能与蛋白质降解产物类型、降解速率及个体差异等因素有关。
Fisher 判别模型按分类能力提取特征,具有很强的数据压缩能力[22-23],本研究死亡时间预测模型效果不理想,考虑Fisher 判别对线性不可分样本无法准确分类,因此需选择更适合的数据分析方法。BP 神经网络作为一种应用广泛的多层前馈神经网络,无需具体的数学模型,同时因其良好的稳定性、准确性和快速运算能力,在解决复杂非线性关系方面具有优势,有很高的实际应用价值[24]。本研究中BP 神经网络模型分析仅在死后5 d 和7 d 各有1 例错判。对比Fisher 判别模型和BP 神经网络模型的外部验证准确率,BP 神经网络模型对本研究死亡时间的预测可达到较为理想的效果,更适用于多指标联合推断死亡时间的研究。
本研究对大鼠样本进行PCA 及OPLS 判别分析发现,蛋白质表达谱随死亡时间的延长呈时序性变化,死亡时间分类预测模型具有较高准确率,与李文晋等[12]前期发现的肝组织蛋白质不同死亡时间变化规律相一致。基于动物样本分析结果,本研究收集人体骨骼肌蛋白质样本,发现不同死亡时间的芯片谱峰存在差异,谱峰含量随死亡时间的延长发生变化。根据谱峰含量对死亡时间进行聚类分析,发现样本在死后4 d 和25 h 两个时间点明显区分,各峰含量亦随死亡时间延长呈现一定变化,说明蛋白质芯片检测技术可应用于动物和人体样本的死亡时间研究。此外,人体样本死亡时间多在25 h 内,时间跨度小,且受死亡原因、现场环境条件和死后保存条件等多种因素影响,信息较为复杂。基于多方面因素的影响,本研究未能建立人体死亡时间推断的数学模型。
综上所述,蛋白质芯片检测技术可快速、准确、高重复性地获取死后不同时间点动物和人体骨骼肌组织相对分子质量为14 000~230 000 的水溶性蛋白质表达谱,结合PCA、OPLS 多维统计方法探索死后蛋白质表达与死亡时间之间的变化规律,建立多种死亡时间推断模型,其中BP 神经网络更适于多指标联合死亡时间推断,可为死亡时间推断提供新的思路和方法。本研究在实验动物模型的制备上尽可能模拟实际情况,如设置多个死后时间点、增加多种死亡原因、尸体冷藏等处理方法,后续将扩大尸体样本的收集,完善相关信息,以期建立基于真实案例下的人体死亡时间蛋白质信息库。

