探究染色质调节因子在胃癌进展中的作用及潜在治疗靶点
马梦璇,张燕飞,祁玉娟
0 引言
胃癌是一种侵袭性疾病,其预后较差,并且通常没有利于早期诊断的特定症状。2018 年,全球有超过 100 万人被诊断出患有胃癌,其中783 000人因胃癌死亡[1]。目前已经开发了多种针对胃癌的治疗方法,但对许多进展期胃癌患者疗效不佳。因此,迫切需要探索新的诊断标志物和肿瘤进展的分子机制,达到胃癌早期诊断与早期治疗的目的[2]。染色质可及性作为与特定位点肿瘤功能相关的基因组特征之一,可通过与转录因子结合调节与癌症进展和侵袭相关的多个基因。最近,研究者开始探索染色质状态变化对癌症发展的影响。染色质可及性失调可能会改变下游癌基因或抑癌基因的转录活性,从而影响恶性肿瘤的进展。染色质重塑是基因调控的关键机制[3]。染色质重塑是所有DNA相关核心细胞过程的经典步骤,一些染色质重塑决定了细胞的存亡,包括与癌症发病和发展有关的细胞过程。过去10年中,染色质重塑在细胞转化和癌症发展中的作用越来越受到关注[4],大规模表观基因组学和转录组学研究结果强调了癌症中染色质重塑因子的频繁突变或失调,表明异常的染色质重塑在癌症发展中起着重要作用[5-6]。
本研究通过生物信息学分析,研究胃癌中染色质调节因子(Chromatin regulator,CR)的表达谱和预后价值,构建基于CR基因的预测模型,探索对治疗胃癌有益的小分子药物。
1 材料与方法
1.1 GEO的数据采集 在GEO (Gene Expression Omnibus)数据库中搜索“Gastric cancer”相关数据表达谱,获得2个微阵列数据研究集(GSE26942和GSE79973)。GSE26942源自 GPL6947平台,GSE79973 源自 GPL570 平台。GSE26942 数据集由 218 个样本组成,包括对照组胃黏膜组织12例,胃腺癌203例,胃间质瘤3例。GSE79973 数据集由 20个样本组成,包括对照组胃黏膜组织10例,胃癌组织10例。见表1。

表1 GEO数据库胃癌基因表达谱数据集(例)
1.2 差异表达基因DEG的筛选 采用GEO数据库中的在线分析工具GEO2R筛选差异基因。R软件limma 包用于筛选DEG。筛选标准为|logFC|>0.2,P值(adj.Pvalue)<0.05,并与染色质调节因子相关基因取交集获取染色质调节因子差异表达基因。为了直观反映基因表达情况,采用R软件中的pheatmap、VennDiagram、ggplot2 R包将染色质调节因子差异表达基因绘制热图火山图。
1.3 DEG的功能富集分析 使用 DAVID生物信息数据库对上述染色质调节因子差异表达基因进行 GO 功能注释和 KEGG 通路分析[7-8]。调整后P<0.05用于阈值筛选,使用R软件中的cluster Profiler、pathview R包将GO和KEGG结果可视化。
1.4 PPI网络构建 将DEGs导入String数据库,置信度>0.9[9],选择智人进行物种选择,形成蛋白质互作网络,使用Cytoscape筛选出PPI网络中的hub基因。本研究以Degree值排序前10位的基因作为蛋白质互作 (PPI)网络中的hub基因。
1.5 构建hub基因预测模型及潜在治疗药物筛选 采用“1.4”中筛选出的hub基因构建胃癌的预测模型,使用ROCR包绘制列线图、Calibration 图及ROC 曲线,并通过Enrichr平台 (https://maayanlab.cloud/Enrichr/)用DSigDB 数据库进行 hub基因和共表达基因的药物筛选。根据P值和调整后的P值选择与差异表达RNA(DEmRNA)相关的前 5种候选药物。表2显示了 DSigDB数据库中排名前 5 位的候选药物[10]。

表2 DSigDB数据库中hub基因的潜在药物
2 结果
2.1 差异基因的获取 基于GSE26942和GSE79973基因集,使用svaR包消除了样本间的批量效应后,获得了差异基因与染色质调节因子取集合,共获得染色质调节因子差异基因137个,其中上调基因93个,下调基因44个,并绘制出热图(图1A)与火山图(图1B),红色代表上调,绿色代表下调。
2.2 差异基因的功能富集分析 为了探索染色质调节因子差异基因对机体细胞组成及代谢活动的影响,本研究对137个差异基因进行了GO和KEGG功能富集分析。结果表明,胃癌上调基因主要包括“碱基切除修复、同源重组、细胞周期、S-腺苷蛋氨酸依赖型甲基转移酶活性、组蛋白甲基转移酶活性、组蛋白结合、核染色质、染色体区域、染色体端粒区域、调节DNA 代谢过程、染色质共价修饰、组蛋白修饰”等,上调的主要KEGG通路为“碱基切除修复、同源重组、细胞周期”(图2A)。下调基因主要包括“修饰依赖蛋白结合、转录辅助因子活性、组蛋白结合、核常染色质、组蛋白甲基转移酶复合体、核染色质、DNA甲基化、组蛋白修饰、染色质共价修饰”等,下调的KEGG代谢通路主要为“胰岛素抵抗”(图2B)。
2.3 PPI网络构建和模块分析 采用String工具构建差异基因的PPI网络,选择物种为homo sapiens,将所得结果导入Cytoscape软件,对PPI网络进行可视化(图3A),置信度>0.9,并采用cyto-Hubba插件筛选出PPI网络中的hub基因。得到前10名hub基因为BRCA2、BARD1、RAD51、NBN、PCNA、TP53BP1、E2F6、RUVBL1、RUVBL2、WDR5(图3B)。
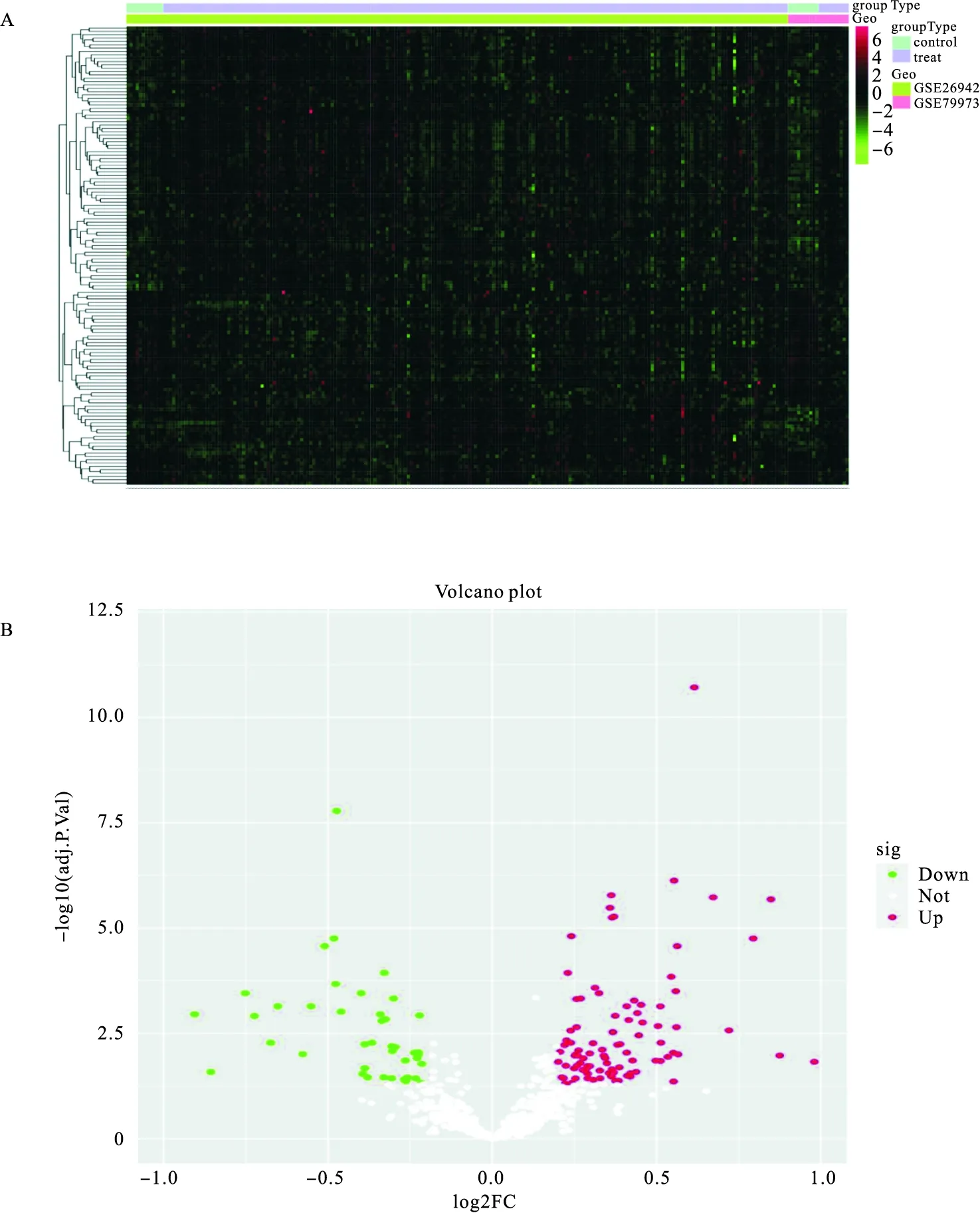
图1 筛选差异基因

图2 DEGs富集分析
2.4 构建hub基因预测模型及潜在治疗药物筛选 采用“2.3”中筛选出的hub基因构建胃癌的预测模型,绘制列线图、Calibration 图及ROC 曲线(图4A-C),由DSigDB数据库可见hub的胃癌预测模型中ROC值为0.953,预测效果良好,其中RUVBL1基因的预测性最佳。并根据hub预测相关潜在治疗药物有氟达拉滨、硝酸钾等。见图4D、表2。

图3 DEGs的蛋白互作网络

图4 ROC曲线与药物预测
3 讨论
近年来,大量研究发现了一些分子标志物与胃癌发生发展的相关性[11-12]。Wu等[13]研究表明,TRERNA1敲低显著降低胃癌细胞迁移、侵袭和转移。TRERNA1消耗减少了体内胃癌的细胞转移,在转录水平调节CDH1作为胃癌进展的关键效应物,提示TRERNA1/CDH1是胃癌治疗的新的潜在靶点[13]。Huang等[14]研究表明,LINC00673可以作为LSD1和EZH2的支架并抑制两者表达,发挥致癌作用,促进胃癌发展进程。Qi等[15]研究显示,AGAP2-AS1过表达促进细胞生长和侵袭。其通过与LSD1和EZH2相互作用并抑制CDKN1A(P21)和E-cadherin转录发挥致癌作用,表明AGAP2-AS1是胃癌患者的潜在诊断标志物和治疗靶点。然而,由于癌症的高度异质性、差异表达基因数量庞大,且染色质调节因子在癌症的发生发展中具有重要作用[16-18],因此,本研究探究了染色质调节因子在胃癌中的作用。
生物信息学分析显示,癌症组织样本较正常组织样本,上调的染色质调节因子基因主要包括:碱基切除修复、同源重组、细胞周期、S-腺苷蛋氨酸依赖型甲基转移酶活性、组蛋白甲基转移酶活性、组蛋白结合、核染色质、染色体区域、染色体端粒区域、调节DNA代谢过程、染色质共价修饰、组蛋白修饰等。上调的KEGG通路:细胞周期、同源重组、碱基切除修复。下调的染色质调节因子基因主要包括:修饰依赖蛋白结合、转录辅助因子活性、组蛋白结合、核常染色质、组蛋白甲基转移酶复合体、核染色质、DNA甲基化、组蛋白修饰、染色质共价修饰等。下调的KEGG代谢通路主要为胰岛素抵抗。
采用cytoHubba插件获得hub基因:BRCA2、BARD1、RAD51、NBN、PCNA、TP53BP1、E2F6、RUVBL1、RUVBL2、WDR5,在胃癌的预测模型中,RUVBL1基因的预测性最佳。RUVBL1具有较弱的ATPase活性[19-20],是TIP60复合物的重要组成部分[21],其伴随和调节转录因子,包括MYC、E2F1和β-连环蛋白(β-catenin),并参与导致致癌的染色质重塑[22]。RUVBL1和β-catenin上调和核定位与癌症进展高度相关,其增强TCF/β-catenin介导的Wnt靶基因转录,因此可能促进癌变[23]。生存结果较差的非小细胞肺癌(NSCLC)患者中也具有较高的RUVBL1和RUVBL2表达[24]。此外,Sun等[25]研究表明,RUVBL1可替代AR信号转导,以促进癌细胞存活和肿瘤耐药性发展。Lone等[26]发现,RUVBL1免疫阳性与肿瘤患者预后不良和复发率较高有关。Zeng等[27]研究表明,敲低RUVBL1可以通过抑制β-连环蛋白信号转导,从而降低癌症细胞增殖和转移,并表明RUVBL1可作为口腔鳞状细胞癌的诊断和预后生物标志物以及治疗靶点。另外,在胃癌的预测模型中,BRCA2与其他基因显示出相反的作用。证据表明,BRCA2基因突变可致胃癌风险增加[28],并且具有BRCA2高表达的胃癌与更好的预后相关[29]。这可能对胃癌具有重要的预后和治疗意义。结果表明,染色质调节因子在胃癌的发生发展中具有重要作用。此外,本研究通过Enrichr平台,采用DSigDB 数据库进行hub基因和共表达基因的药物筛选,其中氟达拉滨、硝酸钾最佳。然而,由于本研究是基于软件统计处理和数据库分析获得的关键基因和药物,其是否能在胃癌中发挥作用仍需进一步的分子生物学实验来验证。
综上所述,染色质调节因子在胃癌的发生发展中具有重要作用,采用染色质调节相关因子构建预后模型效果良好,其中RUVBL1效果最佳,并通过hub基因筛选出了潜在治疗药物,为胃癌的临床治疗提供了辅助指导,但需要更多的临床队列和实验来进一步验证结论。

