澳洲坚果光壳种MiSAD的克隆与表达
杨倩 杨子平 邹明宏 宋喜梅 万继锋 陈 菁 罗炼芳 曾辉


关键词:澳洲坚果;硬脂酰-酰基载体蛋白脱饱和酶;基因克隆;表达分析
中图分类号:S664.9 文献标识码:A
澳洲坚果(M. integrifolia),又称夏威夷果,原产于澳大利亚,在热带和亚热带地区多有种植[1],中国目前已经成为澳洲坚果种植面积最大的国家。澳洲坚果属于山龙眼科澳洲坚果属,主要有4 个种,即M. integrifolia Maiden & Betche、M.tetraphylla L. A. S. Johnson、M. ternifolia F. Mueller和M. jansenii C. L. Gross & P. H. Weston,仅有光壳种(M. integrifolia)和粗壳种(M. tetraphylla)及其杂交种用于商业性栽培。澳洲坚果果仁不仅风味独特,而且具有很高的营养价值,含有丰富的脂肪酸、蛋白质、矿物质元素(锰、铁、镁)、维生素(B6)和微量元素[2]。研究表明,成熟的澳洲堅果中总油脂的含量最高可达80.3%,主要包含油酸、棕榈油酸、棕榈酸、硬脂酸、花生酸、二十碳烯酸、肉豆蔻酸和二十一烷酸等,其中油酸、棕榈油酸和二十碳烯酸是大含量脂肪酸,可以占到总脂肪酸的87.52%[3],澳洲坚果是重要的单不饱和脂肪酸来源植物,也是新兴的木本油料作物,但其遗传转化体系尚未建立,关于澳洲坚果果仁中不饱和脂肪酸生物合成和积累的分子机制研究鲜有报道。
植物硬脂酰-酰基载体脱饱和酶( stearoylacyl-carrier- protein desaturase, SAD)是质体中脂肪酸生物合成途径中的关键酶,能够催化硬脂酰-ACP 在第9 位至第10 位碳原子之间脱饱和形成双键[4],是产生不饱和脂肪酸的关键步骤。因此,植物SAD 酶是产生油酸和多不饱和脂肪酸的前提,大量研究表明,植物SAD 基因能够显著调控植物中饱和脂肪酸和不饱和脂肪酸的比例[5-6]。LIN 等[7]通过转录组测序技术发现澳洲坚果SAD基因家族存在12 个成员,GUMMESON 等[8]和RODR?GUEZ 等[9]早期从澳洲坚果中分离到一个与不饱和脂肪酸生物合成相关的脱饱和酶基因,酶活试验表明澳洲坚果SAD 脱饱和酶能够催化硬脂酰-ACP ( 18:0-ACP ) 和软脂酰-ACP(16:0-ACP)转化成C18:1-ACP 和C16:1-ACP,经过比对分析发现与12 个成员之一的MiFAB2.12一致,但还需进一步验证。因此,本研究开展澳洲坚果光壳种SAD 基因的克隆,并分析其基因结构、理化性质、进化关系和表达特征,为研究澳洲坚果SAD 基因功能和脂肪酸生物合成(积累)分子机制奠定理论基础。
1 材料与方法
1.1 材料
供试材料为澳洲坚果光壳种‘南亚1 号,种植于中国热带农业科学院南亚热带作物研究所的国家热带植物种质资源库——澳洲坚果种质资源圃(中国广东省湛江市)。根取自‘南亚1 号种子萌发的实生幼苗的新鲜幼根;茎、叶取自“南亚1 号”果树上当年新生的嫩叶和嫩茎;花取自‘南亚1 号果树花序上的全部小花混合。分别收集澳洲坚果开花后第45、68、75、86、95、100、117、126、139、146、168、177、188、195、201天的果仁。所有样品采集后,尽快使用液氮速冻,直接用于DNA/RNA提取或者保存于?80℃备用。
Plant Total RNA Isolation Kit Plus 购自天根生化科技(北京)有限公司。MonAmp? 2× MonHI-FIMix 、All-in-One First-Strand Synthesis MasterMix 、Taq SYBR Green qPCR Premix ROXⅠ(NOVA)购自莫纳公司。PCR 产物纯化、凝胶回收试剂盒购自南京诺唯赞生物科技股份有限公司。引物合成与DNA 测序委托广州艾基生物技术有限公司。
1.2 方法
1.2.1 总RNA 提取及cDNA 合成 澳洲坚果叶片总RNA 提取按照Plant Total RNA Isolation KitPlus 方法进行。反转录按照All-in-One First-StrandSynthesis Master Mix 操作。反应体系为:All-in-One First-Strand Synthesis Master Mix 4μL,RNA 模板50 ng~1 μg,dsDNase μL,Nuclease-free water 加至20 μL。反转录程序为:37℃ 10 min,55℃ 30 min,85℃ 1 min。
1.2.2 澳洲坚果SAD 基因全长及cDNA 序列克隆根据澳洲坚果基因组CDS 数据[10],设计扩增SAD编码框的引物(SAD-F:ACAGCAATGGCTCTCAAGCTTA;SAD-R:CCCTTCTACTCAGCTTCCATTCTG)。根据澳洲坚果基因组数据,设计扩增SAD 基因组序列的引物(MiSAD-F51:CGAAAGAAAGAGTGAGTAGGC;MiSAD-R6997:ACAACAATGCCAACGGAG)。PCR 反应体系为:上下游引物各0.5 μL,cDNA 模板1.0 μL,MonAmp? 2×MonHI-FI Mix 10.0 μL,ddH2O 补足至20.0 μL。扩增SAD 编码框的程序为:94℃,预变性5 min;94℃变性30 s,58℃退火30 s,72℃延伸1 min,35个循环;扩增SAD 基因组序列的程序为:94℃,预变性5 min;94℃变性30 s,58℃退火40 s,72℃延伸2 min,35 个循环;终延伸10 min。将PCR产物纯化,连接至T 载体,连接体系为:T 载体0.5 μL,DNA Ligase 0.5 μL,PCR 产物4.0 μL,2×Ligation buffer 5.0 μL,ddH2O 将体系补足至10.0 μL 体系。室温25℃ 5 min 即可完成连接反应。随后转化大肠杆菌DH5α 感受态,以含氨苄青霉素的抗性培养基进行筛选,对阳性克隆测序。
1.2.3 澳洲坚果SAD 生物信息学分析 利用Vector NTI 软件进行多序列比对,以确定扩增的序列为SAD 的基因组序列和编码框。利用DNAMAN 进行开放阅读框(ORF)分析;利用Protparam(http://www.expasy.ch/tools/protparam)在线软件预测蛋白的基本物理化学性质;利用PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)在线软件预测蛋白的二级结构和三级结构; 利用Clustal X 软件多序列比对后,并利用ESPipt3.0( https://espript.ibcp.fr/ESPript/ cgi-bin/ESPript.cgi)在线软件对比对结果可视化;利用SignalP 5.0在线软件预测信号肽;利用MEGA4 软件建立系统进化树。
1.2.4 澳洲坚果SAD 基因表达模式分析 利用Taq SYBR Green qPCR Premix ROXⅠ(NOVA)试剂进行盒荧光定量PCR,罗氏(Roche)LightCycler 480Ⅱ实时荧光定量仪器采集荧光数据。实时荧光定量PCR 反应体系为:SYBR GreenqPCR Mix(2)10.0 μL;特异引物1.0 μL;cDNA模板1.0 μL;ddH2O 补足至20.0 μL。PCR 扩增程序为:94℃预变性3 min;94℃变性10 s,58℃退火30 s,72℃延伸10 s,45 个循环。在每个循环结束后进行荧光信号的采集,所有循环结束后进行溶解曲线分析(65~95℃)。采集数据,计算基因表达量。
2 结果与分析
2.1 澳洲坚果总RNA 提取吸光度检测结果显示,叶片总RNA 的A260/A280 为1.82;其凝胶电泳结果显示,28S 和18S条带清晰,28S 条带亮度约为18S 的2 倍,无弥散条带(图1A)。结果表明提取的总RNA 质量较好,纯度较高,可以用于下一步实验。
2.2 MiSAD 编码框序列的克隆
凝胶电泳结果显示,MiSAD 编码框序列引物扩增的PCR 产物大小约在1000 bp 左右(图1B),与SAD 基因CDS 序列大小接近。测序结果显示,扩增的序列为1224 bp。经分析,扩增片段包含一个最长1191 bp 的编码框,编码396 个氨基酸,命名为MiSAD。经测序后比对,与NCBI 公布的的粗壳种MtSAD(登录号为GU595455.1)及LIN等[7]公布的MiFAB 2.12 序列一致,没有缺失、插入及位点突变。
2.3 MiSAD 基因组序列的克隆
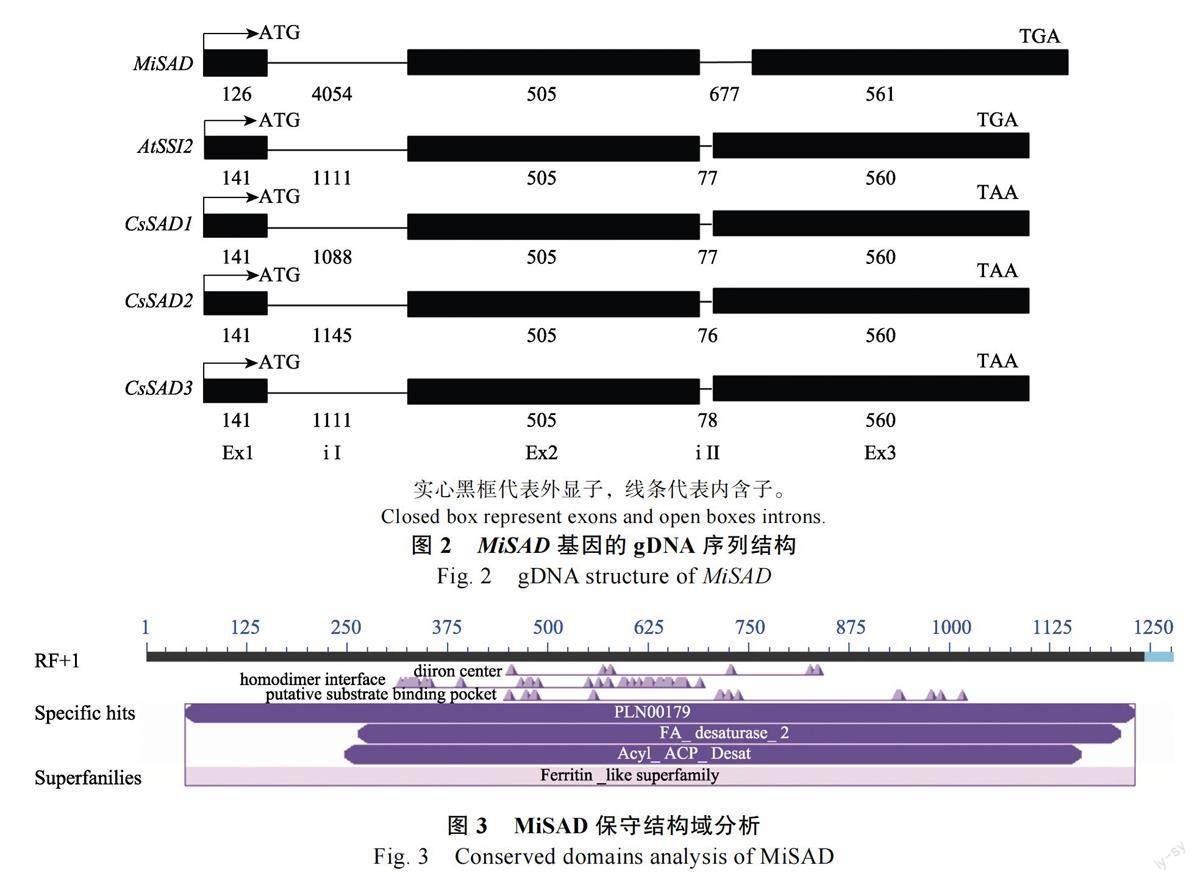
凝胶电泳结果显示,MiSAD 基因组序列引物扩增的PCR 产物大小在6000 bp 左右(图1B),分析发现MiSAD 基因gDNA 序列长度为5923 bp,由3 个外显子和2 个内含子组成(图2)。
比较发现,MiSAD 基因与拟南芥SSI2 基因和亚麻芥SAD 家族基因结构总体相似,都包含3 个外显子2 个内含子(图2)。澳洲坚果的SAD 基因外显子1(Ex1)含126 bp,而拟南芥和亚麻芥的均为141 bp,有细微差异。外显子2 和外显子3长度基本一致,并且序列相似度也较高,为植物SAD 基因的保守区域。其次,澳洲坚果的2 个内含子长度分别为4054 bp 和677 bp,明显大于拟南芥SSI2 和亚麻芥SAD1、SAD2 和SAD3 基因的1088~1145 bp 和76~78 bp。
2.4 澳洲坚果SAD 的理化性质与二、三级结构预测
蛋白理化分析结果显示,澳洲坚果SAD 分子式为C2024H3162N546O598S16,总原子数6346,预测的分子量约为45.22 kDa,蛋白质等电点为5.93,负电荷残基总数(Asp+Glu)为56,正电残基总数(Arg+Lys)为50 个,不稳定系数为40.1,脂肪族氨基酸指数为79.34,总平均亲水性系数为?0.446,推测该蛋白为稳定的亲水性蛋白。
利用NCBI 的保守结构域数据库预测澳洲坚果SAD 蛋白的结构域和保守位点,发现具有2 个保守功能结构域(图3),一个是酰基-ACP 脱饱和酶家族;另一个是类铁蛋白家族。保守区中存在6 个氨基酸的金属离子结合基序、多个酶活性位点及亚基结合位点。
利用在线工具PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)预测澳洲坚果SAD 酶的蛋白二级结构,结果显示,该酶主要以α-螺旋和无规则卷曲为主(图4),并且在第176~179 和第262~265的残基上存在2 个E-X-X-H 组氨酸富集区,为二铁离子提供结合位点,形成SAD 酶的活性中心。三级结构预测中存在螺旋- 转角- 螺旋结构(HTH),与二级结构预测结果一致。SignalP 5.0和DeepLoc-1.0 在线预测结果显示,澳洲坚果SAD序列不存在信号肽,为非分泌的膜结合蛋白。
2.5 澳洲坚果SAD 多序列比对分析和系统进化分析
在NCBI 将推导的氨基酸序列进行blastp 分析发现,发现澳洲坚果SAD 与其他很多物种的SAD 具有较高的同源性,其中与澳大利亚分布的同为山龙眼科的蒂罗花同源性最高达94.2%,与木薯同源性也较高为89.9%,葡萄89.4%,荷花89.1%,油茶88.6%,木油桐88.9%,其他物种的SAD 同源性也均在80% 以上。选取荷花(XP_010242709.1, XP_010245693.1);澳洲坚果(ADE06393.1);葡萄(XP_002274708.2);木薯(XP_021610569.1);乌桕(ABN13874.1);木油桐(ABU50334.1);克萊门柚(XP_006442769.1);山桐子(QDX46951.1);猫爪藤(AHN16186.1,AHN16187.1);咖啡(XP_027164486.1);黄麻(OMO62182.1);木本棉(XP_017648973.1);枣(XP_015870024.1);油茶(QID89772.1);向日葵(CAC80359.1);大豆(AAA92462.1);油橄榄(AAB67840.1);蓖麻(NP_001310659.1);拟南芥(AAL90985.1)等19 个物种的21 个SAD的氨基酸序列,进行多重比对发现,澳洲坚果SAD与其他植物的SAD 序列在功能结构域具有较高的保守性,但在N 端和C 端的氨基酸残基上有较明显的差异(图5)。

