葡萄果粒质量相关性状全基因组关联分析
王慧玲,闫爱玲,王晓玥,刘振华,任建成,徐海英,孙磊
葡萄果粒质量相关性状全基因组关联分析

1北京市农林科学院林业果树研究所,北京 100093;2北京市落叶果树工程技术研究中心,北京 100093;3农业农村部华北地区园艺作物生物学与种质创制重点实验室,北京 100093
【目的】果粒大小是葡萄外观和产量的重要构成因子之一,为受多基因调控的复杂数量性状,挖掘葡萄果粒大小相关性状的关键遗传调控位点和基因,将有助于葡萄产量的提高。【方法】本研究以150份葡萄品种资源为材料,分别于2019年和2020年对葡萄果实单粒重、种子数目和种子质量等进行测定,并结合重测序获得的高密度基因型数据进行全基因组关联分析(genome-wide association study,GWAS),挖掘调控各性状的遗传位点和基因。【结果】各性状在关联群体中呈现广泛的连续变异,变异系数为39.55%—68.89%;在不同年份均服从正态分布,符合数量性状遗传特征;相关性分析表明葡萄果实单粒重、种子数目和种子质量呈显著正相关。全基因组关联分析共检测到150个与果实单粒重显著关联的SNP,在2019年检测到99个SNP,解释表型变异的14.48%—25.59%;在2020年检测到73个SNP,解释表型变异的16.08%—26.83%;其中24个SNP位点在两个年份均检测到,主要位于1号、5号、11号和16号染色体。相较于果实单粒重,检测到的与种子数目显著关联的SNP较少,2019年检测到1个显著性SNP,表型解释率为24.29%;2020年检测到17个显著性SNP,均位于18号染色体。两个年份检测到与种子质量显著关联的SNP分别有1个和2个,位于18号染色体,解释表型变异的23.59%—48.29%。在两年重复检测到SNP位点基因组区域内,根据基因功能注释筛选出11个可能与果实单粒重相关的候选基因,其中包括乙烯信号通路基因(基因ID:VIT_05s0049g00490、VIT_05s0049g00500、VIT_05s0049g00510和VIT_16s0100g00400)、赤霉素信号途径基因(基因ID:VIT_11s0016g04630和VIT_16s0022g02310)、生长素响应蛋白基因(基因ID:VIT_11s0016g05640)和一些重要的转录因子基因(基因ID:VIT_05s0049g00460、VIT_11s0016g05660和VIT_16s0022g02330)。在18号染色体鉴定到与种子含量相关的候选基因(基因ID:VIT_18s0041g01880,编码MADS-box蛋白AGL11),位于该基因上的SNP基因型变化显著影响葡萄果实种子数目和质量。【结论】结合两个年份的表型数据,共检测到150个与果实单粒重显著关联的SNP,主要定位于1号、5号、11号和16号等染色体;检测到19个与种子含量关联的SNP,主要定位于18号染色体。基于基因注释和基因型分析结果,确定了包含VIT_11s0016g04630和VIT_16s0022g02310等在内的11个候选基因可能参与调控葡萄果实单粒重,确定候选基因(基因ID:VIT_18s0041g01880)与种子含量显著相关。
葡萄;果粒大小;全基因组关联分析;候选基因
0 引言
【研究意义】葡萄是世界上种植最广泛的园艺作物之一,全球种植面积约750万hm2,2018年产量为7 780万t,其中36%是鲜食葡萄(http://faostat.fao.org)。果粒大小是鲜食葡萄的重要农艺性状之一,影响着消费者的偏好和接受度,同时决定最终的产量。因此,果粒大小一直是葡萄育种者主要关注的性状之一。阐明果粒大小相关性状的遗传基础,将有助于理解葡萄果实发育的分子调控机制,且为提高传统育种的准确性和效率奠定基础。【前人研究进展】在葡萄中,果粒大小存在广泛的变异。平均而言,成熟时的果实单粒重在0.5—11 g,种子数在0—6个,每个浆果的种子重量在0.03—0.22 g[1]。种子有无与果粒大小密切相关,大多数无核葡萄品种自然条件下果粒偏小[2]。与大多数葡萄农艺性状相同,葡萄果粒大小及其相关性状是由多种基因和环境因素共同作用产生的数量性状[3]。前人利用不同的遗传群体,研究了果粒大小或其相关性状表型变异的遗传决定因素。在以无核品种为亲本的杂交群体中,与果实单粒重相连锁的主效数量性状遗传位点(quantitative trait loci,QTL)主要被定位于第18号染色体上,与已知的无核位点共定位[4-5]。Doligez等[3]利用4个不同的杂交群体,在1号、8号、11号、17号和18号染色体检测到与果实单粒重相连锁的QTL位点,其中位于17号连锁群的QTL位点表型解释率达到31%。CORREA等[6]在‘红宝石无核’和‘无核白’杂交后代群体中,鉴定到2个与果粒大小相关的QTL位点,分别位于2号和18号染色体。同年HOUEL等[7]在微型葡萄(microvine)群体中鉴定到一个新的位于7号染色体的主效QTL位点。而BAN等[8]利用一个具有欧美品种遗传背景的杂交群体,连续4年在11号染色体发现一个稳定的QTL位点。近年来,全基因组关联分析(genome wide association analysis,GWAS)已经广泛应用于各种植物重要性状基因挖掘[9-10]。在葡萄中,GUO等[11]对167份葡萄核心种质进行了低覆盖度的重测序分析,将果实单粒重性状定位到17号、18号和19号等染色体。而FLUTRE等[12]分别在1号、2号、8号、11号、15号和17号等染色体鉴定到与果实单粒重显著关联的基因位点。此外,利用全基因组关联分析,除了18号染色体,ZHANG等[13]在5号、6号、10号和17号等染色体也检测到与种子含量显著关联的基因位点。【本研究切入点】采用不同的群体材料,鉴定到不同的遗传位点信息,说明果粒大小是复杂的数量性状,需要利用遗传背景更广泛的群体进行研究。另外,之前的研究多采用遗传群体,由于群体个数有限,作图精度低,导致定位到的等位基因较少。相较于传统的连锁分析,关联分析是以连锁不平衡为基础,分析标记候选基因的遗传变异与某一群体内目标性状关系的方法。选用不同来源的自然群体作为分析对象,所研究的等位基因更丰富,更容易找到目标性状的多个微效基因,有利于数量性状的基因定位。【拟解决的关键问题】本研究以150份葡萄品种资源群体为材料,结合重测序获得的葡萄全基因组基因型数据,通过对葡萄果粒大小相关性状开展GWAS分析,解析葡萄果粒大小相关性状的遗传基础,挖掘出重要的调控基因位点,提高对葡萄果粒大小形成的遗传结构和分子机制的认识,为进一步克隆调控葡萄果粒大小的关键基因,加速优良葡萄新品种培育及种质改良奠定基础。
1 材料与方法
1.1 试验材料
供试材料选取150份葡萄品种(附表1),该群体具有广泛的果粒大小相关性状变异。供试葡萄植株种植于北京市农林科学院林业果树研究所葡萄资源圃(北纬39°58′,东经116°13′),单臂篱架水平龙干整形,株行距0.8 m×2 m,南北走向,采用简易避雨、地表园艺地布覆盖、滴灌供水和常规病虫害等管理模式,生长期内修剪及肥水管理一致,树势基本相同。
1.2 试验方法
1.2.1 样品采集 于2019年4月初萌芽期采取嫩叶,用液氮速冻保存于-80℃冰箱,用于后续DNA提取。
分别于2019—2020年8—10份进行成熟果实样品采集。果实成熟度依据往年物候期记载和果实可溶性固形物含量(ºBrix≥16)确定。由于机械损伤(埋土防寒)、坐果不良或果实腐烂,每年采集的样本数量各不相同。2019年和2020年分别采集到139份和135份葡萄品种果实样品。针对每个葡萄单株分别在阳面和阴面随机挑取3穗果,从不同位置选取10—20个果实作为一个重复,用于后期性状表型调查,3次重复。
1.2.2 葡萄果粒大小相关性状调查 分别对葡萄果实单粒重(BW)、种子数(SN)和种子质量(SW)进行测量分析,测量方法参照刘崇怀等[14]编著的《葡萄种质资源描述规范和数据标准》。
1.2.3 葡萄叶片DNA提取和全基因组重测序 葡萄叶片DNA提取采用植物DNA提取试剂盒(北京天根生物技术有限公司),并利用琼脂糖凝胶电泳检测DNA的完整性和纯度。检测合格的DNA样品通过Covaris破碎机随机打断成长度为350 bp的片段,经末端修复、加ployA尾、加测序接头、纯化、PCR扩增等步骤完成整个文库制备。构建好的文库通过Illumina HiSeqPE150进行测序。
针对原始测序数据,进行过滤,过滤的过程如下:(1)去除接头(Adapter)序列;(2)去除含N比例大于10%的reads;(3)去除低质量reads(质量值Q≤20的碱基数占整条read的50%以上)。进一步利用比对软件BWA将数据比对到参考基因组(http:// plants.ensembl.org/Vitis_vinifera/Info/Index),利用Picard(Picard: http://sourceforge.net/projects/picard/)对比对结果进行排序并标记重复序列。使用软件GATK获得变异数据集,检测到的变异进一步过滤获得高质量标记:(1)非二等位位点去除;(2)第二等位基因频率(minor allele frequency,MAF)小于0.05的位点去除;(3)缺失率大于0.5的位点去除;(4)杂合比例大于0.8的位点去除。
1.2.4 全基因组关联分析(GWAS) 使用Admixture软件(v1.3)[15]进行群体结构的推断。使用PopldDecay软件(v3.41)[16]计算两两标记间的LD大小(2),绘制连锁不平衡的衰减图。使用gemma软件(v0.98.1)[17]进行GWAS分析,采用GWAS常用模型(GLM、GLM(Q)、MLM(K)、MLM(QK))进行计算。Admixture最优K值对应的群体结构矩阵作为相应模型的Q矩阵,gcta软件计算的样品间亲缘关系矩阵作为相应模型的K矩阵。
1.2.5 候选基因筛选(GO、KEGG注释分析) 根据果粒大小相关性状显著关联的SNP标记在葡萄参考基因组中的物理位置,筛选显著SNP位点上下游LD衰减距离区域范围内的候选基因。利用Ensemble Plants数据库(http://plants.ensembl.org/index.html/)及美国国立生物技术信息中心数据库(https://www.ncbi. nlm.nih.gov/)对候选基因进行功能注释和预测。
1.2.6 数据处理与统计分析 应用Excel 2007和SPSS 13.0软件对性状表型数据进行描述性统计分析和数据分布统计,并运用Pearson相关系数进行性状相关性分析,最低显著水平<0.01。采用SPSS中paired-samplestest进行显著关联标记等位效应分析。
2 结果
2.1 葡萄果粒大小相关性状统计分析
分别于2019年和2020年对150份葡萄品种进行果实单粒重(BW)、种子数量(SN)和种子质量(SW)调查,基本统计分析(表1)表明,2019年关联群体果实单粒重、种子数量和种子质量均值分别为(5.62± 2.37)g、(2.23±1.15)个和(0.34±0.22)g;2020年相应各个性状均值分别为(5.92±2.44)g、(2.20±0.87)个和(0.45±0.31)g;不同年份各性状在关联群体中呈现广泛的连续变异,变异系数为39.55%—68.89%。对各个性状的偏度和峰度进行计算,绝对值均小于1,表明这些性状在不同年份均服从正态分布。结合各自的频率分布直方图可以看出(图1),果实单粒重表型数据基本呈现正态分布,种子数量和种子质量呈现非对称性的双峰分布,并不符合常规的单峰正态分布;各个性状均表现数量性状遗传特征。

表1 葡萄果粒大小相关性状描述性统计

图1 葡萄果粒大小相关性状频率分布
不同年份关联群体果实单粒重、种子数目和质量的Pearson相关分析表明(表2),果实单粒重与种子数目、种子质量在两个年份均呈显著正相关,种子数目和质量在两个年份呈显著正相关,说明各性状之间相互影响,相互协同。此外,相同性状在不同年份也呈现显著正相关,表明各个性状主要受遗传因素影响。

表2 葡萄果粒大小性状相关系数
2019BW:2019年单粒重;2019SN:2019年种子数目;2019SW:2019年种子质量;2020BW:2020年单粒重;2020SN:2020年种子数目;2020SW:2020年种子质量
2019BW: Berry weight in 2019; 2019SN: Seed number in 2019; 2019SW: Seed weight in 2019; 2020BW: Berry weight in 2020; 2020SN: Seed number in 2020; 2020SW: Seed weight in 2020. *:<0.05; **:<0.01
2.2 全基因组关联分析
通过软件PopLDdecay以2降低到全染色体最大值(0.3702)一半时的物理距离作为LD衰减距离(图2-A),计算为1.8 kb。根据连锁不平衡衰减图可见,随着物理距离的增加,2值急剧下降,表明这些群体在演化过程中重组率较大,关联分析所需要的SNP密度更大,但相应能获取更小的候选区间,筛选候选基因更加容易。通过Admixture软件分析,当交叉验证错误率最低时,K值为4,因此,可将亚群根据群体的基因型情况细分为4个。将K=4时所对应的群体结构矩阵作为后续关联分析所用矩阵(图2-B)。

图2 群体连锁不平衡(LD)和群遗传结构分析
连续2年结合果实单粒重、种子数目和种子质量等性状表型数据进行GWAS分析,基于0.01/SNP总数作为显著关联的SNP标记筛选阈值。2019年,共检测到与果粒单重相关的显著SNP有99个,主要位于1号、3号、5号、7号、11号、15号、16号、17号和19号染色体上,表型解释率在14.48%—25.59%(图3-A、附表2);2020年,共检测到与果粒单重相关的显著SNP有73个,主要位于1号、5号、11号、16号和17号染色体,解释表型变异的16.08%— 26.83%(图3-B、附表2)。相较于果粒单重,检测到的与种子数目显著关联的SNP较少,2019年仅检测到1个显著性SNP(图3-C、附表2),表型解释率为24.29%;2020年检测到17个显著性SNP(图3-D、附表2),均位于18号染色体,表型解释率在16.42%—30.48%。两个年份检测到与种子质量显著关联的SNP分别有1个(2019,图3-E)和2个(2020,图3-F),位于18号染色体,解释表型变异的23.59%— 48.29%(附表2)。
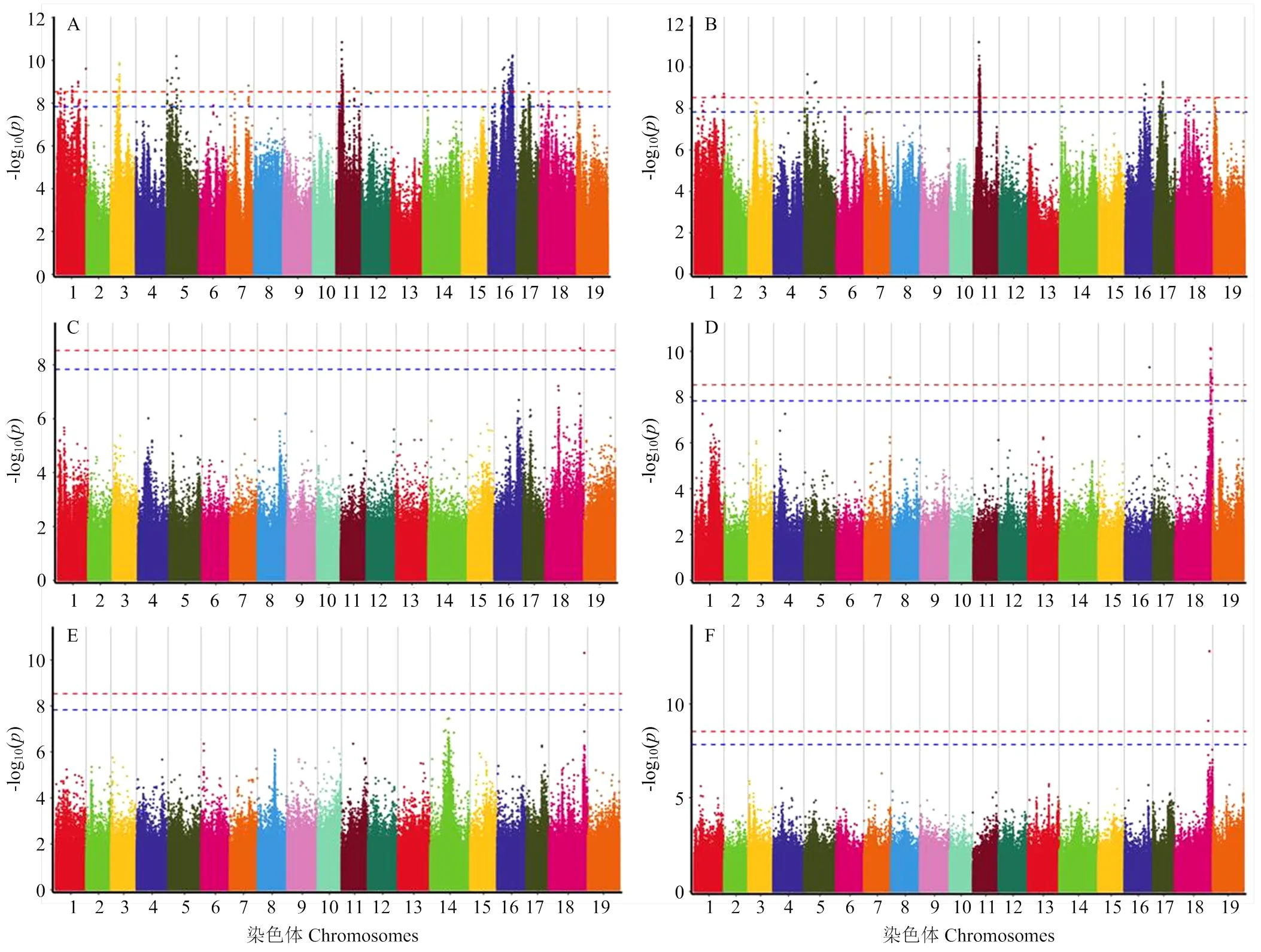
A—B:分别表示2019和2020年葡萄果实单粒重GWAS分析结果;C—D:表示两个年份种子数量GWAS分析结果;E—F:表示两个年份种子质量GWAS分析结果。横坐标为各染色体从小到大排列的物理位置,一个点代表一个SNP位点,纵坐标为p值取-log10后对应数值;红色横虚线位置为0.01/SNP总数的负对数,蓝色虚线为0.05/SNP总数的负对数,高于阈值的点为关联显著位置
为了保证关联位点的可靠性,进一步筛选两个年份同时检测到的显著SNP位点作为后期候选基因挖掘重点区域,由结果可见(表3、附表2),24个与果粒单重显著关联的SNP在两个年份重复被检测到,分别位于1号、5号、11号和16号染色体上。其中1号染色体有1个SNP位点,2019年和2020年的表型解释率分别为23.45%和23.30%;5号染色体上检测到位于7 537 999和9 220 655位置的两个SNP位点,两个年份表型解释率分别为21.59%、22.03%和22.19%、23.47%;11号染色体上有19个SNP在两个年份均被检测到,表型解释率范围16.03%—27.69%;最后两个SNP位点位于16号染色体,14 893 081位点表型解释率分别为18.70%(2019年)和18.45%(2020年);15 790 424位点2019年和2020年表型解释率分别为18.55%和18.45%。与种子数目关联的显著SNP在两个年份同时检测到1个SNP位点,2019年和2020年表型解释率分别为24.29%和25.67%。而两个年份均检测到的与种子质量显著关联的SNP也是1个,表型解释率达到40.11%(2019年)和48.29%(2020年)。
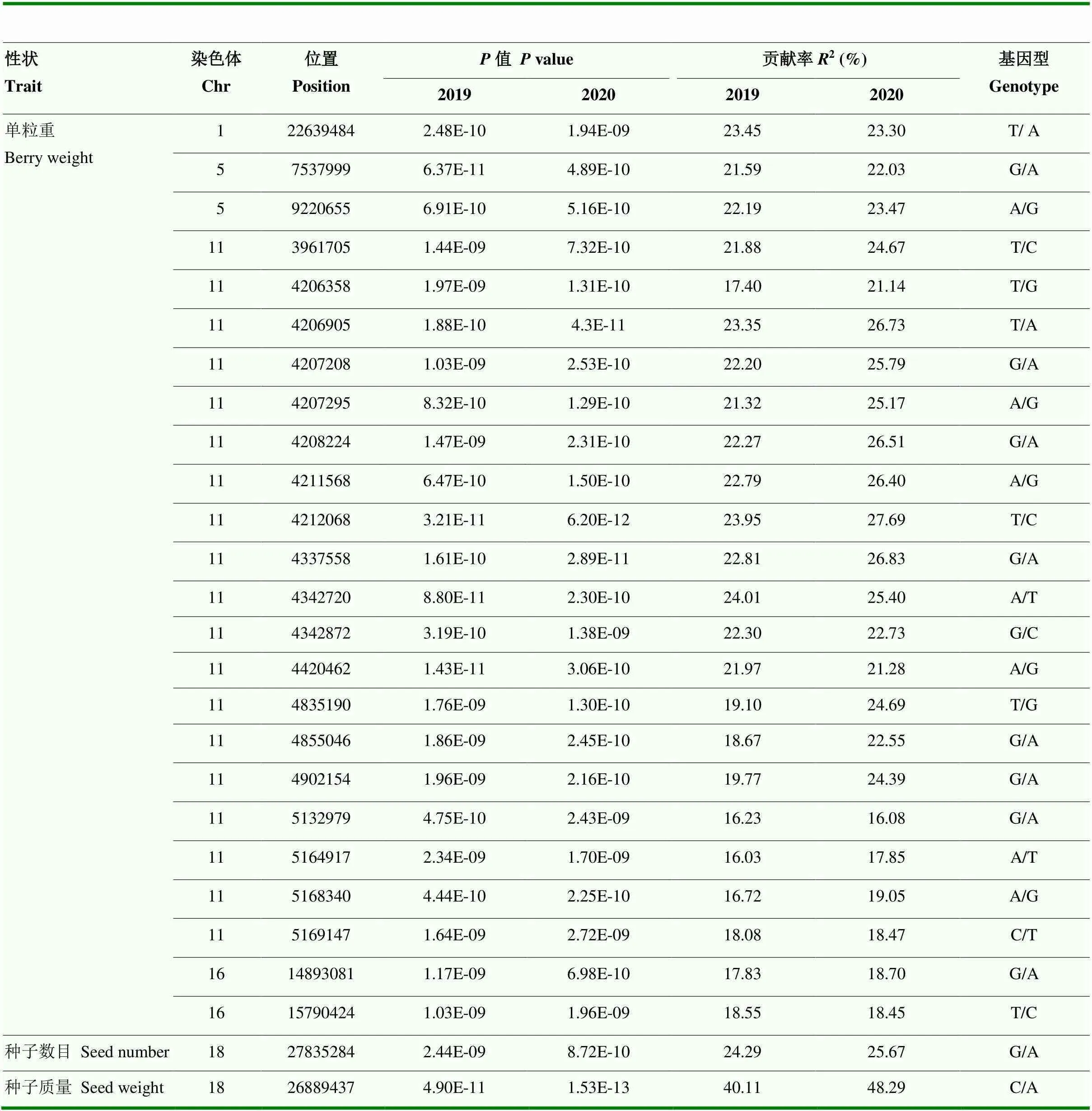
表3 葡萄果粒大小相关性状全基因组关联分析两年同时检测到的重合显著性位点信息
2.3 候选基因筛选
依据该群体的LD衰减距离(图2-A),在26个位点的基因组区段内共检测到76个基因,结合基因功能注释和前人报道,筛选出11个候选基因可能参与果实大小发育过程(表4)。其中与单粒重相关的基因有VIT_01s0150g00260(编码E3泛素蛋白连接酶)、VIT_05s0049g00460(编码转录因子bHLH104)、VIT_05s0049g00490—VIT_05s0049g00510(编码乙烯响应转录因子)、VIT_11s0016g04630(编码DELLA蛋白SLR1)、VIT_11s0016g05640(编码生长素响应蛋白IAA9)、VIT_11s0016g05660(编码转录因子MYB82)、VIT_16s0022g02310(编码赤霉素-20-氧化酶)、VIT_16s0022g02330(编码MADS-box转录因子6)和VIT_16s0100g00400(乙烯响应转录因子ERF027)。与种子数目和质量相关的基因有VIT_ 18s0041g01880(编码MADS-box蛋白AGL11)。

表4 位于检测显著位点区域已发表的可能参与葡萄种子和/或浆果发育的功能候选基因
2.4 候选基因关联标记遗传效应分析
为进一步确定可能的候选基因,针对与候选基因关联的7个SNP位点的等位变异效应进行分析,6个与单粒重性状显著关联的SNP位点在本群体中呈现3种基因型,与种子数量和质量关联的SNP在本群体中检测到2个基因型。由图4可见,每个SNP的等位变异在两个年份的果实大小相关性状均达到显著水平。例如,SNP 5_7537999位点G/A和A/A等位变异在两个年份的平均果实单粒重显著高于G/G等位变异;SNP 18_26889437不同基因型对种子数目和质量均有显著影响。该结果进一步说明与该标记关联的候选基因可能在调控葡萄果粒大小方面发挥重要的作用。
3 讨论
3.1 葡萄果实大小相关性状的遗传特性分析
在杂交后代遗传群体中的研究表明,葡萄果实单粒重呈现丰富的遗传变异,遵循多基因控制的数量性状遗传特征[2-8]。本研究结果显示两个年份果实单粒重性状在群体中均表现出连续的变异,这与之前另外的自然群体研究结果一致[11];并且在不同年份之间该性状表型存在明显的波动,进一步表明葡萄果实单粒重属于复杂的数量性状,可能受到基因和环境因子等多方面调控。此外,HOUEL等[1]调查多个葡萄品种平均单粒重发现,成熟时的果实单粒重在0.5—11 g,这与本研究结果相似。种子数目和种子质量在本研究群体中呈现双峰分布,前期研究者在多个杂交后代群体中发现相似的分布规律,第一个峰主要代表无核个体的分布,而第二个峰代表有核个体的分布,进一步确定葡萄果实种子的有无受一个主效遗传位点SDI控制,而有核个体中种子含量为多基因控制的数量性状[2-5]。

SNPs 1_22639484、5_7537999、11_39611705、11_5164917、16_14893081和16_15790424分别与候选基因VIT_01s0150g00260、VIT_05s0049g00460 —VIT_05s0049g00510、VIT_11s0016g05640—VIT_11s0016g05660、VIT_16s0022g02310—VIT_16s0022g02330关联;SNP 18_26889437位于候选基因VIT_18s0041g01880内;**和*分别表示0.01和0.05显著水平
此外,本研究还发现在本群体中,果实单粒重和种子数目、种子质量在两个年份均表现显著正相关。已有研究表明葡萄果实单粒重和种子含量之间的相关性在不同群体中表现不同,在有核品种内[27-28],无核分离群体内[2-5],以及由有核和无核葡萄育种群体组成的群体内[29],经常观察到果实单粒重和种子含量之间具有正相关关系。但是在另外一些群体中,种子含量和浆果大小之间没有发现显著的相关性[1]。一般认为,果粒大小与种子含量之间的相关性主要来自种子产生的生长调节剂[3]。
3.2 葡萄果实大小相关性状的全基因组关联分析
基于表型数据,本研究两个年份共检测到与葡萄果实单粒重显著关联的SNP位点有150个,分别位于1号、3号、5号、7号、11号、15号、16号、17号和19号等染色体上,其中位于1号、5号、11号和16号染色体上的24个与果粒单重显著关联的SNP在两个年份均检测到,说明这些位点受环境影响较小,可在不同环境下稳定遗传,可能存在调控果实单粒重的重要调控因子。利用不同的杂交后代遗传群体,研究者也分别在1号[3-4]、5号[30]、11号[3,8]染色体上鉴定到稳定的数量性状遗传位点(QTL),与本研究结果一致。此外,还分别在2号、7号、8号、17号、18号等染色体检测到与果粒单重相连锁的QTL位点,其中位于17号连锁群的QTL位点表型解释率较高[3-8]。在本群体中,尽管连续两年均在17号染色体检测到显著性关联位点,但是SNP位置略有差异,并未在2号、8号和18号等染色体检测到显著性位点。但是本群体中检测到新的位于16号染色体的显著关联SNP位点,且其中1个SNP连续两年均被检测到。值得注意的是,本研究中两个年份在11号染色体重复检测到19个显著关联SNP。BAN等[8]利用一个种间杂交群体在11号染色体确定了葡萄果实单粒重主效QTL。DOLIGEZ等[3]和RICHTER等[31]也在欧亚种杂交群体中报道了位于11号染色体上的QTL。这些结果说明在该染色体区域存在重要的调控葡萄果实单粒重的基因。
种子含量作为葡萄的重要性状,研究者已经利用不同的群体确定了位于18号染色体上的该性状主效调控遗传位点SDI[4,26]。在本研究群体中,也在18号染色体检测到与种子数目和质量显著关联的SNP位点,且与SDI位点物理位置一致(表3)。ZHANG等[13]利用199份葡萄种质进行全基因组关联分析,还分别在5号、6号、10号和17号染色体鉴定到与无核显著关联的SNP位点。ZINELABIDINE等[32]还在8号、11号、12号、14号、16号等染色体检测到与种子数目显著关联的基因位点;而在本研究群体中,并未检测到相同的基因位点,这可能与群体大小和不同的遗传背景相关。关于种子数目,本群体中2019年在7号和16号染色体鉴定到新的SNP位点。
3.3 葡萄果实大小相关性状的候选基因预测
针对两年重复检测到的SNP位点基因组区域,进行关键候选基因挖掘和筛选,最终确定11个候选基因可能参与调控葡萄果实大小形成。植物激素可以调节各种植物生长发育过程,包括果实发育和果实大小,以响应环境和内源信号。乙烯作为一种重要的植物激素,通过抑制或者刺激生长在各种植物发育过程中发挥着作用[33]。最近,科学家分离到一种乙烯响应因子(AP2/ERF转录因子,EXCESSIVE NUMBER OF FLORAL ORGANS (ENO)),并报道其参与番茄果实大小的调节[20]。本研究分别在5号和16号染色体显著关联SNP位点区域鉴定到编码乙烯响应转录因子的基因(基因ID:VIT_05s0049g00490、VIT_05s0049g00500、VIT_05s0049g00510和VIT_16s0100g00400)。与这些基因紧密关联的SNP分子标记基因型改变会导致果粒大小显著变化(图4),说明这些基因可能在调控葡萄果实大小方面发挥着重要作用。
此外,本研究在11号染色体显著关联区域鉴定到一个编码DELLA蛋白基因(基因ID:VIT_11s0016g04630)。DELLA蛋白是植物生长发育过程中响应赤霉素(GA)应答途径的关键调控因子,主要行使转录调控因子的功能,几乎参与了植物生长发育的各个重要过程[34]。已有的研究表明,GA在葡萄果实种子形成和果实大小方面发挥着重要的作用,合适浓度的GA处理可以促进葡萄果实增大[35]。在汤普森无核葡萄中,GA信号通过DELLAs、GA受体以及两个GA特异性F-box蛋白传导并发挥作用,DELLA蛋白与不同器官对外源性GA的反应有关[36]。此外,在16号染色体显著性SNP位点区域还鉴定到VIT_16s0022g02310,该基因编码赤霉素-20-氧化酶。在梨果实的研究中,科学家发现赤霉素-20-氧化酶可以通过改变赤霉素合成途径加强GA4合成从而促进坐果及果实发育[24]。结合关联分子标记等位变异效应分析结果,本研究获得的两个基因可能在介导GA调控葡萄果实大小方面具有重要的作用。在11号染色体显著关联区域还鉴定到一个编码生长素响应蛋白(auxin-responsive protein IAA9)的基因(基因ID:VIT_11s0016g05640)。生长素通过调节细胞分裂和细胞伸长,控制植物的生长发育。之前的研究表明,生长素对葡萄果实发育具有重要的调控作用[37]。Aux/IAA作为调控生长素信号通路的主要基因家族之一,也已在番茄中被证明与果实大小相关[22]。
转录因子作为基因表达的调控因子,在植物发育和对激素信号的响应中发挥着重要作用[38]。已有的研究报道bHLH蛋白在肉质果实发育过程中通过调控生长素及赤霉素对于果实大小发挥作用[39]。葡萄中的MYB5b转录因子在番茄中诱导矮化、叶片结构的改变、花形态的改变等,果实和叶片的扫描电镜证实了细胞大小和形状的改变[40]。多个MADS-box转录因子6的基因在其他果树作物中影响果实大小发育[25]。本研究在5号、11号和16号染色体显著关联SNP位点区域分别检测到编码bHLH104、MYB82和MADS-box转录因子6的基因,且与这些转录因子关联的分子标记基因型变化对表型具有显著的影响(图4),推测这些基因可能参与调控葡萄果粒大小形成,但是基因具体的功能需要后期通过转基因和基因敲除等技术验证。
本研究在18号染色体鉴定到与种子含量相关的候选基因(基因ID:VIT_18s0041g01880),该基因已被证明为无核性状主效调控位点SDI的候选基因,并且报道位于该基因上chr18:26889437位置SNP基因型的改变是导致无核的主要原因[26]。在本研究群体中检测到相同位置的SNP位点,该SNP表型解释率达到48%(表3),且该SNP基因型改变显著影响种子含量(图4),可以作为后期无核性状筛选的分子标记。综上,本研究结果进一步证实了葡萄果粒质量相关性状的多基因性质,其受到不同染色体上许多基因位点的调控[2-8]。基于其复杂的遗传基础,需要在单个葡萄基因型中聚合多个有益的QTL等位基因,以通过加性互补机制提高葡萄产量[41]。此外,鉴定到的与表型显著关联的SNP信息,可以为后期通过基因编辑技术定向改良这些遗传位点提供参考依据。
4 结论
连续两年对葡萄果实单粒重、种子数目和种子质量3个相关性状进行全基因组关联分析,共检测到169个与性状显著关联的SNP位点。其中,与果实单粒重显著关联的26个SNP位点在两年被同时检测到,主要位于1号、5号、11号、16号和18号染色体。在重复检测到SNP位点的基因组区段中,筛选出11个可能与果实单粒重相关的候选基因。种子数目和质量性状在两年分别重复检测到1个SNP位点,筛选出与种子数目和质量相关的候选基因(基因ID:VIT_18s0041g01880)。
[1] HOUEL C, MARTIN-MAGNIETTE M L, NICOLAS S D, LACOMBE T, LE CUNFF L, FRANCK D, TORREGROSA L, CONÉJÉRO G, LALET S, THIS P, ADAM-BLONDON A F. Genetic variability of berry size in the grapevine (L.). Australian Journal of Grape and Wine Research, 2013, 19(2): 208-220.
[2] CABEZAS J A, CERVERA M T, RUIZ-GARCÍA L, CARREÑO J, MARTÍNEZ-ZAPATER J M. A genetic analysis of seed and berry weight in grapevine. Genome, 2006, 49(12): 1572-1585.
[3] DOLIGEZ A, BERTRAND Y, FARNOS M, GROLIER M, ROMIEU C, ESNAULT F, DIAS S, BERGER G, FRANÇOIS P, PONS T, ORTIGOSA P, ROUX C, HOUEL C, LAUCOU V, BACILIERI R, PÉROS J P, THIS P. New stable QTLs for berry weight do not colocalize with QTLs for seed traits in cultivated grapevine (L.). BMC Plant Biology, 2013, 13: 217.
[4] MEJÍA N, GEBAUER M, MUÑOZ L, HEWSTONE N, MUÑOZ C, HINRICHSEN P. Identification of QTLs for seedlessness, berry size, and ripening date in a seedless × seedless table grape progeny. American Journal of Enology and Viticulture, 2007, 58(4): 499-507.
[5] COSTANTINI L, BATTILANA J, LAMAJ F, FANIZZA G, GRANDO M S. Berry and phenology-related traits in grapevine (L.): From quantitative trait loci to underlying genes. BMC Plant Biology, 2008, 8: 38.
[6] CORREA J, RAVEST G, LABORIE D, MAMANI M, TORRES E, MUÑOZ C, PINTO M, HINRICHSEN P. Quantitative trait loci for the response to gibberellic acid of berry size and seed mass in tablegrape (L.). Australian Journal of Grape and Wine Research, 2015, 21(3): 496-507.
[7] HOUEL C, CHATBANYONG R, DOLIGEZ A, RIENTH M, FORIA S, LUCHAIRE N, ROUX C, ADIVÈZE A, LOPEZ G, FARNOS M, PELLEGRINO A, THIS P, ROMIEU C, TORREGROSA L. Identification of stable QTLs for vegetative and reproductive traits in the microvine (L.) using the 18 K Infinium chip. BMC Plant Biology, 2015, 15: 205.
[8] BAN Y, MITANI N, SATO A, KONO A, HAYASHI T. Genetic dissection of quantitative trait loci for berry traits in interspecific hybrid grape (×). Euphytica, 2016, 211(3): 295-310.
[9] 张芳, 任毅, 曹俊梅, 李法计, 夏先春, 耿洪伟. 基于SNP标记的小麦籽粒性状全基因组关联分析. 中国农业科学, 2021, 54(10): 2053-2064.doi: 10.3864/j.issn.0578-1752.2021.10.002.
ZHANG F, REN Y, CAO J M, LI F J, XIA X C, GENG H W. Genome-wide association analysis of wheat grain size related traits based on SNP markers. Scientia Agricultura Sinica, 2021, 54(10): 2053-2064. doi: 10.3864/j.issn.0578-1752.2021.10.002. (in Chinese)
[10] 崔承齐, 刘艳阳, 江晓林, 孙知雨, 杜振伟, 武轲, 梅鸿献, 郑永战. 芝麻产量相关性状的多位点全基因组关联分析及候选基因预测. 中国农业科学, 2022, 55(1): 219-232.doi: 10.3864/j.issn.0578-1752. 2022.01.018.
CUI C Q, LIU Y Y, JIANG X L, SUN Z Y, DU Z W, WU K, MEI H X, ZHENG Y Z. Multi-locus genome-wide association analysis of yield-related traits and candidate gene prediction in sesame(L.). Scientia Agricultura Sinica, 2022, 55(1): 219-232. doi: 10.3864/j.issn.0578-1752.2022.01.018. (in Chinese)
[11] GUO D L, ZHAO H L, LI Q, ZHANG G H, JIANG J F, LIU C H, YU Y H. Genome-wide association study of berry-related traits in grape [L.]based on genotyping-by-sequencing markers. Horticulture Research, 2019, 6: 11.
[12] FLUTRE T, LE CUNFF L, FODOR A, LAUNAY A, ROMIEU C, BERGER G, BERTRAND Y, TERRIER N, BECCAVIN I, BOUCKENOOGHE V, ROQUES M, PINASSEAU L, VERBAERE A, SOMMERER N, CHEYNIER V, BACILIERI R, BOURSIQUOT J M, LACOMBE T, LAUCOU V, THIS P, PÉROS J P, DOLIGEZ A. A genome-wide association and prediction study in grapevine deciphers the genetic architecture of multiple traits and identifies genes under many new QTLs. G3: Genes|Genomes|Genetics, 2022, 12(7): jkac103.
[13] ZHANG H, FAN X C, ZHANG Y, JIANG J F, LIU C H. Identification of favorable SNP alleles and candidate genes for seedlessness inL. using genome-wide association mapping. Euphytica, 2017, 213(7): 136.
[14] 刘崇怀, 沈育杰, 陈俊. 葡萄种质资源描述规范和数据标准. 北京: 中国农业出版社, 2006.
LIU C H, SHEN Y J, CHEN J. Descriptors and data standard for grape (L.). Beijing: China Agriculture Press, 2006. (in Chinese)
[15] ALEXANDER D H, NOVEMBRE J, LANGE K. Fast model-based estimation of ancestry in unrelated individuals. Genome Research, 2009, 19(9): 1655-1664.
[16] ZHANG C, DONG S S, XU J Y, HE W M, YANG T L. PopLDdecay: A fast and effective tool for linkage disequilibrium decay analysis based on variant call format files. Bioinformatics, 2019, 35(10): 1786-1788.
[17] ZHOU X, STEPHENS M. Genome-wide efficient mixed-model analysis for association studies. Nature Genetics, 2012, 44(7): 821-824.
[18] SONG X J, HUANG W, SHI M, ZHU M Z, LIN H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase. Nature Genetics, 2007, 39(5): 623-630.
[19] GU C, ZHOU Y H, SHU W S, CHENG H Y, WANG L, HAN Y P, ZHANG Y Y, YU M L, JOLDERSMA D, ZHANG S L. RNA-Seq analysis unveils gene regulation of fruit size cooperatively determined by velocity and duration of fruit swelling in peach. Physiologia Plantarum, 2018, 164(3): 320-336.
[20] YUSTE-LISBONA F J, FERNÁNDEZ-LOZANO A, PINEDA B, BRETONES S, ORTÍZ-ATIENZA A, GARCÍA-SOGO B, MÜLLER N A, ANGOSTO T, CAPEL J, MORENO V, JIMÉNEZ-GÓMEZ J M, LOZANO R. ENO regulates tomato fruit size through the floral meristem development network. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(14): 8187-8195.
[21] UPADHYAY A, MASKE S, JOGAIAH S, KADOO N Y, GUPTA V S. GA3 application in grapes (L.) modulates different sets of genes at cluster emergence, full bloom, and berry stage as revealed by RNA sequence-based transcriptome analysis. Functional & Integrative Genomics, 2018, 18(4): 439-455.
[22] SU L Y, BASSA C, AUDRAN C, MILA I, CHENICLET C, CHEVALIER C, BOUZAYEN M, ROUSTAN J P, CHERVIN C. The auxin sl-IAA17 transcriptional repressor controls fruit size via the regulation of endoreduplication-related cell expansion. Plant and Cell Physiology, 2014, 55(11): 1969-1976.
[23] MORI K, LEMAIRE-CHAMLEY M, ASAMIZU E, MIZOGUCHI T, EZURA H, ROTHAN C. Comparative analysis of common genes involved in early fruit development in tomato and grape. Plant Biotechnology, 2013, 30(3): 295-300.
[24] WANG H B, WU T, LIU J L, CONG L, ZHU Y F, ZHAI R, YANG C Q, WANG Z G, MA F W, XU L F.regulates fruit set and induces parthenocarpy by enhancing GA4content. Frontiers in Plant Science, 2020, 11: 113.
[25] IRELAND H S, YAO J L, TOMES S, SUTHERLAND P W, NIEUWENHUIZEN N, GUNASEELAN K, WINZ R A, DAVID K M, SCHAFFER R J. Apple SEPALLATA1/2-like genes control fruit flesh development and ripening. The Plant Journal, 2013, 73(6): 1044-1056.
[26] ROYO C, TORRES-PÉREZ R, MAURI N, DIESTRO N, CABEZAS J A, MARCHAL C, LACOMBE T, IBÁÑEZ J, TORNEL M, CARREÑO J, MARTÍNEZ-ZAPATER J M, CARBONELL-BEJERANO P. The major origin of seedless grapes is associated with a missense mutation in the MADS-box gene. Plant Physiology, 2018, 177(3): 1234-1253.
[27] FRIEND A P, TROUGHT M C T, CREASY G L. The influence of seed weight on the development and growth of berries and live green ovaries inL. cvs. Pinot Noir and Cabernet Sauvignon. Australian Journal of Grape and Wine Research, 2009, 15(2): 166-174.
[28] WALKER R R, BLACKMORE D H, CLINGELEFFER P R, KERRIDGE G H, RÜHL E H, NICHOLAS P R. Shiraz berry size in relation to seed number and implications for juice and wine composition. Australian Journal of Grape and Wine Research, 2005, 11(1): 2-8.
[29] EBADI A, MOGHADAM J E, FATAHI R. Evaluation of 22 populations achieved from controlled crossing between some seeded × seedless grapevine cultivars. Scientia Horticulturae, 2009, 119(4): 371-376.
[30] FISCHER B M, SALAKHUTDINOV I, AKKURT M, EIBACH R, EDWARDS K J, TÖPFER R, ZYPRIAN E M. Quantitative trait locus analysis of fungal disease resistance factors on a molecular map of grapevine. Theoretical and Applied Genetics, 2004, 108(3): 501-515.
[31] RICHTER R, GABRIEL D, RIST F, TÖPFER R, ZYPRIAN E. Identification of co-located QTLs and genomic regions affecting grapevine cluster architecture. Theoretical and Applied Genetics, 2019, 132(4): 1159-1177.
[32] ZINELABIDINE L H, TORRES-PÉREZ R, GRIMPLET J, BAROJA E, IBÁÑEZ S, CARBONELL-BEJERANO P, MARTÍNEZ-ZAPATER J M, IBÁÑEZ J, TELLO J. Genetic variation and association analyses identify genes linked to fruit set-related traits in grapevine. Plant Science, 2021, 306: 110875.
[33] VANDENBUSSCHE F, VASEVA I, VISSENBERG K, VAN DER STRAETEN D. Ethylene in vegetative development: a tale with a riddle. The New Phytologist, 2012, 194(4): 895-909.
[34] 张文颖, 王晨, 朱旭东, 马超, 王文然, 冷翔鹏, 郑婷, 房经贵. 葡萄全基因组DELLA蛋白基因家族鉴定及其应答外源赤霉素调控葡萄果实发育的特征. 中国农业科学, 2018, 51(16): 3130-3146. doi: 10.3864/j.issn.0578-1752.2018.16.009.
ZHANG W Y, WANG C, ZHU X D, MA C, WANG W R, LENG X P, ZHENG T, FANG J G. Genome-wide identification and expression of DELLA protein gene family during the development of grape berry induced by exogenous GA. Scientia Agricultura Sinica, 2018, 51(16): 3130-3146. doi: 10.3864/j.issn.0578-1752.2018.16.009. (in Chinese)
[35] WU J, ZHONG J H, XU K, WEI Q P, WEI Z L. Effects of exogenous GA3 on fruit development and endogenous hormones in fujiminori grape. Journal of Fruit Science, 2001, 4: 209-212.
[36] ACHEAMPONG A K, HU J H, ROTMAN A, ZHENG C L, HALALY T, TAKEBAYASHI Y, JIKUMARU Y, KAMIYA Y, LICHTER A, SUN T P, OR E. Functional characterization and developmental expression profiling of gibberellin signalling components in. Journal of Experimental Botany, 2015, 66(5): 1463-1476.
[37] 袁华招, 赵密珍, 吴伟民, 于红梅, 钱亚明, 王壮伟, 王西成. 葡萄生长素响应基因家族生物信息学鉴定和表达分析. 遗传, 2015, 37(7): 720-730.
YUAN H Z, ZHAO M Z, WU W M, YU H M, QIAN Y M, WANG Z W, WANG X C. Genome-wide identification and expression analysis of auxin-related gene families in grape. Hereditas, 2015, 37(7): 720-730. (in Chinese)
[38] RIECHMANN J L, HEARD J, MARTIN G, REUBER L, JIANG C Z, KEDDIE J, ADAM L, PINEDA O, RATCLIFFE O J, SAMAHA R R, CREELMAN R, PILGRIM M, BROUN P, ZHANG J Z, GHANDEHARI D, SHERMAN B K, YU G.transcriptionfactors: genome-wide comparative analysis among eukaryotes. Science, 2000, 290(5499): 2105-2110.
[39] MAKKENA S, LAMB R S. The bHLH transcription factorregulates root growth by controlling the size of the root meristem. BMC Plant Biology, 2013, 13: 1.
[40] MAHJOUB A, HERNOULD M, JOUBÈS J, DECENDIT A, MARS M, BARRIEU F, HAMDI S, DELROT S. Overexpression of a grapevine R2R3-MYB factor in tomato affects vegetative development, flower morphology and flavonoid and terpenoid metabolism. Plant Physiology and Biochemistry, 2009, 47(7): 551-561.
[41] TYAGI S, MIR R R, KAUR H, CHHUNEJA P, RAMESH B, BALYAN H S, GUPTA P K. Marker-assisted pyramiding of eight QTLs/genes for seven different traits in common wheat (L.). Molecular Breeding, 2014, 34: 167-175.
Genome-Wide Association Studies for Grape Berry Weight Related Traits

1Institute of Forestry and Pomology, Beijing Academy of Agricultural and Forestry Sciences, Beijing 100093;2Beijing Engineering Research Center for Deciduous Fruit Trees, Beijing 100093;3Key Laboratory of Biology and Genetic Improvement of Horticultural Crops (North China), Ministry of Agriculture and Rural Affairs, Beijing 100093
【Objective】Grape berry size is one of important factors affecting grape appearance and the final productivity. It is a complex quantitative trait regulated by multiple genes. Mining the key genetic regulatory loci and the underlying genes for berry size related traits would help to improve grape yield. 【Method】In this study, 150 diverse grapevine varieties were selected as materials. The berry weight, seed number per berry, and seed weight were measured in 2019 and 2020, respectively. Based on high-density genotype data obtained by resequencing, the genome-wide association studies (GWAS) were carried out to detect significantly associated SNPs and to predict important candidate genes.【Result】The three measured traits exhibited extensive phenotypic variation with 39.55%-68.89% of phenotypic variation coefficients; the phenotypic distribution of the observed three traits in the population showed continuous quantitative genetic characteristics; a significant positive correlation between each trait were observed in two years; based on the phenotypic data collected in two years, a total of 150 significant SNPs were detected for berry weight. In 2019, 99 SNPs were detected, each of which contributed the phenotypic variation from 14.48% to 25.59%; in 2020, 73 SNPs were detected, explaining 16.08%-26.83% of phenotypic variation;among these SNPs, 24 were detected repeatedly in both two years, mainly located on chromosome 1, 5, 11 and 16. Compared with the trait of berry weight, less SNPs significantly associated with the seed number were detected. A significant SNP was detected in 2019, and the phenotypic explanation value was 24.29%; in 2020, 17 significant SNPs were detected, which all located on chromosome 18; 1 and 2 SNPs located on chromosome 18 significantly associated with seed weight were detected in 2019 and 2020, respectively, accounting for 23.59%-48.29% of phenotypic variation. Within the genomic region of SNPs detected repeatedly for two years, 11 candidate genes related to berry weight were screened out based on the functional annotation, including ethylene signal pathway genes (VIT_05s0049g00490, VIT_05s0049g00500, VIT_05s0049g00510 and VIT_16s0100g00400), gibberellin signal pathway genes (VIT_11s0016g04630 and VIT_16s0022g02310), auxin responsive protein gene (VIT_11s0016g05640) and some important transcription factor genes (VIT_05s0049g00460, VIT_11s0016g05660 and VIT_16s0022g02330). A candidate gene VI(encoding a MADS box proteinAGL11) associated with seed content was identified on chromosome 18, and different SNP genotypes on this gene significantly affected the grape berry seed number and weight. 【Conclusion】A total of 150 SNPssignificantly associated with berry weight were detected in two years, mainly located on chromosomes 1, 5, 11 and 16; A total of 19 significant SNPs associated with seed content were detected, mainly located on chromosome 18. Based on the results of gene annotation and genotype analysis, 11 candidate genes that might be involved in the regulation of grape berry weight including VIT_11s0016g04630 and VIT_16s0022g02310 were selected; the candidate gene VIT_18s0041g01880 was determined significantly correlated with seed content.
grape; berry size; genome-wide association studies; candidate gene
10.3864/j.issn.0578-1752.2023.08.011
2022-06-08;
2022-08-24
北京市农林科学院创新能力建设专项(KJCX20200406)、现代农业产业技术体系建设专项(CARS-29)、河北省重点研发计划(21326310D)
王慧玲,E-mail:wanghui198216@126.com。通信作者徐海英,E-mail:haiyingxu63@sina.com。通信作者孙磊,Tel:010-82592156;E-mail:sunlei.bjfu@gmail.com
(责任编辑 赵伶俐)

