基于55K SNP芯片揭示小麦育种亲本遗传多样性
卢茂昂 彭小爱 张 玲 汪建来 何贤芳,,* 朱玉磊,*
研究简报
基于55K SNP芯片揭示小麦育种亲本遗传多样性
卢茂昂2彭小爱2张 玲2汪建来1何贤芳1,2,*朱玉磊2,*
1安徽省农业科学院作物研究所, 安徽合肥 230001;2安徽农业大学农学院, 安徽合肥 230036
为了解不同省份小麦亲本材料间的遗传多样性, 以150份分布于安徽、江苏、河南、四川及山东等省份小麦种质资源为试验材料, 利用小麦55K SNP芯片对其进行遗传多样性分析、聚类分析、主成分分析及群体结构分析。结果表明, 在150份小麦材料中共检测到52,537个SNP位点, 质控后共获得39,422个有效标记, 其中多态性标记为38,135个, 占有效标记数96.74%。多态性标记在亚基因组间分布呈现D (10,450)四川省>山东省>江苏省>安徽省; 聚类分析、主成分分析和群体结构分析结果高度一致, 分群结果与血缘关系、区域来源及育成单位均较为吻合。本研究表明各省份平均多态性信息含量处于中度多态水平, 但材料平均遗传距离较为接近, 仍需引入优质种质资源, 缓解材料同质化情况, 增加小麦应对逆境胁迫能力, 减轻小麦实际生产中的脆弱性及风险性。
小麦; 55K SNP芯片; 育种亲本; 遗传多样性; 群体结构分析
遗传多样性是作物改良目标性状的物质基础[1], 为小麦育种的亲本选配提供理论支撑[2], 也是种质资源研究的热点之一[3]。地方材料将目标性状持续稳定的遗传给下一代需要长时间持续改良[4], 因此育种家更偏爱经过改良的优良品种[5], 以提高产量[6]和缩短育种周期[4]。但育种目标的定向选择降低了栽培品种之间的遗传多样性, 缩小了可用于未来育种研究的种质基础[7], 大量育种家针对小麦种质资源遗传多样性开展研究, 曹廷杰等[8]剖析河南省2000—2013年新审定的95份小麦品种的亲缘关系、李珊珊等[9]以小麦90K SNP芯片对143份河北省推广小麦品种的遗传多样性进行分析以及白彦明等[10]对我国北方小麦品种的原始骨干亲本蚂蚱麦与小白麦衍生系的遗传多样性分析, 结果均表明小麦新品种遗传距离不够丰富, 遗传多样性较低, 需引入新的种质资源, 增加优异基因扩宽遗传基础。因此了解我国小麦大省的种质资源的遗传多样性,对我国粮食安全有着极其重要的意义。
第三代分子标记单核苷酸(single nucleotide polymorphism, SNP)因其相比AFLP[11]和SSR[12]等传统标记, 具有遗传稳定、特异性高和位点丰富等特点, 广泛的存在于所有的动植物中[13]。SNP标记已经广泛的应用在作物全基因组关联分析(genome-wide association study, GWAS)[14]、遗传多样性分析[15]、作物分子身份证构建[16]和群体结构分析[17]等方面。Mourad等[18]对270份冬小麦材料采用基因分型测序, 鉴定出35,000个SNP, 在2D染色体上发现32个SNP标记与耐药性显著性相关; Alemu等[19]在埃塞俄比亚的215个地方材料中发现11,919个SNP, 遗传多样性指数为0.7, 表明其遗传多样性丰富, 在面对生物和非生物胁迫时, 地方材料可以作为重要种质资源; 刘彬等[20]编写perl脚本对251份藜麦材料随机抽取SNP组合, 构建个体分子身份证, 在藜麦种质的溯源和保护起到积极作用; 韩志刚等[21]以148份马铃薯和SNP分子标记进行群体结构分析, 表明148份材料可以分为6个群组, 其中华北平原和国外材料遗传分组相对单一, 需引入新资源, 拓宽遗传背景。
现已相继开发出9K、50K、55K和660K等小麦SNP芯片, 其中55K SNP芯片是中国农业科学院作物科学研究所基于660K SNP芯片结合一千多份本土材料开发出来,其有效标记多态性在不同基因组和同源群的分布优于90K SNP芯片[8], 且前人[6]已证实55K SNP芯片更适宜国内小麦种质材料的研究, 已被广泛应用于小麦抗病[22]、生殖发育[23]及籽粒形态[24]等方面的研究。本研究以150份小麦育种亲本材料为试验材料, 对其进行55K SNP芯片扫描, 意在揭示我国安徽省、江苏省、四川省、山东省和河南省6个小麦大省优质育种亲本遗传多样性和亲缘关系, 以期为我国小麦种质资源保护、亲本选配、遗传研究以及种质资源创新提供理论依据。
1 材料与方法
1.1 供试材料
供试材料150份(附表1), 均由安徽省农业科学院作物研究所提供, 编号从y1到y150, 包括安徽省67份、江苏省43份、四川省12份、山东省11份、河南省11份及其他省份地区6份。2020年10月18日将试验材料秋播于安徽省农业科学院阜南。每份材料种植5行, 行长2 m, 行距25 cm。
1.2 SNP芯片分析
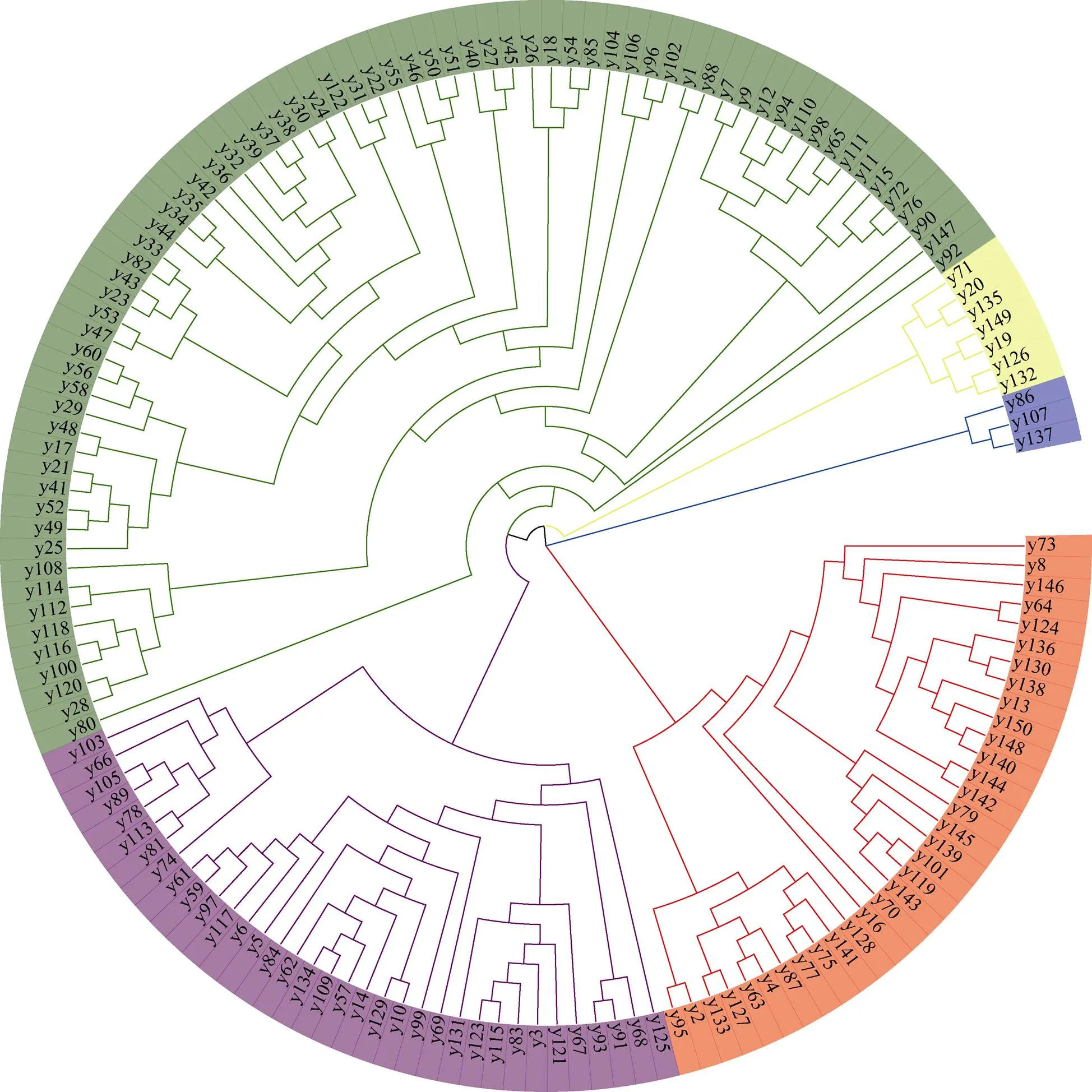
采用SDS法提取籽粒DNA[12], 利用Affymetrix Axiom 55K芯片数据(北京博奥晶典生物技术有限公司)对150份小麦材料进行全基因组扫描, 采用Illumina’s Genome Studio Software进行样本的原始SNP分型。过滤缺失率超过20%, 最小等位基因频率(minor allele frequency, MAF)低于5%的标记, 高质量SNP标记, 用于后续分析。利用Powermarker V3.25 (https://en.freedownloadmanager. org/)软件计算SNP标记的最小等位基因频率(minor allele frequency, MAF)、基因多样性(gene diversity, GD)和多态性信息含量(polymorphism information content, PIC), 采用TASSEL 5.0 (https://tassel.bitbucket.io/)计算材料间遗传距离(identity-by-state, IBS)[25]。依据Botstein等[26]提出的低度多态(0 利用PLINK 1.9 (https://zzz.bwh.harvard.edu/plink)进行主成分分析, 采用STRUCTURE 2.3.4软件对材料进行群体结构分析, burn-in步长设置为10,000和MCMC迭代次数设置为100,000, 设定值为2~10, 对每个值分别进行5次独立重复, 当∆值达到最大时确定最佳类群数目[27]。 采用FastTree 2.1.8 (https://bio.tools/fasttree)最大似然法对其进行聚类分析[28], 并利用evolview (https://www. evolgenius/)对聚类结果进行在线美化。 在供试材料的55K SNP芯片基因型数据中共检测到52,537个SNP位点, 质控后共获得39,422个有效标记, 其中多态性标记为38,135个, 占有效标记数96.74% (图1-A)。多态性SNP位点在21条染色体中的分布数量在752~2478之间, 其中4B染色体最多, 4D染色体最少(图1-B)。A、B及D亚基因组多态性标记数量分别为12,365、15,290和10,450个, 占比分别为32.42%、40.09%及27.48% (图1-C)。除第4部分(4705)和第6部分(4807)同源群多态性SNP位点数量小于5000, 其余各同源群SNP多态性位点数量范围均在5000~6000之间, 分布较为均匀。多态性SNP位点数目在部分同源群的分布频率依次为4<6<1<7<2<3<5。 通过统计38,135个多态性标记发现, 上述SNP的平均最小等位基因频率为0.305, 变幅为0.036~0.500 (图2-A), 最小等位基因频率分布较为集中, 28,353 (74.25%)个多态性标记频率位于0.100~0.400之间; 平均基因多样性为0.399, 变幅为0.070~0.500 (图2-B), 各省份基因多样性最大值均为0.5; 平均多态信息含量为0.315, 变幅为0.068~0.375 (图2-C), 6459 (16.94%)个位点多态性信息含量小于0.25, 处于低度多态, 安徽省多态性信息含量极差最小, 为0.343。各省份中最小等位基因频率、基因多样性及多态性信息含量平均值最大均为安徽省, 分别为0.302、0.396及0.313; 最小均为山东省, 分别为0.237、0.317及0.253。 图1 多态性SNP标记在染色体(A)、同源群(B)和亚基因组(C)上的分布 图2 多态性SNP最小等位基因(A)、基因多样性(B)、多态性信息含量(C)及遗传距离(D)箱线图 为进一步研究两两材料之间的遗传距离, 本研究利用TASSEL 5.0计算材料之间的IBS值, IBS数值大小(0~1)表明基因型相似程度, 数值越趋向于0表明基因型相似程度越大, 共获得11,175个遗传距离值, 范围在0.071~0.531之间, 平均值为0.279 (图2-D)。供试群体间的IBS主要集中在0.2~0.4之间, 为10,101个, 占90.39%; 小于0.2的有1068个, 占9.56%; 大于0.4的最少, 只有6个, 占0.05%。其中18B151品系与扬辐麦7号和浩麦1号之间的遗传距离最小, 均为0.071; 轮选22与绵麦367之间的遗传距离最大为0.531。安徽省、江苏省和四川省供试材料IBS最小值低于0.10表明部分材料之间遗传距离较近, 遗传相似度过高。各省份供试材料遗传距离按从大到小排列顺序依次为: 河南省(0.298)>四川省(0.295)>山东省(0.293)>江苏省(0.284)>安徽省(0.275)。 应用最大似然法构建进化树, 结果表明(图3), 150个材料被划分为5个类群。类群I包含33份(22.00%)供试材料, 主要来自安徽省(11份)、江苏省(10份)及四川省(9份)。其中来自江苏的宁麦13和苏麦11距离最大, 为0.416;淮麦22与18B210的遗传距离较小, 为0.103, 推测和父本都源于江苏的扬麦158有关; 农麦126和苏麦11亲本中都包含宁麦9, 两者遗传距离(0.168)较低。类群II共包含35份(23.33%)供试材料, 主要源于安徽省(18份)和江苏省(11份)。类群II的苏麦10号与类群I的苏麦11双亲均为宁麦9号/扬麦11, 所以两者遗传距离较为接近, 为0.078; 其中皖西麦0638和扬麦22均包含扬麦9号, 两者的遗传距离较为接近, 为0.128。接近一半(48.00%)的优质材料都被划分到类群III, 主要包含安徽省35份、江苏省18份以及山东省11份。材料间的遗传距离在0.073~0.418之间, 平均为0.293。镇麦9号和18B141的遗传距离最大, 为0.418, 推测因为苏麦6号是镇麦9号的母本, 其血统较为复杂, 包含中国安徽(安徽11)、意大利(毛颖阿夫)、中国陕西(丰产3号)和中国江苏(扬麦5号)等血统, 故两者遗传距离较远。类群IV共包含中国江苏省3份、中国安徽省2份、中国四川省以及以色列各1份, 共7份(4.67%)供试材料。此类群亲本来源较为广泛, 未发现材料间有相同的直系亲本。类群V的3份(2.00%)材料来源各不相同, 分别来自中国安徽、江苏以及四川。 图3 供试材料聚类分析图 利用PLINK软件对150份小麦育种亲本材料进行主成分分析, 由图4-A可知前两个主成分分别解释28.26%和19.38%的遗传变异。如图4-B所示, 当=3时Δ最大, 因此将150份材料分为3个亚群(图4-C)。a、亚群III和类群II均主要为安徽及江苏供试材料; 安徽、江苏和四川供试材料占b、亚群I和类群I的绝大部分; c、亚群II和III类群均是以安徽、江苏、山东为主体的群体。聚类分析、主成分分析与群体结构分析结果较为相似, 增加群体结构划分的合理性。 图4 供试材料PCA分析(A)和群体遗传结构分析(B和C) 保护种质资源的可持续利用和遗传多样性是应对反复发生的气候变化和广泛发生的生物胁迫不可替代的手段[19], 同时遗传多样性和种群结构在挖掘优良性状和推进品种商业化上起着及其重要的作用[29]。本研究以安徽、江苏、四川、河南及山东等省份的150份小麦材料作为试验材料, 对其进行小麦55K SNP芯片扫描, 获得38,135个高质量SNP, 其中B组染色体(15,290)最多, D组染色体最少(10,450), 与前人研究一致[30]。普通小麦中A和B基因组存在的时间较长, 经历了更多的基因复制和重组, 积累了更多的突变, D组存在的时间较短, 基因复制和重组较少, 基因编码区较为稳定[31]。除此之外, 大量的早期基因流动可能发生在及其四倍体祖体(AABB)之间, 但不发生在六倍体和(DD)之间[32], 也可能是A、B基因组遗传多样性大于D组的原因之一。 本研究利用小麦55K SNP芯片对150份供试材料进行基因分型, 发现供试材料SNP分子标记平均多态性信息含量为0.315, 处于中度多态, 与刘易科等[2]研究相同。本研究发现河南(0.287)、江苏(0.305)及山东(0.254)供试材料多态性信息含量均小于前人研究[8,33-34]。以上差异究其原因可能为, 前人试验材料数目更多、遗传背景更为广泛、育种过程中长期对某一性状的定向改良[11]以及SNP标记受双等位基因先决条件限制且突变率较为缓慢[35]。 在早期生产水平较为低下时, 长期人工和自然选择造成育成材料等位基因趋于同化, 遗传距离下降[6]。郝晨阳等[36]研究发现我国20世纪50至90年代间的材料平均遗传距离随时间的增加而逐渐减小, 变幅为0.727~0.689, 下降幅度为0.038。同时, 我国小麦新材料选育过程中, 大多围绕大面积推广适应性强的材料为核心进行选育[10]。本研究150份供试材料中35份材料直系亲本来自扬麦系列, 占比23.33%。67份安徽省供试材料中, 7份材料直系亲本为烟农19, 占比为10.45%; 江苏省43份供试材中, 6份材料直系亲本为扬麦11, 占比13.95%; 4份材料直系亲本为宁麦9号, 占比9.30%; 3份来自扬麦9号, 占比6.98%。江苏省供试材料遗传距离较近, 可能是因为其43份供试材料中有25份直系亲本为扬麦系列, 占比58.14%。表明育种材料趋于相同, 且同一育种单位或省份选育新材料时优先考虑现有品系及传统优异材料, 导致小麦材料遗传多样性降低, 降低抗风险能力。同时本研究发现部分材料IBS较近, 但直系亲本并无血缘关系, 原因可能系谱记载过程中并不准确[8]。 聚类分析将150份供试材料分为5个类群, 类群I主要是以安徽、江苏及四川材料为主体, 与主成分分析及群体结构分析结果相同; 类群II的35份材料亲本主要来自淮麦系及扬麦系, 验证前人来自相同地区材料优先聚为一类[2]; 近一半的供试材料被划分到类群III, 72份材料主要来自安徽、江苏及山东, 占比88.87%, 前人研究表明苏北育种家使用大量山东亲本, 皖北地区育种家大量选择山东及河南资源[12], 在类群III亲本谱系图中也有所体现, 丰华8829亲本之一为郑州8329、亿麦9号亲本为郑麦9023及18B187亲本为郑麦112均为河南优质材料, 这可能是54.55%河南材料被划分到类群III的原因; 共7份材料被划分到类群IV, 此类群亲本来源较为广泛, 并未发现直系亲本有所关联; 类群V材料最少, 为3份, 其中18B130亲本辐照06725经过辐射处理发生了某些变化, 与亲本均含四川资源的川麦42和扬麦15聚为一类。 附表 请见网络版: 1) 本刊网站http://zwxb.chinacrops. org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http:// c.wanfangdata.com.cn/Periodicalzuowxb.aspx。 [1] Boakyewaa A G, Badu-Apraku B, Akromah R, Garcia-Oliveira A L, Awuku F J, Gedil M. Genetic diversity and population structure of early-maturing tropical maize inbred lines using SNP markers., 2019, 14: e0214810. [2] 刘易科, 朱展望, 陈泠, 邹娟, 佟汉文, 朱光, 何伟杰, 张宇庆,高春保. 基于SNP标记揭示我国小麦品种(系)的遗传多样性. 作物学报, 2020, 46: 307–314. Liu Y K, Zhu Z W, Chen L, Zou J, Tong H W, Zhu G, He W J, Zhang Y Q, Gao C B. Revealing the genetic diversity of wheat varieties (lines) in China based on SNP markers., 2020, 46: 307–314 (in Chinese with English abstract). [3] Wu X, Li Y, Shi Y, Song Y, Wang T, Huang Y, Li Y. Fine genetic characterization of elite maize germplasm using high-throughput SNP genotyping., 2014, 127: 621–631. [4] Scherlosky A, Marchioro V S, Franco F D A, Braccini A L, Schuster L. Genetic variability of Brazilian wheat germplasm obtained by high-density SNP genotyping., 2018, 18: 399–408. [5] Uddin M S, Boerner A. Genetic diversity in hexaploid and tetraploid wheat genotypes using microsatellite markers., 2009, 18: 65–73. [6] 马艳明, 娄鸿耀, 陈朝燕, 肖菁, 徐麟, 倪中福, 刘杰. 新疆冬小麦品种农艺及产量性状遗传多样性分析. 作物学报, 2020, 46: 1539–1556. Ma Y M, Lou H Y, Chen Z Y, Xiao J, Xu L, Ni Z F, Liu J. Genetic diversity analysis of winter wheat landraces and modern bred varieties in Xinjiang based on agronomic traits., 2020, 46: 1539–1556 (in Chinese with English abstract). [7] Reif J C, Zhang P, Dreisigacker S, Warburton M L, Ginkel M V, Hoisington D, Bohn M, Melchinger A E. Wheat genetic diversity trends during domestication and breeding., 2005, 110: 859–864. [8] 曹廷杰, 谢菁忠, 吴秋红, 陈永兴, 王振忠, 赵虹, 王西成, 詹克慧, 徐如强, 王际睿, 罗明成, 刘志勇. 河南省近年审定小麦材料基于 系谱和SNP标记的遗传多样性分析. 作物学报, 2015, 41: 197–206. Cao T J, Xie Q Z, Wu Q H, Chen Y X, Wang Z Z, Zhao H, Wang X C, Zhan K H, Xu R Q, Wang J R, Luo M C, Liu Z Y. Genetic diversity of registered wheat varieties in Henan Province based on pedigree and single-nucleotide polymorphism., 2015, 41: 197–206 (in Chinese with English abstract). [9] 李珊珊, 易腾飞, 徐渴, 张树华, 赵勇, 杨学举. 河北省小麦品种基于SNP标记的遗传多样性分析. 分子植物育种, 2019, 17: 6850–6859. Li S S, Yi T F, Xu K, Zhang S H, Zhao Y, Yang X J. Genetic diversity of wheat varieties in Hebei Province based on single nucleotidepolymorphism., 2019, 17: 6850–6859 (in Chinese with English abstract). [10] 白彦明, 李龙, 王绘艳, 柳玉平, 王景一, 毛新国, 昌小平, 孙黛珍, 景蕊莲. 蚂蚱麦和小白麦衍生系的遗传多样性分析. 作物学报, 2019, 45: 1468–1477. Bai Y M, Li L, Wang H Y, Liu Y P, Wang J Y, Mao X G, Chang X P, Sun D Z, Jing R L. Genetic diversity assessment in derivative offspring of Mazhamai and Xiaobaimai wheat., 2019, 45: 1468–1477 (in Chinese with English abstract). [11] Arabi M, Shoaib A, Al-Shehadah E, Jawhar M. Genetic diversity within local and introduced cultivars of wheat (L) grown under Mediterranean environment as revealed by AFLP markers., 2019, 63: 25–30. [12] 王升星, 朱玉磊, 张海萍, 常成, 马传喜. 小麦育种亲本材料SSR标记遗传多样性及其亲缘关系分析. 麦类作物学报, 2014, 34: 621–627. Wang S X, Zhu Y L, Zhang H P, Chang C, Ma C X. Analysis of genetic diversity and relationship among wheat breeding parents by SSR markers., 2014, 34: 621–627 (in Chinese with English abstract). [13] Ganal M W, Altmann T, Rder M S. SNP identification in crop plants., 2009, 12: 211–217. [14] Eltaher S, Baenziger P S, Belamkar V, Emara H A, Sallam A. GWAS revealed effect of genotype × environment interactions for grain yield of Nebraska winter wheat., 2021, 22: 2. [15] Bonman J M, Babiker E M, Cuesta-Marcos A, Esvelt-Klos K, Brown-Guedira, Gina, Chao S, See D, Chen J, Akhunov E, Zhang J. Genetic diversity among wheat accessions from the USDA national small grains collection., 2015, 55: 1243–1253. [16] 樊晓静, 于文涛, 蔡春平, 林浥, 王泽涵, 房婉萍, 张见明, 叶乃兴. 利用SNP标记构建茶树品种资源分子身份证. 中国农业科学, 2021, 54: 1751–1772. Fan X J, Yu W T, Cai C P, Lin Y, Wang Z H, Fang W P, Zhang J M, Ye N X. Construction of molecular ID for tea cultivars by using of single-nucleotide polymorphism (SNP) markers., 2021, 54: 1751–1772 (in Chinese with English abstract). [17] Yang X, Tan B, Liu H, Zhu W, Xu L, Wang Y, Fan X, Sha L, Zhang H, Zeng J, Wu D, Jiang Y, Hu X, Chen G, Zhou Y, Kang H. Genetic diversity and population structure of Asian and European common wheat accessions based on genotyping-by-sequencing., 2020, 11: 1157. [18] Mourad A, Ahmed S, Vikas B, Stephen W, Robert B, Jin Y, Ezzat M, Bahy B, El-Wafaa A A, Jesse P. Genome-wide association study for identification and validation of novel SNP markers for Sr6 stem rust resistance gene in bread wheat., 2018, 9: 380. [19] Alemu A, Feyissa T, Maccaferri M, Sciara G, Tuberosa R, Ammar K, Badebo A, Acevedo M, Letta T, Abeyo B. Genome-wide association analysis unveils novel QTLs for seminal root system architecture traits in Ethiopian durum wheat., 2021, 22: 20. [20] 刘彬, 赵雨露, 杨鑫雷, 张建恒, 孙鑫博, 刘晓清, 温晓敏, 耿艳楼, 李悦有, 吕玮, 穆国俊. 251份藜麦种质资源遗传多样性及分子身份证构建. 植物遗传资源学报, 2022, 23: 706–721. Liu B, Zhao Y L, Yang X L, Zhang J H, Sun X B, Liu X Q, Wen X M, Geng Y L, Li Y Y, Lyu W, Mu G J. Genetic diversity of 251 germplasm accessions and construction of molecular ID in quinoa (Willd).,2022, 23: 706–721 (in Chinese with English abstract). [21] 韩志刚, 郝文胜, 谢锐, 郭景山, 伊六喜, 侯建华. 基于全基因组重测序SNP标记的148份马铃薯种质遗传多样性分析. 西北植物学报, 2021, 41: 1302–1314. Han Z G, Hao W S, Xie R, Guo J S, Yi L X, Hou J H. Analysis of genetic diversity of 148 potato germplasm based on SNP markers from whole genome resequencing., 2021, 41: 130–1314 (in Chinese with English abstract). [22] Ye X, Li J, Cheng Y, Yao F, Chen G. Genome-wide association study reveals new loci for yield-related traits in Sichuan wheat germplasm under stripe rust stress., 2019, 20: 640. [23] Tran F, Penniket C, Patel R V, Provart N J, Laroche A, Rowland O, Robert L S. Developmental transcriptional profiling reveals key insightsinto Triticeae reproductive development., 2013, 74: 971–988. [24] Qu X, Liu J, Xie X, Xu Q, Ma J. Genetic mapping and validation of loci for kernel-related traits in wheat (L.)., 2021, 12: 667493. [25] 肖颖妮, 于永涛, 谢利华, 祁喜涛, 李春艳, 文天祥, 李高科, 胡建广. 基于SNP标记揭示中国鲜食玉米品种的遗传多样性. 作物学报, 2022, 48: 1301–1311. Xiao Y N, Yu W T, Xie L H, Qi X T, Li C Y, Wen T X, Li G K, Hu J G. Genetic diversity analysis of Chinese fresh corn hybrids using SNP chips., 2022, 48: 1301–1311 (in Chinese with English abstract). [26] Botstein D, White R L, Skolnick M, Davis R W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms., 1980, 32: 314. [27] 严勇亮, 时晓磊, 张金波, 耿洪伟, 肖菁, 路子峰, 倪中福, 丛花. 春小麦籽粒主要品质性状的全基因组关联分析. 中国农业科学, 2021, 54: 4033–4047. Yan Y L, Shi X L, Zhang J B, Xiao J, Lu Z F, Ni Z F, Cong H. Genome-wide association study of grain quality related characteristics of spring wheat., 2021, 54: 4033–4047 (in Chinese with English abstract). [28] 张亚丽, 韩志刚, 吴昊, 夏云. 应用FastTree2.1.8快速构建HIV-1 pol区基因的最大似然系统进化树. 中国艾滋病性病, 2017, 23: 695–699. Zhang Y L, Han Z G, Wu H, Xia Y. Rapid construction of maximum likelihood tree of HIV-1 pol gene by FastTree2.1.8., 2017, 23: 695–699 (in Chinese with English abstract). [29] Dhillon G S, Singh D, Singh R P, Poland J, Tomar V. Distributed under creative commons CC-BY 4.0 Elucidating SNP-based genetic diversity and population structure of advanced breeding lines of bread wheat (L)., 2021, 9: e11593. [30] Shamseldeen E, Ahmed S, Vikas B, Emara H A,Nower A A,Salem K F M, Jesse P, Baenziger P S. Genetic diversity and population structure of F3:6 Nebraska winter wheat genotypes using genotyping-by-sequencing., 2018, 9: 76. [31] Talbert L E, Smith L Y, Blake K. More than one origin of hexaploid wheat is indicated by sequence comparison of low-copy DNA., 1998, 41: 402–407. [32] Caldwell K S, Dvorak J, Lagudah E S, Akhunov E, Luo M C, Wolters P, Petra W, Wayne P. Sequence polymorphism in polyploid wheat and their d-genome diploid ancestor., 2004, 167: 941–947. [33] 喻俊杰, 金艳, 张勇, 徐辰武. 江苏主栽小麦品种遗传多样性的SSR分析. 麦类作物学报, 2015, 35: 1372–1377. Yu J J, Jin Y, Zhang Y, Xu C W. Analysis on genetic diversity of Jiangsu wheat cultivars using SSR markers., 2015, 35: 1372–1377 (in Chinese with English abstract). [34] 彭芹, 戴双, 郭骞欢, 程敦公, 李豪圣, 刘爱峰, 刘建军, 赵世杰, 宋健民. 1950年以来山东省主推小麦品种的遗传多样性演变. 分子植物育种, 2012, 10: 228–237. Peng Q, Dai S, Guo Q H, Cheng D G, Li H S, Liu A F, Liu J J, Zhao S J, Song J M. The evolution of genetic diversity of wheat varieties released in Shandong province since 1950., 2012, 10: 228–237 (in Chinese with English abstract). [35] Frascaroli E, Schrag T A, Melchinger A E. Genetic diversity analysis of elite European maize (L.) inbred lines using AFLP, SSR, and SNP markers reveals ascertainment bias for a subset of SNPs., 2013, 126: 133–141. [36] 郝晨阳, 王兰芬, 张学勇, 游光霞, 董玉琛, 贾继增, 刘旭, 尚勋武, 刘三才, 曹永生. 我国五十年来育成小麦品种的遗传多样性演变. 中国科学: C辑, 2005, 35: 408–415.Hao C Y, Wang L F, Zhang X Y, You G X, Dong Y C, Jia J Z, Liu X, Shang X W, Liu S C, Cao Y S. Genetic diversity changes of Chinese cultivars in the past 50 years.(Ser C), 2005, 35: 408–415 (in Chinese with English abstract). Genetic diversity of wheat breeding parents revealed by 55K SNP-based microarray LU Mao-Ang2, PENG Xiao-Ai2, ZHANG Ling2, WANG Jian-Lai1, HE Xian-Fang1,2,*, and ZHU Yu-Lei2,* 1Crop Research Institute, Anhui Academy of Agricultural Sciences, Hefei 230001, Anhui, China;2College of Agronomy, Anhui Agricultural University, Hefei 230036, Anhui, China The objective of this study is to identify the genetic diversity among wheat parental materials from different provinces. To reveal the genetic diversity and population structure by using wheat 55K SNP chip, 150 wheat accessions from Anhui, Jiangsu, Henan, Sichuan, and Shandong provinces were analyzed. A total of 52,537 SNP loci were detected in the 150 wheat accessions. 39,422 high quality markers were obtained, of which 38,135 were polymorphic, accounting for 96.74%. The distribution of 38,135 polymorphic markers among the genomes showed the least in D subgenome (10,450), the most in B subgenome (15,290). The average polymorphic information content (PIC) was 0.315, with a variation of 0.068−0.375. The averaged genetic distance of accessions differed based on the origin: Henan > Sichuan > Shandong > Jiangsu > Anhui. The results of cluster analysis, principal component analysis, and population structure analysis were highly consistent, and the clustering results were consistent with the pedigree, regional origin, and breeding group. The study revealed that the average polymorphism information content in each province was at a moderate PIC level, but the average genetic distance of the materials was close. This indicated that the high-quality germplasm resources should still be introduced to alleviate the material homogeneity, so as to increase the ability of wheat to cope with stress and reduce the vulnerability and risk in actual wheat production. wheat; 55K SNP chip; breeding parents; genetic diversity; cluster analysis 10.3724/SP.J.1006.2023.21047 本研究由安徽小麦良种联合攻关(2021-)和国家自然科学基金青年科学基金项目(31901540)资助。 This study was supported by the Anhui Wheat Seed Joint Research Project (2021-) and the National Natural Science Youth Science Foundation of China (31901540). 何贤芳, E-mail: xianfanghe@126.com; 朱玉磊, E-mail: zhuyulei2011@126.com E-mail: lumaoang@126.com 2022-07-03; 2022-10-10; 2022-11-10. URL: https://kns.cnki.net/kcms/detail/11.1809.S.20221109.0850.002.html This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).1.3 主成分分析和群体结构分析
1.4 聚类分析
2 结果与分析
2.1 多态性SNP位点分布
2.2 多态性SNP最小等位基因、基因多样性和多态性信息含量多样性统计


2.3 群体划分多样性分析

3 讨论

