寄主诱导的基因沉默干扰核盘菌致病基因OAH在甘蓝型油菜抗菌核病中的应用
杨一丹 何 督 刘 静 张 岩 陈飞志 巫燕飞 杜雪竹,*
寄主诱导的基因沉默干扰核盘菌致病基因在甘蓝型油菜抗菌核病中的应用
杨一丹1何 督1刘 静2张 岩1陈飞志1巫燕飞1杜雪竹1,*
1湖北大学生命科学学院 / 省部共建生物催化与酶工程国家重点实验室, 湖北武汉 430062;2农业农村部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
油菜是我国第一大油料作物, 菌核病是我国油菜面临的主要病害之一。本试验利用HIGS技术将核盘菌中关键致病基因()的干扰片段转化到甘蓝型油菜中, 获得含有siRNA的转基因油菜植株, 研究HIGS介导的基因沉默对油菜抗菌核病的影响。接种核盘菌后的抗病鉴定结果表明,转基因油菜植株对核盘菌抗性增强; R18、R25和R36株系叶片接菌后病斑部位核盘菌菌丝内的基因表达量均低于野生型, 证明siRNA在转基因油菜中成功表达; 在添加了转基因油菜叶片提取物的培养基上培养的核盘菌扩展面积显著小于野生型, 分别降低35.29%、21.98%、31.53%, 且菌丝生长显著迟缓, 扩展长度较短, 分支少, 生长过程中发生断裂, 生长异常, 将其接种于正常的野生型油菜后, 其致病力明显下降; 对转基因油菜叶片接种核盘菌后观察发现, 叶片上的菌丝扩展较为稀疏, 生长受阻, 侵染垫形成受到抑制, 暗示在油菜中表达基因的干扰片段影响了菌丝生长和扩展; 进一步检测其病斑组织中的草酸含量发现, 接菌36 h、48 h后转基因油菜病斑组织中的草酸含量为391 μg g–1、446 μg g–1, 与野生型相比分别减少54 μg g–1、32 μg g–1。这一研究表明在油菜中表达核盘菌的干扰片段能够降低核盘菌侵染时草酸的积累, 从而增强转基因油菜对核盘菌的抗性水平。本试验利用HIGS技术研究了干扰核盘菌关键致病基因增强油菜对核盘菌的抗性, 为选育油菜抗菌核病品种提供理论基础和种质资源。
甘蓝型油菜; 核盘菌; HIGS; 草酰乙酸乙酰水解酶; 草酸
核盘菌()是一种具有非寄生专一性、侵袭性和坏死性的腐生型真菌, 能够侵害600多种植物, 包括油菜、大豆、向日葵和一些蔬菜作物[1-2]。油菜作为我国最重要的油料作物之一, 常年受到核盘菌的侵害, 每年造成油菜减产10%~20%, 严重的甚至高达80%, 影响油菜的产量和品质[3-5]。目前防治核盘菌的措施主要包括化学农药防治、物理防治、生物防治等, 但由于核盘菌主要是以菌核的形式存在于土壤和病株残体中进行越冬越夏, 能够生存长达7年之久, 且生命力顽强, 难以消除[5-7]。农业上主要以化学药剂防治为主, 然而该方法费时费力, 长时间施用后易使核盘菌产生耐药性, 降低防治效果。因此, 培育抗(耐)菌核病油菜品种是最为经济有效的途径。
核盘菌主要通过分泌细胞壁降解酶、草酸及致病分泌蛋白参与侵染寄主植物, 并发挥重要作用。早期研究结果发现, 利用紫外诱变技术能使核盘菌中的草酸合成受到影响, 导致该突变体的致病能力下降, 证明了草酸在核盘菌致病中的重要作用[8]。病菌中的草酸能够降低寄主病斑部位及周围的pH, 营造适合菌核发育的环境, 抑制寄主抗病相关酶的活性, 从而使病菌产生更多的胞外酶, 加速核盘菌对寄主细胞的降解[9]。草酸还能够螯合寄主细胞中的Ca2+, 阻断寄主细胞的钙离子信号通路, 并且在侵染初期抑制寄主活性氧的爆发、防卫反应和防御信号转导, 同时损伤细胞释放的钙离子浓度, 使生长中的菌丝免受毒性钙离子浓度的影响[10-11]。草酸的合成主要通过草酰乙酸乙酰水解酶(axaloacetate acetylhydrolase, OAH)催化水解草酰乙酸生成草酸[12]。核盘菌中的被证实是OA生物合成的调控枢纽[13]。
最近的研究进展表明, 小RNA诱导的基因沉默可以提高寄主抗性, 扩大抗病资源[14]。这种方法主要是小RNA在转录、转录后和翻译水平上抑制目的基因表达。宿主诱导基因沉默(host-induced gene silencing, HIGS)就是随着该技术发展而来的一种便捷高效的研究手段。该技术主要通过针对病原微生物的目的基因设计干扰片段, 将含有干扰片段的载体导入寄主中进行表达, 产生的dsRNA分子被DCL酶切割成21~24 nt的siRNA, 与寄主中的AGOs蛋白结合形成复合物, 从而靶向沉默病原生物的特定序列, 抑制寄主植物的防卫反应, 通过降解目的基因的mRNA而使该基因的转录水平下降, 进而使寄主植物对病原生物产生抗性。
本试验选取核盘菌侵染油菜的关键致病基因, 利用HIGS技术将的干扰片段导入油菜体内, 靶向降解该基因, 抑制草酸合成, 提高油菜对核盘菌的抗性, 从而创建一批能够干扰核盘菌致病的高抗性油菜株系, 为选育油菜抗菌核病品种提供理论基础和种质资源。
1 材料与方法
1.1 材料
供试品种为甘蓝型油菜Westar品种, 种植于湖北大学温室及培养箱, 培养温度为23℃, 光周期为18 h光培养, 6 h 暗培养。核盘菌菌种为本实验室保存, 活化条件为23℃暗培养。大肠杆菌DH5α、Trans-T1大肠杆菌感受态均为本实验室保存; DNA marker (DL2000)、逆转录酶、反转录试剂盒(TaKaRa Prime Script RT reagent Kit with gDNA Eraser)购买于TaKaRa公司; 植物RNA提取试剂盒购于普洛麦格(北京)生物技术有限公司; 琼脂、乙酰丁香酮、Tris饱和酚购于Sigma公司; MES、甘露醇购于iosharp公司; MS培养基粉末购于武汉天源慧达生物科技有限公司; 无水乙醇、蔗糖、氯化钙等常规试剂购于国药集团化学试剂有限公司; Easy酶、T4-DNA连接酶、2K DNA marker (Trans 2K)购买于全式金生物技术有限公司。
1.2 试验方法
1.2.1 HIGS沉默表达载体的构建 取20~22℃黑暗环境下培养了36 h的核盘菌菌丝, 提取RNA并反转录成cDNA。以cDNA为模板, 选择合适的目的基因干扰序列, 将OAH干涉片段(OAH-s/OAH-as)和中间link序列片段使用引物OAH-sF和OAH- ovlapsR进行PCR扩增; 将正向干涉片段(OAH-s)与中间link序列使用引物OAH-asF和OAH-asR通过PCR重叠延伸技术连接到一起, 之后进行胶回收。
将回收的干涉片段产物克隆至pEasy-Blunt载体上, 转化至大肠杆菌感受态中培养, 挑选克隆进行阳性鉴定。选择pCAMBIA1301载体中的I与I酶切位点进行双酶切, 胶回收线性载体质粒。I与I酶切Ri02s-W载体, 选择I与I酶切位点, 双酶切Ri02as载体后均回收相应的外源片段, 利用T4DNA连接酶连接回收的线性载体质粒, 转化到大肠杆菌感受态中培养。挑取阳性克隆子进行菌液PCR检测。
1.2.2 转基因油菜的获得与鉴定 采用农杆菌遗传转化的方法, 用含有RNAi干扰载体的农杆菌侵染甘蓝型油菜Westar的下胚轴, 经过共培养、选择培养、分化培养转化获得转基因油菜。利用PCR技术检测转基因中油菜植株中潮霉素抗性基因和RNAi干扰片段是否存在, 筛选获得阳性转基因油菜植株。
1.2.3 qRT-PCR表达量分析 各取3片野生型和转基因油菜植株叶片进行接菌, 在接菌24、36、48、60、72 h后取油菜叶片病斑部位核盘菌菌丝, 使用上海生工生物工程有限公司提供的真菌总RNA提取试剂盒提取核盘菌菌丝RNA。利用TaKaRa Bio提供的反转录酶试剂盒, 对抽提后的RNA样品反转录成cDNA。设计q-PCR特异性引物检测基因表达量, 使用CFX ConnectReal-time System荧光定量PCR仪进行分析, 采用2–ΔΔCt法对目标基因进行相对量的差异表达分析, 每个样本设3次生物学重复。
1.2.4 转基因油菜植株抗病性鉴定 离体叶片接菌:将核盘菌在PDA培养基上培养活化2次。各取3片生长6~8周, 生长状态良好、长势一致的油菜叶片。接菌后注意保湿, 并放置于22℃暗培养箱中, 分别在接菌不同时间后进行观察并照相, 使用ImageJ统计其病斑面积, 试验重复3次。
活体茎秆接菌: 选取距离地面20~60 cm处, 生长状态及茎秆大小一致的油菜, 将活化好的核盘菌菌块用保鲜膜包裹, 接种于油菜茎秆上。选择晴朗天气进行接种, 并做好保湿工作。接种4 d后将茎秆剪下照相并用标尺测量病斑直径。
幼苗接菌观察: 配制200 mL PDB培养基, 放入5个核盘菌菌块, 23℃、200转 min–1震荡培养48 h。用纱布过滤菌丝, 再用20 mL PDB培养液重悬24 h。重悬完成后, 用无菌枪头将菌球打碎, 用单层纱布将菌丝过滤, 即菌液制备完成。用定量移液枪将菌液接种在生长10 d左右的油菜叶片上, 在接种后不同时间点进行取样, 同株系油菜叶片各取3片, 试验重复3次。
1.2.5 棉蓝染色 在转基因油菜叶片上接种菌液后, 观察其发病情况。取不同时间点的6片油菜子叶, 放入50 mL的分装管中, 固定液固定24~48 h。用去离子水洗涤后, 棉蓝染色液染色1~2 h, 染色完成后二甲苯透明24 h, 最后制片镜检, 并在Model SZX2-ILLTQ体视显微镜下观察照相。
1.2.6 油菜叶片提取物体外抗真菌 取生长良好、状态一致的野生型和转基因油菜新鲜叶片5 g, 液氮研磨成粉状, 配制buffer提取液(pH调节至6.8~7.0)。如表1所示, 量取30 mL buffer提取液, 混匀后离心20 min, 吸取上清液。去离子水与提取的上清液按1∶1的比例加入到PDA培养基中。将活化后的核盘菌接种至培养基中, 再次活化培养40 h, 置于Model SZX2-ILLTQ体视显微镜下观察, 测量并统计菌丝扩展长度, 试验重复3次。
1.2.7 草酸含量测定 本试验使用上海双赢生物科技有限公司提供的植物(Plant)草酸(OA)酶联免疫吸附测定试剂盒进行测定。
2 结果与分析
2.1 核盘菌致病相关基因表达模式分析
为筛选核盘菌中的关键致病因子, 本试验对已经报道的核盘菌侵染油菜后的转录组数据进一步分析[15], 初步筛选到8个核盘菌坏死诱导分泌蛋白, 利用q-PCR对其进行分析验证。分析结果发现,()基因的表达量显著高于其他致病基因。在接菌24 h时, 核盘菌中()基因的表达显著上升, 接菌48 h时表达量达到最高, 72 h时开始下降(图1)。表明, 在核盘菌侵染油菜过程中,()是核盘菌基因组中关键的致病因子。因此, 选择基因构建RNAi干扰载体。
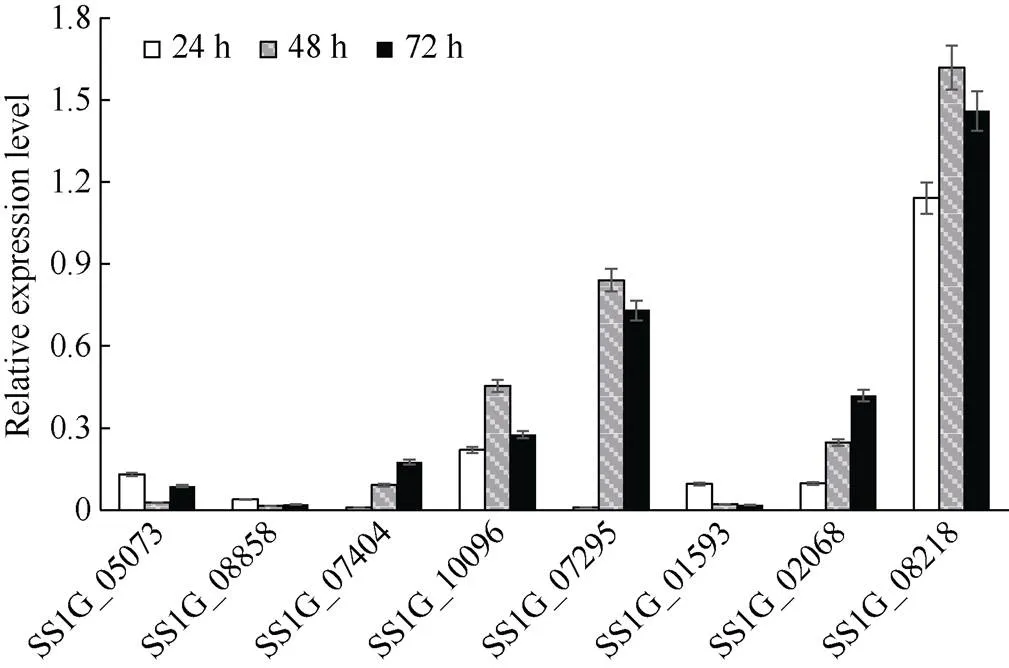
图1 核盘菌侵染野生型油菜后相关致病基因的表达情况
2.2 RNAi载体构建及鉴定
根据NCBI公布的核盘菌基因序列进行分析, 使用Primer 5.0设计引物, 以提取的核盘菌RNA反转录成的cDNA为模板, 扩增出的干扰片段。扩大培养载体菌株, 提取质粒DNA, 以质粒为模板扩增中间Link连接片段, 电泳结果与预期结果一致。
回收目的片段, 并以干涉片段和中间Link片段为模板, 采用PCR扩增方式进行重叠延伸, 将干涉片段和中间Link片段连接到一起, 再次胶回收PCR扩增条带, 连接到载体构建成正向干涉中间载体, 将第一步得到的干涉片段连接到载体上构建成反向干涉中间载体, 转化大肠杆菌DH5α感受态, 挑取单菌落做PCR阳性鉴定。
用I和I同时双酶切pCAMBIA1300和正向干涉中间载体,I和I双酶切反向干涉中间载体, 回收相应片段, 用T4-DNA连接酶连接, 连接产物转化大肠杆菌, 挑取单菌落进行阳性鉴定(图2)。扩大培养后送公司测序, 干涉区域与预期结果一致, 序列正确无突变。将菌株提取质粒后, 转入农杆菌感受态细胞GV3101中, 挑取单菌落进行PCR鉴定, 并于–80℃保存。

图2 OAH干扰终载体菌落PCR检测
M: DL2000; 1~8: 菌落PCR片段。
M: DL2000; 1–8: the colony PCR fragments.
2.3 SS.OAH.RNAi转基因油菜的获得及抗病性鉴定
利用农杆菌转化方法将RNAi干扰载体转化至甘蓝型油菜Westar中, 通过PCR扩增鉴定共获得9个阳性株系。对这些转基因油菜的T0代植株进行菌核病抗性鉴定后筛选出R18、R25、R36三个抗性植株, 自交繁育后获得T2代稳定遗传材料(图3)。
取生长状态良好、大小一致的野生型和T2代转基因油菜的叶片与茎秆, 接种核盘菌进行抗性鉴定。对转基因油菜株系离体叶片接菌36 h后的结果发现, R18、R25、R36转基因株系叶片上的病斑面积明显小于野生型, 其病斑面积分别降低11.68%、14.42%、15.06% (图4-A, B)。活体茎秆接菌4 d后转基因油菜茎秆的病斑直径显著低于野生型, 其分别降低33.07%、38.46%、35.38% (图5-A, B)。表明转基因油菜提高了对核盘菌的抗性。
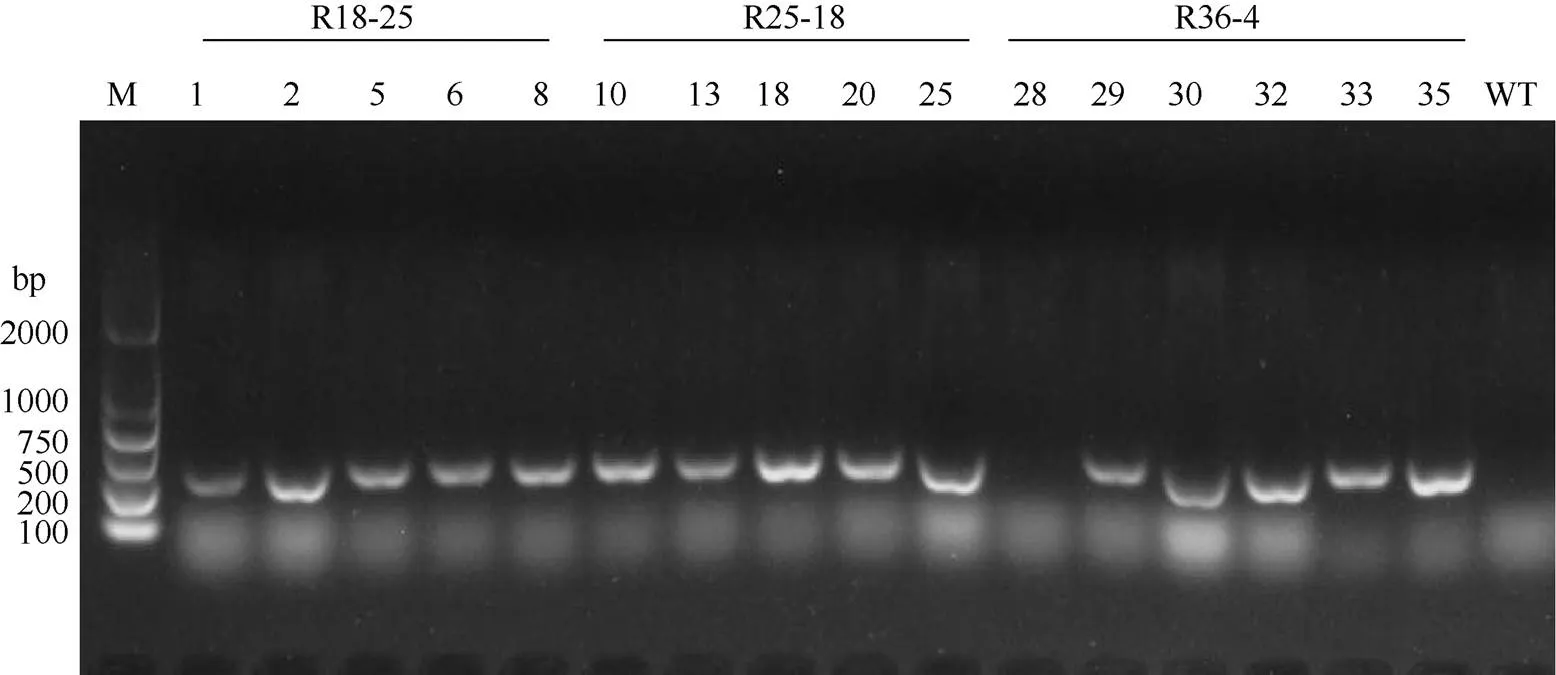
图3 T2代SS.OAH.RNAi转基因植株部分阳性鉴定凝胶电泳图
M: Trans 2K; WT: 阴性对照。M: Trans 2K; WT: the negative control.

图4 T2代SS.OAH.RNAi转基因油菜株系离体叶片接菌36 h病斑面积统计图
WT: 野生甘蓝型油菜; R25、R36、R18为T2代转基因株系材料。
WT: wild; R25, R36, and R18 are T2generationtransgenic lines.
WT: 野生甘蓝型油菜; R25、R36、R18为T2代转基因株系材料。
WT: wild; R25, R36, and R18 are T2generationtransgenic lines.
2.4 SS.OAH.RNAi转基因油菜病斑组织部位菌丝内SsOAH表达量分析
为验证转基因油菜株系抗性的提高是否由于siRNA降解核盘菌基因所导致的, 分别提取野生型与转基因油菜植株接菌24、36、48、60、72 h后病斑部位核盘菌菌丝的RNA, 通过q-PCR分析核盘菌在侵染油菜时核盘菌菌丝体内的基因的表达情况。
分析结果发现, 与WT相比, R18株系叶片病斑组织上核盘菌菌丝中的表达量在接菌24~72 h过程中显著下降, 在接菌36 h时表达量为最低, 72 h时达到峰值; R25和R36株系叶片病斑组织中的表达量在接菌24 h、36 h、48 h、60 h和72 h时均低于野生型。R25株系在接菌60 h时靶基因表达量下降显著, 72 h时达到峰值。R36株系在接菌24 h时靶基因表达量达到峰值, 之后开始逐渐下降, 接菌60 h后开始升高(图6)。表明,转基因株系降低了核盘菌菌丝内基因的表达, siRNA在转基因油菜中成功表达, 靶向降解了基因的表达。
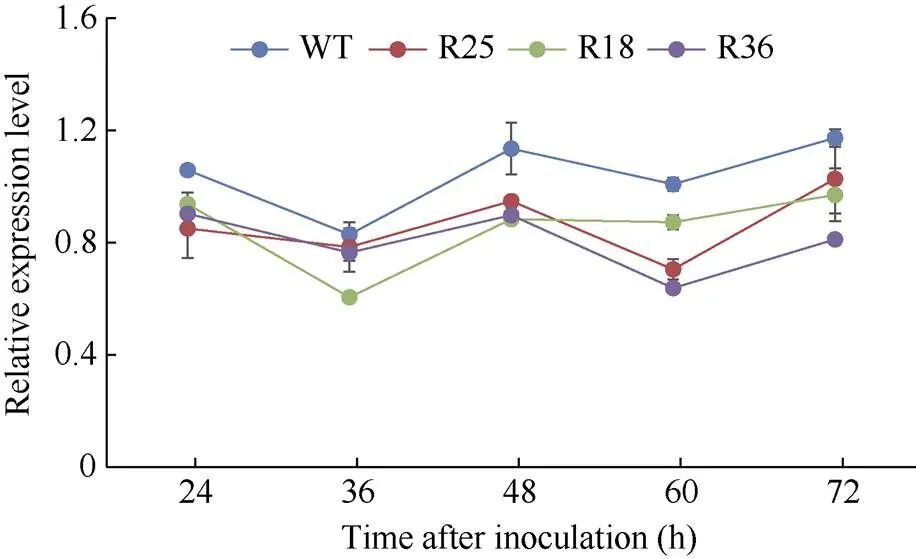
图6 T2代SS.OAH.RNAi转基因油菜叶片接菌后病斑部位核盘菌菌丝内OAH基因的表达模式分析
WT: 野生甘蓝型油菜; R25、R36、R18为T2代转基因株系材料。
WT: wild; R25, R36, and R18 are T2generationtransgenic lines.
2.5 转基因油菜叶片提取物抗病性鉴定
为进一步证实转基因材料对核盘菌的抗性影响, 本试验萃取转基因油菜叶片的提取物培养核盘菌并接种于野生型油菜叶片上, 进一步验证基因材料对核盘菌生长的影响。
在添加了转基因油菜叶片提取物的PDA培养基上培养核盘菌, 培养40 h后观察菌丝扩展面积以及扩展情况(图7)。统计结果发现,转基因材料(R18、R25、R36)叶片提取物制成的PDA培养的核盘菌菌丝的生长受到明显抑制, 扩展面积与野生型相比显著降低, 分别降低35.29%、21.98%、31.53% (图7-A, B1~B5); 在体式显微镜下观察核盘菌菌丝的生长和扩展情况发现, 转基因材料制成的PDA培养基培养的菌丝, 其生长状态与野生型油菜培养的菌丝相比, 扩展长度变短, 菌丝细密并且分支较少。其中R18和R36株系叶片提取物培养的菌丝在生长过程中发生断裂, 且生长异常; R25株系叶片提取物培养的菌丝则生长细密, 扩展受限(图7-B6~B10)。
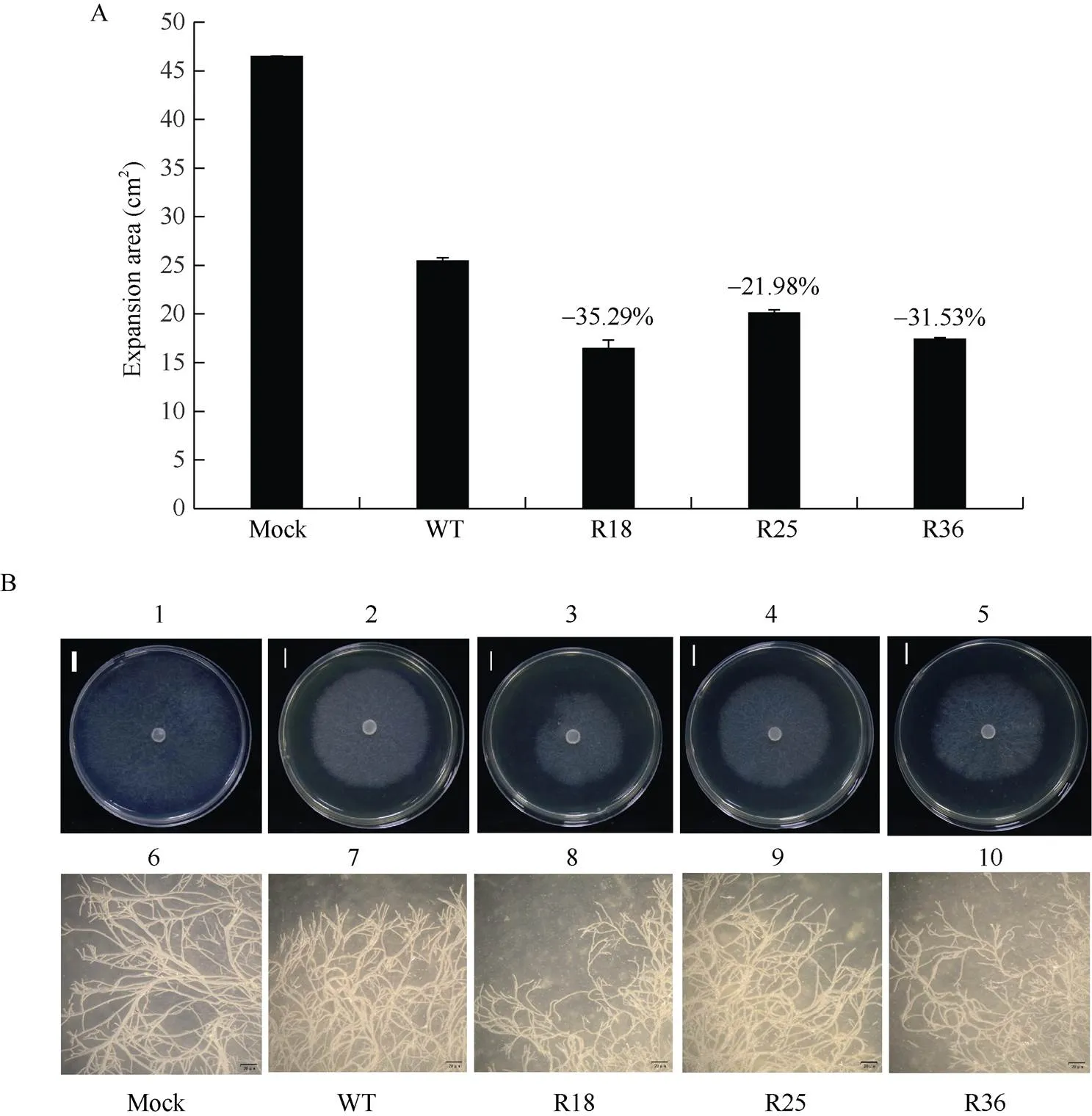
(图7)
WT: 野生甘蓝型油菜; R25、R36、R18为T2代转基因株系材料。Mock: PDA培养基培养的核盘菌; A:添加提取物的PDA培养基培养核盘菌40 h后的菌丝扩展面积统计; B: 标尺为1 cm; B1: 无添加萃取液的PDA活化的菌丝; B2: 添加了野生型油菜叶片萃取液活化的核盘菌; B3~B5为添加了转基因油菜(R18、R25、R36)叶片萃取液活化的核盘菌; B6~B10: 分别对应图B1~B5在体视显微镜下观察到的核盘菌菌丝生长情况; C: 野生型油菜与转基因油菜(R18、R25、R36)叶片提取物制成的培养基培养的核盘菌菌丝接种于野生型油菜后48 h的病斑扩展情况, 标尺为2 cm。
WT: wild; R25, R36, and R18 are T2generationtransgenic lines. Mock:cultured in PDA medium; A: the statistics of the expanded area of the culture disk after adding PDA to the mycelial extract for 40 hours; B: bar: 1 cm; B1: PDA activated hyphae without added extract; B2:activated by wild-type rape leaf extract; B3–B5 addedtransgenic rape (R18, R25, and R36) leaf extract; B6–B10: corresponding to the mycelial growth ofobserved under stereomicroscope in Fig. B1–B5 respectively. C: the expansion ofhyphae cultured in the medium made of wild-type rape and transgenic rape (R18, R25, and R36) leaf extract 48 hours after inoculation in wild-type rape. Bar: 2 cm.
将野生型油菜与转基因油菜叶片提取物制成的培养基培养的核盘菌菌丝分别接种于正常的野生型油菜叶片48 h后发现, 转基因油菜叶片提取物制成的培养基培养的核盘菌致病能力明显下降(图7-C)。表明转基因油菜叶片提取物能够限制核盘菌菌丝的生长和扩展, 影响核盘菌的致病能力。
2.6 棉蓝染色观察转基因油菜叶片接菌部位菌丝生长及扩展情况
进一步观察转基因油菜株系接菌后, 核盘菌菌丝在叶片上的生长及定殖情况。观察结果发现, 在接菌12 h时, 野生型与转基因油菜叶片表面开始出现少量菌丝, 说明菌丝在野生型和转基因材料叶片表面开始定殖。其中R36株系叶片表面的菌丝与野生型无较大差异, 而R18和R25株系叶片表面的菌丝明显较野生型少(图8-A~D); 在接菌24 h时, 野生型和转基因油菜的叶片表面菌丝增多(图8-E~H)。与野生型相比, R18株系叶片上的菌丝分支较少, 侵染垫形成较为迟缓。R25株系叶片的菌丝分支与其他转基因材料相比显著减少, 菌丝生长缓慢, 无侵染垫的形成。R36株系叶片上的菌丝生长异常, 生长缓慢且仅有少量侵染垫形成。说明菌丝在转基因油菜叶片上的生长和侵染垫形成受到了影响(图8-I~L)。在接菌36 h时核盘菌菌丝向外扩展, R18株系叶片上的菌丝扩展稀疏, 形成的圆顶型侵染垫显著低于对照, R25和R36株系叶片上几乎无侵染的形成(图8-M~P)。表明,转基因油菜早期可通过干扰菌丝定殖、侵染垫形成和菌丝扩展来限制核盘菌的入侵。
2.7 草酸含量测定
核盘菌中草酸的合成主要通过草酰乙酸乙酰水解酶(OAH)催化草酰乙酸生成草酸和二氧化碳, 协助侵染寄主植物。因此, 本试验通过测定转基因油菜叶片接菌36 h、48 h时病斑部位组织中的草酸含量, 验证核盘菌中基因的降解是否影响了病斑组织的草酸积累。
由图9可知, 转基因油菜叶片在接菌36 h时病斑部位组织中的草酸含量为391 μg g–1, 与野生型相比减少了54 μg g–1; 在接菌48 h时, 转基因油菜叶片病斑部位组织中的草酸含量为446 μg g–1, 与野生型相比减少了32 μg g–1。结果表明,转基因油菜能够抑制核盘菌侵染时草酸的积累。
3 讨论
已有的研究表明, HIGS技术靶向性高、通用性强、能够沉默多拷贝基因, 在水稻、棉花、大麦、小麦等多种植物中均得到了应用, 能够有效干扰病原菌中的致病基因, 提高寄主对病原菌的抗性水平[16-19]。研究表明, 利用HIGS技术, 在大豆中转入靶向沉默基因的干扰片段后, 导致该基因的相对表达量下降, 提高了转基因大豆菌核病的抗性[20]。另外, Kusum等[21]通过在拟南芥中表达的干扰片段发现, 转基因拟南芥病斑组织上菌丝内的表达量和草酸量积累同时减少, 表明的沉默导致草酸含量降低。以上研究均证明了草酸在核盘菌侵染寄主时发挥的重要作用。
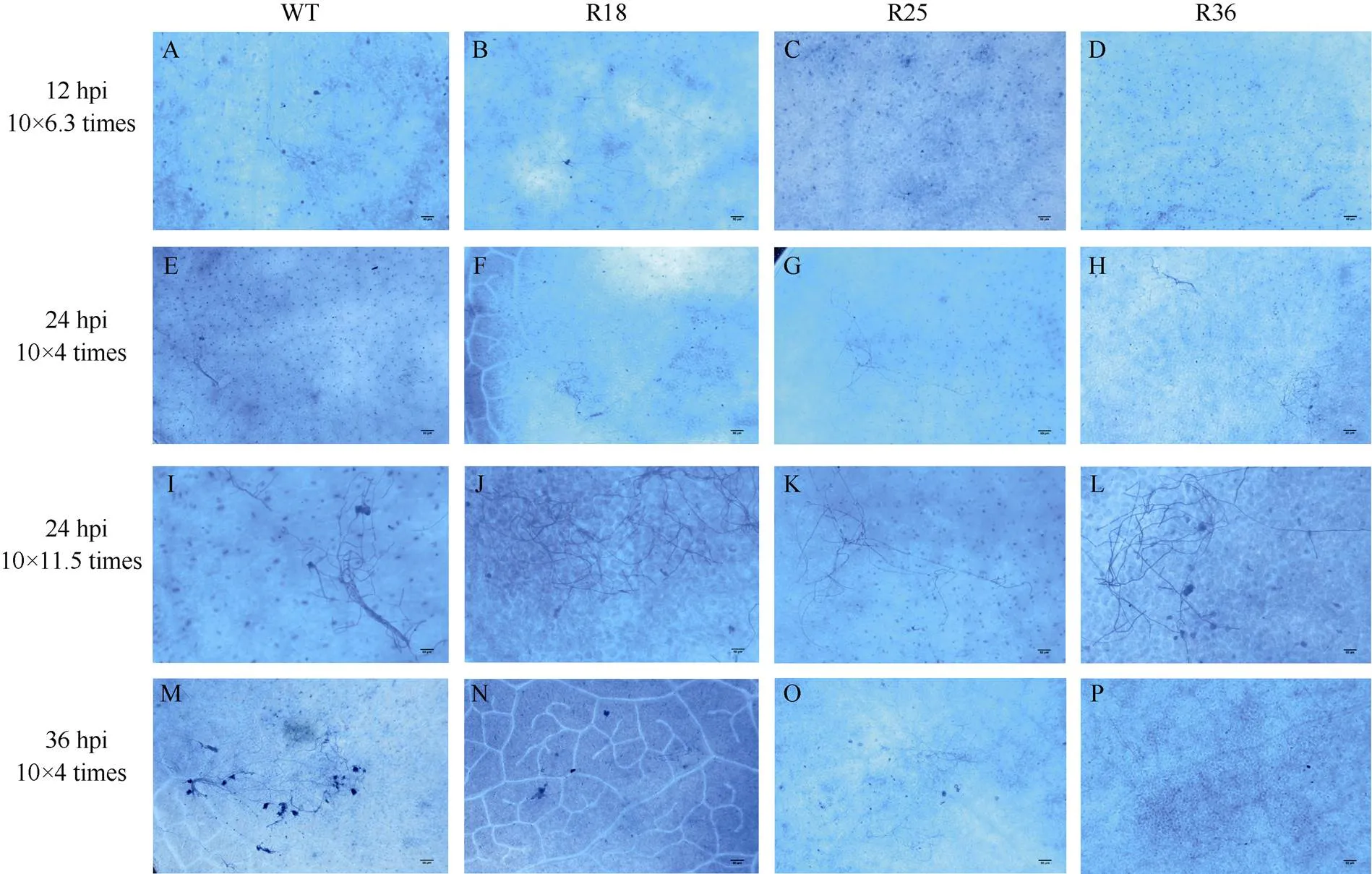
图8 棉蓝染色观察油菜叶片接菌情况
WT: 野生甘蓝型油菜; R25、R36、R18为T2代转基因株系材料。
WT: wild; R25, R36, and R18 are T2generationtransgenic lines.
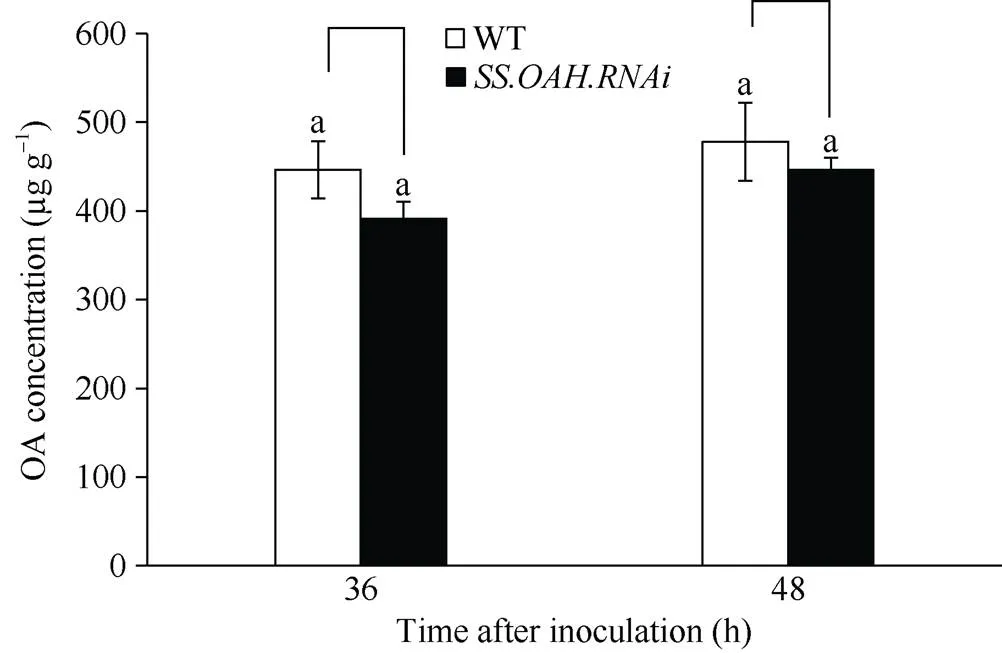
图9 T2代SS.OAH.RNAi转基因植株叶片病斑部位草酸含量测定
字母表示具有统计学意义的差异值, 基于< 0.05的单因素方差分析。
Different lowercase letters represent statistically significant differences at< 0.05 based on one-way ANOVA.
本研究利用HIGS技术, 选择核盘菌的重要致病因子为目的基因, 构建RNAi干扰载体, 创建的转基因油菜植株提高了对菌核病的抗性。前期我们通过q-PCR检测研究未发现转基因油菜叶片体内的表达, 推测插入的干扰片段需要经过核盘菌才能诱导表达。之后通过检测T2代转基因油菜叶片接菌后病斑组织部位核盘菌菌丝中的表达验证了这一猜想。检测结果发现, 转基因植株中的靶基因表达量低于野生型, 推测基因表达量的下降可能是由于油菜体内siRNA的成功转录, 特异性靶向降解所导致。值得注意的是, 核盘菌侵染寄主时基因的表达量虽然有所下降, 草酸积累被抑制, 但抑制效果不佳。推测可能是由于不同的HIGS技术表达系统没有产生足够的沉默信号分子转移至病原菌中, 导致沉默的基因较少,因此抑制草酸合成的能力下降[22-23]。
此外本研究还通过提取野生型和转基因油菜叶片的粗蛋白制成PDA培养基, 从侧面验证了转基因材料的抗病性。棉蓝染色结果也发现, 转基因材料能够影响核盘菌菌丝的生长及扩展, 而提取了转基因油菜的粗蛋白制成的培养基活化的核盘菌菌丝与WT相比, 菌丝生长及扩展均受到了限制。推测RNA干扰片段可能以一种较为稳定的结构存在于转基因油菜组织中。值得注意的是, HIGS技术是将目的基因随机插入植物基因组中, 具有一定的偶然性, 由于植物基因组以及体内基因功能的复杂性, 可能会影响寄主中某个关键基因的表达, 近而影响到植株的整体功能。但由于RNA极易降解, 因此, 干扰片段在转基因油菜体内的存在方式有待挖掘和探究。
另外, HIGS技术整体受到RNAi网络调控, 机制复杂, 目前对于寄主植物中产生的沉默信号分子进入病原菌的机制尚不清楚, 有待进一步深入研究。
4 结论
将编码核盘菌草酸合成的基因的干扰片段利用HIGS技术导入至油菜基因组中, 获得的转基因油菜提高了对核盘菌的抗性, 推测可能是在转基因油菜中转录成功的siRNA靶向降解了的表达, 影响了核盘菌菌丝的正常生长和扩展, 同时抑制了草酸的积累, 从而降低了核盘菌的致病能力。
[1] 李慧, 文李, 刘凯, 官春云. 油菜抗菌核病机制研究进展. 作物研究, 2015, 29(1): 84–90.Li H, Wen L, Liu K, Guan C Y. Research progress on resistance mechanism ofto., 2015, 29(1): 84–90 (in Chinese with English abstract).
[2] Liang X, Rollins J A. Mechanisms of broad host range necrotrophic pathogenesis in., 2018, 108: 1128–1140.
[3] 吴健, 周永明, 王幼平. 油菜与核盘菌互作分子机理研究进展.中国油料作物学报, 2018, 40: 721–729. Wu J, Zhou Y M, Wang Y P. Research progress on molecular mechanisms ofinteraction., 2018, 40: 721–729 (in Chinese with English abstract).
[4] 杨清坡, 刘万才, 黄冲. 近10年油菜主要病虫害发生危害情况的统计和分析. 植物保护, 2018, 44(3): 24–30. Yang Q P, Liu W C, Huang C. Statistics and analysis of oilseed rape losses caused by main diseases and insect pests in recent 10 years., 2018, 44(3): 24–30 (in Chinese with English abstract).
[5] Wu J, Zhao Q, Yang Q, Liu H, Li Q Y, Yi X Q, Cheng Y, Guo L, Fan C C, Zhou Y Z. Comparative transcriptomic analysis uncovers the complexgenetic network for resistance toin., 2016, 6: 19007.
[6] Adams P B, Ayers W A. Ecology ofspecies., 1979, 69: 896–896.
[7] Willetts H J, Wong J. The biology of,.and.with emphasis on specific nomenclature., 1980, 46: 101–165.
[8] Godoy G, Steadman J R, Dickman M B, Dam R. Use of mutants to demonstrate the role of oxalic acid in pathogenicity ofon., 1990, 37: 179–191.
[9] Liang Y, Strelkov S E, Kav N N V. Oxalic acid-mediated stress responses inL., 2010, 9: 3156–3173.
[10] Williams B, Kabbage M, Kim H J, Britt R, Dickman M B. Tipping the balance:secreted oxalic acid suppresses host defenses by manipulating the host redox environment., 2011, 7: e1002107.
[11] Heller A, Witt G T. Oxalic acid has an additional. detoxifying function inpathogenesis., 2013, 8: e72292.
[12] Riou C, Freyssinet G, Fevre M. Production of cell wall-degrading enzymes by the phytopathogenic fungus., 1991, 57:1478–1484.
[13] Evans M V D, Christine S. Oxalate production by fungi: its role in pathogenicity and ecology in the soil environment., 1996, 42: 881–895.
[14] Derbyshire M, Mbengue M, Barascud M, Navaud O, Raffaele S. Small RNAs from the plant pathogenic fungushighlight host candidate genes associated with quantitative disease resistance., 2019, 20: 1279–1297.
[15] Wu J, Zhao Q, Yang Q, Liu H, Li Q, Yi X, Cheng Y, Guo L, Fan C, Zhou Y. Comparative transcriptomic analysis uncovers the complex genetic network for resistance toin., 2016, 6: 19007–19022.
[16] Xu J, Wang X, Li Y, Zeng J, Wang G, Deng C, Guo W. Host-induced gene silencing of a regulator of G protein signalling gene () confers resistance towilt in cotton., 2018, 16: 1629–1643.
[17] Nowara D, Gay A, Lacomme C, Shaw J, Ridout C, Douchkov D, Hensel G, Kumlehn J, Schweizer P. HIGS: host-induced gene silencing in the obligate biotrophic fungal pathogen., 2010, 22: 3130–3141.
[18] Yin C, Jurgenson J E, Hulbert S H. Development of a host-induced rnai system in the wheat stripe rust fungusf. sp., 2011, 24: 554–561.
[19] Zhu L, Zhu J, Liu Z, Wang Z, Zhou C, Wang H. Host-induced gene silencing of rice blast funguspathogenicity genes mediated by the brome mosaic virus., 2017, 8: 241.
[20] McCaghey M, Shao D, Kurcezewski J, Lindstrom A, Ranjan A, Whitham S A, Conley S P, Williams B, Smith D L, Kabbage M. Host-induced gene silencing of aoxaloacetate acetylhydrolase using bean pod mottle virus as a vehicle reduces disease on soybean., 2021, 12: 677631.
[21] Rana K, Yuan J, Liao H, Banga S S, Kumar R, Qian W, Ding Y. Host-induced gene silencing reveals the role ofoxaloacetate acetylhydrolase gene in fungal oxalic acid accumulation and virulence., 2022, 258: 126981.
[22] Nakayashiki H, Nguyen Q B. RNA interference: roles in fungal biology., 2008, 11: 494–502.
[23] Spiering M J, Moon C D, Wilkinson H H, Schardl C L. Gene clusters for insecticidal loline alkaloids in the grass-endophytic fungus., 2005, 169: 1403–1414.
Application of host-induced gene silencing interfering withpathogenic geneinresistance to
YANG Yi-Dan1, HE Du1, LIU Jing2, ZHANG Yan1, CHEN Fei-Zhi1, WU Yan-Fei1, and DU Xue-Zhu1,*
1School of Life Sciences, Hubei University / State Key Laboratory of Biocatalysis and Enzyme Engineering, Wuhan 430062, Hubei, China;2Key Laboratory of Biology and Genetics and Breeding of Oil Crops, Ministry of Agriculture and Rural Affairs, Hubei University, Wuhan 430062, Hubei, China
Rapeseed is the largest oil crop in China, andis one of the main diseases of rapeseed in China. In this experiment, to study the effect of HIGS-mediatedgene silencing on the resistance tointhe interference fragment of the key pathogenic gene() inwas transformed intoby HIGS technique, and the transgenic plants containing siRNA were obtained. The results of disease resistance identification after inoculation withshowed that the resistance oftransgenic rapeseed plants towas enhanced, and the relative expression levels ofgenes inmycelia of R18, R25, and R36 lines were lower than wild type, indicating that siRNA was successfully expressed in transgenic rapeseed. The lesion size ofcultured on the medium supplemented with transgenic rapeseed leaf extract was significantly smaller than wild type, which decreased by 35.29%, 21.98%, and 31.53% respectively. The mycelium grew slowly, the expansion length was shorter, the branches were few, and the growth was abnormal. After inoculating it into normal wild type rapeseed, its pathogenicity decreased significantly. After inoculating the transgenic rapeseed leaves with, it was observed that the mycelium expansion on the leaves was sparse, the growth was blocked, and the infection pad formation was inhibited, suggesting that the interference fragments expressinggene in rapeseed affected the mycelium growth and expansion. Compared with the wild type, the content of oxalic acid in the lesion tissue of transgenic rapeseed was 391 μg g–1and 446 μg g–1at 36 hpi and 48 hpi, which decreased 54 μg g–1and 32 μg g–1, respectively. This study showed that the interference fragment expressingin rapeseed could reduce the accumulation of oxalic acid duringinfection and enhance the resistance level of transgenic rapeseed to. In this experiment, HIGS technique was used to improve resistance toby transforming interference fragments ofgene in rapeseed, and to provide a theoretical basis and germplasm resources for breedingresistant varieties.
;; HIGS; oxalyl acetate acetylhydrolase; oxalic acid
10.3724/SP.J.1006.2023.24126
本研究由农业农村部油料作物生物学与遗传育种重点实验室开放课题基金(KF2022001)和国家自然科学基金项目(31771837)资助。
This study was supported by the Open Project Foundation of Key Laboratory of Oil Crop Biology and Genetics and Breeding of the Ministry of Agriculture and Rural Affairs (KF2022001) and the National Natural Science Foundation of China (31771837).
杜雪竹, E-mail: duxuezhusk@163.com
E-mail: duxuezhusk@163.com
2022-05-26;
2022-10-10;
2022-10-31.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20221028.1836.004.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

