一种用于脑肿瘤分割的改进U 形网络
黄敬腾,李 锵,关 欣
天津大学微电子学院,天津 300072
神经胶质瘤作为一种致死率较高的原发性脑肿瘤,对人们的生产和生活产生严重的影响.在临床中,医生常使用磁共振成像(Magnetic resonance imaging,MRI)观察肿瘤的位置、大小和形态,磁共振成像作为一种成像技术在肿瘤诊断、监测和分割方面发挥着重要作用.国际医学图像计算和计算机辅助干预协会针对脑部肿瘤问题每年举办一次脑部肿瘤分割挑战赛(Brain tumor segmentation,BraTS).BraTS 的脑肿瘤磁共振图像有四个序列模态,分别为T1 加权像(T1 weighted image,T1)、T1ce 像(Contrast-enhanced T1-weighted image1,T1ce)、T2 加权像(T2 weighted image,T2)和液体衰减反转恢复序列(Fluid attenuated inversion recovery,Flair),这些图像模态具备一定的软组织对比度和杰出的功能成像能力,通过多模态融合方法可判断出胶质瘤分割的区域.
神经胶质瘤分割传统上依赖具备丰富经验的医师,这不仅耗时耗力,也会因医师的水平不同、主观因素产生差异.随着技术的发展,胶质瘤等医学疾病的半自动分割、自动分割方法都已经发展起来并作为临床应用的参考工具,其中,随机森林(Random forest)和支持向量机(Support vector machine)等机器学习方法[1-4]有效提高了医学图像分割的分割水平和工作效率,深度学习方法[5-6]的发展更是进一步推动了包括脑肿瘤图像分割在内的各类医学图像分割的发展.
脑肿瘤图像分割任务的挑战主要在于胶质瘤和它的子区域(水肿、增强肿瘤区域、坏疽和健康组织)在外观、位置和形状边界上的多样性和模糊性导致的复杂性.针对这一复杂任务,目前医学上对图像进行分割的方法均基于编码-译码器结构设计自动化分割网络,并使用跳跃连接补充细节特征,但常见跳跃连接补充的特征图为编码器特征提取后的输出特征图,忽略了此过程中的原始细节信息丢失问题.在卷积神经网络中,通道数代表特征数,并影响着网络的性能,因此对于复杂的脑肿瘤图像分割任务而言,常存在着特征不足的问题.基于此,本文提出跳跃连接前置的构想,即令编码器的输入特征图而非输出特征图连接到同分辨率特征图对应的解码器,并设计消融实验验证构想的有效性和泛化性.此外,使用倒残差结构设计编码器和解码器模块,提出前置跳跃连接倒残差U 形网络(FS Inv-Res U-Net),以实现脑肿瘤的精确分割.
1 脑肿瘤分割的方法
1.1 脑肿瘤分割
利用计算机辅助手段分割脑肿瘤图像是当今深度学习的一个研究点,除了常见的基础网络,研究者采用多种新型方法对脑肿瘤区域进行分割.Ali 等[7]基于全卷积网络(Fully convolutional network,FCN)提出Kronecker 卷积用于解决扩张卷积丢失像素点的问题.Cui 等[8]采用生成对抗网络的思想,提出深度学习中新型的神经网络自动分割算法进行语义分割,创新性地提出输入数据的自动编码器学习,有效提升了分割性能.Cheng 等[9]提出了一种新的样本标签校正和标签细化方法,将其引入到三维生成对抗网络.Rahimpour 等[10]则利用知识蒸馏方法实现脑肿瘤图像分割,探索通过跨模态蒸馏方法提高基于 T1 加权 MRI 的脑肿瘤分割性能的能力.这些方法为脑肿瘤分割方法拓宽了研究思路,具有一定参考意义.
此外,对卷积神经网络进行级联也是一种常见方法[11],级联网络常使用第一级网络对脑肿瘤图像进行粗分割获得粗略分割效果,粗分割结果作为第二级网络的输入进行细分割并获得精细化分割效果.Zhang 等[12]提出多步级联的方式,利用脑肿瘤区域之间的包含关系,单一网络针对脑肿瘤的特定区域进行分割,并将多个单一网络进行级联,但是级联方式大幅提高了网络的参数量和计算量,增加了网络的复杂度,也并未进一步考虑到跳跃连接(Skip connection)在分割网络中的重要性.
1.2 跳跃连接和倒残差结构
3D U-Net[13]在编码阶段和解码阶段分别提取图像特征和恢复图像,在两者之间采用跳跃连接为编码器补充原始信息,通过跳跃连接,可将先前得到的一部分特征图按通道级联或相加而达到特征重用的效果.Huang 等[14]提出一种密集连接方法,使用跳跃连接密集地将特征图进行连接,这一思想采纳了跳跃连接和残差结构的长处,使用最少的参数加强了特征图之间的重用和传递.Zhang等[12]设计了一个块级跳跃连接的跨级联 V-Net,在级联V-Net 的两个编码器之间、两个解码器之间设置跳跃连接以实现特征重用,提高了分割效果.Wu 等[15]提出一个跳跃连接U-Net 网络,将下采样之后的原始特征图与同分辨率的解码器特征图相加,通过添加跳跃连接捕获了更多特征并收敛到更好的最优值.Ma 等[16]提出一个用于遥感图像分割的MSCA-Net,将空洞卷积和跳跃连接融入到U-Net,与U-Net++[17]不同的是,U-Net++在跳跃连接处使用合适的卷积层以实现密集连接,而MSCANet 则利用下采样实现密集连接.
跳跃连接在3D U-Net 中发挥着重要的作用,将低分辨率特征输入到高级别卷积层以补充细节信息,有利于处理复杂边界挑战的医学图像分割任务.但在上述的网络设计中,特征图经过每层编码器特征提取后,通过跳跃连接补充到对应解码器,其补充的特征细节相较原始特征已有偏差,这不利于解码器的恢复图像细节;同时密集的跳跃连接虽然不增加网络参数量,却大幅地提高网络的计算负载,增加了计算的时间成本.
为了缓解梯度消失问题,He 等[18]提出残差结构,通过创建短连接,使梯度可以不间断地流动,从而允许参数在网络深处更新.Sandler 等[19]利用残差思想提出倒残差结构,通过通道宽度的拓展实现网络性能的提升.在此基础上,Yu 等[20]使用倒残差结构代替池化层以提升信息提取能力.Zhao等[21]在倒残差结构基础上提出多尺度倒残差卷积网络以学习不同尺度的故障特征,保证故障特征提取的完整性.Zhang 等[22]提出深度倒残差模块以高维特征表示低维特征数据,从而缓解数据特征数量少的挑战.倒残差结构通过提升网络宽度以实现充分学习和拓展特征图信息,它不仅可以缓解梯度消失问题和网络退化问题,也能够弥补特征不足问题,实现网络性能的大幅提升.
2 改进网络的设计
在医学图像分割任务中,多数网络[23-24]基于编码-解码器的思想设计,尤其是以3D U-Net 为基础架构进行改进.在原始3D U-Net 的编码阶段,输入图像数据经过三次下采样降低输入图像尺寸,通过编码器提取图像特征,提取到的特征在解码阶段经过三次上采样和解码器处理逐步恢复图像.在相同尺寸大小的特征图之间,使用跳跃连接补充原始图像信息,但是传统3D U-Net 并未进一步考虑原始图像细节的补充,为此本文提出前置跳跃连接并设计了前置跳跃连接倒残差U 形网络结构(Front-skip connection inverted residual U-Net),通过跳跃连接将每层编码器的输入特征图连接至对应解码器.
2.1 FS Inv-Res U-Net 架构图
本文设计提出的前置跳跃连接倒残差U 形网络FS Inv-Res U-Net 如图1 所示.
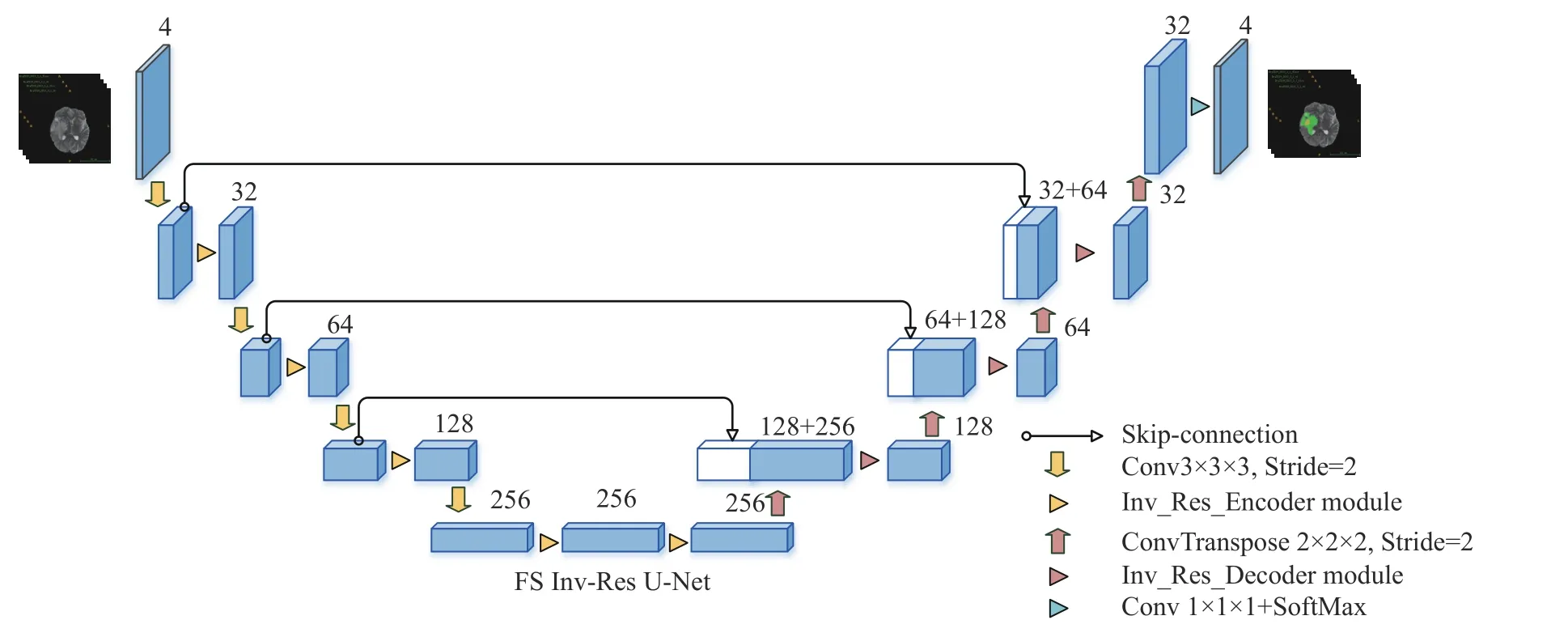
图1 前置跳跃连接倒残差U 形网络的架构图Fig. 1 Architecture of the front-skip connection inverted residual U-shaped network
FS Inv-Res U-Net 网络共设计了4 次下采样和4 次上采样,在编码器和解码器之间使用了前置跳跃连接进一步补充原始细节,同时分别设计了下采样卷积模块和编码器模块,上采样卷积模块和解码器模块.在编码阶段,输入特征图经下采样模块处理后尺寸逐步降低,对应的特征图数量加倍;同理,在解码阶段,输入特征图经上采样模块处理后逐步恢复相应的尺寸大小,特征图数量经解码器模块处理后减小,最后特征图经1×1×1 卷积和Softmax 函数处理后输出.
2.2 前置跳跃连接
在3D U-Net 设计中,特征图随着每一层的卷积、正则化和激活函数处理之后,其含有的细节信息与初始的细节信息都会有所偏差.对于跳跃连接而言,其设计本质是将原始特征的细节信息补充到对应解码器,即在解码器阶段,通过跳跃连接补充的特征图越原始,解码器越容易达到更优的分割效果,因此提出前置跳跃连接,将每一层编码器获得的最原始特征图以跳跃连接的方式补充到对应同分辨率编码器.
DenseNet、3D U-Net 等传统方式通过使用下采样或上采样方式调整到相同大小的特征图分辨率,最终达到密集连接的效果.不同于这种密集连接方式,前置跳跃连接发生在同分辨率尺寸的特征图之间,在不增加网络模型复杂度的前提下通过对连接起点的调整以达到提高网络性能的提升.
2.3 倒残差模块
本文设计的倒残差模块如图2 所示.在FS Inv-Res U-Net 编码器阶段,输入到编码器模块的特征图首先经3×3×3 卷积模块提取特征,之后经过跨卡同步归一化和ReLU 激活函数处理,得到结果后重复上述处理.

图2 倒残差模块.(a)倒残差编码器模块;(b)倒残差解码器模块.Fig. 2 Encoder and decoder modules: (a) inverted residual encoder module;(b) inverted residual decoder module
需要注意的是,第2 个的3×3×3 卷积输出通道数是输入通道数的3 倍,这样将输入特征图以高维特征表示可以实现特征信息的拓展.经过学习和提取高维特征之后,第3 个3×3×3 卷积将倒残差模块的输出通道数恢复到模块输入通道数,也同样采用跨卡同步归一化和ReLU 激活函数处理.与此同时,倒残差编码器模块直接使用了短跳跃连接以实现恒等映射.
倒残差解码器模块也有类似处理过程,但因输入和输出通道数不同,解码器模块使用了1×1×1 卷积实现恒等映射.由于残差结构使用的是先降维后升维的“瓶颈形”设计思想,而本文采用的是先升维后降维的“纺锤形”设计思想,故而称其为倒残差模块.
2.4 损失函数
对于三维磁共振图像的脑肿瘤分割任务,待分割的区域仅占整个输入空间的一小部分,这种目标区域和背景区域的分布占比不平衡问题,也影响着分割网络的性能.
为了解决目标区域和背景区域占比不平衡的问题,广义损失函数(Generalized dice loss,GDL)[25]通过引入权重改善了Dice loss 对检测小目标不利的问题.
式中:wl为每类的权重,pln为预测第l类中体素n的值,tln则为相应的真实标注值,L和N分别为类别总数和体素总数.
3 结果与讨论
3.1 实验数据
基于MICCAI 脑肿瘤分割比赛BraTS2018 和BraTS2019[26-27]的训练集,进行脑肿瘤分割网络的训练和学习,将学习到的网络参数用于处理不含真值的BraTS2018 和BraTS2019 验证集,并把分割好的结果上传到公开网站CBICA Image Processing Portal 进行验证,验证结果按区域分为增强型肿瘤(Enhanced tumor,ET)、全肿瘤(Whole tumor,WT)和肿瘤核心(Tumor core,TC)3 类.
BraTS2018 训练集共有285 个病例,其中210个HGG(High-grade glioma)病例和75 个LGG(Lowgrade glioma)病例,验证集共有66 个无真值病例.BraTS2019 训练集共有335 个病例,其中259 个HGG病例和76 个LGG 病例,验证集共有125 个无真值病例.每个病例有T1、T2、T1ce 和Flair 四个模态,每个模态有155 张切片,一张切片的大小是240×240,在推断时填充到160 张切片并将这四种模态的160 张切片作为三维数据整体输入到网络模型中,根据医师标定的标签作为真值对网络模型进行训练和学习,肿瘤分割的标签包括背景(标签0)、坏死和非增强肿瘤(标签1)、瘤周水肿(标签2)和增强性肿瘤(标签4).
3.2 实现细节
实验环境为CPU Intel® Core i9-9900X 3.5GHZ,Ubuntu16.04 操作系统,4 张GTX2080Ti(11GB)的显卡.基于PyTorch 深度学习框架,设置Batchsize为8,最大迭代次数为900,采用He 初始化、ReLU激活函数和跨卡同步归一化,并选择Adam 优化器进行参数的迭代优化,初试学习速率设置为10-3,并设置权重衰减为10-5,学习率随着epoch 的增加而逐渐减小(poly 学习率衰减法).
在训练网络模型时,训练数据集被随机裁剪为128×128×128 分辨率作为输入数据,同时使用了以下的数据增强手段:以 0.5 的概率在轴向、冠状和矢状平面上随机镜像翻转;在[-10°,+10°]范围内随机旋转和[0.9,1.1]范围内的随机强度偏移.
3.3 评价指标
从网络分割精度考虑,采用多指标综合评判方法,分割精度方面采用BraTS 比赛评价指标体系中的相似系数(Dice similarity coefficient)和豪斯多夫距离(Hausdorff95 distance).
式中:TP、FP 和FN 分别为真正例、假正例和假负例,相似系数表示实验分割结果与医师标定标签(真值,GT)的相似度,相似度越高则细分精度越好.
式中:P、T分别为网络预测的肿瘤区和真实标注区域的体素集,p、t分别为两个体素集中的体素点,d(p,t)是两个体素点的距离,sup 和inf 分别为取上限函数和取下限函数.豪斯多夫距离表示分割结果与真值之间的最大距离,绝对值越小,分割效果越吻合.
3.4 消融实验
为了验证前置跳跃连接的有效性和泛化性,本文采用3 个常见脑肿瘤分割网络(DMF Net、HDC Net、3D U-Net)[13,23-24],分别加入前置跳跃连接进行改进,并在BraTS2018 和BraTS2019 的两个验证集上进行消融实验.在此基础上,本文还设置了FS Inv-Res U-Net 的消融实验.
3.4.1 前置跳跃连接
前置跳跃连接的消融实验如表1 所示.由Bra-TS2018 的消融实验可知,前置跳跃连接对3 个基础网络的多数性能指标有提升,如ET、TC 区域的Dice 指标,其中DMF Net+FS 在ET 区域的Dice 指标提升了0.73%,3D U-Net+FS 在WT、TC 区域的Dice 指标分别提升了约0.29%和1.17%,在ET、WT区域的豪斯多夫距离也有优化效果.由BraTS2019的消融实验可知,在加入前置跳跃连接后,3 个网络在ET 区域的Dice 指标分别提升了0.21%、0.02%和0.49%;在WT 区域的Dice 指标分别提升了0.07%、0.48%和0.43%;在TC 区域的Dice 指标分别提升了0.26%、1.06%和0.78%,这充分表明前置跳跃连接的有效性和泛化性.尤其是3D U-Net+FS 在ET区域的Dice 指标突破了78%,且3 个区域的平均Dice 值达到了最优指标的83.55%,远远超过同类的先进网络.
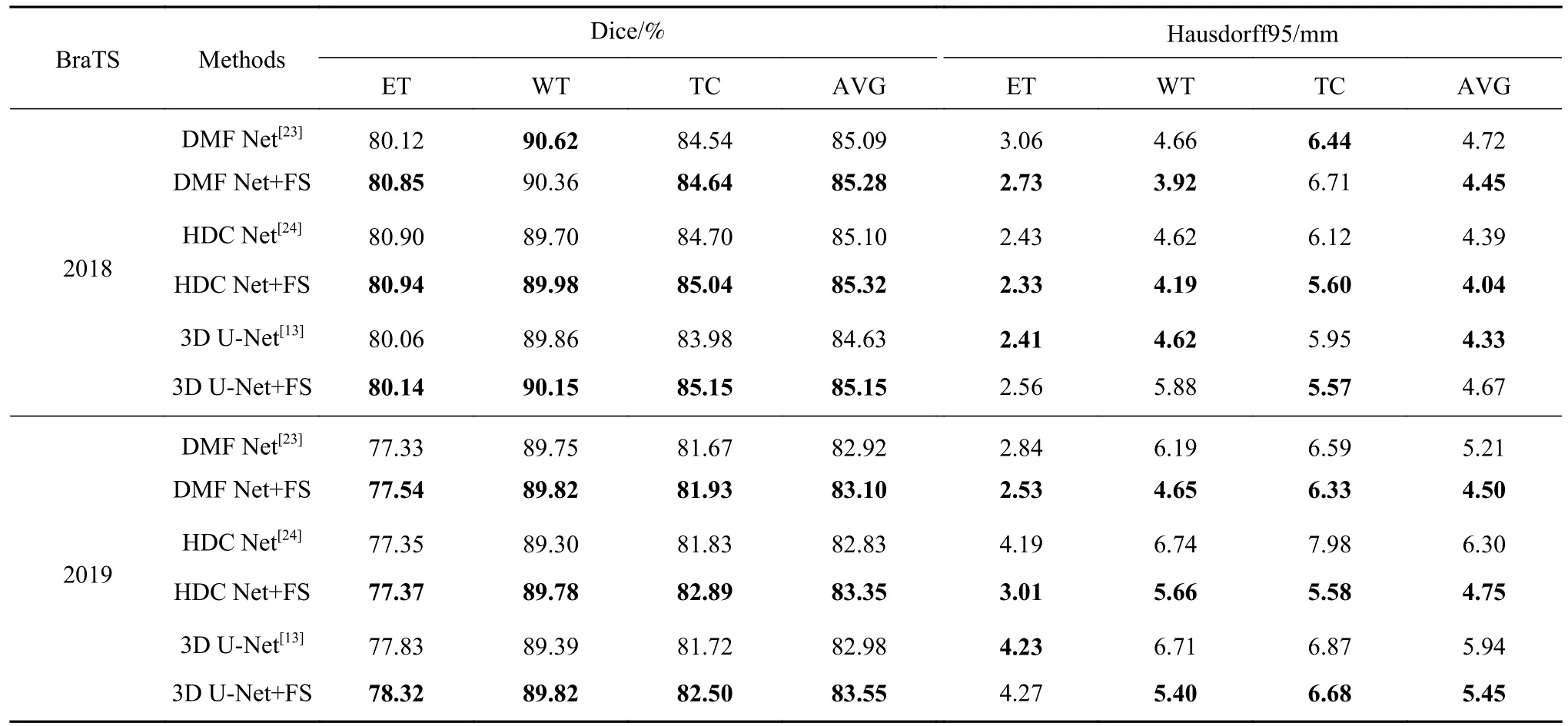
表1 前置跳跃连接的消融实验Table 1 Ablation experiments of front-skip connections
图3 展示的是前置跳跃连接的消融实验分割图,采用BraTS19_CBICA_AUW_1 病例,三维坐标是(80,131,77).图4 展示的是FS Inv-Res U-Net 的消融实验分割图.如图3 和图4 的左3 列分割图所示,在加入前置跳跃连接之后,各网络都降低了分割图中的分类错误,也缓解了子区域大小与真值大小不一致的问题,这些分割图表明前置跳跃连接的有效性与可泛化能力.
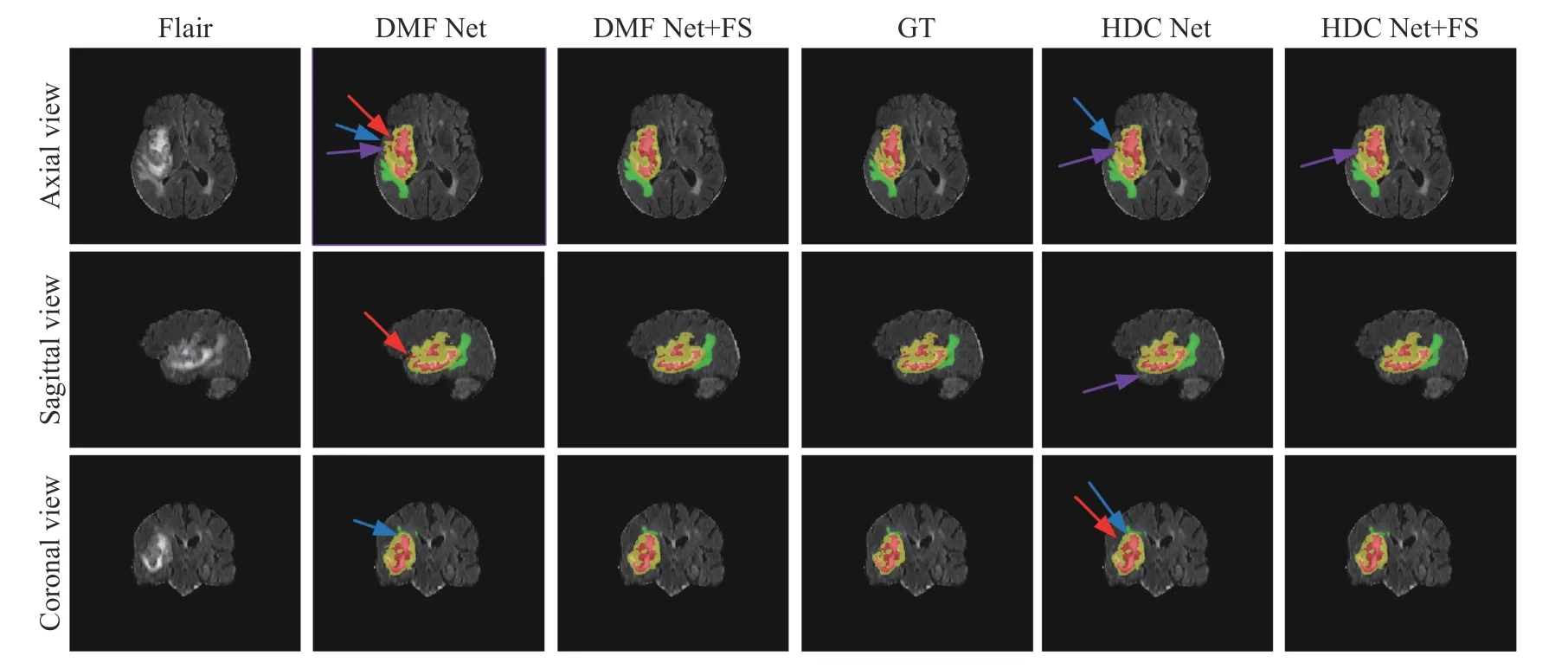
图3 前置跳跃连接的消融实验(FS 表示前置跳跃连接,GT 表示标准真值,红色箭头表示错误分类区域,蓝色箭头表示边界信息丢失,紫色箭头表示子区域与真值区域大小不同)Fig. 3 Ablation experiment of front-skip connections (FS indicates pre-skip connections,GT indicates ground truth,red arrows indicate misclassified regions,blue arrows indicate loss of boundary information,and purple arrows indicate subregions with different sizes than ground truth regions)
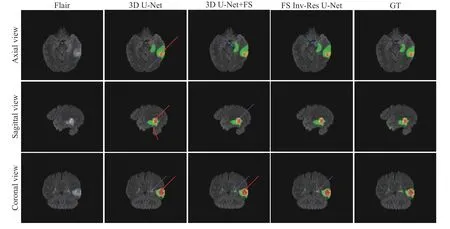
图4 FS Inv-Res U-Net 的消融实验(FS 表示前置跳跃连接,GT 表示标准真值,红色箭头表示错误分类区域,蓝色箭头表示边界信息丢失,紫色箭头表示子区域与真值区域大小不同)Fig. 4 Ablation experiments of FS Inv-Res U-Net (FS indicates the front-skip connection,GT indicates the ground truth,red arrows indicate misclassified regions,blue arrows indicate loss of boundary information,and purple arrows indicate subregions with different sizes than ground truth regions)
3.4.2 FS Inv-Res U-Net 消融实验
FS Inv-Res U-Net 的消融实验如表2 所示,本文选取3D U-Net 作为基础网络设计出FS Inv-Res U-Net,分别在数据集BraTS2018 和BraTS2019 的验证集上验证前置跳跃连接和FS Inv-Res U-Net网络的有效性.在加入前置跳跃连接之后,3D UNet 在BraTS2018 和BraTS2019 上的验证集Dice 指标都有提升,分别是0.08%、0.29%和1.17%以及0.49%、0.43%和0.78%.豪斯多夫距离在BraTS2019验证集上WT 区域和TC 区域都有改善,分别是1.31 mm 和0.19 mm,在BraTS2018 上仅有TC 区域减少了0.38 mm,表明了前置跳跃连接在常见3D U-Net 上的提升效果主要针对Dice 指标的3 个区域,对豪斯多夫距离的改善表现在3 个区域的平均值上.相较于3D U-Net 与前置跳跃连接的组合,本文提出的FS Inv-Res U-Net 的Dice 指标和豪斯多夫距离指标在BraTS2018 上的3 个区域分别提高了0.09%、0.15%和0.3%,减小了0.21、1.11 和0.07 mm,在BraTS2019 上的ET、TC 的Dice 指标分别提升了0.06%和0.51%,豪斯多夫距离分别减小了0.27 mm 和0.41mm.
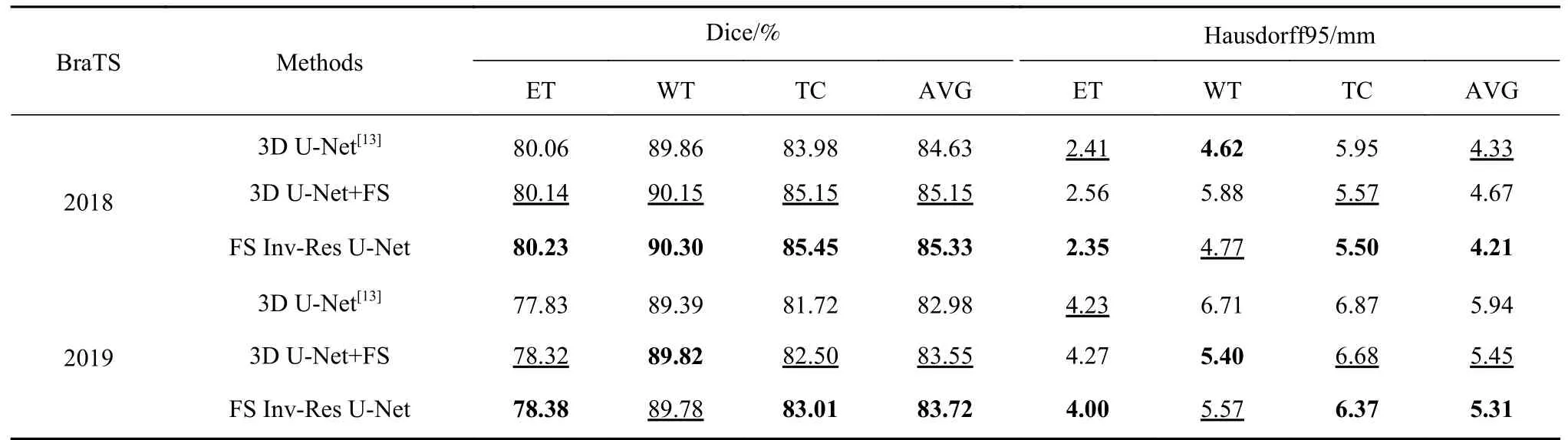
表2 FS Inv-Res U-Net 的消融实验Table 2 Ablation experiments of FS Inv-Res U-Net
图4 展示的是FS Inv-Res U-Net 消融实验分割图,采用的是BraTS19_CBICA_BGT_1 病例,三维坐标是(168,153,54).其第2 列3D U-Net 分割图依旧存在着标签分类错误和子区域与真值区域大小不同的问题,其中标签分类错误问题在横断位、矢状位、冠状位都存在.在加入前置跳跃连接之后,标签分类错误问题大大降低,但仍存在子区域大小和真值区域大小不一致现象.由本文设计的FS Inv-Res U-Net 分割图(第4 列)可知,虽然仍存在边界信息损失和区域不一致问题,但问题仅出现在其中的两个状位且影响较小,分类错误问题得到大大缓解,这表明FS Inv-Res U-Net 分割图更加接近真值.
3.5 网络对比
FS Inv-Res U-Net 与其他先进网络对比表如表3所示,本文分别选择了BraTS2018、BraTS2019 数据集上具有代表性的先进方法作为对比网络.
由表3 可知,FS Inv-Res U-Net 在BraTS2018 验证集上的Dice 指标ET、WT 和TC 区域都取得了较优结果,低于BraTS2018 比赛的第一名NVDLMED 团队[31]约1.5%、0.38%和0.57%,但在豪斯多夫距离指标的ET、TC 区域和3 个区域的平均值上,FS Inv-Res U-Net 都取得了最优值,相较于NVD-LMED 团队减小了1.47、1.35 和0.75 mm,这表明FS Inv-Res U-Net 网络的先进性,具备出色的分割性能.在BraTS2019 验证集上,FS Inv-Res U-Net 取得了3 个区域Dice 平均值的最优性能,高于BraTS-2019 比赛的第二名Zhao 团队[35]约0.42%,尤其ET区域的Dice 指标明显超过其他先进方法,超过Zhao团队2.98%,豪斯多夫距离指标的WT 和TC 区域都取得了次优结果,综合性能较为优异.CA Net 仅在TC 区域取得了最优的Dice 结果,但在ET 区域落后FS Inv-Res U-Net 网络约2.48%,WT 区域落后FS Inv-Res U-Net 网络约1.28%,且CA Net 的3个区域Dice 均值比FS Inv-Res U-Net 低了0.55%.

表3 FS Inv-Res U-Net 与其他先进网络的对比Table 3 Comparison of FS Inv-Res U-Net with other state-of-the-art networks
图5 展示的是多个病例下的不同分割方法的分割对比图,从左到右分别是Flair 序列、真值、DMF Net、HDC Net 和FS Inv-Res U-Net,从上到下是BraTS2019 训练集中的5 个随机病例.图5 中DMF Net 的分割图出现较多的分割错误,其中Bra-TS19_CBICA_AUW_1 分割图出现了3 类分割错误,HDCNet 的分割图出现的错误大多是分割标签分类错误,但是FS Inv-Res U-Net 更加地接近真值分割图.
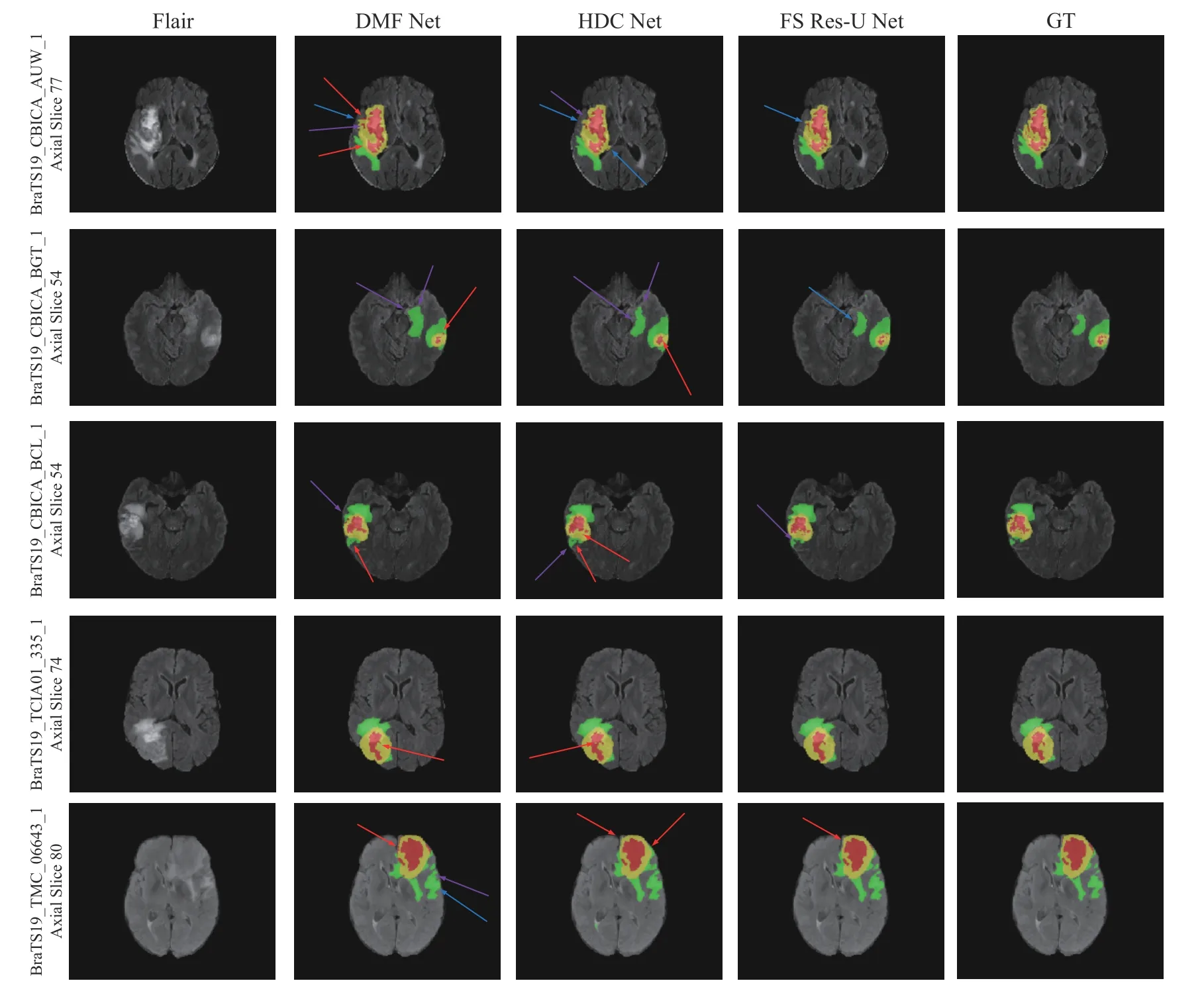
图5 不同方法的分割图对比(红色箭头表示错误分类区域,蓝色箭头表示边界信息丢失,紫色箭头表示子区域与真值区域大小不同)Fig. 5 Comparison of different methods’ segmentation maps (Red arrows indicate misclassified regions,blue arrows indicate loss of boundary information,and purple arrows indicate subregions with different sizes than ground truth regions)
4 结论
本文通过对脑肿瘤分割方法的深入分析和对前置跳跃连接的实验探究,在探讨了前置跳跃连接可行性的基础上,设计出前置跳跃连接倒残差U 形网络(FS Inv-Res U-Net).前置跳跃连接的消融实验表明该构想的有效性和泛化性,也表明其具有即改即用的特点.同时FS Inv-Res U-Net 网络在BraTS 验证集上获得了检验,BraTS2018 的验证结果在增强型肿瘤、全肿瘤和肿瘤核心上的Dice值分别是80.23%、90.30%和85.45%,豪斯多夫距离分别是2.35、4.77 和5.50 mm;BraTS2019 的验证结果在增强型肿瘤、全肿瘤和肿瘤核心上的Dice值分别是78.38%、89.78%和83.01%,豪斯多夫距离分别是4、5.57 和6.37 mm,表明FS Inv-Res U-Net网络可用于精确分割脑肿瘤.但同时由于提出的前置跳跃连接并未跳出编码器-解码器结构限制,为了验证进一步前置跳跃连接的泛化性,在未来工作中,可将前置跳跃连接改进FCN 等其他适用网络架构,且本文提出的前置跳跃连接倒残差U形网络是一个较为基础的网络模型,未来也会引进其他高效卷积模块到该网络中.

