滇龙胆居群遗传多样性和遗传结构分析
沈 涛 ,虞 泓*,王元忠
1.玉溪师范学院化学生物与环境学院,云南 玉溪 653100
2.云南大学生态学与环境学院云百草实验室,云南 昆明 650091
3.云南省农业科学院药用植物研究所,云南 昆明 650200
分子标记可直接检测DNA 分子上的遗传变异,是探索药用植物遗传多样性与居群遗传结构的重要工具[2]。近年单核苷酸多态性(single nucleotide polymorphism,SNP)因其在基因组中位点多、分布广,具有较好的多态性和较高的遗传稳定性,被国内外研究者所关注[11-13];全基因组测序有助于获取高质量SNP 数据,但与农作物相比多数药用植物研究面临基因组测序基础薄弱、分析缺乏模式物种及有效参考基因、后续SNP 标记开发难度大等问题[14-16]。近年GBS(genotyping-by-sequencing)简化基因组(reduced-representation genome sequencing,RRGS)技术的发展成为解决上述问题的重要途径[17-18]。Otto 等[19]通过GBS 技术确定了药用植物母菊Matricaria chamomillaL.的遗传结构,并通过全基因组关联作图鉴定了与植株花期、药效成分积累相关的SNP 位点。Qiao 等[7]基于GBS技术与SNP 标记,分析了四川、青海等地川西獐牙菜Swertia mussotiiFranch.的遗传结构,同时结合分布区环境数据,探讨了物种遗传多样性形成的可能机制。此外,张笑[20]则利用GBS 技术结合物种生态位模拟,开展绞股蓝Gynostemma pentaphyllum(Thunb.)Makino 居群遗传学与进化历史研究。以上报道表明GBS 与SNP 技术相结合的分析策略,可为传统药用植物资源多样性评价提供新的方法和研究思路。
滇龙胆Gentiana rigescensFranch.ex Hemsl.为传统保肝中药龙胆的主要植物来源,云南及周边地区是其药材主产地[21-22]。作为云南边疆少数民族山区脱贫攻坚和乡村振兴的特色药用植物,开展滇龙胆种质资源多样性评价对其药用资源的永续利用及科学保护具有重要意义。当前滇龙胆资源学研究主要集中于化学评价[23-25],种质资源遗传多样性与调查评估鲜有报道[26-27]。国内学者利用核糖体DNA(rDNA)ITS 序列和多个叶绿体DNA(cpDNA)序列片段,初步探讨了云南滇龙胆野生居群的遗传变异与分化,发现滇龙胆对环境适应性较强,不同居群间遗传关系较复杂[26-27]。滇龙胆分布区跨越横断山区与云贵高原,区域内从南至北伴随海拔逐渐升高,生境气候条件也呈现出较大差异[28];不同环境条件滇龙胆居群的遗传多样性及其变化有待探明。
本研究以生长于横断山区和云贵高原的野生滇龙胆为材料,基于GBS 简化基因组测序和SNP 分子标记,研究滇龙胆居群遗传多样性与遗传结构;探讨异质环境对该物种遗传分化的影响作用。研究结果为滇龙胆种质资源多样性评价,野生资源保护及遗传资源的发掘利用提供理论依据。
1 材料与仪器
1.1 材料
2019—2020 年对云南、贵州和四川的19 滇龙胆居群147 株健康野生植株进行采样;所有样品经云南大学虞泓教授鉴定为滇龙胆G.rigescensFranch.ex Hemsl.。采样居群10 个分布于横断山区,9 个分布于云贵高原;采样信息详见表1。居群内采样个体间距离不小于10 m;个体数较少的居群采样4~5 株,其余居群采样数>9 株;每株个体采摘带叶柄新鲜无病害叶10~15 片,去除叶面灰尘后迅速放入独立的分子材料袋内,包埋于洁净的变色硅胶中干燥保存。凭证标本保存于云南省农业科学院药用植物研究所标本馆,居群信息详见表1。
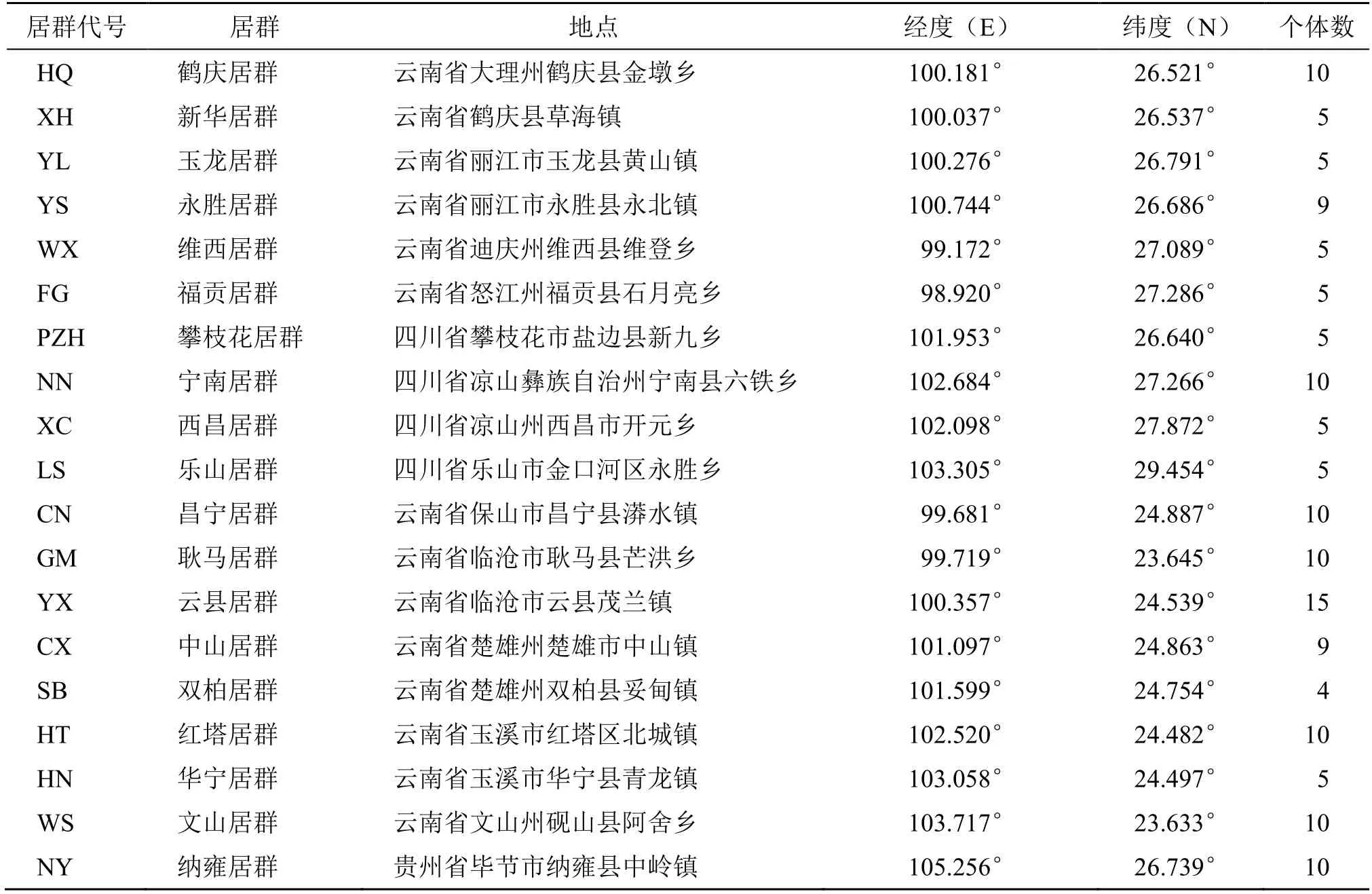
表1 滇龙胆居群采样信息Table 1 Sampling information of G.rigescens populations
1.2 仪器
Agilent 2100 Bioanalyzer 型生物分析仪(美国安捷伦公司)、NanoPhotometer N60 型微量分光光度计(德国因普恩公司)、Invitrogen Qubit Flex 型Qubit荧光光度计(美国赛默飞公司)、VeritiPro 型PCR仪(美国赛默飞公司)。
2 方法
2.1 样品DNA 提取
采用CTAB 法,从低温保存的干燥叶片中提取滇龙胆测序所需基因组DNA[11,29]。测序前,提取的DNA 均使用1.00%的琼脂糖凝胶进行电泳,结合微量分光光度计和Qubit荧光光度计检测DNA的浓度(OD)和纯度[30]。
百年风雨,沧桑巨变,故宫从辉煌到离乱再到新生的路途,又何尝不是中华民族百年起伏的投影与写照?凤凰涅槃,浴火重生,中华民族伟大复兴的征途,将由我们写就,让我们昂首阔步,勇敢前行。
2.2 文库构建与测序
滇龙胆基因组信息较缺乏,因此研究采用无参考基因的GBS 技术进行测序。文库构建与测序主要参考Sonah 等[31]的方法。建库流程主要包括DNA酶切、接头链接、片段筛选、PCR 文库富集及纯化等流程[32]。测序工作由Illumina NovaSeq 6000 测序平台完成,测序策略为双端测序(paired-end,PE),每端150 bp;以上GBS 测序工作在广州基迪奥生物科技有限公司内完成。
2.3 数据过滤与SNP 挖掘
SNP 分析前为保证数据质量需对测序后原始数据进行数据过滤并减少噪音;数据过滤过程中,含有接头(adapter)、无法识别碱基(N 碱基)或含有较大比例低质量碱基的片段(reads)均需要处理和剔除[20]。以上分析由数据质控软件fastp(Version:0.18.0)完成。利用BWA(Version:0.7.12)软件进行对比;使用变异检测软件GATK(Version:3.4.46)进行群体SNP 检测,并对原始数据的质量进行过滤;在此基础上用ANNOVAR(Version:2.0)分析基因组数据中的遗传变异,并对检测出的变异进行功能注释;使用VCFtools(Version:0.1.13)依据文献设定参数对数据再次进行数据质量过滤[7];最终获得高质量的SNP 数据集,用于滇龙胆的居群遗传学分析。
2.4 遗传多样性与遗传结构分析
选取近年文献报道较多的指标[7,9],利用perl 脚本与样品SNP 信息计算居群的观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、多态信息含量(polymorphic information,PIC)、Shannon 多样性指数(shannon’s diversity index,I)、Nei’s 多样性指数(Nei’s gene diversity,Nei’s)、核苷酸多样性(nucleotide diversity,π)、遗传分化系数(genetic differentiation index,FST)和基因流(gene flow,Nm)。
采用 Stack(Version 1.43)软件与邻接法(neighbor-joining methods,NJ)构建进化树;GCTA(genome-wide complex trait analysis)软件用于主成分分析(principal component analysis,PCA);利用ADMIXTURE 软件(Version 1.3.0)结合SNPs 数据集和最大似然法(maximum-likelihood methods,ML)对滇龙胆居群的遗传结构进行推算;预先设定K值为1~10,选取最低交叉验证错误率对应的K作为最优分群方法,用于推测居群个体可能的分组。
2.5 环境因子分析与Mantel 检验
利用ArcGIS 提取19 个居群对应的水分、热量、UV-B 辐射等环境因子(表2)数值分析横断山区与云贵高原滇龙胆分布区的环境差异。环境变量中生物气候变量(Bio01~Bio19)和活动积温(10AAT)分别由WorldClim(https://worldclim.org)与中国科学院资源环境与数据中心(https://www.resdc.cn/)提供,UV-B辐射相关数据源自glUV 网站(https://www.ufz.de/gluv/)。Mann-Whitney U 检验用于不同分布区环境因子的差异比较;基于差异显著的环境因子计算不同居群间的环境距离(Environmental distance,ED),利用居群经纬度信息计算居群间地理距离(Geographical distance,GD)[33-34];本研究中ED 和GD 均为欧式距离。Mantel 检验用于分析居群间FST与地理距离、环境距离间的相关性[34]。Mann-Whitney U检验由IBM SPSS Station 25.0 计算;Mantel 检验在XLSTAT(Version 2019.2.2)中完成;其余统计分析由SIMCA(Version 14.1)完成。
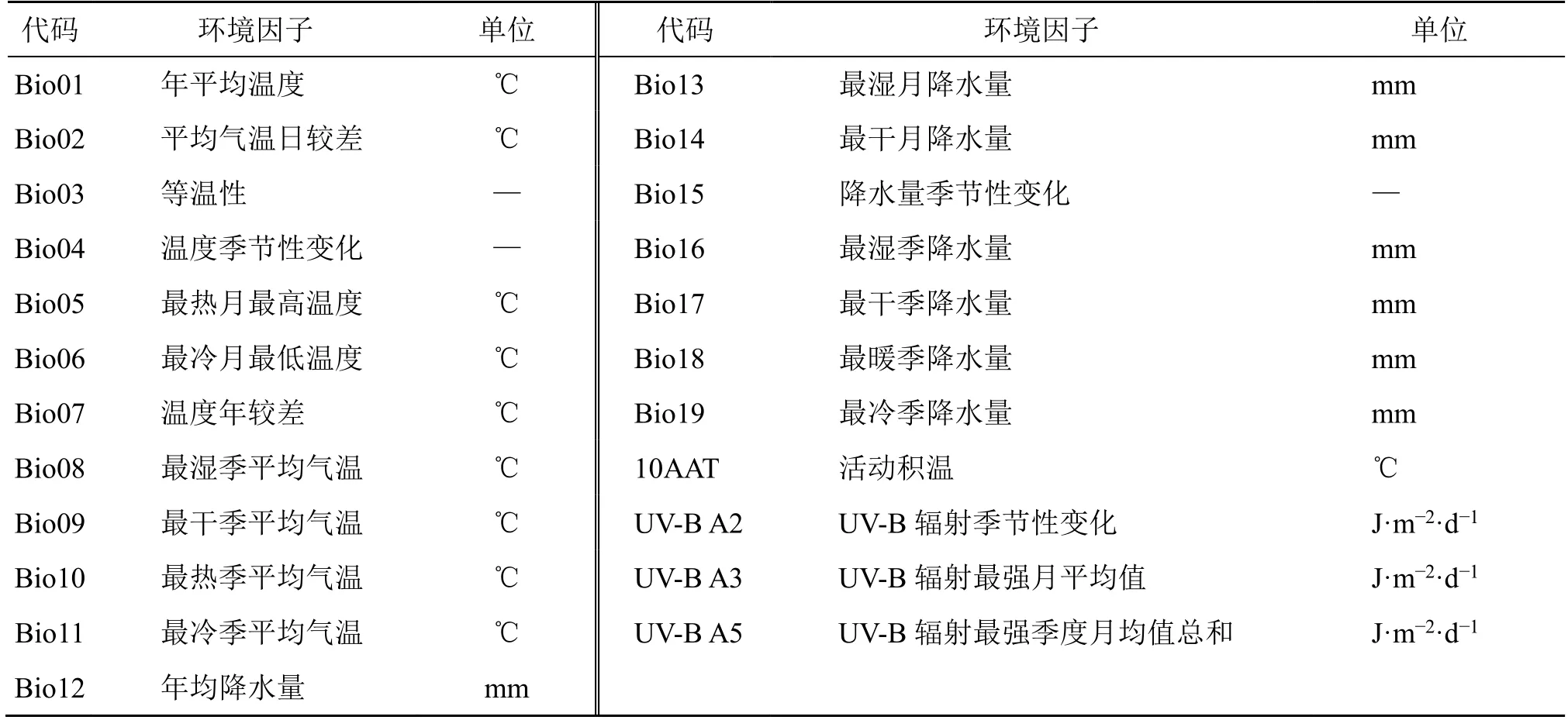
表2 研究涉及环境因子Table 2 Environment factors used in the study
3 结果与分析
3.1 测序数据统计结果
滇龙胆GBS 简化基因组测序结果统计显示,数据经过滤后,数据集测序质量值Q20(碱基错误率<1.00%)平均值为97.81%,测序质量值Q30(碱基错误率<0.10%)平均值为93.55%,平均CG 含量为41.07%,N 碱基含量为0.00%,表明测序结果出错率低,数据总体质量较高。测序样品高质量序列(HQ clean reads)条数均值为10 923 810,中位数为9 527 980,每份样品平均测序序列数据量约1.55 Gb。
3.2 滇龙胆居群遗传结构
基于筛选后的SNPs 数据集,运用邻接法构建19 个居群147 株滇龙胆的系统树。系统树分析结果显示(图1),不同地理来源的样品可大致分为2 个分支。第1 分支主要是生长在云贵高原的滇龙胆,包括滇中的中山(CX)、双柏(SB)、红塔(HT)居群,滇西昌宁(CN)、耿马(GM)、云县(YX)居群以及贵州西北部的纳雍(NY)居群部分个体;此外四川乐山(LS)居群的滇龙胆也被聚到第1 个分支中。第2 分支主要为云贵高原以北地区分布的滇龙胆;包括滇西北福贡(FG)、维西(WX)、鹤庆(HQ)、新华(XH)、玉龙(YL)、永胜(YS)居群,川西南的攀枝花(PZH)、宁南(NN)和西昌(XC)居群;除上述地区的滇龙胆,滇中华宁(HN)聚居及滇东南文山(WS)居群的样品也被聚到第2分支中。系统树显示,同一居群的样品基本聚在一起,相近地理区域的样品多聚为一类,分布于云贵高原和滇西北横断山区的植株聚为不同分支,表明上述地区的滇龙胆在遗传上呈现出较明显的地域分异。
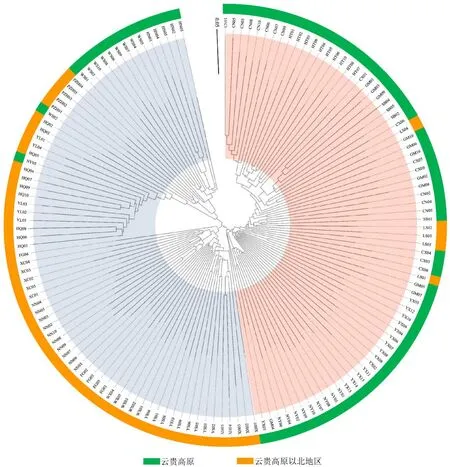
图1 滇龙胆19 个居群147 株个体基于SNPs 数据集聚分析Fig.1 Cluster analysis of 147 individuals in 19 populations of G.rigescens based on the SNPs data set
PCA 三维得分图显示(图2),所有样品依据得分大致聚为4 组。第I 组样品主要由云南鹤庆(HQ)、玉龙(YL)2 个居群的滇龙胆构成;第II 组样品主要由云南新华(XH)、永胜(YS)、福贡(FG)、维西(WX)及四川西昌(XC)、宁南(NN)居群的植株构成;第 III 组样品主要为四川攀枝花(PZH)、云南玉溪(HN)和文山(WS)居群的植株;第IV 组样品为云南南昌宁(CN)、临沧(YX、GM)、楚雄(CX、SB)、玉溪(HT)及四川乐山(LS)、贵州纳雍(NY)的居群的滇龙胆构成。
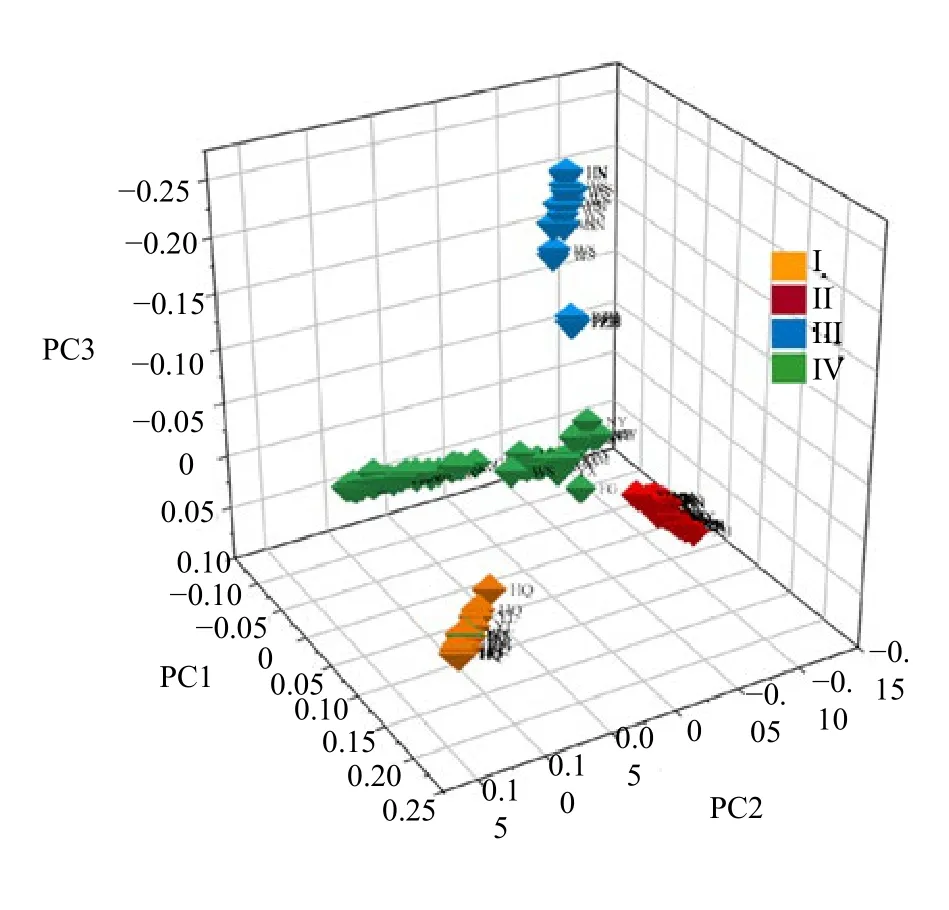
图2 滇龙胆19 个居群147 株个体SNPs 数据集主成分分析Fig.2 PCA of 147 individuals in 19 populations of G.rigescens based on SNPs data set
19 个滇龙胆居群的遗传结构分组研究发现,随K值增加模型交叉验证误差逐步降低;当K为4 时误差值达到谷值,因此K=4 是最优分群方法(图3)。
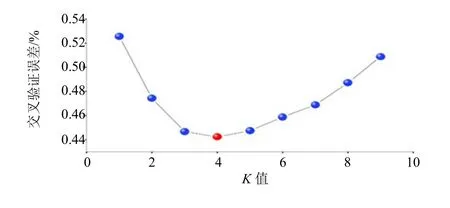
图3 滇龙胆19 个居群147 株个体SNPs 数据集遗传结构最佳K 值筛选Fig.3 Optimum K value selection for genetic structure of 147 individuals in 19 populations of G.rigescens based on SNPs data set
基于最优分组结果发现,19 个居群的基因型从地理上呈现出明显的地域特征(图4)。4 个亚群中,红色基因型个体主要存在于鹤庆(HQ)和玉龙(YL)2个居群中;橙色基因型个体主要生长在滇西北和川西南地区(攀枝花居群除外)。保山昌宁、楚雄双柏、玉溪红塔及四川乐山居群中蓝色基因型占主导地位;四川攀枝花、云南华宁和云南文山3 个居群植株均以绿色基因型为主。云南耿马、云县及贵州纳雍居群具有明显的种质混杂,其组成可能来源于多个理论祖先亚群。
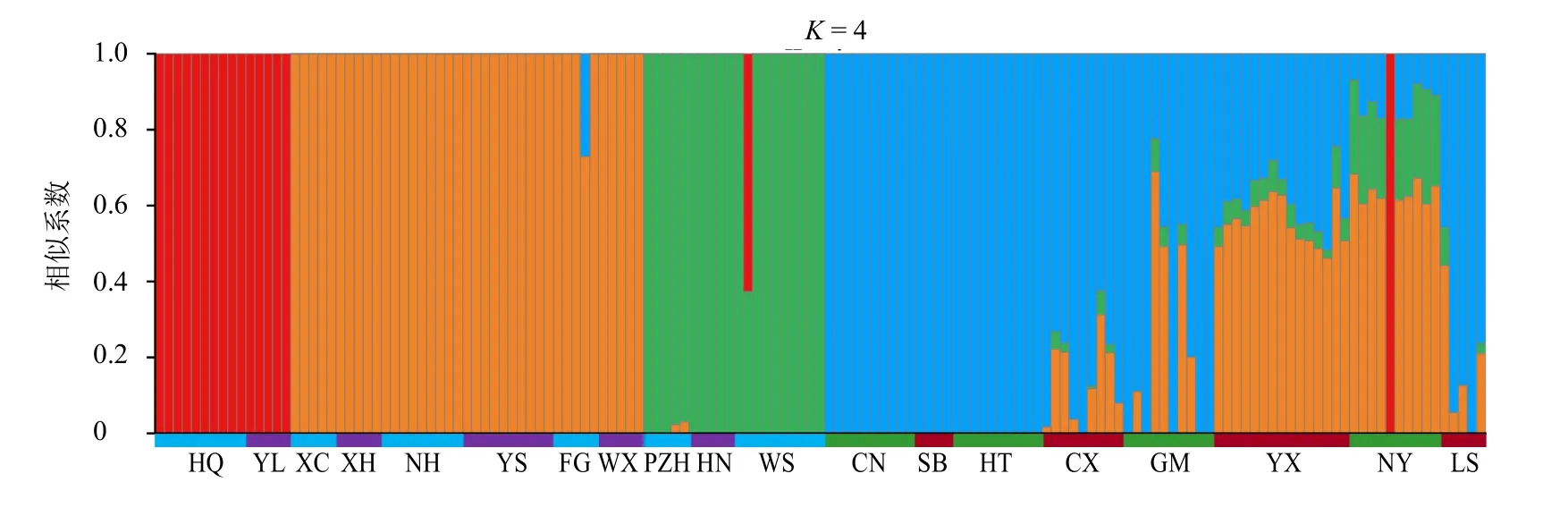
图4 滇龙胆19 个居群的遗传结构分组 (K=4)Fig.4 Genetic structure analysis of 19 Gentiana rigescens population (K=4)
综合以上分析结果认为以滇西昌宁(CN)—黔西纳雍(NY)一线为界,滇龙胆居群遗传结构在云贵高原和横断山区间呈现出明显的南北差异。依据研究样品地理来源和简化基因组分析结果,19 个居群的滇龙胆有4 种基因型,并可划分为北部(共计10 个居群)和南部(共计9 个居群)2 个组。北部组主要分布在横断山区(滇西北和川西南),组内基因型I 和基因型II 占比较高;南部组主要分布于云贵高原,组内基因型III 和基因型IV 占比较高。
3.3 居群遗传多样性
3.3.1 横断山区居群遗传多样性 对分布于横断山区的10 个滇龙胆居群遗传多样性参数进行计算,结果显示(表3),10 个居群6 个遗传参数均值分别为Ho=0.051(数值0.040~0.070),He=0.098(数值0.085~0.111),PIC=0.077(数值0.067~0.088),I=0.144(数值0.125~0.163),Nei’s=0.114(数值0.100~0.133),π=1.175×10-3(数值0.823×10-3~1.625×10-3)。
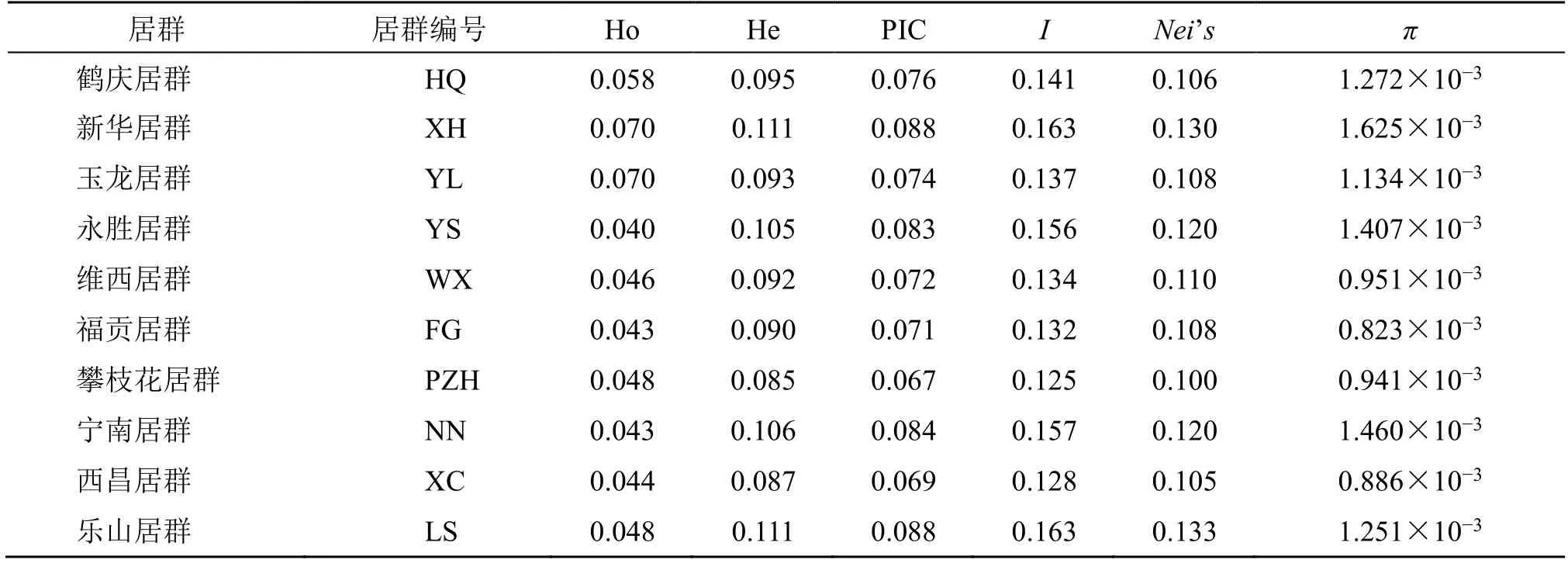
表3 横断山及周边地区滇龙胆10 个居群的遗传多样性参数Table 3 Genetic diversity parameters of 10 population of G.rigescens in Hengduan Mountains and its surrounding areas
3.3.2 云南贵高原居群遗传多样性 分布于云贵高原的滇龙胆居群遗传多样性参数统计结果显示(表4),9 个居群遗传参数均值分别为:Ho=0.052(数值0.042~0.072),He=0.110(数值0.088~0.141),PIC=0.088(数值0.071~0.113),I=0.164(数值0.132~0.211),Nei’s=0.127(数值0.098~0.161),π=1.677×10-3(数值1.209×10-3~2.261×10-3)。
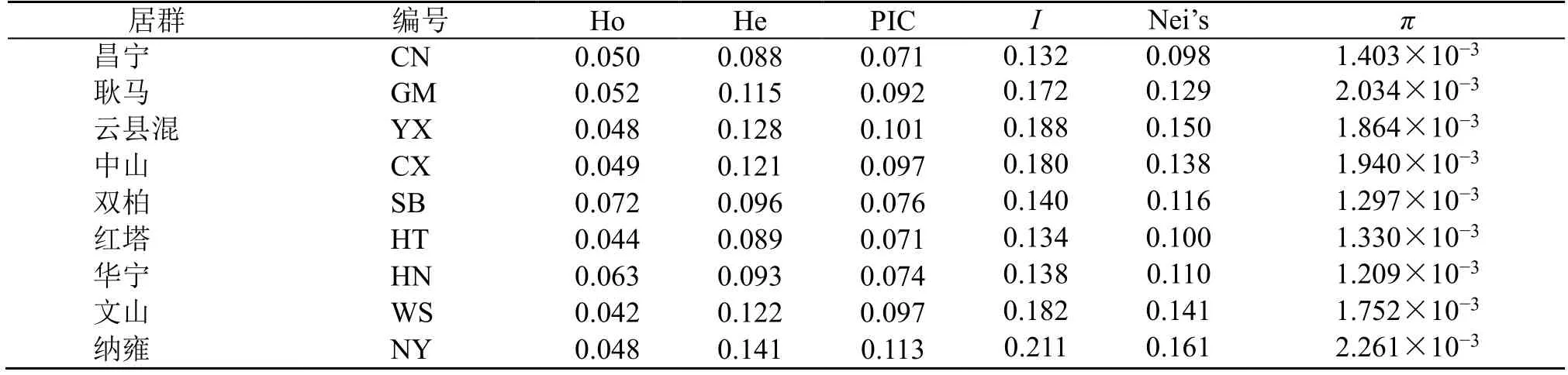
表4 云贵高原滇龙胆9 个居群的遗传多样性参数Table 4 Genetic diversity parameters of nine population of G.rigescens in Yunnan-Guizhou Plateau
19 个居群遗传多样性参数总体计算显示,Ho=0.037,He=0.268,PIC=0.223,I=0.426,Nei’s=0.279,π=1.399×10-3。综合比较横断山区居群和云贵高原居群He、No、I、Nei’s、PIC 和π6 个遗传多样性参数的数值变化,并对2 组数据进行Mann-Whitney U 检验(图5)。均值和中位数比较发现,横断山区滇龙胆居群的遗传多样性较云贵高原居群低,其中π值组间差异显著(Mann-Whitney U 检验P<0.05)。
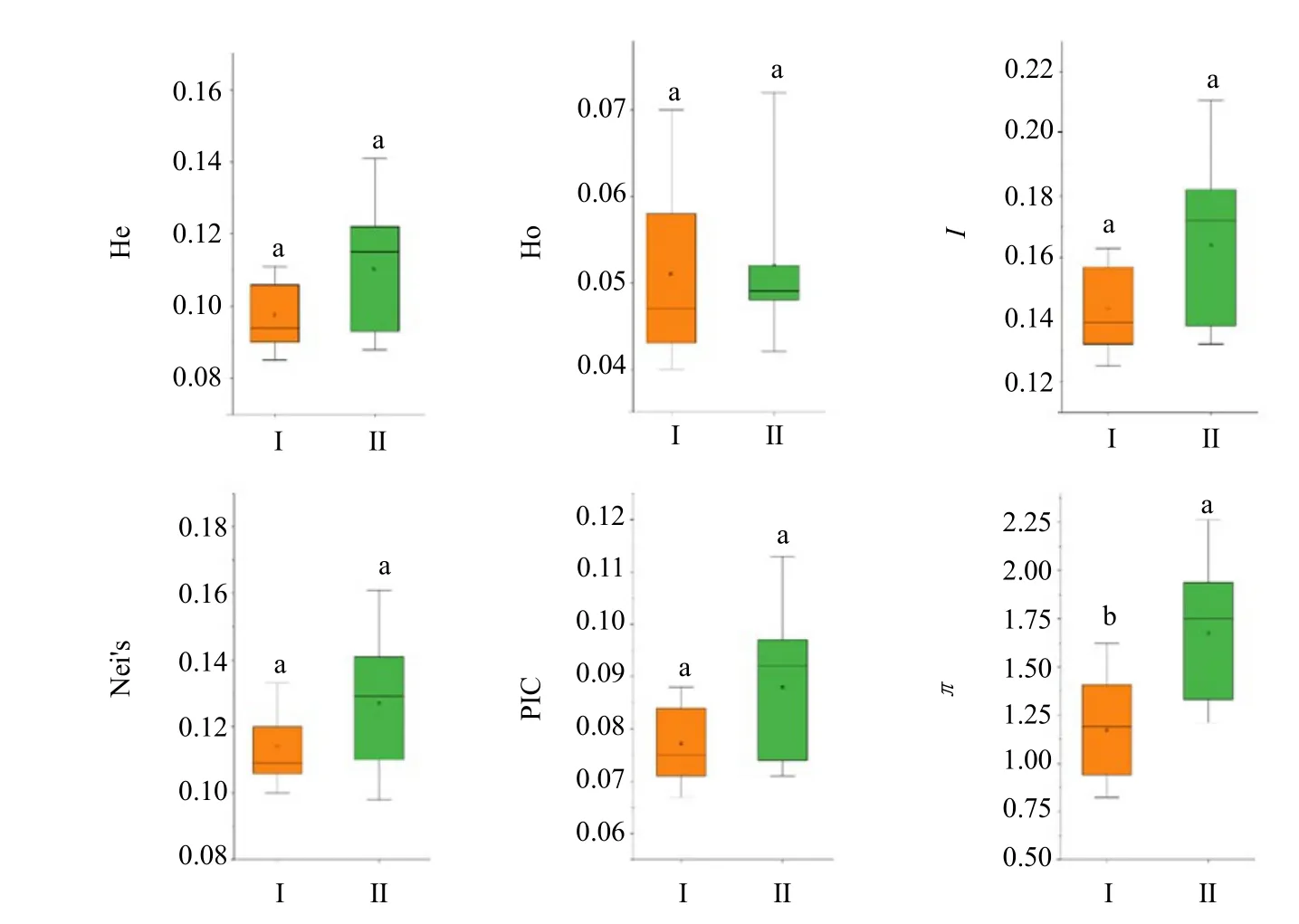
图5 横断山区与云贵高原滇龙胆居群遗传多样性比较Fig.5 Genetic diversity comparison between the Hengduan Mountains population and the Yunnan-Guizhou Plateau population of G.rigescens
3.4 遗传分化与基因流
3.4.1 横断山区居群 横断山及周边地区10 个滇龙胆居群FST的均值为0.471,FST数值变化范围0.163~0.580,鹤庆(HQ)居群与玉龙(YL)居群间FST值最小,攀枝花(PZH)居群与玉龙(YL)居群间FST值最大(表5)。除鹤庆、玉龙2 个居群,横断山区滇龙胆群间总体呈现出中等或较高水平的遗传分化。
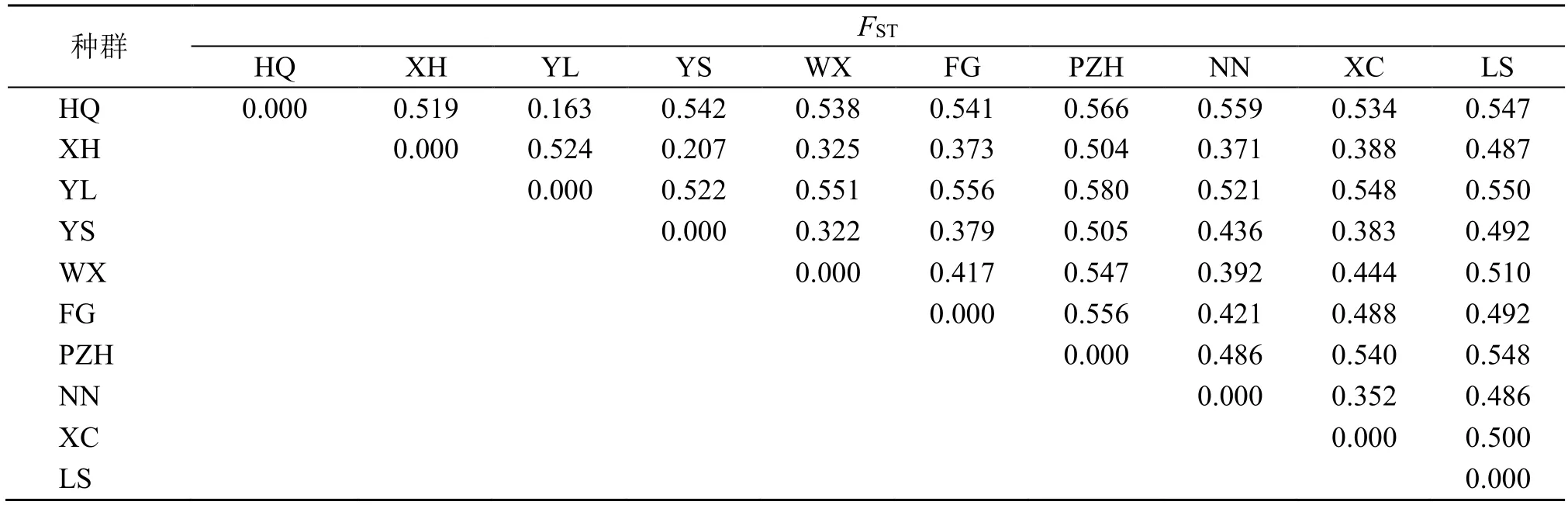
表5 横断山及周边地区滇龙胆10 个居群的FST 数值Table 5 FST value between 10 population of G.rigescens in Hengduan Mountains and its surrounding areas
Nm计算显示(表6),横断山区10 个居群间Nm数值介于0.181~1.279;仅鹤庆(HQ)居群与玉龙(YL)居群间Nm值大于1.000,其余44 对居群的Nm值均小于1.000。Nm数据分析显示,除滇西北少数居群外,横断山区大部分居群间的基因交流均受到不同程度的阻隔。
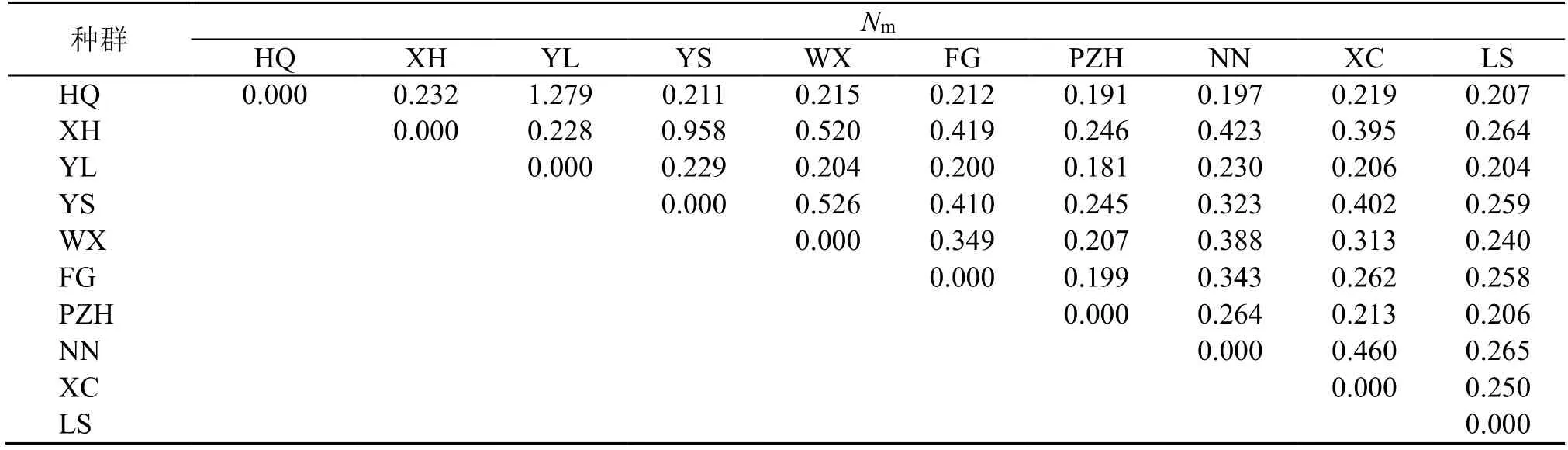
表6 横断山及周边地区滇龙胆10 个居群的Nm 数值Table 6 Nm value between 10 population of G.rigescens in Hengduan Mountains and its surrounding areas
3.4.2 云南贵高原居群 云贵高原9 个滇龙胆居群FST均值为0.422,FST数值0.227~0.576,中山(CX)居群与双柏(SB)居群间FST值最小,南华(NH)居群与昌宁(CN)居群间FST值最大。贵州纳雍的居群(NN)与云贵高原其它地区的居群呈现出不同程度的遗传分化,居群间FST值在0.373~0.495(表7)。
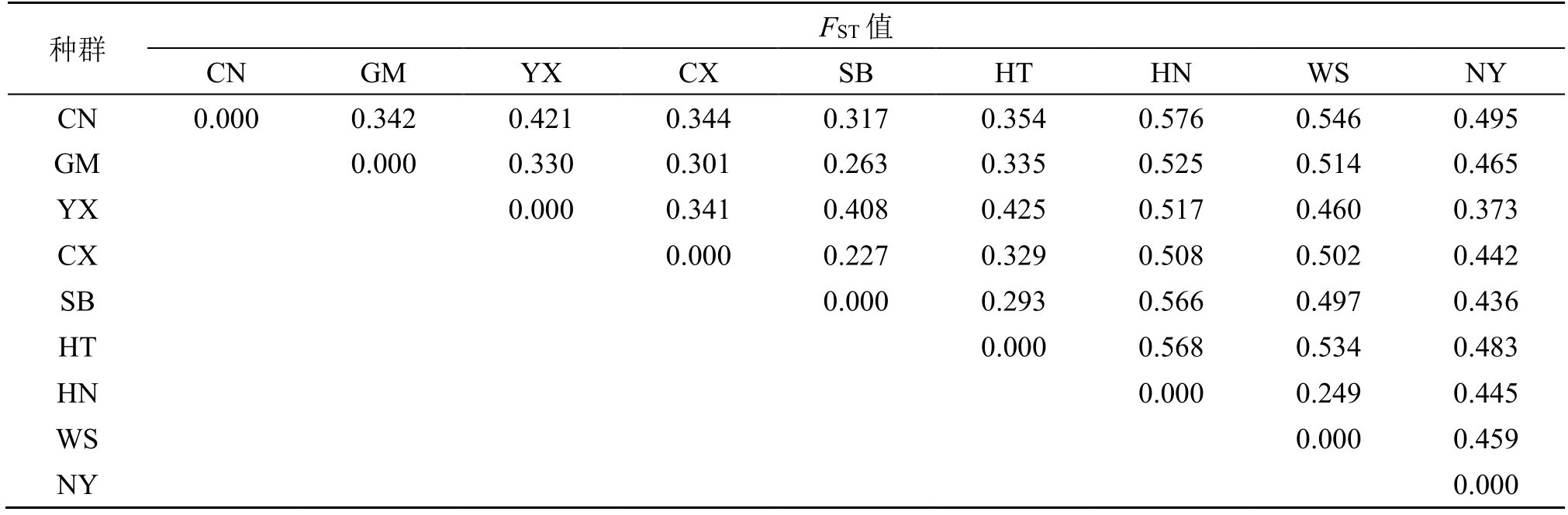
表7 云贵高原滇龙胆9 个居群的FST 数值Table 7 FST value between nine population of G.rigescens in Yunnan-Guizhou Plateau
Nm分析显示(表8),云贵高原9 个居群间Nm值变化范围:0.184~0.853;3 对居群Nm值小于0.200,13 对居群Nm值小于0.300,6 对居群群Nm值小于0.400,10 对居群Nm值小0.600,仅有4 对居群Nm值在0.603~0.853。贵州境内滇龙胆居群与云南境内滇龙胆居群间Nm值多小于0.400;滇中华宁居群与滇东南文山群间Nm值较高为0.754;综合比较发现,滇龙胆居群间基因流在滇中与滇西地区较强。
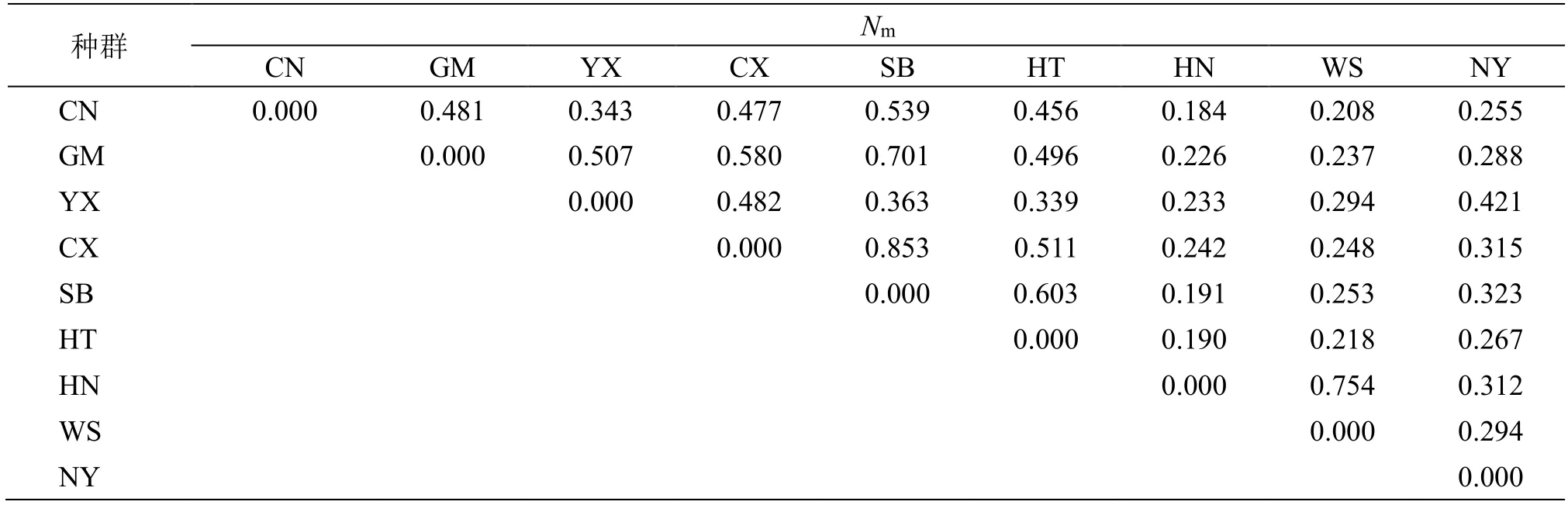
表8 云贵高原滇龙胆9 个居群的Nm 数值Table 8 Nm value between nine population of G.rigescens in Yunnan-Guizhou Plateau
3.4.3 遗传变异分析 滇龙胆19 个居群的遗传变异分析显示(表9),遗传变异主要来源于居群内,占总变异的86.99%;剩余13.01%的变异来源于居群间。
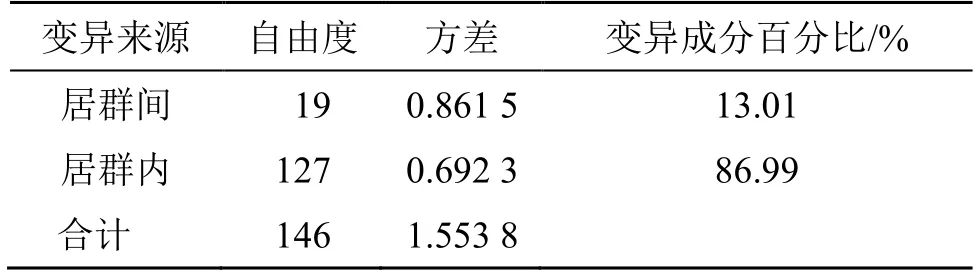
表9 滇龙胆19 个居群的分子变异分析Table 9 AMOVA results of 19 population of G.rigescens
3.5 地理距离、环境距离对居群遗传分化的影响
3.5.1 横断山区与云贵高原环境差异分析Mann-Whitney U 检验结合分布区23 个环境因子数据(表2)的统计分析显示,横断山区与云贵高原滇龙胆生境在热量指标和UV-B 辐射两方面存在显著差异(表10),温度季节性变化(Bio 04)、最冷月最低温度(Bio 06)、温度年较差(Bio 07)、活动积温(10AAT)、UV-B 辐射季节性变化(UV-B A2)、UV-B 辐射最强月份平均值(UV-B A3)以及UV-B 辐射最强季度月均值总和(UV-B A5)在居群间差异显著(P<0.05)。
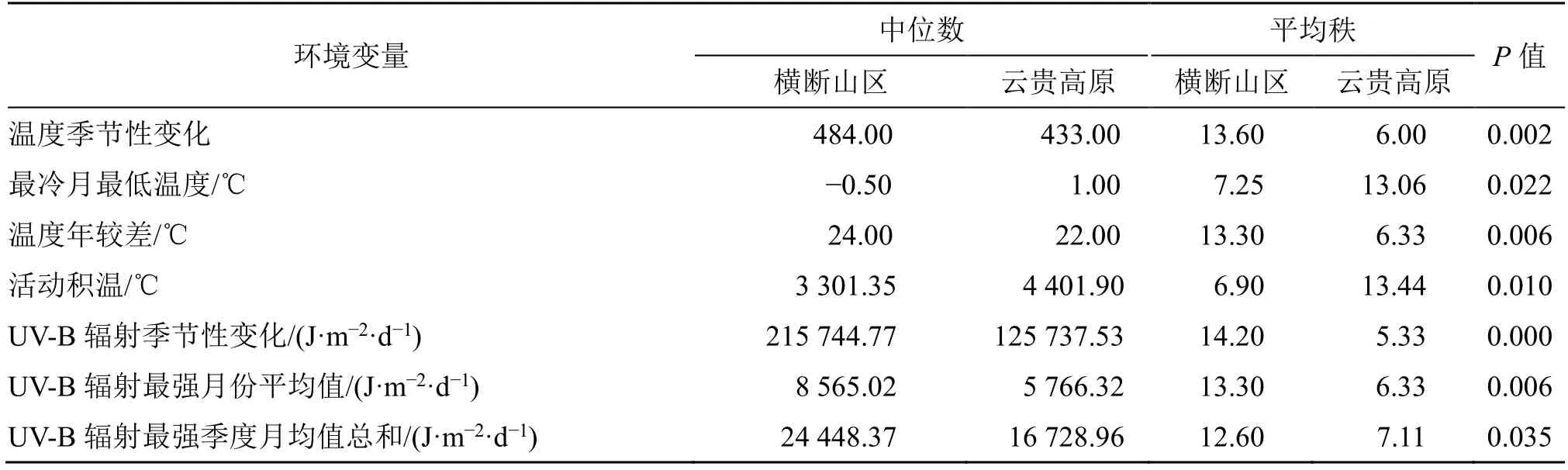
表10 横断山区与云贵高原差异显著的环境变量Table 10 Environmental variables with significant differences between Hengduan Mountains and Yunnan-Guizhou Plateau
对上述差异显著的7 个环境变量进行PCA,结合双标图显示(图6),7 个环境变量均远离圆心,表明上述环境变量可作为区分不同居群生境的特征变量;7 个变量进一步划分为3 组,Bio 04 与Bio 07为第1 组(均在第1 象限),Bio 06 与10AAT 为第2 组(均在第2 象限),所有UV-B 辐射变量为第3组(均在第3 象限)。综合上述,Bio 04、Bio 07、Bio 06 等7 个变量能较好反映19 个居群的生境差异与环境特征,适用于计算居群间环境距离。
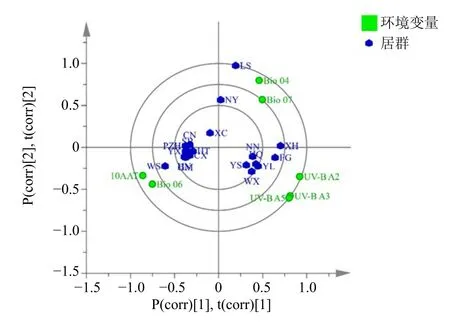
图6 基于7 个环境变量的PCA 双标图Fig.6 Biplot based on PCA of seven environment variables
3.5.2 Mantel 检验 Mantel 检验显示(表11),地理距离(GD)与居群FST相关性不显著(P>0.05);环境距离与FST显著相关(P<0.05)。为进一步分析影响居群遗传分化的主要环境因子,分别用表10 中的4 个热量指标和3 个UV-B 辐射指标计算环境距离,并与居群FST进行Mantel 检验。结果显示(表11),居群间UV-B 辐射指标与FST呈极显著正相关(P<0.01),热量指标与FST相关性不显著(P>0.05)。以上结果表明,横断山区和云贵高原滇龙胆居群间的遗传分化可能与生境UV-B 辐射变化有关。

表11 遗传距离与地理距离、环境距离的Mantel 检验 (基于斯皮尔曼相关系数)Table 11 Mantel tests for the correlation between genetic distance and geographic distance,environmental distance(Spearman’ s coefficient)
4 讨论
4.1 滇龙胆遗传结构与遗传多样性
基于GBS 简化基因组测序,对分布于云南、四川、贵州的19 个居群147 株个体居群遗传结构和遗传多样性进行分析。发现滇龙胆野生居群基因型丰富,其中四川乐山(LS)、贵州纳雍(NY)、云南楚雄(CX)、临沧(YX、GM)等地居群存在种质混杂。19 个居群依据遗传结构从地理上大致可划分为北部、南部2 个组。北部组居群主要分布于滇西北横断山区及川西南山区,南部组居群主要分布于云贵高原。
北部组与南部组各居群FST平均值分别为0.471、0.422;物种FST值与线叶龙胆G.lawrenceivar.farreri(I.B.Balfour) T.N.Ho 较接近,高于多花龙胆G.striolataT.N.Ho、阿墩子龙胆G.atuntsiensisW.W.Smith,低于钻叶龙胆G.haynaldiiKanitz[35-36]。遗传变异分析显示滇龙胆个体间遗传多样性较高,种内变异有13.01%源自居群间,其余86.99%均来自居群内。该结果与龙胆科药用植物线叶龙胆和川西獐牙菜较相似[7,37]。居群间Nm数值变化分析显示,横断山区各居群Nm值在0.181~1.279,云贵高原各居群Nm值在0.184~0.853。结合赵宗苹等[27]的研究结果认为,滇龙胆居群间存在一定的遗传分化和基因流,但不同分布区居群间遗传分化程度及基因流强度存在差异。
基于I、Nei’s、π等参数的分析结果,与龙胆属其他物种对比发现,滇龙胆遗传多样性较分布于青藏高原和横断山区的线叶龙胆、六叶龙胆G.hexaphyllaMaxim.ex Kusnez.、三叶龙胆G.ternifoliaFranch.低[37-38],与麻花艽G.stramineaMaxim.和粗茎秦艽G.crassicaulisDuthie ex Burk.较接近[11,39]。将19 个居群依据其地理分布进行划分发现,横断山居群与云贵高原居群的遗传多样性具有差异;分布于横断山区的滇龙胆遗传多样性总体低于分布于云贵高原的滇龙胆。
4.2 生境差异对居群遗传分化的影响
物种多样性及特异种质的形成通常与地理隔离或特殊生境适应有关[40-41]。利用Mantel 检验分析了滇龙胆居群间遗传距离与地理距离、环境距离的相关性;结果显示居群FST数值的变化与地理距离相关性不显著(P>0.05),而与环境距离紧密相关(P<0.05)。该结果暗示环境隔离(isolation by environment,IBE)可能在驱动滇龙胆群遗传分化方面发挥了重要作用。
较复杂的地形可能对居群间种子流和花粉流造成影响,使物种的遗传分化与地理距离不相关[42]。滇龙胆为虫媒花,繁育系统为兼性异交型[43];种子有蜂窝状网纹,仅适合短距离风媒传播[28]。横断山区内部及横断山与云贵高原间的居群可能因高大山脉或河流、峡谷阻隔,影响植株授粉及种子传播;导致居群间遗传分化与地理距离不相关。此外,槭树Acer ginnalaMaxim.、青杨Populus cathayanaRehd.、无患子Sapindus mukorossiGaertn.等物种研究表明对于分布较广,分布区地貌复杂、生境条件差异大的物种,居群水平的遗传分化更多与分布区环境差异形成的选择压有关[42,44-45]。滇龙胆分布区北部属云贵高原与青藏高原的过渡区,海拔总体较高;分布区南部多为向西下降的盆地,海拔相对较低[46]。分布区地势变化总体呈现高纬度与高海拔相结合,低纬度与低海拔相一致的特点,进一步加剧了南北分布区的UV-B 辐射强度差异[46-47]。分布于青藏高原的麻花艽研究显示,改变生境UV-B 辐射强度可对植株叶片厚度、呼吸强度、光合作用等造成影响[48-50];同时麻花艽在长期进化过程中也形成对UV-B 辐射强度变化的适应机制[48]。通过Mantel检验结合简化基因组数据初步分析推测UV-B 辐射变化可能是驱动滇龙胆南部、北部居群遗传分化的重要环境因素。目前UV-B 辐射对滇龙胆生理、生态的影响尚未见报道。对于生长于高海拔山区的龙胆属植物,生境UV-B 辐射渐变如何驱动滇龙胆种内遗传分化,上述遗传变异对环境的适应意义及对药材质量的影响仍有待后续深入研究。
利益冲突所有作者均声明不存在利益冲突

