汉麻聚酮合酶基因家族成员鉴定与表达分析
朱雪雯,米要磊,孟祥霄,陈伟强,王思凡,曹 雪,陈士林,徐志超,苏 畅,孙 伟*,万会花*
1.中国中医科学院中药研究所,中药鉴定与安全性评估重点实验室,北京 100700
2.东北林业大学生命科学学院,黑龙江 哈尔滨 150040
3.深圳市药品检验研究院,广东 深圳 518057
聚酮类化合物(polyketide,PK)是一类由细菌、真菌、放线菌及植物产生的具有重要生物活性的天然次生代谢产物,它们在植物与环境相互作用以及植物生长发育方面发挥着重要的生物学意义[1]。聚酮合酶(polyketide synthases,PKSs)是催化聚酮化合物合成过程中的关键酶,该类酶可催化起始底物酰基辅酶A(CoA)与丙二酰-CoA 缩合形成一系列的聚酮类化合物骨架。PKSs 根据其蛋白结构和催化机制的不同分为I、II 和III 型[2]。I 型和II 型PKS主要存在于微生物中。I 型PKS 是以模块形式存在的复合酶,每个模块含有多个催化功能域,每个模块负责完成一次链延伸反应,这些模块包含不同的活性位点,以至于每个酶催化的反应均具有不同的活性[3];II 型PKS 是一组多酶复合物,主要催化多环芳族聚酮化合物的生物合成[2]。III 型PKS 广泛存在于植物中,是一类同源二聚体蛋白,主要由查耳酮合酶(chalcone synthase,CHS)和类CHS 超家族组成[4-5]。目前基于基因组水平的PKS基因家族已在多种被子植物中(例如水稻、玉米、香蕉、大豆和陆地棉等)被鉴定[4,6-11],且它们的研究主要集中在苯丙烷两大代谢分支类黄酮和木脂素的生物合成中。例如,砀山酥梨PbPKS 参与梨果实石细胞和木质素的合成[12],从而决定梨果实品质;而CHS作为花青素生物合成的关键酶已经在多种植物中被鉴定。植物中已发现的III 型PKS 根据其催化产物结构的区别可分为 CHS 以及类 CHS 超家族(chalcone synthase-like,CHSL)包括2-吡喃酮合酶(2-pyrone synthase,2-PS)、茋类合酶(stilbene synthase,STS)、联苄基合酶(bibenzyl synthase,BBS)、吖啶酮合酶(acridone synthase,ACS)、二苯甲酮合酶(benzophenone synthase,BPS)、苯戊酮合酶(valerophenone synthase,VPS)、苯亚甲基丙酮合酶(benzalacetone synthase,BAS)以及橄榄醇合成酶(olivetol synthases,OLS)等[6]。
汉麻Cannabis sativaL.又名火麻、胡麻和线麻,是大麻科(Cannabinaceae)大麻属CannabisL.一年生草本植物。汉麻具有重要的经济和药用价值,其干燥成熟果实即火麻仁可润肠通便,滋阴养血[13],其花和苞片中富含的萜酚类化合物,具有镇痛、抗肿瘤、抗炎等作用。其中大麻二酚(cannabidiol,CBD)和精神活性物质四氢大麻酚(tetrahydrocannabinol,THC)的结构、生物合成和生物活性已经得到了深入的研究[14-16]。CBD 作为非精神活性物质,其巨大的药用潜力正逐步被挖掘[17]。汉麻中萜酚类化合物的生物合成途径主要来自脂肪酸途径和萜类途径。OLS 作为新型PKS,是汉麻萜酚类化合物前体物质合成的关键酶[18]。OLS 与橄榄酸环化酶(olivetolic acid cyclase,OAC)将丙二酰辅酶A(malonyl-CoA)与己酰辅酶A(hexanoly-CoA)缩合生成2,4-二羟基-6-戊基-苯甲酸(olivetolic acid,OA)。OA 和香叶基二磷酸(geranyl diphosphate,GPP)在大麻醇酸合成酶(cannabigerolic acid synthase,CBGAS)的催化下合成汉麻酚萜类化合物的主要前体物质大麻醇酸(cannabigerolic acid,CBGA)。此外,汉麻中的PKS 还参与多种次生代谢产物如类黄酮和茋类等的生物合成[19]。但是目前在全基因组层面对汉麻PKS基因家族的分析还未有报道,因此对汉麻中PKS基因家族的物理化学性质,基因和蛋白结构特征以及基因的表达模式等信息的挖掘,对汉麻中萜酚类化合物以及其他次生代谢产物的合成有着重要的意义。
本研究利用生物信息学工具,对汉麻基因组中聚酮合酶基因家族进一步挖掘与分析。根据CsPKS基因家族成员的同源性和基因结构特征,探究CsPKS基因家族在进化过程中的复制事件,结合基因的表达模式探讨功能上是否存在部分或者完全冗余;同时研究CsPKSs基因在染色体上的分布情况,从而说明CsPKS基因家族的规模在进化过程中经历了收缩或者扩张;通过分析启动子区域窥探基因的表达模式,为揭示CsPKSs 功能提供了方向,为进一步研究汉麻中酚萜类化合物以及类黄酮的生物合成机制和CsPKSs 在汉麻中的生理功能奠定基础,并对开发新聚酮化合物药用资源提供理论基础。
1 材料与方法
1.1 CsPKS 家族成员的获取
从NCBI(https://www.ncbi.nlm.nih.gov)网站上获取雌性汉麻CBDRx 的全基因组及注释文件(accession number:GCA_900626175.1),应用TBtools[20]软件提取汉麻蛋白序列,于 Uniport(https://www.uniprot.org/)网站进行预测,预测得到的所有PKS 家族成员,进一步去除重复转录本,利用TBtools 软件获得CsPKS 家族的gene ID 所对应的蛋白序列。同时从Uniport 获得拟南芥和水稻的PKS 家族成员,利用BlastTP 工具比对汉麻CBDRx的蛋白序列文件,以E值<1×10-5获得汉麻CsPKS序列,去冗余后与上述方法进行比较,获得最终的汉麻 PKS 家族成员。使用生物信息学工具ExPASy-ProSite(http://web.expasy.org/ protparam/)对CsPKSs 蛋白的相对分子质量和等电点进行预测。利用softberry 网站(http://www.softberry.com/cgi-bin/programs/proloc/protcomppl.pl)进行亚细胞定位预测。
1.2 CsPKSs 蛋白序列的保守型和系统发育树分析
利用DNAMAN 将CsPKSs 与其他物种的PKS氨基酸序列进行保守结构域的同源性比对。使用MEGA6.0[21]通过邻接(neighbour-joining,NJ)方法构建系统发育树,并进行重复1000 次的Bootstrap检验。利用 MEME(http://meme-suite.org/tools/meme)网站预测保守基序Motif(参数设为10)。用TBtools 软件对CsPKSs基因进行外显子和内含子、保守结构域和系统进化树三者可视化处理。
1.3 CsPKSs 基因的染色体定位及共线性分析
使用TBtools软件解析汉麻的基因组注释文件,确定每个PKS基因的位置信息并绘制其所对应的染色体物理位置图。从NCBI 网站下载拟南芥、汉麻、水稻和番茄的全基因组数据,使用TBtools 软件对其共线性关系进行可视化处理,并找到PKS基因的同源基因对。
1.4 CsPKSs 基因顺式作用元件预测
使用PlantCare(http://www.plantcare.co.uk/)网站对CsPKSs基因顺式作用元件进行预测,并使用TBtools 软件对其可视化构图。
1.5 CsPKSs 蛋白二级和三级结构预测
通过SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)在线预测蛋白质二级结构。利用Alphafold[22]构建CsPKSs 蛋白三维结构模型。首先分析靶蛋白序列并进行多序列比对,随着深度神经网络学习预测原子对的距离和扭转分布,通过梯度下降的蛋白特异性电位来构建靶蛋白结构[23]。使用的工具包括:HHblits[24](3 次迭代,E=1×10-3);PSI-BLAST v.2.6.0 nr 数据集[25](3 次迭代,E=1×10-3);BioPython v.1.65[26];用于结构可视化的PyMol 2.2.0[27]。
1.6 CsPKSs 基因表达模式分析
根据已公布的9 个不同栽培品种(Black Berry Kush(BB)、Sour Diesel(SD)、Black Lime(BL)、Canna Tsu(CT)、Cherry Chem(CC)、Terple-3(T3)、Mama Thai(MT)、Valley Fire(VF)、White Cookies(WC)雌株腺毛转录组数据(PRJNA498707)以及商业品种Dinamed kush CBD autoflowering(Diku)的花、叶、苞片、茎、种子和根的转录组数据(Accession No.:bract:SMAN16122880~SAMN16 122882;stem:SAMN16122883~ SAMN16122885 ;flower :SAMN16122886~ SAMN16122888 ;leaf :SAMN16122889~SAMN16122891)计算CsPKSs基因的表达量(FPKM),并运用TBtools 软件绘制热图,进行基因聚类和差异表达分析。具体比对以及计算方法如下:使用star 软件将RNA-seq 数据比对至参考基因组,然后使用rsem 软件包中的rsem-calculateexpression 工具对比对结果进行定量分析,获得表达量FPKM 值。
1.7 CsPKS 基因表达
根据进化关系以及RNA-seq 数据对本研究中筛选的CsPKS1、CsPKS4-5和CsPKS9基因进行实时荧光定量 PCR(quantitative real-time PCR,qRT-PCR)验证。汉麻Diku 品种的花、苞片、种子、根、叶和茎的RNA 提取使用RNAperp Pure Plant Kit试剂盒(北京天根有限公司),采用TransScript®One-Step gDNA Removal and cDNA Synthesis SuperMix 试剂盒(北京全式金公司)逆转录合成cDNA,使用StarLighter SYBR Green qPCR Mix 试剂盒(北京启衡星公司)在Rotor-Gene Q(QIAGEN公司,德国)上进行qRT-PCR 检测,反应程序:95 ℃酶激活5 min,95 ℃变性 30 s,58 ℃退火 20 s,72 ℃延伸 15 s,循环次数40,用2-ΔΔCt法计算基因相对表达量[28]。相关引物见表1,内参基因选择EF1α[29]。本实验进行了3 次生物学重复。

表1 qRT-PCR 引物Table 1 Primers for qRT-PCR
2 结果与分析
2.1 CsPKS 家族成员的获取
从NCBI 网站获取汉麻的全基因组及注释文件(accession number:GCA_900626175.1),利用TBtools 软件提取汉麻蛋白序列,将PlantTFDB 网站获得的拟南芥和水稻PKSs 蛋白序列与汉麻蛋白序列数据进行比对,去除重复转录本后,共筛选得到CsPKS基因家族成员9 个,依次命名为CsPKS1~CsPKS9,其编码氨基酸长度为356~412 aa;蛋白相对分子质量的大小为39 285.38~45 282.99;等电点介于5.52~8.07。亚细胞定位预测9 个基因全部定位于细胞质(表2)。

表2 CsPKS 基因家族成员基本信息及特征Table 2 Information and characteristics of CsPKSs
2.2 CsPKS 序列的保守型和系统发育树分析
利用DNAMAN 软件将9 条CsPKS 氨基酸序列与GenBank 数据库中其他植物PKS 氨基酸序列进行比对分析(图1)。CsPKS 氨基酸序列与金鱼草Antirrhinum majusL.(BAE80511)、紫花苜蓿Medicago sativaL.(P30074)、虎杖Polygonum cuspidatumL.(ABK92282)中的氨基酸序列具有较高的相似度。CsPKS 具有植物III 型PKS 超家族三联体活性中心位点Cys164、His303 和Asn336(图1)[30],其中Cys164 是聚酮链形成过程中的亲核活性位点,通过共价键与起始底物分子和反应中间体结合,而His303 和Asn336 则起着稳定或活化起始底物和延伸底物的作用。同时CsPKSs 氨基酸序列具有CHS 典型的活性位点[31],如Arg38、Phe49、Thr50、Thr132、Gly211、Gly216 等。起“Gatekeeper”作用的2 个氨基酸残基Phe 215 和Phe265 在大多数CsPKSs 中高度保守,但CsPKS5 和CsPKS8 中Phe256 分别被Tyr 和Ile 取代,与其他PKS 存在较为明显的差异。

图1 PKS 氨基酸序列比对Fig.1 Alignment of PKSs
为了阐明CsPKS 与其他植物物种PKS 之间的进化关系,对CsPKS 与拟南芥、水稻、苜蓿等植物的PKS、CHS、CHSL、STS 和VPS 等进行了系统发育树分析。系统发育重建后发现,CsPKS蛋白共划分为3 个组,分别命名为group 1、group 2 和group 3(图2)。针对9 个CsPKS,发现第1组共包含CsPKS2、CsPKS3 和CsPKS4 3 个成员,其中CsPKS2 和CsPKS3 聚为一支后与NsCHSL聚在一起,CsPKS4 与AtPBSB 聚为一支。第一组PKS 多与模式植物如拟南芥、水稻和烟草等关系密切;第2 组包含5 个CsPKS,分别是CsPKS1、CsPKS 5~8,该组5 个成员与VvSTS1、RpBAS和HlVPS 关系较为密切,其中CsPKS5 和CsPKS8聚为一支;CsPKS1、7 和6 聚为一支后与啤酒花的VPS 聚为一支;第3 组仅包含CsPKS9,其与啤酒花CHS 同源。
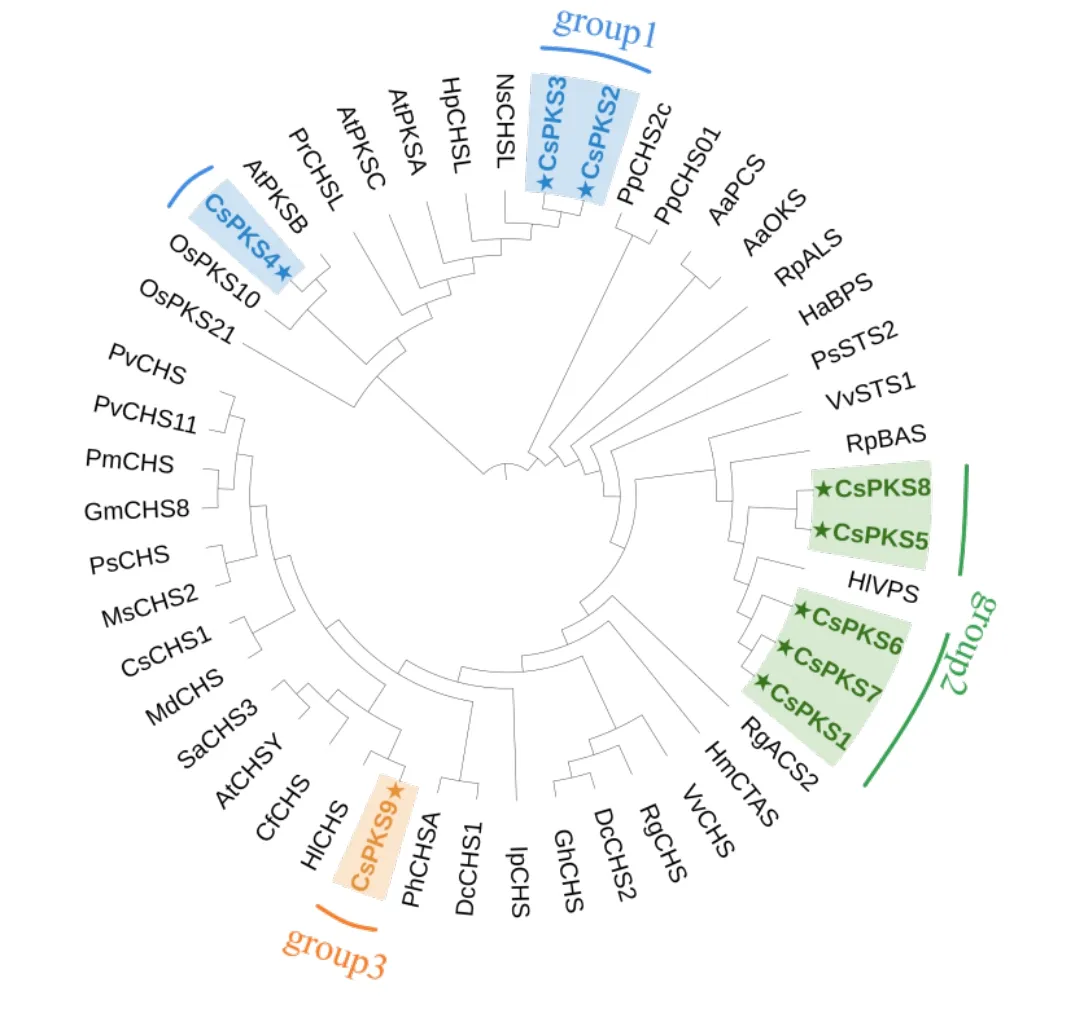
图2 CsPKS 家族基因系统发育树及其分类Fig.2 Phylogenetic analysis of PKSs from C.sativa and other plant species
CsPKSs基因结构分析显示(图3)所有的CsPKSs基因中仅有1 个内含子这与其他植物III 型PKS基因的结构较为保守相符。裸子植物和被子植物中,除已报道的金鱼草外,其余已报道III 型PKS基因均含有1 个内含子且该内含子位置保守[31-33]。CsPKS2和CsPKS3在3’端无非翻译区(untranslated region,UTR),CsPKS4和CsPKS6内含子较长,分别为1187、2075 bp。通过保守基序分析发现,

图3 CsPKS 的基因结构及蛋白质保守基序分析Fig.3 Gene structure and conserved motif analysis of CsPKS
Motif1、Motif2、Motif4、Motif5、Motif7、Motif8、Motif9 存在于所有CsPKSs 中,Motif3 和Motif6 存在于CsPKS1、CsPKS5、CsPKS6、CsPKS7、CsPKS8、CsPKS9 中,Motif10 存在于CsPKS2、CsPKS3、CsPKS4 中。
2.3 CsPKS 基因的染色体定位及共线性分析
通过对CsPKS 家族成员染色体进行定位分析(图4),发现该家族9 个成员分布在Chr1、Chr3、Chr5、Chr7、Chr10 号染色体上。其中,CsPKS1、CsPKS6和CsPKS7成簇位于10 号染色体运用比较基因组学的方法和共线性原理挖掘汉麻与双子叶模式植物拟南芥、番茄以及单子叶模式植物水稻之间的共线性关系。图5 中高亮区域显示PKS 家族在3个物种间的同源基因对,汉麻和拟南芥中,一共发现2 对同源基因(CsPKS4和AtPKSB,CsPKS1或7和AtCHSY),其中CsPKS1和CsPKS7为一对串联重复基因,且氨基酸序列完全一致;在汉麻和水稻中,一共发现了1 对同源基因(CsPKS1或CsPKS 7和OsCHS1);汉麻和番茄中,一共发现2 对同源基因(CsPKS4和SlPKSB,CsPKS9和NM_001247104.2)。
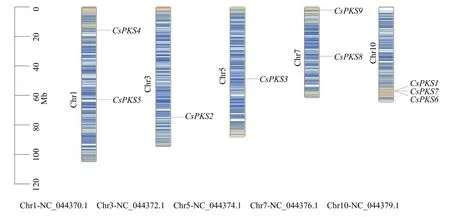
图4 CsPKS 基因染色体定位Fig.4 Chromosomal locations of CsPKS genes in C.sativa

图5 汉麻与拟南芥、水稻和番茄的共线性分析Fig.5 Collinear relationship of C.sativa with respect to Arabidopsis thaliana,Oryza sativa and Solanum lycopersicm
2.4 CsPKS 基因顺式作用元件预测
提取CsPKS 基因的翻译起始密码子(ATG)上游的2 kb 作为启动子序列,对CsPKS基因家族成员顺式作用元件进行预测和可视化分析(图6),结果发现,核启动元件、光响应元件存在于汉麻所有PKS基因的启动子区域。CsPKS1、CsPKS6、CsPKS7中顺式作用元件的数目和种类均较多,CsPKS3所含顺式作用元件数目最少,且干旱诱导元件仅存在于CsPKS3中。CsPKS基因启动子区的顺式作用元件种类及分布具有多样性。
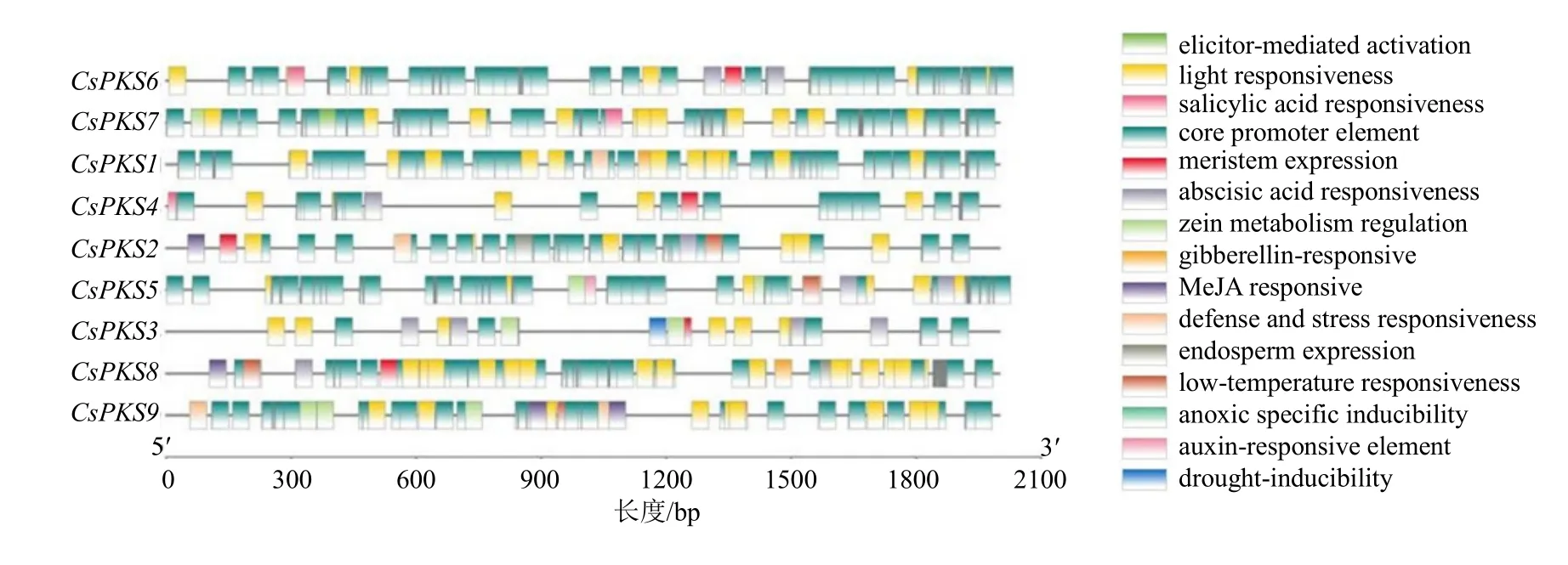
图6 CsPKS 家族基因的启动子区顺式作用元件Fig.6 Cis-acting elements within the promoters of CsPKS genes
2.5 CsPKSs 蛋白二级和三级结构分析
蛋白质二级结构预测结果如表3 所示,CsPKSs的α-螺旋介于38.11%~46.56%;延伸链和β-折叠在整个二级结构中占比较少,分别为14.61%~18.45%和5.85%~8.50%;随机卷曲介于32.88%~62.40%。使用Alphafold 对目前已鉴定功能的OLS(CsPKS1/CsPKS7)和CsPKS9 蛋白进行三维建模(图7)并通过Pymol 软件实现蛋白构象的可视化。AlphaFold 的核心组件是一个卷积神经网络,它通过对Protein Data Bank(PDB)数据库中的蛋白结构深度学习来预测蛋白质残基的Cβ原子对ij之间的距离(dij)[23]。OLS 和CsPKS9 蛋白主要由α-螺旋和无规则卷曲形成活性口袋组成,Asn330、His257、Cys157、Phe208 和Phe259 为OLS 活性位点:Asn330、His257 和Cys157 是OLS 活性中心位点,Phe208 和Phe259 为OLS 起“Gatekeeper”作用的两个氨基酸残基;Asn336、His303、Cys167、Phe215 和Phe265 为CsPKS9 活性位点:Asn336、His303 和Cys167 是CsPKS9 活性中心位点,Phe215 和Phe265 为CsPKS9起“Gatekeeper”作用的2 个氨基酸残基。

图7 CsOLS (CsPKS1 或CsPKS7) 和CsPKS9 蛋白三维结构Fig.7 Three-dimensional structure of CsOLS and CsPKS9

表3 CsPKS 蛋白二级结构Table 3 Predicted protein secondary structures of CsPKS
2.6 CsPKS 基因表达模式分析
为探究CsPKS基因的表达模式,对汉麻品种Diku 的花、苞片、种子、根、叶、茎6 个不同组织和9 个不同汉麻品种的腺毛转录组数据进行基因表达差异分析,并绘制热图。研究发现CsPKS9在花中表达量最高,苞片和叶中表达量均较高,茎中表达较低,种子和根中几乎不表达;CsPKS1、CsPKS7在茎中表达量较低,种子和根中几乎不表达,花和苞片中表达较高,且在苞片表达量高于花;CsPKS5在花和苞片中表达量较低,在其他组织中不表达;其余基因在6 个组织中都几乎不表达。早期的研究显示CsPKS1和CsPKS7为汉麻中萜酚类化合物合成途径的关键酶基因[17]。通过热图聚类可以看出,在汉麻9 个不同品种的腺毛中(图8),CsOLS(CsPKS1和CsPKS7)在所有品种的腺毛中都高度表达,CsPKS5和CsPKS9在所有腺毛中也均有较高的表达,其次是CsPKS6。CsPKS2、CsPKS3、CsPKS4和CsPKS8在所有品种中表达量很低或者不表达。

图8 CsPKS 基因的表达模式分析Fig.8 Expression pattern of CsPKS genes in different tissues of Diku and trichomes of different varieties
2.7 qRT-PCR 验证CsPKS 基因表达
为了进一步验证转录组数据的表达分析结果,本研究利用qRT-PCR 验证OLS(CsPKS1/7)、CsPKS4-5、CsPKS9在汉麻Diku品种不同组织部位的表达情况(图9)。结果显示OLS、CsPKS5、CsPKS9在汉麻花和苞片中的表达量比在其他组织部位的表达量高(P<0.05),与转录组结果一致。由于CsPKS4基因整体表达量极低,其qRT-PCR 结果与转录组趋势略有不同。

图9 CsPKS 在不同组织中的相对表达水平Fig.9 Relative gene expression of the CsPKS in different tissues
3 讨论
PKS 是聚酮化合物合成过程中的关键酶,在植物生长发育、抗逆以及植物与环境之间的信息交互等方面起着重要的作用[1]。基于基因组水平的PKS基因家族成员已在多种植物中被鉴定[7],目前水稻和蔷薇科梨[12]中含有数量最多的PKS 成员,达27个,而在其他被子植物中PKS 的数量基本在4-20个之间(香蕉20 个[9]、玉米14 个[8]、大豆14 个[10]、悬钩子10 个[11]和兰花5 个[34]等)。通过基因组水平的研究发现,PKS 在植物基因组中的数量差异可能与物种是否存在古老的全基因组复制或者近期的串联重复复制有关[12]。本研究在汉麻基因组中共鉴定得到9 个CsPKS基因,其中CsPKS1、CsPKS7和CsPKS6在基因组上成簇存在,CsPKS1和CsPKS7核酸序列相似度为99.40%,氨基酸序列相似度为100%;CsPKS6与CsPKS7的核酸序列相似度为69.1%,并且它们具有相似的保守基序和保守结构域分布。Gao 等[35]通过对1 个野生品种的汉麻深度测序,发现其发生了3 次近代全基因组复制事件和一次大规模的基因复制事件,因此推测全基因组复制事件可能是产生CsPKS1和CsPKS7基因对的直接原因。
CsPKS 在汉麻类黄酮和酚萜类化合物的生物合成中发挥着重要的作用。CsPKS 在酚萜类化合物合成途径中以己酰辅酶A(hexanoyl-CoA)和丙二酰辅酶A(malonyl-CoA)作为底物生成中间产物丁烯酮辅酶A(tetraketide-CoA);CsPKS 在类黄酮合成途径中以香豆酰辅酶A(p-coumaroyl-CoA)和丙二酰辅酶A 为底物生成柚配基查耳酮。CsPKS1/7与Taura 等[18]克隆鉴定的汉麻酚萜类化合物合成途径基因OLS为同一基因,因此CsPKS1/7是参与汉麻酚萜类化合物生物合成的关键酶基因。另外,CsPKS9与Raharjo等[36]在汉麻中克隆的参与汉麻类黄酮化合物合成的CHS基因序列一致,这也表明了CsPKS9是汉麻类黄酮合成的关键酶基因。CsPKS1/7 和CsPKS6 在系统发育树中与HlVPS 聚类[37],以异戊酰辅酶A(isovaleryl-CoA)和异丁基辅酶A(isobutyryl-CoA)作为偏好性底物。在汉麻与拟南芥以及水稻的共线性分析中发现CsPKS1/7与AtCHSY和OsCHS1同源,而AtCHSY 和OsCHS1已报道的催化产物是柚配基查耳酮,这也说明了PKS 在进化过程中功能产生了分化。在系统发育树中CsPKS9 与其它多种植物的CHS 聚类,且在共线性分析中CsPKS9 和番茄SlCHS 同源,它们都参与了柚配基查耳酮的生成,这也表明CHS 作为类黄酮合成的关键酶,其功能是相对保守的。值得一提的是,CsPKS9 不仅可以香豆酰辅酶A(p-coumaroyl-CoA)作为底物,也可以异戊酰辅酶A 或异丁基辅酶A 作为底物分别生成间苯二酮和间苯异丁苯甲酮[38],这也表明CsPKS9 具有VPS 活性。
CsPKS2、CsPKS3 和CsPKS4 与NsCHSLK、AtPKSA 和 AtPKSB 聚为一支,研究发现烟草NsCHSLK 在花药发育中,特别是对花粉粒外壁合成中起促进作用,它的mRNA 的表达仅限于小孢子和绒毛细胞且NsCHSLK 启动子表现出花药特异性[39];在拟南芥花药的孢子发育过程中,AtPKSA和AtPKSB 在绒毛膜细胞中存在组织特异性和时空特异性表达,其参与了孢粉蛋白的合成,被分类为花药特异性 CHS like(anther specific chalcone synthase-like,ASCL)蛋白[36,40]。由于本研究中涉及的汉麻为雌性植株,不存在花药的发育过程,所以转录组中CsPKS2、CsPKS3和CsPKS4基因的表达量很低甚至不表达。因此,CsPKS2、CsPKS3和CsPKS4基因可能在雄性大麻花药发育过程中发挥作用。另外,CsPKS5 和CsPKS8 与STS 聚为一支,因此这2 个PKS 可能参与汉麻茋类化合物的生物合成。CsPKS 启动子的激素诱导和抗逆胁迫类顺式作用元件数量较多,表明PKS 基因家族在汉麻生长发育过程中,尤其是在响应胁迫中可能具有重要的调控作用[41]。
植物中聚酮类化合物的积累以及关键III 型PKS 基因的表达往往具有一定的组织特异性。例如,GhCHS 主要在陆地棉Gossypium hirsutumLinn.的纤维中表达,与纤维素的生物合成密切相关[42];凹缘金虎尾Malpighia emarginataL.中的MeCHS 的表达在果实成熟过程中显著上调[43];HlVPS基因在啤酒花蛇麻腺中特异性表达,直接影响苦味酸的生物合成[37]。汉麻雌花和苞片是酚萜类化合物等次生代谢产物的主要合成场所[36],而与这些次生代谢产物密切相关的基因比如CsPKS1、CsPKS5-7和CsPKS9在花和苞片以及不同品种的汉麻腺毛中都高度表达。
PKS 是聚酮类化合物的合成关键酶和限速酶。本研究通过生物信息学,对CsPKS在全基因组水平上进行了鉴定与分析,对该家族成员的物理特性、基因结构、保守基序等进行分析,并探究了汉麻与其他物种间PKS 的进化关系,确定了各基因在汉麻的组织表达特异性,为探明CsPKS基因功能提供理论支撑,并为寻找潜在的汉麻中聚酮类合成的关键酶提供理论依据。
利益冲突所有作者均声明不存在利益冲突

