肉牛剩余采食量相关瘤胃及粪便微生物特征比较分析
张岩峰 丁燕玲 马应 周小南 杨朝云 史远刚 康晓龙
(宁夏大学农学院,银川 750021)
在哺乳动物消化道中,存在大量微生物,人类体内微生物是其体细胞数量的10 倍,预示它们编码的独特基因比人类自身的基因组多100 倍[1]。这一比例在牛上更加显著,牛肠道菌群数目约是其体细胞的120 倍[2],这反映了微生物在将饲料转化为肉和奶等畜产品过程中的重要作用。当微生物组中某些特定的菌群失调时,则会引起宿主发生疾病、影响发育生长[3-4]。因此,把微生物组作为一个特殊表型去预测复杂性状是可行的,例如家畜的饲料利用效率[2,5]。此外,研究发现消化道的微生物组成与宿主基因型存在关系[5-6],证明微生物群落的构成不仅依赖于环境和饮食,而且还依赖于其宿主基因型。反刍动物的生产中,肠道微生物菌群与饲料的消化利用率的关系已经得到了证实[7-8]。在养殖过程中,饲料成本约占总成本的70%-80%[9]。提高饲料效率有助于提高饲养效益并减少环境污染[10],选育高饲料效率的肉牛能够增加肉牛养殖的可持续发展。
剩余采食量(residual feed intak,RFI)是指动物的实际饲料采食量与基于增重率和体型的预测采食量之间的差值。自Koch 等[11]提出后,经过多年的研究和实践,RFI 已经成为了衡量家畜能量效率的主要指标[12],这主要是因为RFI 具有中等遗传力,且在遗传上与生长和体型无关[13]。LRFI 的动物在饲料利用方面是高效的,这表现在尽管采食量较低,但在生长方面没有损失,具有良好的生产潜力[14]。虽然对造成RFI 的特定生物学机制尚未完全了解,但目前的文献已经确定了与饲喂方式、甲烷排放、蛋白周转率、消化时间、活动方式等的关系[15-18]。据估计,RFI 中大约14%的生物学变异与消化过程差异有关[19],瘤胃微生物在营养物质消化过程中承担着重要作用,已有证据表明瘤胃菌落特征与RFI之间存在联系[20]。
以往的研究表明,通过改变瘤胃微生物组来提高饲料利用效率存在可行性。为了探究肉牛RFI 与其胃肠道微生物的相关性,本研究选取极端RFI 个体,屠宰后收集个体瘤胃内容物和粪便样本进行宏基因组测序。之后对测序数据进行基因预测,物种注释,功能注释等分析,以此来明确不同RFI 个体中微生物的特征与差异,进一步对其进行功能注释分析,以期探究胃肠道微生物与肉牛RFI 表型之间的关系,为筛选与RFI 表型相关标志微生物提供参考。
1 材料与方法
1.1 材料
选取30 头健康状况良好,月龄(12±1 months)相近的安格斯去势公牛,试验牛只均来自宁夏石嘴山市某养殖场,其初始体质量为(266.7±35.7)kg。试验牛按照标准程序饲养,按照专利号CN201610301797.6 的饲料配方进行饲喂,饲喂周期为81 d。试验结束后,根据RFI 表型值选择RFI最高和最低的极端个体各5 头,HRFI 组的RFI 值为1.12,0.69,0.53,0.47,0.47;LRFI 组的RFI 值为-0.97,-0.82,-0.77,-0.76,-0.73。采用颈动脉放血进行屠宰,分离出消化道,取出瘤胃内容物固、液相混匀采集2 份,在肠道末端采集粪便2 份,分装于50 mL 冻存管,于-80℃冷冻保存备用。本研究的动物处理程序均通过宁夏大学动物伦理委员会的伦理审查和批准(NXUC20200618)。
1.2 方法
1.2.1 DNA 提取 按照CTAB 法标准化操作流程提取粪便和瘤胃液中的DNA,将提取的DNA 至于-80℃冷冻保存备用。
1.2.2 测序文库的构建 将检测合格的DNA 样品通过超声处理成350 bp 大小的片段,再对DNA 片段进行末端抛光,A 尾处理,添加测序接头,PCR 扩增,纯化PCR 产物等操作构建文库,并通过Agilent2100 Bioanalyzer、Q-PCR 检测文库质量。将检测合格的文库在Illumina PE150 平台上完成测序,得到原始数据。相关数据存放在NCBI 数据库中,登录号为PRJNA752224。
1.2.3 测序数据预处理 使用Readfq(V8,https://github.com/cjfields/readfq)测序后的原始数据进行预处理并采用Bowtie2 软件(version2.2.4,http://bowtiebio.sourceforge.net/bowtie2/index.shtml)去除牛源的污染,得到用于后续分析的有效数据。
1.2.4 Metagenome 组装和基因预测分析 对上一步得到的粪便和瘤胃液的有效数据使用SOAPdenovo软 件(V2.04,http://soap.genomics.org.cn/soapdenovo.html)进行组装,组装后采用MetaGeneMark(V2.10,http://topaz.gatech.edu/GeneMark/)进行基因预测,使用CD-HIT 软件(V4.5.8,http://www.bioinformatics.org/cd-hit/)去冗余,使用Bowtie2(version2.2.4)进行比对得到用于后续分析的非冗余基因(Unigenes),计算得到样本中的丰度信息。
1.2.5 物种注释分析 使用DIAMOND 软件(v0.9.9.110,https://github.com/bbuchfink/diamond/)将Unigenes 与NCBI 的NR 数据库中进行比对,获得每个基因的物种注释信息,并结合基因丰度表,获得不同分类层级的物种丰度表。
1.2.6 功能注释分析 使用DIAMOND 软件将Unigenes 与KEGG、eggNOG、CAZy 数据库进行比对,统计得出相关基因的功能注释和丰度分析。利用R对上述结果进行数据的可视化。
2 结果
2.1 RFI分组结果
对高低剩余采食量两组数据进行统计分析,结果表明,HRFI 组与LRFI 组的剩余采食量差异有统计学意义,HRFI 组剩余采食量显著高于LRFI 组(P<0.05),即HRFI 组每天比LRFI 组多采食1.46 kg,占LRFI 组日均采食量的12%;而两组平均日增重无显著差异(P>0.05)。由于与前期文章属于同一批样本,详细表型统计见已发表文献[21]。
2.2 基因预测和丰度分析
通过对数据预处理和组装后,能够得到用于后续处理的有效数据。经质控过滤,分别从粪便样FB.H 和FB.L 及瘤胃液样LWY.H 和LWY.L 中获得了63 514.59、63 953.72、65 840.72 和65 736.44 Mb有效数据。有效数据中测序错误率小于1%(Q20)的reads 达到97%以上,表明测序数据可靠性较高。对FB.H、FB.L、LWY.H 和LWY.L 进行组装后分别得到962 128 234、1 281 607 466、1 206 843 305和1 311 619 661 bp 的Scaftigs。基因预测分别得到1 470 490、1 951 578、1 822 963 和1 763 022 条ORFs,去冗余后,完整基因所占比例均达到了22%以上,表明后续物种注释和基因功能分析有较高的可靠性。各样本基本信息统计见表1。
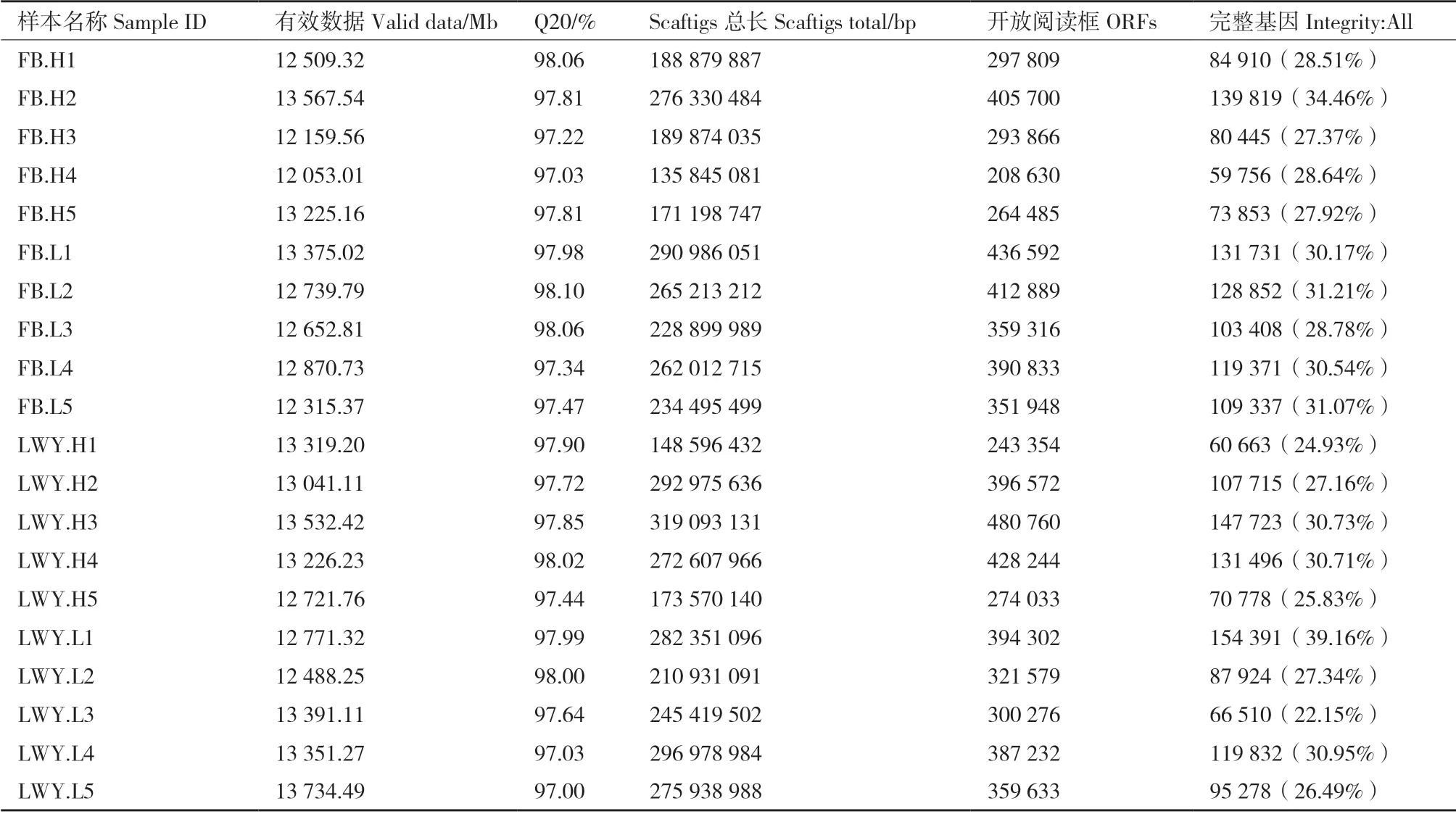
表1 各样本基本信息统计表Table 1 Basic information statistics of each sample
基于基因丰度表,通过随机抽取不同数目的样本,可以获得不同数目样本组合间的基因数目,由此构建和绘制了Core 和Pan 基因的稀释曲线,以研究样本间的基因数目的相似性。随着抽取样本的增加,样本核心基因(core-genome)数目逐渐降低,在样本数达到4 之后非冗余基因数目就达到平衡(图1-A),说明样本之间核心基因的相似性很高;同时随着样本增加,样本的泛基因组(pan-genome)数目不断增加,并最终达到平衡(图1-B),说明本试验采集的样本数量多,足以覆盖到机体胃肠道的全部微生物。
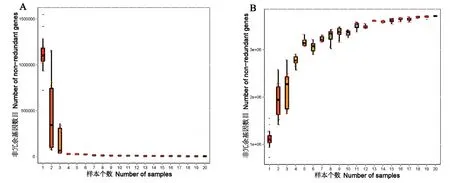
图1 core-pan 基因稀释曲线Fig.1 core-pan gene rarefaction curves
韦恩图可以很好的解释不同组间样本的基因分布情况,尤其是不同组间的共有基因及特定基因的信息。粪便中共有基因为67.23%,HRFI 组特异基因占23.18%,LRFI 组特异基因仅有9.59%(图2-A)。瘤胃液中共有基因在两组间高达80.03%,HRFI 组特异基因占10.13%,LRFI 组特异基因为9.84%(图2-B)。

图2 不同RFI 组瘤胃及粪便样中基因数目比较Fig.2 Comparison of rumen and fecal gene numbers in different RFI groups
为了进一步探究不同组之间基因数目差异,绘制箱型图展示不同RFI 组个体在瘤胃及粪便样本中的基因数目差异,如图3所示,在粪便中HRFI、LRFI 组基因数目无差异(P>0.05),但在瘤胃液中HRFI 组基因数目显著高于LRFI(P<0.05)。

图3 不同RFI 组间基因数目差异箱图Fig.3 Box plot of the differences in gene number among RFI groups
2.3 物种注释
微生物样本基因组的分类组成及物种注释,可以更好地分析不同个体间微生物的差异[22]。本研究中,从不同分类层级的相对丰度表出发,选取在两组中相对丰度最高的Top10 微生物种类,并将其余物种设置为Others,进行组间及组织间菌群种类在门属水平的差异特征。在门水平上,粪便中的物种分布无差异(P>0.05),拟杆菌门相对丰度最高,为优势菌门,其在HRFI、LRFI 组的相对丰度分别为37.49%、37.16%;其次是厚壁菌门,在HRFI、LRFI 组的相对丰度分别为31.52%、31.37%(图4-A)。瘤胃液中优势菌门与粪便中相似,且HRFI的相对丰度丰度高于LRFI 组,如拟杆菌门在HRFI组相对丰度为36.07%,显著高于LRFI 组的26.97%(P<0.05);同样的趋势也体现在厚壁菌门,HRFI 组相对丰度为16.24%,显著高于LRFI 组的12.54%(P<0.05)。
在属水平上,粪便中HRFI、LRFI 组Top10 微生物的相对丰度无差异(P>0.05),第一优势属为拟杆菌属,HRFI、LRFI 组分别占10.37%、10.18%;普雷沃菌属在HRFI、LRFI 组的相对丰度仅为3.79%、3.89%。与粪便组相比,瘤胃液中的普雷沃菌属是最大优势属,HRFI 组与LRFI 组存在显著差异(P<0.05),普雷沃菌属的相对丰度由HRFI 组的24.08%下降到LRFI 组的18.76%,其他优势属均有相同的下降趋势(图4-B)。

图4 门水平和属水平的物种相对丰度Fig.4 Relative abundances of species at phylum level and genus level
为了筛选HRFI、LRFI 组具有显著差异的生物标志物(biomarker),通过LefSe(LDA Effect Size)分析得到差异物种的LDA 值分布图(图5)。在粪便中,筛选得到的Biomarker 只在LRFI 组出现,分别为丹毒丝菌目(Erysipelotrichales)、丹毒丝菌纲(Erysipelotrichia)、Turicibacter属、八迭球菌属(Sarcina)。在瘤胃组,筛选得到的Biomarker 分布在HRFI 组,与粪便中恰好相反。LDA score ≥4共有11 个,分布在种水平有7 个,由于种水平对微生物组的贡献有限,相关研究主要集中于种以上水平微生物特征[23]。根据上述结果,可作为粪便或瘤胃组中潜在Biomarker 的有甲烷杆菌纲(Methanobacteria)、瘤胃球菌属(Ruminococcus)、古细菌科(Paludibacteraceae)、甲烷短杆菌属(Methanobrevibacter)。

图5 粪便和瘤胃液差异物种的LDA 值分布图Fig.5 Distribution of LDA values for species with differences in fecal and rumen fluid
2.4 功能注释
为了深入了解微生物群落的功能多样性,基于KEGG、eggNOG、CAZy 数据库对去冗余后的ORFs进行了各数据库level1 层级的功能注释。KEGG 通路分析如图6-A 所示,在粪便中,HRFI、LRFI组的新陈代谢(Metabolism)分别占所有通路的50.67%、48.94%,遗传信息处理(genetic information processing)分别占所有通路的23.90%、22.67%。在瘤胃液中HRFI、LRFI 组中的新陈代谢分别占所有通路的30.17%、23.96%,遗传信息处理分别占所有通路的14.49%、12.16%。对粪便、瘤胃液微生物的六大代谢通路进行分析对比,可以得到粪便中注释到通路上基因大于瘤胃液的(P<0.05)。进一步对组间功能差异的LEfSe 分析,在瘤胃液中LRFI 组最具代表性的是K07451(mcrA)、K01185(lysozyme)、K03283(HSPA1s)、K07198(AMPK)、K07893(RAB6A)、K07874(RAB1A)、K08857(NEK1_4_5)、k13412(CPK)、k02334(dpo)、k07901(RAB8A)(图7-A)。如图6-B 所示,eggNOG 功能注释佐证了KEGG 的分析结果,粪便中功能基因的相对丰度显著高于瘤胃液中功能基因的相对丰度(P<0.05)。在eggNOG 数据库的富集中,富集的功能类别主要集中在翻译、核糖体结构和生物生成,复制、重组和修复,碳水化合物的运输和代谢,氨基酸的运输和代谢(图7-B)。碳水化合物活性酶(CAZymes)数据库经常用于解密与碳水化合物酶相关的宏基因组序列的功能关联。由图6-C 可以看出糖苷水解酶类(GHs)的含量在各组中占据绝对优势,分别占总碳水化合物酶的59.25%、58.15%、55.61%、48.61%;其次为糖苷转移酶类(GTs)、多糖裂解酶类(PLs)、糖水化合物脂酶类(CEs)和碳水化合物结合模块(CBMs);辅助模块酶类(AAs)相对丰度最低。利用LefSe 分析筛选显著差异的功能Biomarker 时,LDA score>4的有3 个,分别是LRFI 瘤胃液中的GH24,HRFI瘤胃液中的GH43,LRFI 粪便中的GH13(图7-C)。

图6 KEGG、eggNOG、CAZy 功能注释图Fig.6 Functional annotation map of KEGG,eggNOG,and CAZy

图7 KEGG、eggNOG、CAZy 差异功能的LDA 值分布图Fig.7 Distribution of LDA values for KEGG,eggNOG,and CAZy differential functions
3 讨论
3.1 不同RFI组物种组成差异和生物标记物
本研究分析结果显示,无论在门水平还是属水平,粪便中微生物的物种组成总比瘤胃液中拥有更少的差异性,这是由于瘤胃微生物群落是饲料利用效率中的核心,可以通过发酵活性调节宿主可获得的能量[24],而肠道末端的粪便虽然来源于十二指肠上端,但随着食糜从一个部位转移到另一个部位,微生物群落多样性会发生显著变化[25]。本研究中,粪便样中的微生物在HRFI、LRFI 组之间差异不显著,表明粪便中细菌群落与RFI 表型相关性较低,因此,粪便样本能否作为瘤胃液的替代物对RFI 相关微生物群落进行分析及早期筛选,有待更多实验数据的支撑,这与Noel 等[26]的结论一致。在瘤胃液中,LRFI 组微生物的相对丰度小于HRFI 组,较低的相对丰度出现在饲料利用效率更高的群体。已有的文献中也得到了类似的结果,较低的微生物组基因含量和分类单元丰富度与较高的饲料效率紧密相关[27];研究人员通过分析不同奶牛产奶量的瘤胃细菌群落发现,与低产奶牛相比,高产奶牛的瘤胃细菌丰度和菌种均匀度较低[28]。高效的瘤胃微生物组成多样性及相对丰度较低[27-28],可能产生的相关分解代谢物范围较小,意味着较少的微生物代谢损耗。
HRFI、LRFI 组在门水平最丰富的微生物均为拟杆菌门和厚壁菌门,但粪便中的厚壁菌门是瘤胃液中的两倍左右。厚壁菌门拥有能够分解纤维素并产生机体可以直接吸收利用的短链脂肪酸的能力[29],厚壁菌门的显著差异可能是不同部位消化效率差异的原因之一。在瘤胃液中,拟杆菌门在HRFI组占比36.07%,高于LRFI 组的26.97%,而拟杆菌门主要负责蛋白质的水解,碳水化合物的降解以及氨基酸向乙酸盐的发酵[30],这可能与HRFI 消耗更多的饲料有关。在属水平上,粪便中和瘤胃液中的物种组成在属水平有显著差异。粪便中第一优势属为拟杆菌属,而瘤胃液中相对丰度最高的为普雷沃菌属,这与前人的研究结果一致[31-32],但相对丰度低于之前的研究,可能是研究物种、日粮条件、环境及研究方法等不同引起。
利用LEfSe 对不同RFI 组间微生物物种差异进行分析,当LDA 值≥4 时,认为该物种是生物标记物(Biomarker)。丹毒丝菌纲(Erysipelotrichia)丰度在粪便中组间具有显著差异,且在LRFI 有较多的富集,这与McGovern 等[33]的研究相符,关于该菌种的相关研究较少,尚不清楚其具体作用,但有研究者在研究人的肥胖与菌种关系时,发现该菌种在肥胖组中有较高丰度[34]。此外,瘤胃液中差异最显著的是甲烷杆菌纲,在HRFI 组有较多的富集,Lam等[35]的研究也有相同的发现。有研究通过35 个国家的32 个动物物种的742 个样本中前肠微生物群落组成,发现优势细菌的特征较差,但甲烷菌在世界范围内高度保守[36]。因此,可以推测甲烷菌的这种差异可能与RFI 相关,甲烷杆菌纲在HRFI 组的较高丰度表明HRFI 组肉牛采食量较高的同时也更容易产生甲烷,这个结果符合前人的研究[37]。据报道,具有不同RFI 的肉牛的能量代谢显著不同[38],其中牛产生甲烷所需的能量是其摄入能量的6%[39],这导致宿主摄入能量的大量流失。甲烷菌的差异可能造成消化效率的差异,进而影响RFI 的高低。在HRFI 组还发现了沙眼衣原体,这是能够引起许多疾病的病原菌,也会引起一些亚临床症状[40],造成机体消化效率降低,可能是导致RFI 较高的原因之一。
3.2 不同RFI组的微生物功能注释
在KEGG 数据库的富集中,新陈代谢的基因数量最多,其次是遗传信息处理,环境信息处理,细胞过程、人类疾病和生物体系统。通过LEfSe 分析筛选组间具有显著差异的功能Biomarker,在瘤胃液中HRFI 组、LRFI 组,粪便中HRFI 组都发现了区分不同组的Biomarker。这些Biomarker 在菌落的生长过程发挥不同的作用。研究人员发现mcrA基因可以分辨不同甲烷菌的种群[41],本文中的差异可能预示HRFI、LRFI 组中的甲烷菌属于不同的种群。有研究人员发现热休克70 kD 蛋白基因的纯合性与肥胖高度相关[42]。AMP 依赖的蛋白激酶(AMPK)是至关重要的细胞能量传感器,可以调节全身的代谢能量平衡[43]。Ras 通路是一个十分明确的通路,它能够在级联放大后激活转录因子,调控细胞的生长分化[44]。这些功能上的差异表明肠道微生物作为一个整体时会呈现出一定的差异,这有助于深入理解微生物对宿主的影响。在瘤胃液中HRFI 组,K07133(dUTP 焦磷酸酶)有显著富集,dUTP 焦磷酸酶(dUTPase)能够催化dUTP 水解为dUMP 和PPi,从而防止复制过程中尿嘧啶掺入DNA 影响遗传稳定,该酶对细菌的生长繁殖非常重要[45]。另外有K02335(DNA 聚合酶I)、K05349(β-葡糖苷酶)、K01992(ABC-2 型运输系统渗透酶蛋白)在瘤胃液中HRFI 组显著富集。在粪便中HRFI 组,K01955(氨基甲酰磷酸合酶大亚基)、K01915(谷氨酰胺合成酶)、K00764(氨基磷酸肌苷转移酶)、K01951(GMP 合成酶)、K02355(延展因子G)有显著富集,但在粪便中LRFI 组却没有发现Biomarker,可能与粪便中HRFI 组和LRFI 组功能注释无差异有关。
肠道微生物编码的碳水化合物活性酶在将饲料中复杂的碳水化合物分解成可被宿主肠上皮吸收的成分中起着至关重要的作用[46]。本研究的结果显示,HRFI、LRFI 组碳水化合物活性酶的多样性无差异,但各碳水化合物活性酶的相对丰度却存在显著差异。糖苷水解酶类是瘤胃液中相对丰度最高的一类酶,这与张慧敏等[47]在海子水牛的结果一致。因为普雷沃菌属是牛瘤胃中GHs 的主要贡献者[48],瘤胃中糖苷水解酶的相对丰度与普雷沃菌属的相对丰度有很大的相似性。糖苷水解酶类能够水解复杂碳水化合物的糖苷键,有助于微生物降解纤维素、半纤维素和淀粉[49]。HRFI 组CHs 相对丰度高于LRFI 组的原因可能是过多糖苷水解酶加速微生物对饲料中纤维素的降解,造成机体的空腹感增强,从而导致采食量增加,RFI 升高。为了补充胃肠道微生物群的功能注释分析,使用eggNOG 数据库对胃肠道微生物组的直系同源群(orthologous groups,OGs)进行了功能分析,但较多富集的同源蛋白并未表现出差异,一些差异显著的同源蛋白在微生物中表达水平普遍较低,但在相对丰度上却存在显著差异,由于目前的研究较少,没有足够的证据证明微生物中同源蛋白的相对丰度会通过微生物间接影响宿主的消化利用和机体生长。
4 结论
本研究发现瘤胃液与粪便样本微生物结构组成差异显著。粪便及瘤胃中的优势菌门均为拟杆菌门和厚壁菌门;粪便中的优势属为拟杆菌属,瘤胃液中的优势属为普雷沃菌属。粪便中的丹毒丝菌纲、瘤胃液的甲烷杆菌纲可作为肉牛LRFI 个体筛选的潜在生物标志物。瘤胃及粪便微生物可部分解释肉牛RFI 差异,同时微生物基因功能上的差异也有助于理解肉牛RFI 相关的表型变异。

