舒尼替尼耐药肾癌细胞中circTMBIM6表达变化及其与miR-19a-3p/PPARα轴的调控关系
艾尼瓦尔·艾木都拉,张骥驰,张瑞丽,阿不来提·买买提明
1 新疆医科大学第一附属医院肿瘤中心,乌鲁木齐 830054;2 新疆医科大学第一附属医院泌尿中心
肾癌是人类泌尿系统最具侵袭性的恶性肿瘤之一,其中以肾细胞癌(RCC)最为常见,约占所有肾癌的85%[1]。研究显示,约33%的RCC 患者在初诊时就发现有转移病灶,最终发展为转移性肾细胞癌(mRCC)[2]。舒尼替尼是mRCC 一线治疗方案,初始反应率高达47%,但9~12 个月后往往发生耐药和肿瘤进展,提示对舒尼替尼获得性耐药[3]。目前关于mRCC 对舒尼替尼耐药的机制仍未明确,深入了解RCC的耐药机制有助于为mRCC患者提供更有效的治疗策略。研究表明,环状RNA(circRNA)可介导微小RNA(miRNA)调控靶基因信使RNA(mRNA)表达。circRNA可作为miRNA的分子海绵,与miRNA相互作用来调控信号通路相关基因的表达,从而影响肿瘤细胞的增殖、迁移、侵袭、凋亡及调节肿瘤耐药。但cirRNA 影响舒尼替尼耐药的分子机制鲜有报道。跨膜Bax抑制剂的母体6(TMBIM6)是一种钙离子通道样蛋白,可降低内质网(ER)膜表面表达的钙离子稳态,它在许多癌症类型中上调,参与了多种癌症的发生,而TMBIM6 在肾RCC 中表达和对舒尼替尼耐药性的影响尚无相关研究。本研究通过体外试验观察circTMBIM6 在舒尼替尼耐药RCC 细胞中的表达,并通过生物信息学预测和体外实验证实circTMBIM6 调控的 miRNA 和 mRNA,探讨 circTMBIM6在RCC细胞舒尼替尼耐药性的作用机制,以期为RCC的靶向治疗提供新的靶点。
1 材料与方法
1.1 材料 人肾透明细胞癌A498、Caci-1、780-O细胞株购于中科院上海细胞库。舒尼替尼购于大连美仑生物技术有限公司。DMEM 培养基、PBS、胎牛血清购于以色列BioInd 公司,0.25%胰蛋白酶+0.02%EDTA购于美国Biosharp公司,蛋白定量试剂盒购于北京索莱宝科技有限公司,抗体均购自英国Abcam 公司,ECL 显影液购自美国 Biosharp 公司,CCK-8 试剂盒购于江苏凯基生物技术股份有限公司。RNA 提取试剂盒购自超研生物科技有限公司(上海);Lipofectamine 2000 购于美国 Invitrogen 公司,双荧光素酶检测报告系统试剂盒、miR-19a-3p mimics 及阴性对照购于美国Promega 公司。野生型和突变型的circTMBIM6 的3′UTR、PPARα mRNA 的3′UTR 双荧光报告质粒购自上海基因股份有限公司。凝胶成像仪购自美国Bio-Rad 公司。酶标仪购于美国Thermo。
1.2 细胞的培养
1.2.1 正常RCC 细胞的培养 取A498、Caci-1、780-O细胞,加入含10%胎牛血清、青霉素100 U/mL和链霉素 100 µg/mL 的 DMEM 培养液,置于 37 ℃、5%CO2培养箱中常规培养,每48 h 更换培养液。当细胞融合达到90%时,0.25%胰酶消化后传代,取3代细胞进行后续实验。
1.2.2 舒尼替尼耐药细胞RCC 的培养 取A498、Caci-1、780-O 细胞,加入含10%胎牛血清、1%青霉素—链霉素的DMEM 培养液,加入终浓度为2.5µmol/L 的舒尼替尼,置于37 ℃、5%CO2培养箱中继续培养,开始耐药筛选。倒置显微镜下观察细胞的生长状态,待细胞生长至覆盖瓶底约90%左右时,按1∶2进行传代并冻存细胞备用。采用低浓度梯度法,分别以2.5、7.5、12.5、20.0 µmol/L 舒尼替尼培养细胞。在同一药物浓度作用细胞大约传代15 代左右稳定后增加药物浓度,最终建立舒尼替尼耐药细胞A498/S、Caci-1/S、780-O/S。
1.3 舒尼替尼耐药和正常RCC细胞circTMBIM6表达检测 采用qRT-PCR法。使用TRIzol试剂分别提取三种耐药细胞和正常RCC细胞中的总RNA,使用逆转录试剂盒将总RNA 转录成cDNA。采用SYBR Green PCR 试剂盒 CFX ConnectTMreal-time PCR 检测系统上进行qPCR。实时荧光定量PCR 采用两步法PCR 扩增标准程序。qPCR 引物及内参照U6逆转录引物由上海博尚生物技术有限公司设计合成。引物序列:上游 5′-TGTGAAGACAAAGGTGTTTTTGA-3′,下游:5′-TCAATATCAGGGAGCCCAAG-3′。反应条件:预变性95 ℃ 30 s;95 ℃ 5 s,60 ℃ 30 s,共40个循环。采用2-ΔΔCt法计算目的基因的相对表达量。实验重复3次,取平均值。
1.4 A498、Caci-1、780-O 细胞 PPARα 蛋白检测采用 Western blotting 法。取 A498、Caci-1、780-O 细胞,加入RIPA 裂解液提取细胞总蛋白,BCA 蛋白定量试剂盒测定蛋白浓度。取40 µg 总蛋白进行电泳,转膜后5%脱脂牛奶封闭2 h。洗膜3 次,加入PPARα 及 GAPDH 一抗,4 ℃孵育过夜。洗膜 3 次,加入二抗室温孵育2 h,洗膜3 次。ECL 化学发光显像。采用Image J 图像分析软件,以β-actin 为内参,计算PPARα与内参蛋白灰度值的比值,获得PPARα蛋白相对表达量。
1.5 circTMBIM6 靶 向 miRNA 和 RNA 的 预 测 及验证
1.5.1 miR-19a-3p 与 circTMBIM6、PPARα mRNA靶向关系的预测 采用circRNA interactome 数据库,预测 miR-19a-3p 与 circTMBIM6 的 3′非翻译区(3′UTR)是否可能存在互补结合。采用Targetscan数据库,预测 miR-19a-3p 与 PPARα mRNA 的 3′UTR是否存在直接结合。
1.5.2 miR-19a-3p 与 circTMBIM6、PPARα mRNA结合位点的验证 采用双荧光素酶报告基因实验。以pmirGLO 为载体,构建包含结合位点的野生型及突变型 circTMBIM6 3′UTR 和 PPARα mRNA 3′UTR质粒,然后转染至A498、Caci-1、780-O 细胞,同时分别转染 miR-19a-3p mimics 或 miR-NC 阴性对照,检测双荧光素酶报告基因活性。
1.5.3 circTMBIM6 对 miR-19a-3p 表达的调控作用观察 将A498、Caci-1、780-O 细胞分为抑表达组与空白对照组,以Lipofectamine2000 分别转染sicircTMBIM6、si-NC+miR-NC。采用qRT-PCR 法检测细胞miR-19a-3p的相对表达量,检测步骤同1.3。
1.5.4 miR-19a-3p 对 PPARα mRNA 和蛋白表达的调控作用观察 将A498、Caci-1、780-O 细胞分为miR-19a-3p 过表达组、miR-19a-3p 抑表达组与空白对照组,以Lipofectamine 2000 分别转染miR-19a-3p mimics、miR-19a-3p inhibitor、si-NC+miR-NC。24 h后收集各组细胞,采用qRT-PCR 法检测细胞PPARα mRNA、Western blotting法检测PPARα蛋白,检测步骤同1.3。
1.5.5 circTMBIM6 通 过 miR-19a-3p 靶 向 调 控PPARα 表达的作用观察 将A498、Caci-1、780-O 细胞分为阴性对照组、miR-19a-3p抑制组、circTMBIM6抑制组,分别转染si-NC+miR-NC、si-NC+miR-19a-3p inhibitor、si-circTMBIM6+miR-NC、si-circTMBIM6+miR-19a-3p inhibitor。 采 用 qRT-PCR 和 Western blotting法检测PPARα mRNA 和蛋白表达,检测步骤同1.3。
1.6 舒尼替尼耐药RCC 细胞中circTMBIM6 与miR-19a-3p/PPARα 轴的调控作用观察 将A498/S、Caci-1/S、780-O/S 细胞分为阴性对照组、miR-19a-3p 抑制组、circTMBIM6 抑制组,分别转染si-NC+miR-NC、si-NC+miR-19a-3p inhibitor、si-circTMBIM。加入100 µL CCK-8 试剂,37 ℃孵育2 h,上酶标仪测量各孔450 nm 处的OD 值,计算细胞增殖抑制率(%)=[(OD对照孔-OD实验孔)/OD对照孔]×100%。实验重复3次,取平均值。
1.7 统计学方法 采用SPSS18.0 统计软件。符合正态分布的计量资料以表示,两组间比较采用t检验,多组间比较采用One-way ANOVA 法。P<0.05为差异有统计学意义。
2 结果
2.1 舒尼替尼耐药与正常RCC细胞circTMBIM6表达比较 A498、Caci-1、780-O 细胞circTMBIM6 相对表达量分别为 1.00 ± 0.11、3.42 ± 0.32、3.64 ±0.63,A498/S、Caci-1/S、780-O/S 细胞 circTMBIM6相对表达量分别为3.64±0.71、7.91±0.80、9.47±0.78。与正常 RCC 细胞比较,A498/S、Caci-1/S、780-O/S 细胞 circTMBIM6 表达水平均升高(P均<0.01)。
2.2 circTMBIM6 与 miR-19a-3p 和 PPARα 靶向关系的预测结果 Starbase 数据库预测miR-19a-3p 与circTMBIM6 的 3′UTR 存在结合区域,Targetscan 数据库预测 miR-19a-3p 与 PPARα mRNA 的 3′UTR 存在潜在的结合区域。见表1、2。

表1 miR-19a-3p与circTMBIM6 3′UTR结合位点的预测结果
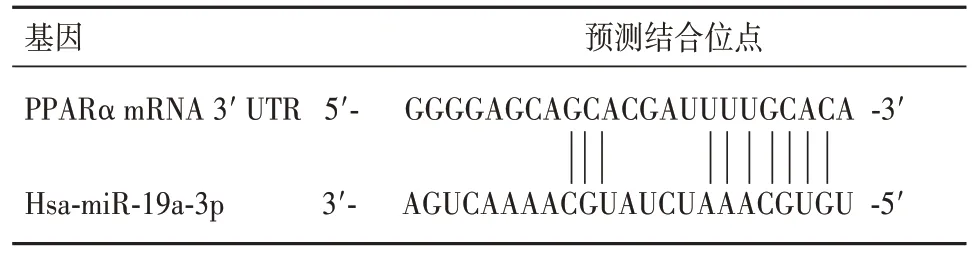
表2 miR-19a-3p与PPARα 3′UTR结合位点的预测结果
2.3 miR-19a-3p 与 circTMBIM6、PPARα mRNA 靶向结合的验证结果 miR-19a-3p mimics+circTMBIM6 WT 组荧光素酶活性低于circTMBIM6 WT+miR-NC 组(P<0.05),miR-19a-3p mimics+circTMBIM6 MUT组与circTMBIM6 MUT+miR-NC组荧光素酶活性无统计学差异(P>0.05),说明circTMBIM6-WT 能够与miR-19a-3p 结合,见表3。转染miR-19a-3p mimics 可以降低野生型 PPARα mRNA 3′UTR 荧光素酶报告基因活性(P<0.05),但对突变型PPARα mRNA 3′UTR 报告基因活性无显著影响(P>0.05),提示PPARα是miR-19a-3p的靶点,见表4。
表3 miR-19a-3p对circTMBIM6 3′UTR荧光素酶报告基因活性调控()

表3 miR-19a-3p对circTMBIM6 3′UTR荧光素酶报告基因活性调控()
注:与miR-NC组比较,*P<0.05。
circTMBIM6-WT 1.02±0.28 0.44±0.48*组别miR-NC组miR-19a-3p mimics组circTMBIM6-MUT 0.97±0.28 0.88±0.14
表4 miR-19a-3p对PPARα mRNA 3′UTR荧光素酶报告基因活性调控()

表4 miR-19a-3p对PPARα mRNA 3′UTR荧光素酶报告基因活性调控()
注:与miR-NC组比较,*P<0.05。
组别miR-NC组miR-19a-3p mimics PPARα-WT 1.12±0.25 0.41±0.24*PPARα-MUT 1.24±0.45 1.11±0.17
2.4 circTMBIM6 对miR-19a-3p 表达的影响 三种RCC 细胞中,circTMBIM6 抑表达组 miR-19a-3p 表达均高于空白对照组(P均<0.05)。见表5。
表5 circTMBIM6对miR-19a-3p表达的影响()

表5 circTMBIM6对miR-19a-3p表达的影响()
注:与空白对照组比较,*P<0.05。
组别786-O 2.13±0.64*0.49±0.13 miR-19a-3p circTMBIM6抑表达组空白对照组A498 4.12±0.65*0.15±0.57 Caki-1 3.37±0.35*0.34±0.21
2.5 miR-19a-3p 对 RCC PPARα mRNA 及蛋白表达的影响 三种RCC 细胞中,PPARα mRNA 及蛋白水平在抑表达组>对照组>过表达组(P均<0.05),见表6、7。
表6 miR-19a-3p对PPARα mRNA表达的影响()
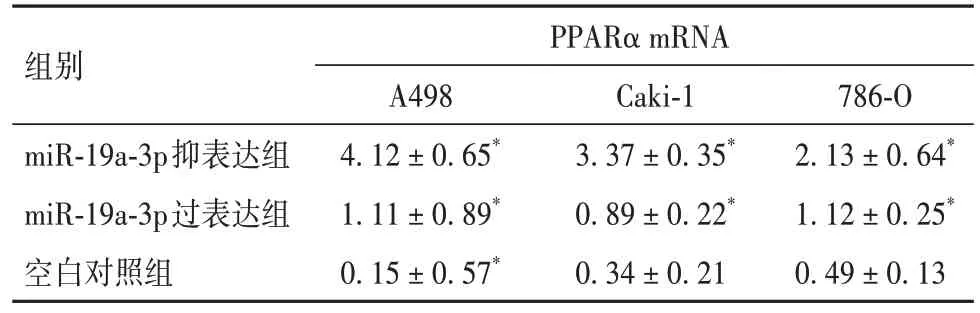
表6 miR-19a-3p对PPARα mRNA表达的影响()
注:与空白对照组比较,*P<0.05。
组别PPARα mRNA miR-19a-3p抑表达组miR-19a-3p过表达组空白对照组786-O 2.13±0.64*1.12±0.25*0.49±0.13 A498 4.12±0.65*1.11±0.89*0.15±0.57*Caki-1 3.37±0.35*0.89±0.22*0.34±0.21
表7 miR-19a-3p对PPARα蛋白表达的影响()

表7 miR-19a-3p对PPARα蛋白表达的影响()
注:与空白对照组比较,*P<0.05。
组别PPARα蛋白miR-19a-3p抑表达组miR-19a-3p过表达组空白对照组786-O 3.13±0.44*0.42±0.22*1.23±0.11 A498 6.20±0.21*1.34±0.27*3.24±0.34 Caki-1 4.37±0.35*0.77±0.14*2.34±0.21
2.6 舒尼替尼耐药RCC 细胞中circTMBIM6 通过miR-19a-3p 靶向调控PPARα 表达 在舒尼替尼耐药 RCC 细胞系中,miR-19a-3p 抑制组 PPARαmRNA和蛋白表达较阴性对照组上调,circTMBIM6 抑制组PPARα mRNA和蛋白表达较阴性对照组下调(P均<0.05)。见表8、9。
表8 舒尼替尼耐药RCC细胞中circTMBIM6通过miR-19a-3p对PPARα mRNA表达的影响()
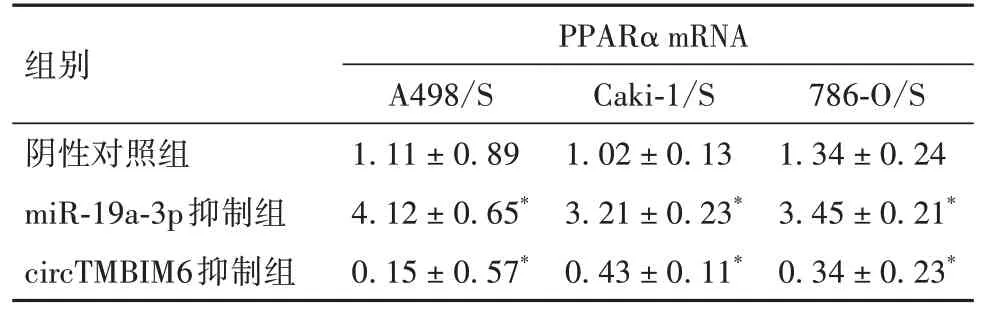
表8 舒尼替尼耐药RCC细胞中circTMBIM6通过miR-19a-3p对PPARα mRNA表达的影响()
注:与阴性对照组比较,*P<0.05。
786-O/S 1.34±0.24 3.45±0.21*0.34±0.23*组别阴性对照组miR-19a-3p抑制组circTMBIM6抑制组PPARα mRNA A498/S 1.11±0.89 4.12±0.65*0.15±0.57*Caki-1/S 1.02±0.13 3.21±0.23*0.43±0.11*
表9 舒尼替尼耐药RCC细胞中circTMBIM6通过miR-19a-3p对PPARα蛋白表达的影响()

表9 舒尼替尼耐药RCC细胞中circTMBIM6通过miR-19a-3p对PPARα蛋白表达的影响()
注:与阴性对照组比较,*P<0.05。
786-O/S 2.67±0.33 6.32±0.43*1.55±0.89*组别阴性对照组miR-19a-3p抑制组circTMBIM6抑制组PPARα蛋白A498/S 2.23±0.53 4.50±0.60*1.23±0.23*Caki-1/S 2.79±0.23 5.11±0.76*0.96±0.64*
2.7 circTMBIM6 通 过 miR-19a-3p/PPARα 轴 对RCC 细胞舒尼替尼耐药性的影响 circTMBIM6 抑制组细胞增殖抑制率高于miR-19a-3p 抑制组和阴性对照组(P均<0.05)。见表10。
表10 各组舒尼替尼耐药RCC细胞增殖抑制率比较(%,)
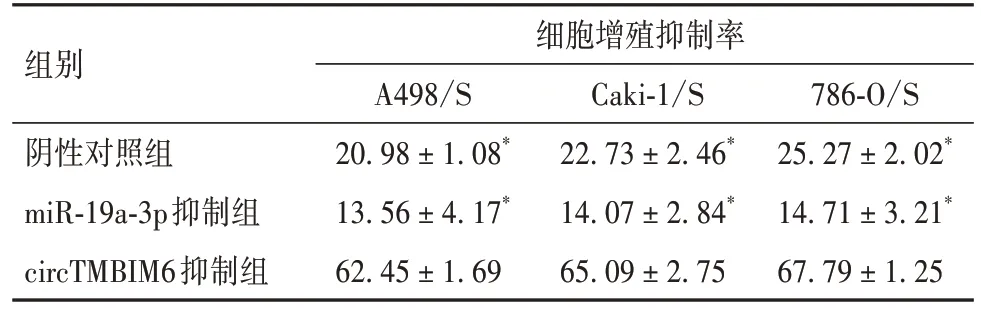
表10 各组舒尼替尼耐药RCC细胞增殖抑制率比较(%,)
注:与circTMBIM6抑制组比较,*P<0.05。
组别细胞增殖抑制率A498/S 20.98±1.08*13.56±4.17*62.45±1.69 786-O/S 25.27±2.02*14.71±3.21*67.79±1.25 Caki-1/S 22.73±2.46*14.07±2.84*65.09±2.75阴性对照组miR-19a-3p抑制组circTMBIM6抑制组
3 讨论
近年来,以舒尼替尼为首的酪氨酸激酶抑制剂的研究在mRCC 治疗方面取得了很大进展[4]。舒尼替尼可以有效抑制肿瘤血管生成,最终抑制肿瘤生长,但耐药性是制约疗效的瓶颈。目前对于肾癌临床无有效治疗方法,其分子生物学基础及调控机制尚不完全清楚。因此,我们迫切需要了解肾癌的发病及耐药机制,从而为mRCC 患者提供更有效的治疗策略。
circRNA 作为 miRNA 和 lncRNA 之后新的研究热点,对肿瘤的影响逐渐被认识。由于其特殊的结构,circRNA 比lncRNA 更稳定,更适合于肿瘤的诊断和治疗。circRNA 是由前体RNA 剪接而形成的环状结构,主要由编码蛋白质基因的外显子产生。circRNA 作为一种调控RNA,可以促进基因转录、调节选择性剪接、抑制miRNA 成熟、促进蛋白—蛋白相互作用和miRNA 海绵等多种功能。此外,circRNA-miRNA轴可能是肿瘤治疗的潜在靶点并在肿瘤的诊断和预后中具有巨大潜能。TMBIM6 又称Bax-1 抑制剂,MBIM6 基因位于 12p13.12 号染色体上,是一种位于内质网膜上的阳离子(Ca2+载体),包含6 个跨膜Bax 抑制剂基序。TMBIM6 在乳腺癌、肺癌、前列腺癌、鼻咽癌和肝癌等多种肿瘤类型中表达上调,并改变葡萄糖代谢和激活钠—氢交换器来促进癌症转移[5-6]。研究发现,TMBIM6 缺失或敲除会抑制原发肿瘤的生长。此外,TMBIM6 可上调mTORC2 的激活,并刺激糖酵解、蛋白质合成、脂质合成基因和糖基化蛋白的表达。研究发现,TMBIM6拮抗剂可以阻止TMBIM6 与mTORC2 结合,降低mTORC2 活性,还可以调节TMBIM6 泄漏的钙离子,进一步抑制肿瘤的形成和进展[7]。DOYCHEVA 等[8]发现,TMBIM6 是内质网应激诱导凋亡的一种新的调节因子。根据KIM等[9]的研究,TMBIM6是一种跨膜Bax 抑制剂,可以参与顺铂诱导生殖毒性的分子机制;对野生型和TMBIM6 缺陷小鼠模型注射顺铂后,突变型小鼠内质网应激水平和蛋白质羰基化水平高于野生型小鼠。经慢病毒转染的突变小鼠TMBIM6 表达后,睾丸和血清中的各项指标均可恢复,提示TMBIM6 可通过诱导HO-1 来保护顺铂致睾丸毒性。尽管许多证据支持TMBIM6参与了多种癌症的发生,但TMBIM6 在RCC 耐药进展中作用的分子机制的文献较少。我们研究TMBIM6在RCC对靶向药物舒尼替尼耐药中的作用,有一定的前沿意义。本研究结果显示,对RCC细胞系A498、Caki-1、780-0进行耐药培养后,耐药RCC细胞中的circTMBIM6表达显著上升,提示circTMBIM 在RCC 细胞对舒尼替尼的耐药过程中发挥作用。
cirRNAc 作为miRNA 海绵,可以竞争性地结合miRNA,从而影响miRNA 的活性和下游靶基因的表达,这一机制已在多种肿瘤中得到证实,但是RCC对舒尼替尼耐药的机制尚未清楚。故本研究首先探讨circTMBIM6 调控的下游miRNA 及其靶基因。本研究通过 Starbase 数据库预测 circTMBIM6 3′UTR 中存在miR-19a-3p 目标碱基序列,通过生物信息学软件Targetsan 预测 PPARα 可能为 miR-19a-3p 的潜在靶基因,并采用双荧光素酶报告实验进行了进一步的证明。后续通过qRT-PCR 验证,结果显示,转染si-circTMBIM6 降低miR-19a-3p 表达水平,进一步揭示circTMBIM6竞争性结合miR-19a-3p并升高其表达。
miR-19a-3p 属于miRNA-17-92 基因簇。它位于人类染色体13q31.3,具有重要的生物学功能。研究显示,miR-19a/b 能够靶向肿瘤抑制因子MXD1,调控胃癌细胞的转移,通过PITX1 下调影响转录激活的PDCD5 促进细胞恶性,并预测胃癌的不良预后。YANG 等[12]发现,miR-19a-3p 在小鼠乳腺癌细胞中可下调M2表型巨噬细胞的表达,而miR-19a-3p在脊椎动物中广泛保守。miR-19a-3p mimic 基因转染RAW264.7 巨噬细胞后,Fra-1 基因的表达明显受到抑制(Fra-1基因作为支持乳腺癌侵袭发展的原癌基因),并降低下游基因Fra-1、VEGF、STAT3 和PS的表达,同时通过荧光素酶报告系统验证miR-19a-3p与Fra-1的结合活性。由此可见,miR-19a-3p可下调M2 巨噬细胞的M2 表型,在上调Fra-1 表达、诱导M2 巨噬细胞极化中发挥重要作用。FENG 等[13]分别构建miR-19a-3p 模拟质粒和空白对照质粒,转染DU145细胞,发现过表达miR-19a-3p能够抑制SOX4蛋白表达,从而抑制DU145 细胞的迁移和侵袭,说明miR-19a-3p 通过SOX4 抑制前列腺癌细胞的迁移、侵袭和EMT。
本研究结果显示,转染miR-19a-3p inhibitor 可在蛋白及mRNA 水平负向调控PPARα。舒尼替尼耐药的RCC细胞中circTMBIM6表达显著上升,并通过与miR-19a-3p结合,促进PPARα 表达。由于本课组前期研究发现敲低PPARα 通过激活NF-κB 信号通路促进舒尼替尼对RCC 细胞的药物敏感性[14],提示PPARα可能参与耐药调控过程。
为了探讨 circTMBIM6/miR-19a-3p/PPARα 轴在RCC 舒尼替尼的耐药过程中调控机制。进一步探讨这两个非编码RNA 是否可以调节PPARα 的表达影响RCC 对舒尼替尼敏感性,我们将si-NC+miRNC、si-NC+miR-19a-3p inhibitor、si-circTMBIM6+miR-NC 分别转染到舒尼替尼耐药的RCC 细胞,结果显示,抑制 miR-19a-3p 后,PPARα 基因和蛋白表达显著上调,细胞增殖抑制率下降,细胞活性增强,RCC 细胞对舒尼替尼的耐药性增加;而抑制circRNA-TMBIM6 可 上 调 miR-19a-3p 表 达 ,抑 制PPARα 基因和蛋白表达,细胞增殖抑制率升高,细胞存活率降低,从而提高RCC 细胞对舒尼替尼的敏感性。
综上所述,本研究通过生物信息学分析和进一步验证试验,初步显示circTMBIM6通过海面吸附作用 miR-19a-3p,影响PPARα 表达,调控 RCC 对舒尼替尼的敏感性。因此,circRNA TMBIM6可能是潜在的RCC耐药治疗新靶点。

