中华绒螯蟹翻译起始因子4G基因在幼体发育阶段的mRNA表达模式与适应性进化分析
吴海霞,时昕晔,2,周开亚,严 洁,李 鹏
(1.南京师范大学生命科学学院,江苏 南京 210023) (2.南京师范大学海洋科学与工程学院,江苏 南京 210023)
真核翻译起始因子(eukaryotic translation initiation factors,eIFs)是一类调控起始密码子周围功能核糖体复合物组装的蛋白质,在真核细胞蛋白质翻译起始过程中发挥重要作用. 真核生物的基因表达受基因转录和翻译水平的调节;在翻译级联的四个阶段中,起始是关键的速率限制步骤,因此eIFs是蛋白质生物合成的关键调节剂[1]. 但随着研究的深入,发现eIFs的表达异常会导致一些基因无法正常翻译,致使细胞恶变、肿瘤、神经退行性疾病的发生[2]. 例如,eIFs家族与恶性肿瘤的发生、发展和预后关系密切,主要体现在基因表达水平的变化上,已有研究在子宫内膜癌中观察到eIF4G、peIF2α、eIF3H的显著过表达,eIF3C表达显著降低[3].
目前eIFs家族成员共有20余种,通常以复合物的形式实现其生物学活性[4];其中对eIF2、eIF3、eIF4、eIF5和eIF6的研究较多. eIF4家族由eIF4A、eIF4B、eIF4E、eIF4G等亚基组成(称作eIF4F),真核细胞蛋白质合成通常从该复合物与mRNA 5′端的7-甲基化鸟嘌呤核苷帽状结构(m7GpppN)特异性结合开始[4]. eIF4G作为组装eIF4F复合物的骨架蛋白,与RNA解旋酶eIF4A有两个结合位点,与mRNA帽结合蛋白eIF4E、核糖体结合复合物eIF3、促分裂原激活的蛋白激酶1(mitogen-activated protein kinase interacting kinase,MNK1)和聚腺苷酸结合蛋白(poly-A-binding protein,PABP)各有一个结合位点[5-6]. eIF4G与PABP相互作用以使mRNA有效环化,从而增强eIF4F复合物的帽结合活性以及60S与40S核糖体亚基的连接[7]. eIF4G与eIF4E结合,增加eIF4E介导的帽识别和eIF4F复合物的帽结合活性;eIF4G与eIF3的结合有助于将40S核糖体亚基带入eIF4F复合体[8]. 此外,eIF4G将43S预启动复合物(PIC,包括40S核糖体亚基、eIF3复合物和相关因子)募集到mRNA 5′非翻译区(terminal untranslated region,UTR),随后启动扫描寻找起始密码子[9].
从酵母、植物到哺乳动物,迄今已在许多物种中鉴定出eIF4G,其一般具有3个典型结构域:MIF4G、MA3和W2/eIF5C结构域,均为由α螺旋组成的HEAT重复片段结构. 其中哺乳动物细胞具有3种同工型,即eIF4GI、eIF4GII和p97/NAT1/DAP-5[10]. eIF4GI和eIF4GII都具有eIF4A、eIF4E、eIF3和PABP的结合位点,但eIF4GI与eIF4GII的浓度在不同细胞中有所不同,通常eIF4GI的表达浓度高于eIF4GII[11]. 有研究发现eIF4G基因表达水平的上升与细胞恶性转化、抑制凋亡有关,而表达水平下降与营养缺乏、热休克、缺氧等因素有关[12]. Das等报道eIF4G可以通过充当细胞mRNA代谢的衔接分子,总体上调整mRNA的功能状态[13]. Orellana等研究表明,当缺乏合成代谢刺激时,脂多糖LPS诱导的蛋白质合成减少与核糖体效率和eIF4E·eIF4G组装体降低有关,因此发育过程涉及核糖体数目和翻译因子丰度降低,以及eIF2α磷酸化增加[14]. 红细胞特异性RNA结合蛋白RBM38与eIF4G相互作用,促进mRNA的翻译,从而动态控制哺乳动物红细胞的生成过程[15]. Ghosh和Lasko研究发现,果蝇eIF4GI基因敲低并不影响雄性生育能力,而eIF4GII在精子发生过程中的作用却很明显;这些蛋白质在双重敲除后,发现其在精子发生早期功能冗余[16]. 以上所有发现均证明eIF4G家族在蛋白质合成中起关键调节作用,从而调控机体的代谢、生长、发育和繁殖过程.
中华绒螯蟹(Eriocheirsinensis)属于真蟹类方蟹科,其形态在发育早期经历从似虾型向蟹型进化的“短尾化(Brachyurization)”过程[17]. 短尾化期间,蟹的外部形态发生显著变化,包括腹部退化、折叠在头胸甲下方并插入到步足之间或特殊腔内[18]. 目前,对短尾化变态的分子机制研究甚少,主要是对相关基因表达量变化的探究[19-22],比较功能基因在幼体不同发育阶段或不同部位的表达差异;以及中华绒螯蟹幼体发育阶段的比较转录组学分析[23],筛选了1016个差异表达基因参与变态、感官知觉和免疫过程;但尚未研究eIF4G基因在甲壳动物变态发育过程的作用,且未从进化生物学方面来探讨相关基因是否在甲壳动物进化过程中发生适应性进化. 本研究利用RACE(Rapid amplification of cDNA ends)技术克隆中华绒螯蟹eIF4G基因全长cDNA序列(命名为EseIF4G),运用生物信息学方法分析eIF4G基因的理化性质、功能结构,并通过qRT-PCR(Quantitative real-time PCR)方法探究该基因在中华绒螯蟹幼蟹10个发育时期的mRNA表达差异情况,以及从进化生物学角度对中华绒螯蟹EseIF4G进行选择压力分析,为进一步探究该基因在中华绒螯蟹早期发育过程的分子进化机制以及十足目甲壳动物的短尾化变态发育研究提供参考.
1 材料与方法
1.1 实验样品采集
实验材料取自江苏泰州兴化中堡镇中华绒螯蟹养殖场,取仔蟹Ⅰ期腹部组织约30 mg,加入RNA保护试剂,使用无RNase的手术剪剪碎混匀后保存于-80 ℃备用. 另取中华绒螯蟹受精卵(oosperm,O)、溞状幼体Ⅰ期(the first zoea stage,Z1)、溞状幼体Ⅱ期(the second zoea stage,Z2)、溞状幼体Ⅲ期(the third zoea stage,Z3)、溞状幼体Ⅳ期(the fourth zoea stage,Z4)、溞状幼体Ⅴ期(the fifth zoea stage,Z5)、大眼幼体期(megalopa stage,M)、仔蟹Ⅰ期(the first crab stage,J1)、仔蟹Ⅱ期(the second crab stage,J2)、仔蟹Ⅲ期(the third crab stage,J3)整体组织样品各约30 mg,加入RNA保护试剂,剪碎匀浆后于-80 ℃保存备用.
1.2 EseIF4G基因cDNA全长扩增
按RNeasy Mini Kit(Qiagen,Germany)说明书提取总RNA,使用BioPhotometer核酸和蛋白质定量仪(Eppendorf,Germany)检测RNA的浓度和纯度(OD260/OD280). 采用Super SMARTTMPCR cDNA Synthesis Kit(Clontech,USA)合成cDNA,用于后续RACE实验克隆EseIF4G基因,以及实时荧光定量PCR(qRT-PCR)检测.
根据课题组前期构建的中华绒螯蟹抑制性消减杂交文库[20]中获得的一个492 bp的表达序列标签(Expressed sequence tags,EST)序列(GenBank登录号:GE329190)设计RACE反应特异引物(见表1). 以仔蟹I期腹部组织cDNA为模板,用EseIF4G基因特异性引物和通用引物10×Universal Primer A Mix(UPM)进行RACE PCR扩增. 5′RACE和3′RACE产物进行切胶纯化和载体连接,连接产物转入Escherichiacoli细胞DH5α(TaKaRa,中国大连)并经蓝白斑筛选,选取3个阳性克隆送往生工生物工程(上海)股份有限公司测序.
1.3 EseIF4G基因序列分析
将拼接获得的EseIF4G基因全长cDNA序列用NCBI(http://www.ncbi. nlm.nih.gov/)进行cDNA全长完整性验证、开放阅读框查找(open reading frame,ORF). 使用ExPASy服务器(https://web.expasy.org/)[24]的ProtParam和ProtScale工具分析EseIF4G蛋白的理化性质和疏水性. 使用SignalP程序(http://www.cbs.dtu.dk/services/SignalP)[25]预测信号肽,MotifScan程序(http://myhits.isb-sib.ch/cgi-bin/motif_scan)查找motif,SMART软件(http://smart.embl-heidelberg.de/)[26]分析结构功能域,PSORT II软件(http://psort.hgc.jp/form2.html)进行亚细胞定位,TMHMM v.2.0软件(http://www.cbs.dtu.dk/services/TMHMM-2.0/)[27]预测跨膜区. 使用在线软件PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)预测二级结构,SWISS-MODEL(http://www.expasy.org/swissmod/SWISS-MODEL.html)预测三级结构[24]. 使用MEGA X软件构建邻接树(Neighbor-joining tree,NJ tree)来验证EseIF4G蛋白在进化过程中与其他同源蛋白的关系[28].

表1 本研究所用的主要引物Table 1 Primers used in this study
1.4 实时荧光定量PCR(qRT-PCR)检测
用Prime Primer 5软件设计引物qβ-actin-F和qβ-actin-R(表1)用于qRT-PCR检测,选择β-actin基因(GenBank登录号:HM053699.1)作为内参基因,其引物qβ-actin-F和qβ-actin-R引自文献[29]. 使用SYBR®Premix Ex-TaqTMII试剂盒(TaKaRa,中国大连)在ABI Step One PlusTM仪器上检测EseIF4G基因在中华绒螯蟹幼体10个不同发育阶段的mRNA表达情况. PCR反应和实验样本均设计3次重复,采用2-ΔΔCt方法计算相对表达量[30]. 用SPSS 25.0软件进行单因素方差分析和Tukey’s法多重比较,以(平均值±标准差)表示,显著性差异水平以α=0.05表示,图中用不同的小写字母标注.
1.5 适应性进化分析
1.5.1 序列获取
为研究中华绒螯蟹EseIF4G基因的适应性进化,选择了其他13种代表性节肢动物物种的eIF4G基因序列加入分析,涉及甲壳亚门、螯肢亚门和六足亚门,并选择黑头软口鲦(Pimephalespromelas)作为外群. 部分物种的eIF4G基因序列从NCBI数据库直接下载,或从NCBI下载的基因组中用近缘物种的序列执行本地blast获取,本研究所用的直系同源基因在NCBI上的登录号和本地获取所用物种基因组信息见表2.

表2 本研究所用物种基因或基因组信息Table 2 Species genes or genomes information used in this study
1.5.2 构建系统发育树
使用PRANK软件对上述获取的eIF4G基因序列进行密码子水平的比对[31]. 使用Gblocks软件过滤比对后序列,删除一些低质量或高变异度的序列区域,仅留进化保守的区域用于后续分析[32]. 使用IQ-TREE自动测试并选择最佳替代模型,构建最大似然树(maximum likelihood tree,ML tree)[33]. 该系统发育树同时也被用于构建后续选择压力分析的树文件. 从TimeTree(http://www.timetree.org/)获取该15个物种的系统发育关系[34].
1.5.3 检测特异性位点
选择其中6种甲壳动物,将其编码区序列翻译为氨基酸序列,PRANK比对后使用FasParser软件鉴定中华绒螯蟹EseIF4G基因的特异性位点(Segregating sites)[35].
1.5.4 选择压力分析
非同义替代率(Non-synonymous substitution rate,dN)与同义替代率(Synonymous substitution rate,dS)的比值ω(dN/dS),通常作为检测选择压力的标准,其中ω>1,ω=1和ω<1分别代表基因受到正选择(Positive selection),中性选择(Neutral selection)和纯化选择(Purify selection). 本研究使用PAML 4.9软件包的CODEML程序,选择位点模型(Site model)、分支模型(Branch model)和分支位点模型(Branch-site model)进行进化分析[36].
使用位点模型检测eIF4G基因在节肢动物数据集中受正选择的位点,比较M8a和M8模型,利用似然率检测方法(Likelihood ratio teats,LTRs),计算2ΔlnL值与自由度之间的卡方分布,若P<0.05,则两个模型之间有显著差异[37]. 在M8模型中,利用经典贝叶斯方法BEB(Bays empirical bays)计算位点的后验概率(Posterior probability,PP),若PP>0.95,则被认为是潜在的正选择位点[38]. 在分支模型中,首先比较one-ratio和free-ratio模型以确定支系是否具有不同的进化速率;其次,将中华绒螯蟹末端分支设为前景支,比较 one-ratio 模型与two-ratio模型来判断前景分支是否受正选择. 使用分支位点模型检测某一进化谱系受正选择作用影响的位点,本研究中同样将中华绒螯蟹的末端分支设为前景支进行分析,若Ma0和Ma模型差异显著且ω>1时,接受正选择模型Ma,同时所检测的正选择位点PP>0.95,则被认为是正选择位点.
此外,使用Datamonkey(http://www.datamonkey.org/)中的固定效应似然法(fixes effects likelihood,FEL)、单一似然祖先计数法(singe likelihood ancestor,SLAC)和FUBRA(Fast unconstrainted bayesian approximation)检测正选择位点,设置FEL和SLAC显著水平小于0.2,FUBAR后验概率值大于0.8[39]. 最后,整合PAML和Datamonkey筛选的正选择位点,将两种方法以上检测到的位点视为显著的正选择位点,并标注在模拟的中华绒螯蟹eIF4G蛋白三维结构中.
2 结果与讨论
2.1 中华绒螯蟹EseIF4G基因cDNA全长序列的克隆与分析
通过RACE PCR扩增与测序获得EseIF4G基因序列片段,使用DNAStar 7.1软件拼接获得的各序列,获得中华绒螯蟹EseIF4G基因的cDNA全长序列为3 769 bp(图1),GenBank登录号为KF199900. 其包括568 bp的5′-UTR、822 bp的3′-UTR和2 379 bp的ORF. 其编码的蛋白质具有792个氨基酸,分子量为89.6 kDa,理论等电点pI为6.07. EseIF4G蛋白是不稳定蛋白和非分泌型蛋白,没有明显的疏水性区域,没有跨膜区且不具有信号肽,定位于细胞核可能性高达91.3%.
推导的氨基酸序列进行同源比对分析后发现,本实验克隆的EseIF4G蛋白与其他物种显示出较高的同源性,例如与克氏原螯虾(Procambarusclarkii,GenBank登录号:ASW35070.1)eIF4G蛋白的同源性高达84%,与南美白对虾(P.vannamei,GenBank登录号:XP_027234070.1)eIF4G2-like蛋白的同源性为86%,与灰眼雪蟹(C.opilio,GenBank登录号:KAG0719495.1)eIF4G2蛋白的同源性为83%,与普通卷甲虫(A.vulgare,GenBank登录号:RXG61596.1)eIF4G2蛋白的同源性为60%等.

3′RACE引物以方框标注,5′RACE引物以波浪线标注,ORF扩增引物以双下划线标注;MIF4G、MA3和W2/eIF5C结构域被分别标注为阴影,推导的加尾信号用粗体和下划线表示,终止密码子以“*”标注.图1 中华绒螯蟹EseIF4G基因的cDNA全长序列和推导的氨基酸序列结构Fig.1 The full-length cDNA and predicted amino acid sequence of EseIF4G gene from Eriocheir sinensis
用SMART分析EseIF4G蛋白的结构域,结果表明EseIF4G蛋白含有MIF4G结构域、MA3结构域和W2/eIF5C结构域,前两个结构域是eIF4G蛋白家族共有的典型结构域. 用Motif Scan搜索EseIF4G蛋白的motif,结果表明EseIF4G蛋白可能还含有如下功能基序:6个N端糖基化(Asn糖基化)位点、2个环腺苷酸(cyclic adenosine monophosphate,cAMP)磷酸化位点、11个酪蛋白激酶II(casein kinase II,CK2)磷酸化位点、7个N-酰基化(N-myristoylation)位点、8个蛋白激酶C(protein kinase C,PKC)磷酸化位点、1个NLS_BP核定位信号和1个富含Gln区等. 使用PSIPRED预测EseIF4G蛋白的二级结构,结果表明EseIF4G蛋白包含30个α螺旋,无β-折叠和31个无规则卷曲. SWISS-MODEL对EseIF4G蛋白进行同源建模,显示该蛋白与模板3l6a.1.A蛋白的匹配度最高,构建的蛋白三维结构图如图2所示,与PSIPRED预测结果一致,该蛋白为全α蛋白.
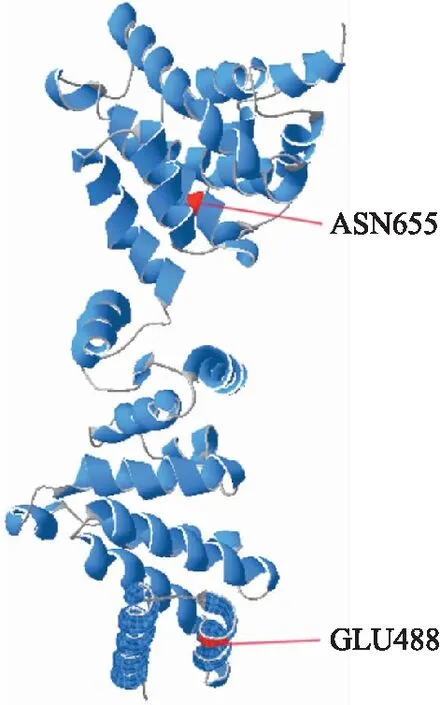
图中蓝色代表α-螺旋,浅灰色代表无规则卷曲,红色标注受正选择的位点.图2 中华绒螯蟹EseIF4G蛋白的三维结构Fig.2 Predicted three-dimensional structure ofEseIF4G from Eriocheir sinensis
2.2 多序列比对和系统发育分析
为了更好地研究EseIF4G与其他同源蛋白的关系,从NCBI上下载了包括脊椎动物和无脊椎动物在内的22个不同物种的eIF4G蛋白序列,使用MEGA X软件基于邻接法构建了NJ系统发育树(图3). 系统发育分析显示,EseIF4G与无脊椎动物分支(Crustacea+Insecta+Merostomata,甲壳纲+昆虫纲+肢口纲)聚在了一起;同时,中华绒螯蟹与南美白对虾(P.vannamei)、普通卷甲虫(A.vulgare)形成了甲壳纲分支,该分支与昆虫纲分支和肢口纲分支完全分离.
2.3 中华绒螯蟹EseIF4G基因在其不同幼体发育阶段的mRNA表达模式
利用qRT-PCR技术对EseIF4G基因在中华绒螯蟹其不同幼体发育阶段的mRNA表达进行检测,结果如图4所示. 以受精卵期的基因表达量作为参照,EseIF4G基因在中华绒螯蟹10个不同的幼体发育阶段广泛表达. 总体上,EseIF4GmRNA表达量在受精卵、溞状幼体Ⅰ、Ⅱ、Ⅳ期相对较高,相反在溞状幼体Ⅴ期、大眼幼体期、仔蟹Ⅰ、Ⅱ期相对较低.EseIF4G基因的表达量在短尾化期间显著波动.EseIF4G基因在溞状幼体Ⅱ期表达量最高,而在溞状幼体Ⅴ期和仔蟹Ⅱ期表达量最低(P>0.05). 值得注意的是,在Z5期急剧下降至最低后,EseIF4G基因表达量在大眼幼体期和仔蟹Ⅰ期逐步增加(P<0.05).

每一枝上的数值表示Neighbor-Joining bootstrap值;氨基酸序列号在物种名之后,不同分支被不同颜色覆盖;各物种eIF4G蛋白的结构域在对应分支后方显示.图3 基于邻接法构建的EseIF4G及其同源蛋白的聚类树状图Fig.3 Phylogenetic tree of EseIF4G and its homologs bases on neighbor-joining method

O:受精卵,Z1:溞状幼体I期,Z2:溞状幼体Ⅰ期,Z2:溞状幼体Ⅱ期,Z3:溞状幼体Ⅲ期,Z4:溞状幼体Ⅳ期,Z5:溞状幼体Ⅴ期,M:大眼幼体期,J1:仔蟹Ⅰ期,J2:仔蟹Ⅱ期,J3:仔蟹Ⅲ期. 以卵期基因的表达量为参照,采用(平均值±标准差)表示,不同的小写字母表示不同发育阶段表达量的差异水平显著(P<0.05),内参基因β-actin,n=3.图4 中华绒螯蟹EseIF4G基因在其不同幼体发育阶段的相对表达量Fig.4 The relative expression level of EseIF4G gene in different larval development stages of Eriocheir sinensis
2.4 中华绒螯蟹EseIF4G基因的适应性进化分析
2.4.1 系统发育关系
利用包括中华绒螯蟹在内的14种节肢动物以及外群黑头软口鲦的eIF4G基因序列,基于最大似然法构建了系统发育树. 结果如图5显示,eIF4G基因树分为三个主要的进化枝:甲壳亚门进化枝(Crustacea clade)、六足亚门进化枝(Hexapoda clade)和螯肢亚门进化枝(Hexapoda clade). 比较eIF4G基因树(图5-a)和物种树(图5-b)后,发现甲壳动物eIF4G基因树与物种树的拓扑结构略有差异. 在物种树中,鳃足纲的大型溞(D.magna)和颚足纲的鹅颈藤壶(P.pollicipes),与其余甲壳动物聚成一个大的进化枝;而在基因树中,大型溞和鹅颈藤壶,与六足亚门昆虫纲的两个物种聚为一个进化枝;其余的进化枝(例如软甲纲、螯肢亚门)的拓扑结构完全一致.

a:基于eIF4G基因密码子序列构建的最大似然树;b:从TimeTree下载的节肢动物的物种树.图5 节肢动物和外群eIF4G基因的系统发育树Fig.5 Phylogenetic tree of eIF4G genes from arthropods and outgroup species
2.4.2 特异性位点检测
对中华绒螯蟹EseIF4G蛋白的特异性位点检测结果如图6所示,位点97、457为特异性位点,分别位于MIF4G结构域和MA3结构域. 位点97处的氨基酸由谷氨酰胺(Gln,Q)变为精氨酸(Arg,R),位点457处的氨基酸由赖氨酸(Lys,K)变为精氨酸(Arg,R).

图中特异性位点用红色标记.图6 中华绒螯蟹EseIF4G基因的特异性位点Fig.6 Segregating sites of EseIF4G gene in Eriocheir sinensis
2.4.3 选择压力分析
为研究中华绒螯蟹EseIF4G基因是否受到正选择作用,首先假设系统发生树所有分支有同一个ω值,one-ratio模型结果显示ω值为0.035 5,显著小于1,表明该基因整体上受纯化选择作用(表3). Free-ratio模型假定系统发生树上每一支和节点都具有独立的ω值,发现free-ratio模型显著优于one-ratio模型,提示不同支系具有不同的进化速率. 在分支模型的two-ratio中,将中华绒螯蟹的末端分支设置为前景支,其余分支设为背景支,two-ratio模型拟合效果并没有明显优于one-ratio模型(P=0.440),表明中华绒螯蟹EseIF4G基因与其他近缘甲壳动物有相似的进化速率(表3). 其次,使用位点模型对EseIF4G基因检测,M8模型明显优于M8a模型(P<0.01),但是没有检测出后验概率大于0.95的正选择位点(表3). 最后,使用分支位点模型,将中华绒螯蟹设置为前景支,发现Ma模型和Ma0模型之间没有显著差异(P>0.05).

表3 基于分支模型、位点模型和分支位点模型对EseIF4G基因的选择压力分析Tablele 3 Selective pressure analysis of EseIF4G gene based on branch model,site model and branch-site model
在Datamonkey网站上选用SLAC、FEL和FUBAR 3种方法检测,设置SLAC和FEL显著水平为0.2,FUBAR后验概率为0.8. 分析结果显示,FEL检测出3个正选择位点(448、512和655),FUBAR检测出 2个正选择位点(448和655),而SLAC没有检测出正选择位点(表4). 将PAML和Datamonkey 2种方法检测的结果整合起来,EseIF4G基因检测出2个正选择位点(448和655),标注在eIF4G蛋白三维结构中,如图2所示.

表4 PAML和Datamonkey鉴定EseIF4G基因的正选择位点Table 4 Positively selected sites of EseIF4G gene by PAML and Datamonkey
eIF4G是eIF4F复合体的重要组成部分,在翻译过程中可作为组装mRNA、核糖体亚基和帽结合蛋白的骨架蛋白. 本研究克隆了中华绒螯蟹EseIF4G基因的全长cDNA序列. 系统发育分析发现,EseIF4G蛋白序列具有高度保守的结构域,其在无脊椎动物和脊椎动物的整个物种中高度保守.
中华绒螯蟹胚胎与幼体发育包括受精卵、5个溞状幼体期、1个大眼幼体期和3个仔蟹期,此后幼蟹经过多次蜕皮发育为成蟹. 在溞状幼体Ⅴ期至仔蟹Ⅰ期,中华绒螯蟹幼体形态发生显著变化,即“短尾化”过程. 短尾化之前,蟹类幼体呈类似虾的体型;短尾化过程中,形态结构变化过程分为两个截然相反的过程:分化和整合. 一些部位高度分化(尤其是附肢)或融合在一起(尤其是腹神经节);某些部位的功能得到加强,例如胸部;某些部位的功能减弱了,例如腹部[18]. 短尾化过程完成后,蟹的显著形态变化减少,但体型随蜕壳而不断变大. 本实验中,在中华绒螯蟹幼体发育的10个阶段均检测到EseIF4G基因表达,表明EseIF4G基因可能在中华绒螯蟹的幼体发育过程中起调节作用. 其次,随着幼蟹的形态变化,EseIF4G基因的表达量也随之波动,在溞状幼体Ⅴ期(Z5)突然下降,然后在大眼幼体期(M)显著升高,这个显著变化暗示EseIF4G基因可能在中华绒螯蟹的短尾化过程中起调节作用. 此外,中华绒螯蟹幼体经历生活环境由咸淡水转为淡水的过程:从受精卵到大眼幼体期前期生活在江河口或沿海水域等咸淡水中,而后在大眼幼体后期则迁移到淡水中[21,40]. 中华绒螯蟹EseIF4G基因表达变化的趋势与其生活史对应,说明EseIF4G基因在中华绒螯蟹适应生活环境的渗透压调节方面有一定作用. 已有研究表明,中华绒螯蟹EjsHSP90、EjsANK和EjsPCNA基因在仔蟹Ⅰ期腹部组织的表达量均高于在大眼幼体期腹部组织[19-20],EsScr和EsAntp基因在溞状幼体Ⅴ期的表达量最低且显著高于在大眼幼体期[21]. 上述基因的表达模式的变化与EseIF4G基因高度相似,验证了EseIF4G基因随着幼蟹的形态变化和生活环境的迁移,可能参与中华绒螯蟹幼蟹的早期发育,特别是短尾化过程.
筛查中华绒螯蟹EseIF4G蛋白的特异性位点,发现位点97、457为特异性位点,分别位于MIF4G结构域和MA3结构域. eIF4G的中间结构域称为MIF4G结构域,已被证明介导多种蛋白质-蛋白质相互作用,例如与翻译起始因子eIF4A和eIF3、RNA的结合. 同时,除了eIF4F复合体外,其他含MIF4G结构域的蛋白质还参与了mRNA成熟、监视、翻译和降解等信号通路[41]. eIF4G的C端结构域也被称为MA3结构域,具有调控功能,可与ATP依赖性RNA解旋酶eIF4A结合以促进翻译起始;程序性细胞死亡4蛋白(PDCD4)也具有两个MA3结构域,与eIF4G-MA3竞争结合eIF4A,从而抑制蛋白质的合成[42]. 因此推测这两个特异位点可能与中华绒螯蟹的幼体发育阶段的mRNA组装、翻译、降解相关,但需要进一步的实验验证.
基于one-ratio模型的PAML选择压力分析显示ω值为0.035 5,显著小于1,表明该基因整体上受纯化选择作用;free-ratio与one-ratio比较显示不同的节肢动物谱系具有不同的进化速率. 进一步,将中华绒螯蟹的末端分支设为前景支的分支模型和分支位点模型,以及整体水平上的位点模型都没有检测到正选择信号,一定程度上说明中华绒螯蟹EseIF4G基因进化保守,纯化选择在该基因进化过程中起关键作用,与其他节肢动物具有相同或类似的功能. 但在选定的节肢动物中,使用Datamonkey的FEL和FUBAR方法,共同鉴定到两个正选择位点(448和655),分别位于MA3和W2结构域. W2结构域是eIF4G蛋白羧基末端小却保守的结构域,因包含两个保守的色氨酸得名;充当潜在的翻译调节剂参与到eIF2B、eIF5与eIF2亚基,eIF4G与蛋白激酶MNK1的相互作用[43]. 结果表明,这些位点在节肢动物eIF4G蛋白进化中起重要作用,调控mRNA翻译过程和蛋白质相互作用,可能参与各种节肢动物外部形态、生理活动等的差异调控. 未来可通过定向诱变等实验探明EseIF4G基因在中华绒螯蟹幼体发育阶段发挥的作用,为深入理解中华绒螯蟹短尾化的遗传学基础提供证据.
3 结论
本研究首次克隆了中华绒螯蟹EseIF4G基因全长cDNA序列,其编码蛋白具有MIF4G、MA3和 W2/eIF5C 3个保守结构域,在进化上与甲壳动物最近,并且在无脊椎动物长期进化过程中高度保守.EseIF4G基因在中华绒螯蟹幼体发育各个阶段广泛表达,在溞状幼体Ⅴ期表达量最低,随后在大眼幼体期表达量显著升高,可能参与调控中华绒螯蟹的短尾化和渗透压适应过程. 同时,检测到eIF4G蛋白两个中华绒螯蟹特异性位点和两个节肢动物潜在正选择位点,这些可能是调控mRNA翻译和蛋白质互作的位点,参与节肢动物生长、发育等过程. 本研究为进一步探索甲壳动物幼体短尾化调控机制提供了基础资料.

