石油污染土壤富集前后细菌群落组成和共现网络分析
余天飞,柳晓东,艾加敏,王佳敏,郭一丹,刘鑫慧,姜影影,邓振山
石油污染土壤富集前后细菌群落组成和共现网络分析
余天飞,柳晓东,艾加敏,王佳敏,郭一丹,刘鑫慧,姜影影,邓振山*
(延安大学生命科学学院,陕西 延安 716000)
为了探究石油污染土壤中细菌群落在富集过程中的演替规律,试验采用平板划线法、菌落PCR和高通量测序技术,分析了富集前后细菌群落结构、共现网络和核心菌属组成,并对富集后体系中的微生物进行分离鉴定,筛选石油降解菌.研究表明富集体系中可培养微生物隶属于34个属53个种,其中3个为潜在新种微生物,OS33和OS62-1具有降解石油的能力.高通量测序结果显示,在门分类水平上,富集前后丰度较高的菌门均为Proteobacteria和Actinobacteria,富集后两菌门的丰度可达到97.98%,占据绝对优势;丰度较高菌属由和转变为Unspecified和,标志微生物转变为与石油降解有关的.细菌群落共现网络在富集后,网络结构进一步简化且更加稳定,核心微生物转变为与石油降解有关的、、、、,且非石油降解菌P35可协同石油降解菌OS33降解石油.
石油污染;富集培养;共现网络;迪茨氏菌;土壤;高通量测序;群落结构
石油工业目前正面临石油污染物修复和提高石油采收效率两个主要问题.由于石油所含成分复杂,处理难度较大,因此石油已被列入优先控制的污染物[1].陆地和水生生态系统中的石油污染,主要应用微生物修复技术进行处理,其中微生物的降解作用,被认为在石油污染物消散过程中发挥重要作用[2].细菌是石油污染物降解最主要、最活跃的类群[3],因此,获得高效率石油降解菌,是应用生物处理石油污染的基础.
在自然环境土壤中,具有降解碳氢化合物能力的微生物仅占微生物群落的0.01%,而在石油污染环境中这一数值可以增加到1%~10%[4].虽然石油污染环境中的石油降解微生物丰度有所增加,但是直接通过石油污染土壤进行石油降解菌的筛选仍然较为低效.富集培养在功能微生物筛选过程中发挥着重要作用,通过富集培养,已经筛选到诸多石油降解微生物,如、、、、、、、、、和[5].研究环境中微生物群落组成的传统方法主要为:平板划线分离法、变性梯度凝胶电泳法(DGGE)和基因芯片等,但是这些方法难以精确显示微生物的群落组成,而高通量测序技术则能够高分辨率揭示微生物群落多样性和优势群体[6].Huang等[7]基于高通量测序技术,对比分析土壤在发生石油污染前后细菌群落组成时发现,石油污染后Proteobacteria和Actinobacteria微生物丰度显著升高,其中丰度较高的属为:、、、和,核心菌属转变为与石油降解相关的、、、和,与多环芳烃代谢相关的基因丰度也有所增加.
单一微生物在土壤环境中往往由于各种因素限制,难以高效降解石油,因此构建石油降解菌群逐渐被大家所关注.Chen等[8]利用sp. YC-X 2、sp. YC-X 7、sp. YC-X 4三株石油降解菌构建了一个菌群,7d内实现了49.22%的降解效率.Zhang等[9]将和(真菌)按照1:1的比例进行复配,在pH值为9.0的条件下,7d石油降解效率可达80%,均高于单一菌株的降解效率.目前,所构建的菌群也都集中在石油降解菌之间的简单复配.然而石油污染环境中的微生物类群绝大多数为非石油降解菌,有研究表明,这些非石油降解菌可产生表面活性剂,增加石油的溶解度,从而提高石油降解菌降解效率[10];Hu等[11]研究发现非石油降解菌产生的次生代谢产物也可以提高石油降解菌相关酶的活性,从而提高降解效率.然而在构建石油降解菌群时,对石油降解菌和辅助菌的选择上缺少相关理论指导,导致所构建菌群作用效果不稳定.
富集培养作为分离石油降解菌的中心环节之一,在高浓度石油和营养物质的刺激下,微生物群落会进行相应演替,可最大程度模拟自然环境下对石油降解菌株的选择,并且可为构建菌群提供理论指导.基于此,本研究以石油为唯一碳源,对石油污染土壤中的微生物进行富集培养,采用平板划线法和菌落PCR法,研究了富集后体系中可培养微生物群落组成,并从中筛选出石油降解菌.采用16S扩增子高通量测序技术,揭示了富集前后细菌群落的多样性,构建了细菌群落共现网络,分析了富集前后核心菌属组成.以高通量测序结果为指导,测试了菌株石油降解效率.并使用GC-MS对单独培养和共同培养后残余原油组分进行分析.本研究将为构建石油降解菌群提供菌种资源,为微生物修复石油污染提供理论参考.
1 材料与方法
1.1 样品采集
于2020年7月在陕西省榆林市靖边县延长石油公司油井采集原油和石油污染土壤(108°34′58″E,37°4'28''N,半干旱大陆性季风气候),油样中烷烃含量59.04%(/),芳香烃含量13.81%(/),沥青质含量20.93%(/),蜡质含量2.54%(/).石油污染土壤取自距离井口2m,深度为20~30cm处的土壤,石油含量为1328.78mg/kg,样品在4℃下使用无菌采样袋运回实验室,-80℃保存.
1.2 菌株的分离、鉴定
1.2.1 富集和分离 将石油污染土壤按1%(/)比例添加到ERM培养基中,在28℃,180r/min条件下培养15d后,将富集培养液分成两部分,一部分用于高通量测序,一部分用于分析可培养微生物群落组成.分析可培养微生物群落组成主要步骤为:①取一定量富集培养液进行梯度稀释,然后将30μL稀释液分别涂布在蛋白胨酵母粉葡萄糖培养基(Peptone Yeast Glucose broth,PYG)、R2A培养基(R2A Agar Medium,R2A)、完全培养基(Complete Medium,CM)3种培养基上,28℃,培养3d.每个梯度设置3个重复;②为避免挑取大量相同种微生物,选择培养基中菌落分布较为均匀的平板,根据菌落形态、颜色、生长时间等差异,挑取培养基上不同的单菌落,转接到相应的培养基中培养.
用于富集培养的ERM培养基配方为(g/L):原油10.0,(NH4)4SO41.0,NaNO32.0,KH2PO45.0,MgSO4·7H2O 0.3,NaCl 5.0,pH值7.5,121℃灭菌15min.
用于分离纯化的PYG、R2A和CM培养基的配方分别为:PYG培养基(g/L):蛋白胨 5.0,酵母粉 0.2,葡萄糖 5.0,牛肉膏 3.0,NaCl 0.5,MgSO4·7H2O 1.5,pH值7.5,121℃灭菌15min.
R2A培养基(g/L):酵母粉 0.5,酸水解酪蛋白胨 0.5,月示蛋白胨0.5,葡萄糖0.5,淀粉0.5,K2HPO4·3H2O 0.3,MgSO4·7H2O 0.024,丙酮酸钠0.3,pH值 7.5,121℃灭菌15min.
CM培养基(g/L):NaCl 80.0,酪氨酸 7.5,蛋白胨 5.0,酵母粉 1.0,柠檬酸钠 3.0,MgSO4·7H2O 20.0,KH2PO40.5,pH值7.5,121℃灭菌15min.
1.2.2 菌株16S rRNA基因扩增及测序 采用菌落PCR法对所分离的菌株进行鉴定,首先使用无菌牙签挑取已纯化菌株的单菌落,加入装有5.0μL溶液Ⅱ(0.2mol/L NaOH,1%SDS)的PCR管中,95℃裂解5min,待裂解完成后,加入95μL的ddH2O,颠倒混匀,以此作为扩增16S rRNA基因模板,PCR引物为27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R (5'-GGTTACCTTGTTACGACTT-3'),扩增体系为2×Taq Master Mix(Dye plus) 25.0μL,27F和1492R各2.0μL,模板DNA 2μL,ddH2O 19μL,反应条件为:预变性94℃,5min;变性94℃,30S;退火54℃,30S;延伸72℃,30S,终延伸72℃,3min;循环数:30.扩增结果用1.0%琼脂糖凝胶,90V水平电泳30min检查扩增结果,PCR产物送至擎科生物科技公司(西安)进行测序,测序结果在Ezbiocloud数据库(https://www. ezbiocloud.net/identify)进行16S rRNA序列比对,使用MEGA-X软件中N-J法构建菌株系统发育进化树[12].
1.2.3 石油降解菌的筛选 2,6-二氯靛酚(DCPIP)是一种人工合成电子受体,氧化态为蓝色,还原态为无色.微生物在生长过程中产生还原性物质,能够将氧化态DCPIP还原为还原态的DCPIP,从而使培养基从蓝色变为无色,因此可用来评估菌株在以石油为唯一碳源的培养基中生长情况[13].具体而言,将所分离的菌株接种到PYG培养基中,28℃,180r/min,过夜培养,然后在4℃,12000r/min条件下,离心15min,收集菌体,用ERM(无石油)培养基调节菌体密度至OD600=1.0.最后在48孔板中加入ERM培养基(无石油)800μL、DCPIP溶液(37.5μg/mL)100μL、菌体悬液80μL、灭菌石油5μL,28℃,180r/min,培养7d后,观察培养基颜色,无色表示菌株具有石油降解能力,蓝色表示菌株没有石油降解能力.
1.3 石油污染土壤富集前后高通量测序
1.3.1 测序样本预处理 取富集后的培养液100.0mL,4℃、12000r/min离心30min,收集沉淀,以富集时所加入的石油污染土壤为对照,测序样本使用干冰运送至深圳微科盟生物科技公司进行16S扩增子测序.
1.3.2 样本16S扩增子高通量测序 测序样本基因组DNA提取、16S基因扩增和测序均委托深圳微科盟生物科技有限公司完成.16S扩增区域为V3~V4,扩增引物为338F(5'-ACTCCTACGGGAG- GCAGCAG-3')和806R(5'-GGACTACHVGGGTW- TCTAAT-3'),采用IIIumina NovaSeq平台进行双末端测序.
1.3.3 测序数据处理 高通量测序数据经过barcode拆分后获得有效序列,使用Qiime 2软件中的DADA 2插件对所有样品的原始序列进行质控、去噪、拼接和去除嵌合体,以99%核酸相似性聚类,形成操作分类单元(OTU),选取OTU代表序列,与Greengenes Database 13_8数据库进行比对获得物种注释信息[14].采用LEFSe分析法(LDA>4.0),对样品特征微生物进行标记[15].
1.4 富集前后微生物共现网络构建
基于样本各OTU之间Spearman系数构建共现网络.首先将OTU丰度表中出现率小于2/3的OTU剔除;然后使用R语言中“psych”程序包,采用“Spearman法”计算OTU之间相关性系数,如果OTU之间的值大于0.6且值小于0.05,则认为两者之间存在显著相关,反之则不相关.在值矩阵中记为“0”;随后使用Gephi 0.9.2软件进行网络可视化,网络中圆点代表一个OUT,节点大小按OTU的丰度进行设置,以门分类水平进行节点着色,并统计各节点之间的平均度;最后使用Cytoscape 3.9.0软件中MCODE插件,对网络进行拓扑计算[7,16].
1.5 室内摇瓶试验
以高通量测序结果为指导,将所筛选的石油降解菌和非石油降解菌的菌体密度调节至OD600= 1.00,在20.0mL的ERM中接种2.0μL菌体悬液,28℃,180r/min培养7d,使用红外测油仪(北京华夏科创,OiL-480)测量石油含量,残余石油组分使用GC-MS (Agilent,5975C)进行分析.接种方式分为3种.即为:单独培养和共同培养.以上试验设置3个平行对照.
1.6 数据分析
采用Origin 2020软件进行数据整理及绘图,采用SPSS 22.0软件进行显著性和单因素方差分析(<0.05).
2 结果与分析
2.1 富集体系中微生物分离鉴定及石油降解菌筛选
石油污染土壤中微生物经过富集后,从中共分离纯化得到173株细菌,依据16S rRNA序列结果分为53个种,其中PYG培养基共分离得到32种微生物,R2A培养基共分离得到22种微生物,CM培养基共分离得到19种微生物.在MEGA-X软件中采用N-J法构建菌株系统发育进化树(图1).这些菌株分布于Actinobacteria,Proteobacteria,Firmicutes和Deiococcus-Thermus 4个菌门,隶属于34个属.其中隶属于Firmicutes主要为,大多数由CM培养基分离得到.Proteobacteria中主要为、、和;隶属于Actinobacteria种类最多,主要为、和.其中编号为E-05、OS52和OS49-1-1菌株与M3、NSG39和DSM9035的16S rRNA序列相似性分别为97.57%、97.71%和97.54%,可初步认定这3株菌为潜在的3个新种.原油降解测试中,接种OS33、OS62-1的培养基颜色由蓝色变为无色,可初步认为OS33、OS62-1具有石油降解能力.

图1 富集体系中可培养微生物群落结构
红色圆圈表示该菌株从相应的培养基分离得到
2.2 富集前后细菌群落多样性
2.2.1 富集前后细菌群落α多样性分析 采用高通量测序技术,对富集前后体系中细菌群落进行16S扩增子测序.扩增区域为:V3~V4.质控后得到的有效序列数为562157,以99%核酸相似性形成操作分类单元(OTU),去除嵌合体后,得到OTU代表序列数目为5184.从稀释曲线来看,Shannon多样性指数曲线趋于平坦,说明测序数据量足够,可反映样本中绝大多数微生物信息.从反映微生物群落α多样性指数:chao1,faith_ PD,shannon,simpson指数来看,富集后体系多样性指数均小于富集前的体系(图2).以上结果说明石油污染土壤中细菌群落经过富集后,微生物的群落多样性会降低.
2.2.2 富集前后细菌群落组成 如图3A所示,所有样品共检测到24个菌门,其中5个菌门为共有菌门,分别为:Actinobacteria、Proteobacteria、Firmicutes、Bacteroidetes和Chloroflexi. Actinobacteria和Proteobacteria为富集后体系中丰度最高菌门,两者共占总丰度的97.98%,分别占52.25%和45.73%,比富集前体系中分别高7.05%和19.25%.其余菌门在富集前体系中有着更高的丰度,如Firmicutes和Chloroflexi.此外在富集前体系中检测到的18个菌门(Acidobacteria、TM7、Gemmatimonadetes、Deferribacteres、Cyanobacteria、Verrucomicrobia、Tenericutes、Nitrospirae、Planctomycetes、Armatimonadetes、Crenarchaeota、Fibrobacteres、Chlorobi、MVP_21、OP11、BRC1、FBP、OD1)在富集后的体系中未被检测到,而Euryarchaeota只在富集后的体系中被检测到.以上结果表明,石油污染土壤的细菌群落经过富集培养后,在门水平上发生了显著变化.
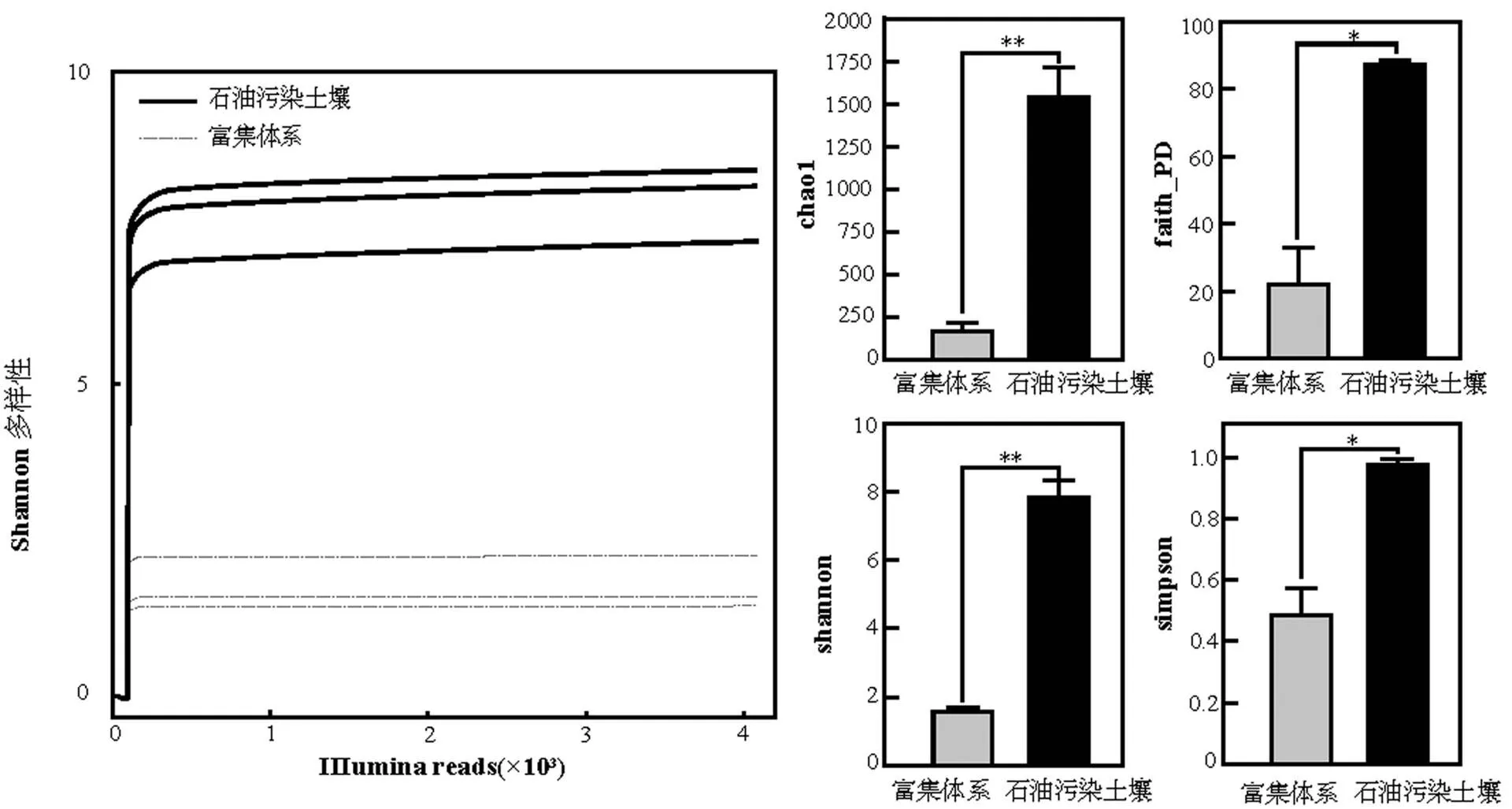
图2 富集前后细菌群落α多样性
*、**和***表示差异显著(检验,<0.05,<0.01和<0.001)
在属水平的群落组成如图3B所示,富集后体系的主要群落为:、Unspecified_、、和,而富集前体系中的主要微生物类群为:、、和.富集后体系中丰度排名前30的菌属里,有12个属的细菌类群被分离得到,如:、、、.其中8个属隶属于Actinobacteria,11个菌属隶属于Proteobacteria,这一结果与采用平板划线法研究富集体系中微生物群落组成的结果一致.
为了进一步研究富集体系中的特有细菌类群,采用LEFSe(LDA阈值>4.0)对样品中特有类群进行分析.结果显示,共有38个细菌类群存在显著差异,有33个细菌类群在富集前的体系中存在显著差异(图3C).然而,在富集后的体系中,仅有5个细菌类群存在显著差异,如:Dietziaceae和Idiomarianceae.
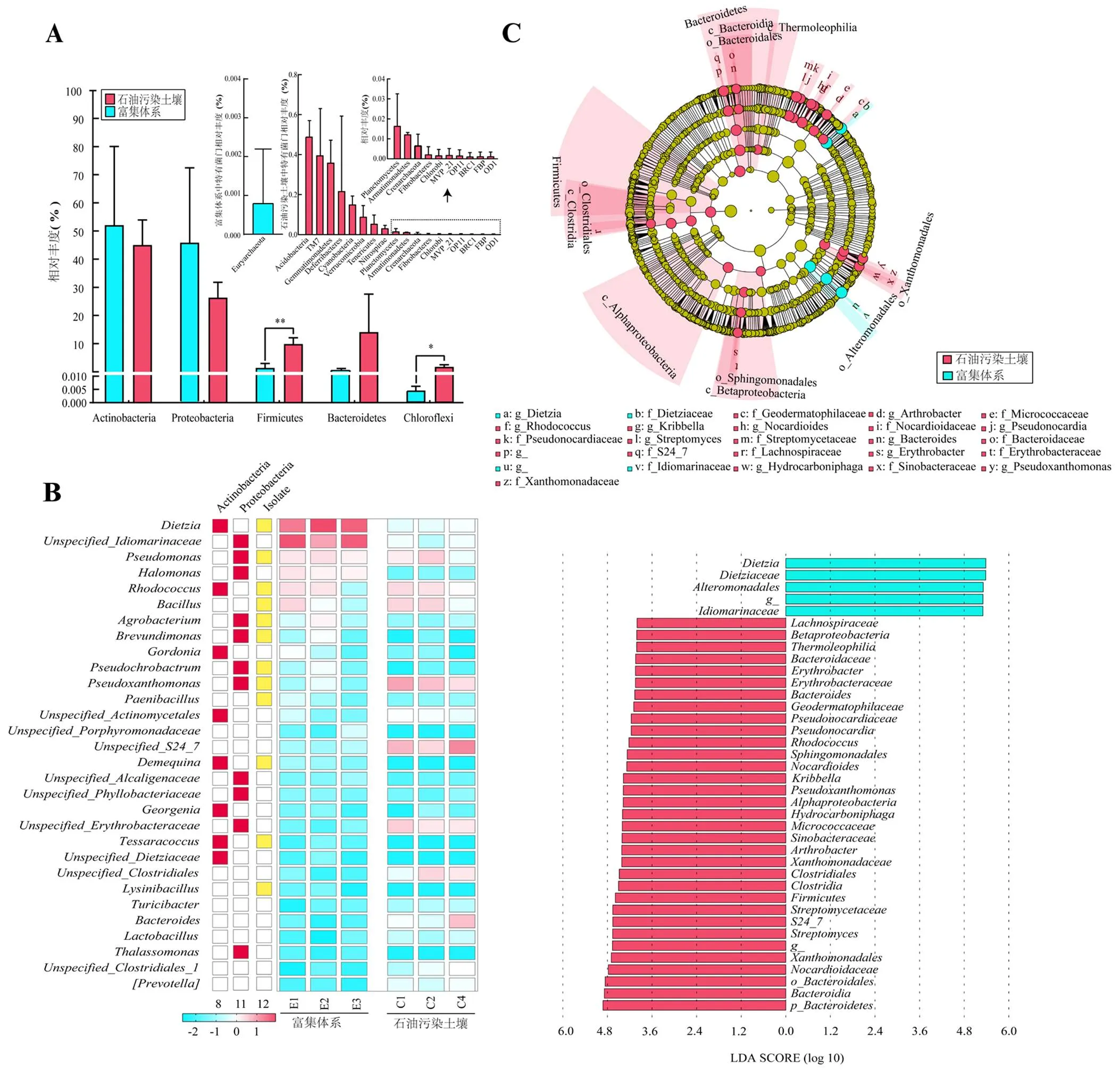
图3 富集前后细菌群落组成
(A)为在门分类水平上,富集前后样品中细菌群落相对丰度.(B)为在属分类水平上,相对丰度为前30的细菌群落相对丰度热图.红色矩形表示该菌属隶属于变形菌门或放线菌门.黄色矩形表明该菌属的微生物已被分离到.(C)为微生物类群LEfSe分析(LDA阈值:4.0 )
2.3 富集前后细菌群落共现网络转变
富集前后细菌群落共现网络如图4A、B所示,富集后体系共现网络的连通性低于富集前,网络更加简单.为进一步评估共现网络的稳定性,对网络中各节点平均度进行统计(图4C),富集后体系共现网络的平均度高于富集前体系(富集后为3.273,富集前为2.477).对网络进一步拓扑分析,如图4D、E所示,在富集后体系中,有18个属节点和38个环节被解析出来,低于富集前(17个属节点和44个环节).以上结果表明,石油污染土壤中的细菌群落经过富集培养后,微生物网络进一步简化,细菌群落之间的联系更加紧密,网络结构更加稳定.
富集后体系中相关性最高的5个核心菌属分别为:、、、、,均为丰度前30的菌属;富集前的体系中相关性最高的8个核心菌属分别为:、、、、、、、,只有、是丰度前30的菌属,其余6个菌属虽然在样本中丰度较低,但其对维持共现网络的稳定性发挥重要作用.核心菌属作为整个共现网络的基石,如果没有这些菌属,整个微生物共现网络也会随之崩溃[17-18].

图4 富集前后共现网络分析
(A)和(B)分别为富集后和石油污染土壤中微生物共现网络,节点大小与微生物相对丰度成正比,节点着色以门分类水平进行.(C)为共现网络各节点平均度统计.(D)和(E)分别为富集后和石油污染土壤中微生物共现网络k核分解.节点的颜色表现为核心度的分布,靠近中心的节点具有较高的核心度
2.4 室内摇瓶试验
根据富集后核心菌属组成,选择OS33为石油降解菌,P35为辅助菌,经过7d降解测试,原油含量如图5A所示,单独接种P35和OS33石油含量分别为(8506.26±253.43)mg/L和(2887.08±253.43)mg/L,共同培养的石油含量为(1435.79±322.43)mg/L.残余原油组分如图5B所示,OS33可降解中长链烷烃,在P35协同下,可进一步增加对中长链烷烃的利用,以上结果说明非石油降解菌P35可协同石油降解菌OS33降解石油.

图5 培养基中原油含量及组分
菌株处理之间不同字母表示其之间具有显著性差异(<0.05)
3 讨论
从石油污染的环境中分离石油降解菌,是目前获得石油降解菌的主要方法之一.由于缺乏具有特异性互补底物的微生物,故利用几种石油降解菌构建的菌群其降解效率往往会受到限制[19].本研究以石油为唯一碳源,对石油污染土壤中的微生物进行富集培养,从中分离得到34个属53个种的微生物,并且得到3个潜在新种微生物.结合高通量测序结果,丰度为前30的菌属仅分离得到13个属,然而丰度较高的未分离得到,相关研究表明,为中度嗜盐微生物[20-21],常与生物降解有关[22],并且可降解石油组分中的单环芳烃[23].针对高通量测序结果中丰度较高,但未分离得到的可培养微生物,应继续了解该菌属的生长特性,选择更加适合该菌属生长的培养基,进一步丰富可培养微生物的多样性.
采用高通量测序技术研究了石油污染土壤富集前后细菌群落组成,结果表明细菌群落在富集前后Proteobacteria和Actinobacteria中的微生物为主要类群,但在富集后两菌门的丰度达到97.89%,占据绝对优势,并且隶属于Actinobacteria中的为富集后体系中标志微生物,这与Huang等[7]采用高通量测序技术研究石油对土壤中细菌群落影响的结果一致.Proteobacteria作为类群最大、种类最多的微生物类群,在原油污染的陆地和沿海湿地中广泛分布[24],其中Gammaproteobacteria的微生物类群与石油烃的降解有关[25],如和,此外Actinobacteria中、也是经常报道的石油降解菌属[26].
构建了富集前后细菌群落共现网络,并对核心网络进行解析,富集前体系中有3个核心菌属与石油降解相关,分别为:[27-28]、[29]和[30-31],其中微生物在降解石油方面有较好的效果,已被广泛报道.富集后体系中的核心菌属[32-33]和[34]为石油降解菌属,其中产鼠李糖脂类表面活性剂已被广泛报道,而生物表面活性剂可增加石油在水中的溶解度,可协同石油降解菌降解石油[10].常应用在重金属修复方面[35],在对石油污染土壤的理化性质研究发现,土壤发生石油污染往往也伴随着重金属污染和盐碱化[36].以上结果进一步说明,在高浓度石油和营养物质的作用下,石油污染土壤中的微生物群落得以简化,微生物朝着与石油降解有关的类群转变.
作为富集体系中丰度最高的菌属,却未出现在最核心的菌属中,而等丰度较低的菌属却成为核心菌属,的很多微生物能够降解链状烷烃、单环芳烃、多环芳烃和蜡质[3,37],室内摇瓶试验发现非石油降解菌P35能够协同石油降解菌OS33降解石油,因此下一步将采用响应面分析法优化菌株的复配比例,进行室内模拟修复试验,评价菌群稳定性,应用于实际场地.
4 结论
4.1 石油污染土壤中细菌群落经过富集培养后,可培养微生物隶属于34个属53个种,其中有3个为潜在新种微生物,OS33OS62- 1为主要石油降解菌.
4.2 石油污染土壤中细菌群落经过富集培养后,丰度较高的优势菌属由、、和转变为、Unspecified_和,并且共现网络更加简单,结构更加稳定,核心菌属转变为与石油降解相关、、、、.
4.3 非石油降解菌P35可协同石油降解菌OS33降解石油.
[1] Varjani S J,Upasani V N. Carbon spectrum utilization by an indigenous strain ofNCIM 5514: Production,characterization and surface active properties of biosurfactant [J]. Bioresource Technology,2016,221:510-516.
[2] Truskewycz A,Gundry T D,Khudur L S,et al. Petroleum hydrocarbon contamination in terrestrial ecosystems-fate and microbial responses [J]. Molecules,2019,24(18).
[3] Meckenstock R U,Boll M,Mouttaki H,et al. Anaerobic degradation of benzene and polycyclic aromatic hydrocarbons [J]. Journal of Molecular Microbiology and Biotechnology,2016,26(1-3):92-118.
[4] Aislabie J,Saul D J,Foght J M. Bioremediation of hydrocarbon- contaminated polar soils [J]. Extremophiles,2006,10(3):171-179.
[5] Varjani S J. Microbial degradation of petroleum hydrocarbons [J]. Bioresource Technology,2017,223:277-286.
[6] Wang L,Huang X,Zheng T L. Responses of bacterial and archaeal communities to nitrate stimulation after oil pollution in mangrove sediment revealed by Illumina sequencing [J]. Marine Pollution Bulletin,2016,109(1):281-289.
[7] Huang L,Ye J,Jiang K,et al. Oil contamination drives the transformation of soil microbial communities: Co-occurrence pattern,metabolic enzymes and culturable hydrocarbon-degrading bacteria [J]. Ecotoxicology and Environmental Safety,2021,225:112740.
[8] Chen J,Yang Q,Huang T,et al. Enhanced bioremediation of soil contaminated with viscous oil through microbial consortium construction and ultraviolet mutation [J]. World Journal of Microbiology and Biotechnology,2011,27(6):1381-1389.
[9] Zhang X,Kong D,Liu X,et al. Combined microbial degradation of crude oil under alkaline conditions byandsp [J]. Chemosphere,2021,273:129666.
[10] Ghorbani M,Hosseini M,Najafpour G,et al. Synthesis and characterization of rhamnolipid biosurfactant produced byPTCC 1340 for emulsification of oil sludge in oil storage tank [J]. Arabian Journal for Science and Engineering,2022,47:216-219.
[11] Hu B,Wang M,Geng S,et al. Metabolic exchange with non-alkane- consumingSLG510A3-8improves n-alkane biodegradation by the alkane degradersp. strain DQ12-45-1b [J]. Applied and Environmental Microbiology,2020,86(8):e02931-19.
[12] Deng Z,Jiang Y,Chen K,et al. One biosurfactant-producing bacteriasp. A-8and its potential use in microbial enhanced oil recovery and bioremediation [J]. Frontiers in Microbiology,2020,11(247):1-8.
[13] Guerra A B,Oliveira J S,Silva-Portela R C B,et al. Metagenome enrichment approach used for selection of oil-degrading bacteria consortia for drill cutting residue bioremediation [J]. Environmental Pollution,2018,235:869-880.
[14] Desantis T Z,Hugenholtz P,Larsen N,et al. Greengenes,a chimera- checked 16S rRNA gene database and workbench compatible with ARB [J]. Applied and Environmental Microbiology,2006,72(7):5069-5072.
[15] Langille M G,Zaneveld J,Caporaso J G,et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences [J]. Nature Biotechnology,2013,31(9):814-821.
[16] 李 东,肖 娴,孙 波,等.水热增加下黑土细菌群落共生网络特征 [J]. 微生物学报,2021,61(6):1715-1727.
Li D,Xiao X,Sun B,et al. Co-occurrence network of bacterial communities in mollisol soils under increasing hydrothermal conditions [J]. Acta Microbiologica Sinica,2021,61(6):1715-1727.
[17] Zhao Y,Duan F A,Cui Z,et al. Insights into the vertical distribution of the microbiota in steel plant soils with potentially toxic elements and PAHs contamination after 60years operation: Abundance,structure,co-occurrence network and functionality [J]. Science of the Total Environment,2021,786:147338.
[18] Banerjee S,Schlaeppi K,Van Der Heijden M G A. Keystone taxa as drivers of microbiome structure and functioning [J]. Nature Reviews Microbiology,2018,16(9):567-576.
[19] Souza E C,Vessoni-Penna T C,De Souza Oliveira R P. Biosurfactant-enhanced hydrocarbon bioremediation: An overview [J]. International Biodeterioration & Biodegradation,2014,89:88-94.
[20] Pandiyan K,Kushwaha P,Bagul S Y,et al.sp. nov.,a moderately halophilic bacterium isolated from beach soil in India [J]. International Journal of Systematic and Evolutionary Microbiology,2021,71(1):1-9.
[21] Ramezani M,Pourmohyadini M,Nikou M M,et al.sp. nov.,a novel halophilic bacterium isolated from a hypersaline wetland [J]. International Journal of Systematic and Evolutionary Microbiology,2020,70(12):6098-6105.
[22] Guadie A,Gessesse A,Xia S.sp. strain A55,a novel dye decolorizing bacterium from dye-uncontaminated Rift Valley Soda lake [J]. Chemosphere,2018,206:59-69.
[23] Widdel F,Rabus R. Anaerobic biodegradation of saturated and aromatic hydrocarbons [J]. Current Opinion in Biotechnology,2001,12(3):259-276.
[24] King G M,Kostka J E,Hazen T C,et al. Microbial responses to the Deepwater Horizon oil spill: from coastal wetlands to the deep sea [J]. Annual Review of Marine Science,2015,7:377-401.
[25] Zhou Z,Liu Y,Pan J,et al. Gammaproteobacteria mediating utilization of methyl-,sulfur- and petroleum organic compounds in deep ocean hydrothermal plumes [J]. The ISME Journal,2020,14(12):3136-3148.
[26] Wang X B,Chi C Q,Nie Y,et al. Degradation of petroleum hydrocarbons (C6-C40) and crude oil by a novelstrain [J]. Bioresource Technology,2011,102(17):7755-7761.
[27] Wang Y,Nie M,Diwu Z,et al. Toxicity evaluation of the metabolites derived from the degradation of phenanthrene by one of a soil ubiquitous PAHs-degrading strainFF [J]. Journal of Hazardous Materials,2021,415:125657.
[28] Bhatia S K,Gurav R,Choi Y K,et al.sp. YHY01a microbial cell factory for the valorization of waste cooking oil into lipids a feedstock for biodiesel production [J]. Fuel,2021,301:121070.
[29] Kumar A,Chandra R. Biodegradation and toxicity reduction of pulp paper mill wastewater by isolated laccase producingAKRC03 [J]. Cleaner Engineering and Technology,2021,4:100193.
[30] Yadav S,Kim J S,Lee S S. Alkanindiges hydrocarboniclasticus sp. nov. Isolated from crude oil contaminated sands and emended description of the genus[J]. Current Microbiology,2021,78(1):378-382.
[31] Bogan B W,Sullivan W R,Kayser K J,et al.gen. nov.,sp. nov.,an obligately hydrocarbonoclastic,aerobic squalane-degrading bacterium isolated from oilfield soils [J]. International Journal of Systematic and Evolutionary Microbiology,2003,53(Pt 5):1389-1395.
[32] Miri S,Perez J A E,Brar S K,et al. Sustainable production and co-immobilization of cold-active enzymes fromsp. for BTEX biodegradation [J]. Environmental Pollution,2021,285:117678.
[33] Kim H W,Jo J H,Kim Y B,et al. Biodegradation of polystyrene by bacteria from the soil in common environments [J]. Journal of Hazardous Materials 2021,416:126239.
[34] Zhao L B,Wang X X,Li C,et al. Isolation and characterization of a phosphate-solubilizing bacteriumsp. PSB13from petroleum-contaminated drill cuttings [J]. Advanced Materials Research,2014,955-959:407-410.
[35] Zhu Y,He X,Xu J,et al. Insight into efficient removal of Cr(VI) by magnetite immobilized withsp. JLT12: Mechanism and performance [J]. Chemosphere,2021,262:127901.
[36] Fu X,Cui Z,Zang G. Migration,speciation and distribution of heavy metals in an oil-polluted soil affected by crude oil extraction processes [J]. Environmental Science-Processes & Impacts,2014,16(7):1737-1744.
[37] Hoskeri R S,Mulla S I,Shouche Y S,et al. Biodegradation of 4-chlorobenzoic acid byPA01NC [J]. Biodegradation,2011,22(3):509-516.
Bacterial community composition and co-occurrence network before and after enrichment of oil-contaminated soil.
YU Tian-fei,LIU Xiao-dong,AI Jia-min,WANG Jia-min,GUO Yi-dan,LIU Xin-hui,JIANG Ying-ying,DENG Zhen-shan*
(School of Life Sciences,Yan'an University,Yan'an 716000,China).,2022,42(8):3858~3866
Enrichment culture is one of the most important aspects of isolating petroleum degrading microorganisms,in which the microbial community undergoes corresponding succession. The bacterial community structure,co-occurrence network and core genus composition before and after the enrichment were conducted with the streak plate method,colony PCR and Illumina-MiSeq high-throughput sequencing technology to investigate the succession pattern of bacterial communities in oil-contaminated soil during the enrichment process. The microorganisms in the enriched system were isolated and identified. We observed that the culturable microorganisms in the enriched system belong to 34 genera and 53 species,among which 3 are of potential new species,andOS33 andOS62-1are able to degrade oil. High-throughput sequencing results show that Proteobacteria and Actinobacteria are the main phylum,whenever it is before or after enrichment. But after enrichment,these two phylums are of absolutely dominant groups and the abundance is up to 97.98%. At the genus level,the dominant genuses are transformed from,,andto,Unspecified_and,the core genera are altered tofor degrating petroleum. The co-occurrence networks of bacterial communities are more simplified and stable after enrichment. The core microorganisms are altered to,,,andwhich are related to petroleum degradation. The non-petroleum degrading bacteriaP35can cooperate with the oil degrading bacteriaOS33to degrade petroleum.
petroleum pollution;enrichment cultivation;co-occurrence networks;;soil;high-throughput sequencing;community structure
X53,Q93
A
1000-6923(2022)08-3858-09
2022-01-04
陕西省教育厅专项科研计划项目(21JK0992);延安市科技专项经费(2019-27,203010105);陕西省教育厅服务地方专项计划项目(16JF029);延安大学博士科研启动项目(YDBK2019-43);延安大学2021年研究生教育创新计划项目(YCX2021080)
* 责任作者,教授,zhenshandeng214@163.com
余天飞(1998-),男,陕西安康人,延安大学硕士研究生,主要从事资源与环境微生物学研究.

