CT影像组学模型及深度学习技术预测肺腺癌EGFR突变
黄栎有,徐璐,温林春,王延花,李智勇
肺腺癌是肺癌中发病率最高的组织学类型[1]。随着分子靶向药物研究的不断发展,近年来酪氨酸激酶抑制剂(tyrosine kinase inhibitors,TKIs)如吉非替尼等药物已广泛用于肺腺癌的临床治疗,患者获得了比化疗更长的无进展生存期[2]。表皮生长因子受体(epidermal growth factor receptor,EGFR)基因表型决定了患者是否能够接受TKIs治疗[3]。如何快速有效的检测EGFR基因突变就变得格外重要。目前临床上主要通过内窥镜及穿刺活检来检测患者EGFR基因突变。但活检仅能采集局部少量肿瘤组织,无法反映肿瘤整体的异质性,容易出现因采集部位不佳或采集样本过少而无法正确检测出突变结果的情况。此外,活检术对患者身体条件及肿瘤大小、位置等均有较高要求,而且高昂的检查费用也限制了其在临床上的普及。CT扫描作为临床上常用的影像学检查技术,为分析肿瘤异质性提供了一种非入侵式的方法。已有研究表明,肺癌CT图像中的纹理特征与基因表达具有相关性,能够预测EGFR基因突变[4-5]。而深度学习技术通过强大的端对端学习能力,已经在多种疾病的诊断中展现出远超传统分类模型的诊断能力[6-8]。因此,本研究中对比分析了传统影像组学模型和深度学习模型对肺腺癌患者EGFR突变的预测效能,旨在探寻基于CT扫描图像预测肺腺癌EGFR基因突变的最佳方法。
材料与方法
1.研究对象
回顾性搜集2018年1月-2021年5月在本院确诊为肺腺癌的228例患者的CT影像资料。其中,EGFR突变型 116例,野生型112例。纳入标准:①经病理证实为原发肺腺癌;②具有EGFR基因检测结果;③CT检查前未接受任何治疗。排除标准:①肿瘤直径小于3.0 mm;②CT图像质量不佳;③合并其它恶性肿瘤。
2.检查方法
使用Siemens Somatom Definition AS(101例)或Philips Brilliance 64 CT机(127例)行胸部CT扫描。扫描参数:120 kV,210 mA,螺距0.9,扫描层厚1~5 mm。分别采用B40f和iDose标准重建算法进行图像重建,重建层厚1~2 mm。
3.影像组学分析方法
图像分割及特征提取:使用MRIcroGL软件对CT影像进行重采样,重采样后图像的像素大小为1 mm×1 mm×1 mm。由2位具有7年以上CT诊断经验的放射科医师分别在肺窗(窗宽1500 HU,窗位-600 HU)图像上采用单盲法各自独立在所有肿瘤层面进行ROI的勾画,并将获得的病灶容积ROI(VOI)保存为掩模数据(图1)。分别采用一阶统计、灰度共生矩阵、灰度游程矩阵和小波变换等方法对VOI进行特征提取,共得到395个影像组学特征。采用组内相关系数(intraclass correlation coefficient,ICC)对两位医师提取的特征进行一致性检验,以ICC值大于0.7表示一致性较好。

图1 男,53岁,左肺腺癌。a)横轴面、矢状面和冠状面CT图像显示左肺内病灶3个方位的特征;b)沿病灶边缘逐层手动勾画ROI后,横轴面、矢状面和冠状面CT图像显示ROI(红色区域)的三方向观;c)ROI掩模图像的三方向观,白色ROI像素值为1,其余像素值为0。
影像组学建模:对纹理特征进行归一化和标准化后,删除与其它纹理特征相关系数大于0.9的特征。按照7:3的比例将所有病灶随机划分为训练集和验证集。采用最小绝对收缩和选择算子(least absolute shrinkage and selection operator,LASSO)和5折交叉验证对训练集数据进行特征筛选,并通过对所筛选特征及其权重系数的线性组合来构建每例患者的影像组学评分(Radscore)。
使用Wilcoxon检验分别对训练集和验证集中EGFR突变型组和野生型组的Radscore进行比较,以P<0.05为差异具有统计学意义。绘制验证集中Radscore的受试者工作特征曲线(receiver operating characteristic curve,ROC),计算曲线下面积(AUC)。
机器学习模型的建立和验证:分别建立随机森林(random forest,RF)和支持向量机(support vector machine,SVM)机器学习模型。在训练集中采用5折交叉验证法分别对2种机器学习模型进行训练,并计算它们在验证集中的AUC。
4.深度学习分析方法
数据预处理:通过对CT图像像素矩阵与对应ROI掩模数据进行矩阵乘法,使ROI体素外像素值为0,保证深度学习分析区域与影像组学ROI一致。对ROI进行裁剪,去除周围全零区域得到最终图像数据。
数据增广:深度学习训练集和验证集的划分与影像组学训练集和验证集划分完全一致。由于样本量较小,我们将训练集中每个病灶的所有层面图像均作为输入样本,共计将2873帧图像输入到2D残差网络(ResNet)深度学习网络中进行训练,并通过对图片进行随机20度旋转和左右、上下镜像进一步扩充训练集数据至理论上限459680帧。在验证集中选取每个病灶直径最大层面的图像作为验证集数据,共68帧图像。
ResNet和迁移学习:将每帧图像的大小重置为224×224×3像素并输入ResNet网络。迁移学习技术能将某个网络中学习到的知识迁移到另一个具有共通性的网络中。本研究利用迁移学习技术将Image-Net上训练好的预训练模型参数作为模型初始参数迁移到ResNet网络中。ImageNet是拥有128万帧自然图像的大型数据库,官方(https://download.pytorch.org/models)提供了基于该数据库的ResNet预训练模型。通过迁移学习ImageNet上的模型参数能够间接扩大训练数据,从而提高神经网络的性能。
深度学习模型的建立和验证:选择交叉熵作为模型的损失函数,对训练集数据进行循环训练,以交叉熵损失最小的循环作为最佳深度学习模型。计算此深度学习模型在验证集中的AUC,并比较其与其它3种影像学组模型的AUC的差异。
5.统计学处理
采用pytorch1.04深度学习库搭建深度学习网络,并利用visdom模块绘制损失(loss)曲线。采用R语言(Version 3.5.1)对患者资料进行统计学分析。对患者年龄进行正态性检验,符合正态分布,故其数据资料采用均值±标准差来表示,采用两独立样本t检验进行组间比较。采用皮尔逊χ2检验比较2组间性别构成的差异。以P<0.05为差异有统计学意义。采用corrplot软件包进行相关性特征删除,采用glmnet软件包进行LASSO数据降维,利用pROC软件包绘制模型或参数的AUC,并采用Delong检验比较各模型间AUC的差异。
结 果
1.人口学资料的比较
EGFR突变组患者年龄为39~80岁,平均(60.55±8.65),野生组患者年龄为42~83岁,平均(62.59±9.13)岁,组间差异无统计学意义(t=1.729,P=0.085)。突变组中男47例、女69例,野生组中男67例、女45例;两组间性别构成的差异具有统计学意义(χ2=6.377,P=0.012)。
2.影像组学分析
一致性分析结果显示,在两位医师各自提取的肺癌病灶的395个影像组学特征中有306个特征的观察者间一致性较好(ICC>0.7),其中参与建模特征的ICC值见表1。

表1 参与建模的纹理参数的一致性检验结果
自306个一致性良好的组学特征中删除相关系数大于0.9的特征,剩余122个特征。经过5折交叉验证得到LASSO回归中参数λ值与模型均方误差的关系图(图2a)。选择λ值为LASSO回归均方误差最小值加一倍标准差时(0.071)作为最佳LASSO回归参数,最终从剩余的122个纹理特征中筛选出9个纹理特征(图2b)。得到的Radscore计算方程:Radscore=0.02375800-LLH_DifferenceVariance×0.03231060+LLH_InverseVariance×0.11277837-LHH_Autocorrelation×0.25185757-HLL_Autocorrelation×0.04808175+HHH_Correlation×0.06753695-sigma_2mm_ClusterProminence×0.10374467-sigma_3mm_ClusterTendency×0.09305346+sigma_5mm_ClusterShade×0.09416770-square_ClusterTendency×0.19429391。在训练集和测试集中样本的Radscore数据分布差异如图3所示,训练集和验证集中EGFR突变组的Radscore均显著高于未突变组(训练集:Z=-6.771,P<0.001;验证集:Z=-3.793,P<0.001)。
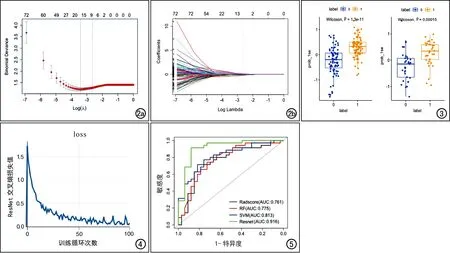
图2 LASSO模型参数图。 a)5折交叉验证得到参数λ值,第1条虚线为LASSO均方误差最小值对应的λ值,第二条虚线为最小误差1倍标准差对应的λ值; b) 纹理特征的LASSO系数曲线,在均方误差最小值1倍标准差处绘制虚线得到9个纹理特征。 图3 Rad-score预测EGFR突变箱式散点图。Label 0代表野生型,label 1代表突变型。 图4 ResNet 损失函数随epoch的变化曲线,显示损失值在前10个epoch迅速下降,在50个epoch后趋于稳定。 图5 四种模型在验证集中的ROC曲线图,以Resnet的AUC最大。
基于筛选出的9个纹理特征,在训练集中训练得到RF和SVM 2种机器学习模型。RF模型中参数设置:决策树数量500,二叉数2;SVM模型参数:惩罚因子C值=1,sigma=0.143。
Radscore、RF和SVM三种模型在训练集上和验证集中对EGFR突变的诊断效能指标值见表2。在测试集中3种模型的AUC值均超过0.7,表现出良好的诊断效能。在验证集中以SVM模型的AUC最大,但与RF和Radscore模型的差异无统计学意义(Z=-0.975,P=0.330;Z=1.700,P=0.089)。

表2 四种模型ROC分析结果
3.深度学习模型的效能
训练集中输入数据的batch size=32,学习率为0.0001,循环次数为100 epoch。结果显示在前10个epoch交叉熵损失迅速下降,在50个epoch后趋于稳定(图4),第73个epoch时交叉熵损失最低(0.12),得到最佳模型。
在验证集中,深度学习方法获得的RseNet模型的AUC、敏感度和特异度均高于另外3种影像组学模型(表2、图5),ResNet与Radscore、RF模型间AUC的差异具有统计学意义(Z=-2.178,P=0.031;Z=-2.051,P=0.043),与SVM模型间AUC的差异无统计学意义(Z=-1.627,P=0.106)。
讨 论
本研究建立并比较了多种基于CT图像影像组学特征建立的预测模型,评估其预测肺腺癌患者EGFR突变的能力。建立的Radscore模型(一种线性模型)证实了通过分析CT图像能够预测肺腺癌患者EGFR基因突变。通过对线性模型、机器学习模型以及深度学习模型进行比较,来筛选预测EGFR突变的最佳模型算法。本研究结果显示,深度学习模型的预测表现明显优于其它预测模型,表明基于CT影像组学分析能够预测EGFR突变,深度学习技术可以有效提高模型的预测准确性。
相对于传统人工阅片,影像组学通过对图像进行定量分析,能够利用不同分析方法从图像中提取多维度影像组学特征。其中一些特征可以反映肉眼难以观察到的肿瘤内部异质性信息[9]。影像组学已被应用于疾病的诊断以及疗效和预后的预测等[10-12]。近年来,一些研究尝试建立影像组学与相应基因的联系。Tu等[13]基于CT图像提取的93个纹理特征构建肺癌EGFR突变预测模型,在训练集和验证集中的AUC分别为0.762和0.775;吴琦等[14]从396个纹理特征中筛选出11个最佳特征构建logistic回归模型来预测肺腺癌EGFR基因突变状态,训练集ROC曲线的AUC为0.83 ;罗燕等[15]联合影像组学特征和吸烟状态建立logistic回归模型,预测肺腺癌EGFR突变的AUC为0.673。本研究中Radscore模型预测肺腺癌EGFR突变的AUC为0.761,与上述研究结果相符。
既往数研究大多未考虑模型算法的影响,仅通过简单线性模型对EGFR突变进行预测,诊断结果并不尽如人意。本研究中利用机器学习算法建立多种模型并比较其诊断效能,旨在提高影像组学方法预测EGFR基因突变的准确性,寻找最佳模型算法。本研究中基于提取的CT影像组学特征建立了Radscore、RF和SVM三种模型,在验证集中的AUC分别为0.761、0.775和0.813,以SVM模型的诊断效能最高。虽然SVM模型与Radscore和RF模型间的差异无统计学意义,但我们认为该问题通过进一步扩大样本将得到解决。SVM算法通过搜索对空间最佳划分的超平面进行特征分类,对数据维度和样本数量的依赖性较小。已有研究发现相较于传统回归模型,SVM算法能够有效提升模型的预测性能,如彭涛等[16]研究结果表明基于MRI影像组学特征建立的SVM模型对前列腺癌的诊断效能优于线性回归模型;李琳等[17]研究结果表明相较于logistic回归模型,SVM模型可以更好地预测肝癌患者的预后。本研究结果与上述研究结果类似。
深度学习作为人工智能领域的一项重要技术,可以通过神经网络自动对输入图像进卷积,并利用梯度下降算法完成模型学习,最终实现高精度预测。数据处理过程中仅需使用者定义肿瘤ROI,而无需进行特征提取、特征选择和模型构建等传统影像组学的复杂程序。已有将深度学习技术应用于医学图像的诊断中,并得到了媲美诊断医师级别的准确性[18]。本研究中,利用深度学习方法构建的ResNet网络在验证集中的AUC、敏感度、和特异度分别为0.916、0.879和0.914,均高于另外3种通过传统方法建立的影像组学模型。本研究结果提示,在预测肺腺癌EGFR突变方面,深度学习技术较传统方法构建的预测模型更具优势。
本研究的局限性:①为单中心研究且样本量相对较小,研究结果可能存在偏倚;②人工勾画ROI,人为因素可能对特征提取造成误差;③采用的模型算法有限,需进一步扩大算法种类。
综上所述,基于CT图像的影像组学模型能够较好的预测肺腺癌EGFR突变,其中以深度学习模型的诊断效能更优,为EGFR检测提供了一种有效的无创性检测技术。

