ZBED6基因敲除猪脾脏转录组测序数据分析
赵海东,陈平博,韦燕佩,邬明丽,易晓华,杜敏杰,魏泽辉*,孙秀柱*
(1.西北农林科技大学 动物科技学院,陕西 杨凌 712100;2.成都中科奥格生物科技有限公司,四川 成都 610000)
基因编辑技术因其能够定向改造动物基因组相关信息的强大功能受到研究者的追捧,其在建立疾病模型、培育新型转基因动物品种和治疗人类疾病等尖端科研领域具有划时代的作用。随着相关研究的不断推进,基因编辑动物上存在的某些问题也逐渐引起了社会的关心与担忧,其中对基因编辑动物“早衰”和“抵抗力低”等问题尤为重视。随着基因编辑动物技术的日渐成熟和群体规模的不断扩大,对基因编辑动物环境适应能力的研究显得尤为重要,同时这也是基因编辑动物是否具有市场推广价值的重要评价环节。
锌指蛋白6(zinc finger BED-type containing 6,)基因是近年来新发现的一个重要的转录因子,该基因位于猪基因第一内含子区段,其调控模式倾向于与宿主基因共用调控区,本身仅含有一个外显子,在哺乳动物当中较为保守。同时,基因在哺乳动物中具有能够保守性调控IGF2(Insulin-like growth factor 2)的功能,通过结合内含子区段的GCTCG序列调节表达的效率,提升猪产肉量,以期能够创制一个高产肉猪新品种。潘登科通过CRISPER/Cas9技术获得了基因敲除猪群体,并对其在肌肉发育、血液生化和心脏发育等方面进行了相应的研究。但其环境适应能力和免疫机能是否健康、健全均未从分子层面加以明确。脾脏是哺乳动物集体重要的免疫器官,能够作为免疫球蛋白的成熟器官参与机体免疫功能。因此,本研究拟通过对-SKO和WT组基因编辑猪脾脏组织进行转录组测序,通过生物信息学方法对其分子层面的差异表达基因进行分析,获得基因编辑猪免疫功能是否正常的相关分子层面数据资料,以期为基因编辑动物安全性评价和环境适应能力评价提供科学依据。
1 材料与方法
1.1 主要试剂、仪器
Trizol购自Takara公司。主要仪器有PCR仪(伯乐),凝胶成像系统(伯乐),微量分光光度计(赛默飞)。
1.2 样品采集与敲除验证
试验用基因单等位基因敲除巴马小型猪()4份脾脏组织样本和半同胞野生型巴马小型猪()3份脾脏组织样本由成都中科奥格生物科技有限公司潘登科团队馈赠。基因敲除猪采用CRISPR/Cas9技术制备,在其外显子区产生1 bp 缺失使得基因发生移码突变达到对该基因的敲除,通过PCR扩增结合sanger测序验证敲除位点,使用正向引物为: 5'-GCTTTGTTAGCGTCTGATGCC-3',反向引物为:5'-AGGTTACAAATTGCCCGCCA-3'。
1.3 RNA提取、质检与cDNA文库构建
组织样品收集并保存于液氮中,采用Trizol法对7个脾脏样本进行RNA提取,使用NanoDrop 1000和凝胶电泳对RNA的质量进行检验,RNA样本检验合格后,送上海生工生物科技有限公司进行文库构建及测序。文库构建完成后对建成文库的质量进行检测,文库质量合格后,利用Illumina 1.9测序平台对7个样本进行双端150 bp高通量测序。
1.4 转录组测序数据处理与分析
1.4.1 质控与比对 对测序的原始数据通过FastQC进行质量评估,通过Trimmomatic进行质量剪切,得到相对准确的有效数据。使用HISAT2将样本有效数据比对到参考基因组上,统计Mapping信息。采用Qualimap根据比对结果进行均一性分布检查、基因组结构分布等分析。利用BEDTools进行基因覆盖率统计分析和染色体上测序序列分布等分析。
1.4.2 表达差异分析 使用StringTie和已知的基因模型对测序所得数据进行基因表达量的计算,对测序数据获得的表达数量,考虑到基因长度、测序深度和样本的不同,采用TPM (Transcripts Per Million)对基因表达量进行估算。针对-SKO 组和对照组脾脏测序数据,使用DESeq2进行基因表达差异分析获得表达具有显著差异的基因集(<0.05),采用Z-score对差异表达数据进行归一化,采用hclust对差异表达数据进行层级聚类,对获得的结果使用pheatmap进行可视化。获取组间基因表达FDR值小于0.05的基因集,通过模块表达分析对基因的表达模式进行鉴定。
1.4.3 差异表达基因的KOG分类、GO分类和 KEGG富集分析 基于对-SKO型和WT型猪脾脏组织以FDR<0.05位条件获得差异表达基因,利用topGO软件对获得的差异表达基因进行GO注释并进行可视化,对获得的差异表达基因使用clusterProfiler进行KEGG通路和KOG分类富集分析与可视化。
2 结果与分析
2.1 RNA-seq 原始测序数据质量控制
通过Sanger测序验证敲除位点正确,存在1 bp缺失造成基因移码(图1)。针对-SKO 组和对照组脾脏进行测序,每个样本测序获得6 Gb以上数据,并使用FastQC对样本的测序数据质量进行可视化评估,包括原始序列、过滤后序列、质控率和Q30等数据(表1)。测序数据的过滤后数据比率和Q30数据比率均超过90%,满足后续生物信息学分析需求。

图1 ZBED6基因敲除位点sanger测序验证
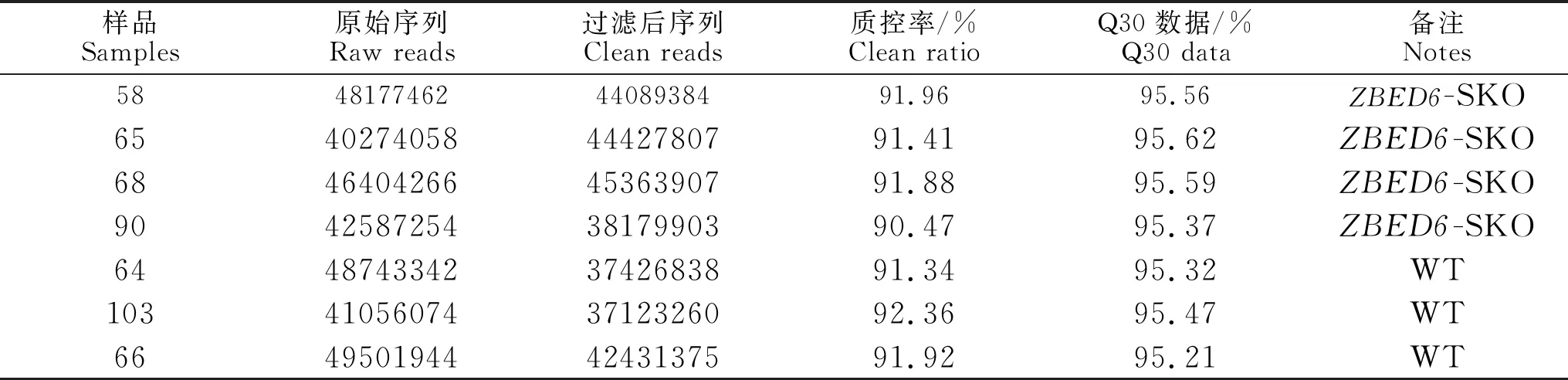
表1 测序数据质量控制
2.2 差异表达基因的筛选
以TPM为对象筛选-SKO组和WT组,以<0.05为条件获取显著差异表达基因集,共获得下调基因85个,上调基因62个,对获得的结果使用pheatmap进行可视化(图2)。通过FDR值小于0.05对差异表达基因进行校正,获得上调基因5个,下调基因4个,分别是Interleukin 4 receptor()、Dephospho-CoA kinase domain containing()、Family with sequence similarity 91 member A1()、Zinc finger protein 140()、ATPase H+ transporting V1 subunit D()、Endonuclease V()、Zinc finger protein333 ()、Procollagen-lysine,2-oxoglutarate 5-dioxygenase 2()、--211859(novel gene)(表2)。对差异表达基因进行模块表达分析分为4个模块,其中2个上调模块和2个下调模块(图3)。上调模块为Subcluster 2(和)和Subcluster 3(和),下调模块为Subcluster 1(和)和Subcluster 2(和)(表3)。

表2 Q检验差异表达基因

表3 差异表达基因模块信息

图2 差异基因表达量聚类热图
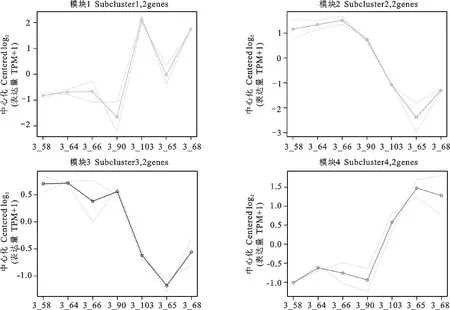
图3 差异基因各模块表达趋势折线图
2.3 差异表达基因的KOG分类、GO分类和KEGG富集分析
对获得的9个差异表达基因进行比对和功能注释。在GO分析体系中,对前20条GO富集进行可视化,发现3个基因注释于GO:0034641 : cellular nitrogen compou,2个基因注释于GO:0051186 : cofactor metabolic process,2个基因注释于GO:0006575 : cellular modified amino,但均未发现显著富集GO功能(图4)。在KEGG分析中,富集到20条通路,其中ssc00280:Valine, leucine and isoleucine degradation通路显著性为0.09,ssc04530:Tight junction通路显著性为0.16,未富集到显著差异代谢通路信息(图5)。通过对KOG富集分析发现Coenzyme transport and metabolism、General function prediction only、Function unknown和Posttranslational modification, protein turnover, chaperones被富集,但并未富集到显著KOG信息(图6)。

图4 差异表达基因 GO 功能分类图

图5 差异表达基因 KEGG功能分类图

图6 差异表达基因 KOG 功能分类图
3 讨 论
随着基因编辑动物育种群体和基因编辑动物实验模型群体的不断扩展,关于基因编辑动物安全评价的相关研究逐渐被重视。一方面,在基因编辑动物生产的长期实践当中,成功率极低,且基因编辑动物个体可能存在一定程度的异常风险;另一方面,基因编辑动物群体在长期饲养过程中,其繁殖能力、体况外貌和健康指标等均与野生型动物不存在明显差异。一定程度上,基因编辑动物在经过重编程的过程中有可能出现相当大比例的“重启失败”现象,但在成功获得健康基因编辑动物后,研究者对于其免疫机能和机体健康方面的研究则极少涉及。基因编辑操作对动物是否会产生潜在的免疫影响尚不得而知。
6基因作为一个近年来新发现的转录因子,是一个重要的瘦肉型家畜新品种创制靶标,众多研究者通过基因编辑手段对其功能机制进行阐明与验证。6基因的作用机制主要依赖于靶向结合2基因内含子区GCTCG序列产生对IGF2及其下游通路的调控作用,进而影响猪肌肉发育相关功能。一方面唐雨婷通过敲除6基因猪的模型研究发现,敲除6基因能够上调血液中IGF2的水平;另一方面Liu等通过编辑猪2基因上的6结合位点后发现其能够直接促进猪的肌肉发育水平。
脾脏是动物体重要的免疫器官,针对脾脏基因表达模式的研究将为基因编辑动物免疫功能是否较预期存在问题获得相对具有说服力的数据支持。据此,本研究收集6基因敲除猪脾脏样本,通过转录组测序手段,对其差异表达基因进行相关生物信息学分析,共发现9个差异表达基因(FDR<0.05),其中上调基因5个,下调基因4个,分别是4、、911、140、61、、333、2、--211859,在同等饲养条件下,6-SKO组较其半同胞或全同胞WT型对照组差异表达基因数量极为有限,并未发现能够显著影响脾脏免疫机能的差异表达基因。
针对差异表达基因进行分析。IL4R是一个重要的炎症因子受体,其表达量上调表明基因编辑猪在可能处于一定程度上的应激状态,但其并未产生其他相关因子的变化,这种变化仍需在后续大群体样本中进行检测和持续关注。同时IL4具有肌肉发生相关功能,其受体的上调可能与脾脏组织因IGF2上调导致的生长发育变化应答有关。基因是大脑相关过程的调节因子,与帕金森病的发生具有一定的相关性,并未有研究发现其与动物免疫机能之间的关系。911作为一个超甲基化基因,其表达量的异常可能与克隆猪在重编程过程中表观遗传产生的某种特殊变化有关,同时该基因功能与细胞增殖存在一定的相关性。140基因是锌指蛋白家族的转录因子,其功能与骨髓增生异常存在一定的相关关系,但其在本研究中6基因敲除猪脾脏中表达下调的机制尚不明确。61基因与细胞自噬存在一定的相关性,是真核细胞广泛存在的液泡型ATP酶的一个亚单位,对其抑制能够起到抑制肿瘤细胞生长、侵袭和转移的作用。Nakadera等发现61参与抑制mTOR可改善肝脂肪变性引起的自噬小泡酸化损伤。Wang等研究发现61参与TFEB介导的胰腺炎发生过程中的溶酶体自噬相关生物学功能。ENDOV是一种细胞内DNA损伤保护因子,它能够识别和切割含有次黄嘌呤的DNA,其切割位点为损伤碱基次黄嘌呤下游3′端的第二个磷酸二酯键,在嗜热细菌基因组稳定性中具有重要的作用,其在动物中可能具有较为相似的功能,该基因的下调可能与基因编辑动物DNA的稳定性具有一定的关系,也可能参与部分细胞癌变的过程,但仅仅这一个基因的表达变化并不足以导致基因编辑猪遗传稳定性上的障碍。333基因也是锌指蛋白家族的转录因子,具有ZFN家族保守性结构,尚未有文献报道其相关功能,因此异常表达在6基因敲除猪脾脏中的意义尚不得而知。2基因与纤维化疾病之间存在显著关系,其作为赖氨酸羟化酶2的编码基因,直接参与器官纤维化的形成。该基因广泛参与多种生物学功能,一定程度上该基因高表达组织具有一定的纤维化风险,但在本研究中6基因敲除猪脾脏中2基因低表达的意义尚不明确。--211859基因作为一个新基因,其功能未见报道。在对差异表达基因的GO聚类、KEGG聚类和KOG聚类分析中均未发现显著富集的生物学信息与功能,一定程度上表明在6基因敲除后,其脾脏代谢并未发生较大的变化,其免疫功能并未产生所担心的基因编辑动物“免疫力差”等问题,但不可否认,本研究仅对6这一个基因编辑猪群体进行检测,其功能在基因编辑动物的大概念上是否适用尚需长期和扩展观测。
4 结 论
通过转录组测序技术对6-SKO和WT猪脾脏进行鉴定,共发现9个差异表达基因(FDR<0.05),在GO分析、KEGG分析和KOG分析中均为发现明显的能够支持基因编辑猪免疫缺陷问题的相关证据。因此,6基因敲除猪在免疫功能上一定程度未发现明显的因基因编辑相关操作所造成的“免疫功能障碍”相关问题,为开展基因编辑动物育种提供相应的理论依据。

