10个马铃薯新育成品种的SSR分析
张海龙, 于 卓, 祁 娜, 于肖夏, 李景伟, 岳 东
(内蒙古农业大学农学院, 呼和浩特 010019)
SSR(Simple Sequence Repeat)标记呈共显性,具有多态性位点丰富、重复性高等优点[1],因其操作简单、成本低,在作物种质资源评价、分子标记辅助育种、遗传图谱构建和遗传关系分析等方面已得到广泛应用[2]。吴立萍等[3]用14对多态性高的SSR引物对54份马铃薯品种遗传关系进行分析,共扩增出多态性条带229个,从DNA分子水平上揭示了供试品种间的亲缘关系。本试验拟对10个马铃薯新品种的基因组DNA进行SSR分析,在分子水平上,明确10个马铃薯新品种的遗传差异性,为下一步作为马铃薯亲本进行杂交改良和新品种保护利用提供依据。
1 材料与方法
1.1 试验材料
供试材料为内农薯1号(NNS-1)、内农薯2号(NNS-2)、内农薯3号(NNS-3)、内农薯4号(NNS-4)、内农薯5号(NNS-5)、内农薯6号(NNS-6)、红彩5号(HC-5)、紫彩1号(ZC-1)、紫彩2号(ZC-2)和紫彩3号(ZC-3),均由内蒙古农业大学于卓教授带领的马铃薯育种团队新近育成品种(系)。
1.2 试验地概况
试验材料于2020年5月在内蒙古农业大学试验基地进行种植,该试验地位于111°45′ E~45°50′ N,海拔1 069 m,年降水量350~400 mm,无霜期约145 d。土质为沙壤质土,pH值在7.8~8.2之间,土壤肥力中等,具有灌溉条件。每种材料单行种植50株,株距25 cm,行距75 cm,播种深度约12 cm,种子级别为原种。
1.3 测定方法
1.3.1DNA提取与检测
在马铃薯苗期,随机取各材料顶端新鲜叶片,用北京天根公司生产的DNA试剂盒提取基因组DNA。取各材料DNA原液5 μL,用1.0%琼脂糖凝胶电泳对各材料进行纯度检测,将达到试验要求的DNA稀释至50 ng/μL,置于-40 ℃冰柜中留存备用。
1.3.2SSR引物来源及筛选
试验所用引物均从NCBI网站上公布的马铃薯SSR特异性引物中筛选获得,由上海生物工程有限公司负责合成。将各试验材料基因组DNA作为模板,对80对SSR适宜引物进行PCR扩增、电泳,从中选出多态性高、位点稳定、明亮的SSR引物10对(表1),用于后续SSR分析。

表1 筛选出的10对SSR适宜引物及序列Table 1 10 pairs of SSR-appropriate primers and sequences screened
1.3.3SSR-PCR扩增体系及程序
SSR-PCR扩增体系:体系总体积20 μL,含样品DNA(50 ng/μL)2.0 μL,1.4 μL dNTPs(2.5 mmol/L),10×PCR Buffer(Mg2+)2.1 μL,TaqDNA聚合酶(5 U/μL)0.2 μL,上游和下游引物各0.6 μL,ddH2O 13.1 μL。
扩增程序:在Bio-Rad Mycycler Thermal Cycler上进行扩增,程序为:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火45 s,72 ℃延伸90 s,循环5次;94 ℃变性30 s,54 ℃退火45 s,72 ℃延伸90 s,循环35次;72 ℃延伸10 min;4 ℃下终止反应。
1.3.4扩增产物检测
将5 μL变性剂加入PCR扩增产物中,在94 ℃下变性5 min,储存于4 ℃冰箱备用。每种材料吸取变性后的PCR扩增产物5 μL点样,用100 bp的DNA Marker作对照,电泳恒定功率70 W,电泳时间75 min。电泳结束后,将电泳胶板取下,于固定液内固定16 min;固定结束后用蒸馏水漂洗2~3 min,于2.0%的硝酸银溶液中染色15 min,再用蒸馏水速漂4~6 s;置显影液中进行显色,待DNA条带显色清晰为止。晾干胶板,观察记录条带位点和标记数目、照相[4-5]。
1.3.5数据处理
利用0/1赋值法[13]统计电泳胶板上扩增出的SSR多态性位点,用“1”表示条带清晰,“0”表示没有条带,“-”表示缺失的条带,最终生成“1”/“0”的原始二元数据矩阵,不统计没有多态性的位点,多态性条带位点百分率(P)计算公式为:
P(%)=(K/N)×100%
式中:K为多态性条带位点总数,N为条带位点总数[14]。用DPS(Data Processing System)软件中的类平均聚类法(UPGMA)计算各品种间的遗传距离,并对各品种进行聚类分析。
2 结果与分析
2.1 供试材料基因组DNA纯度检测
由图1可见,各材料基因组DNA电泳条带清晰、明亮、无拖尾和降解现象,表明各试验材料的DNA纯度高,完全达到SSR-PCR扩增实验的要求。
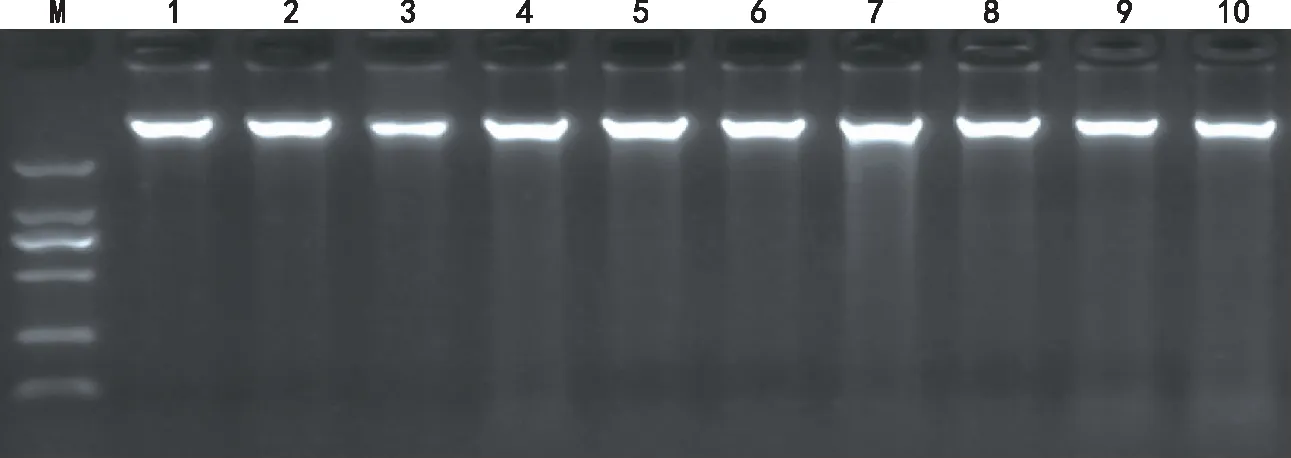
注:M为DL 2000;1为NNS-1;2为NNS-2;3为NNS-3;4为NNS-4;5为NNS-5;6为NNS-6;7为HC-5;8为ZC-1;9为ZC-2;10为ZC-3。下同。 图1 马铃薯新品种基因组DNA电泳检测结果Fig.1 Results of DNA electrophoresis testing for genome of new potato varieties
2.2 马铃薯新品种基因组DNA的SSR扩增
利用筛选出的10对SSR适宜引物对各试验材料基因组DNA进行PCR扩增、电泳并统计其多态性位点条带,结果(表2、图2和图3)表明,共有139个条带稳定、清晰的SSR标记位点,其中含有SSR多态性位点条带117个,多态性位点占总标记位点的84.17%,平均每对引物扩增出11.7个多态性条带位点。引物S 189和S 25能很好地将10个马铃薯新品种清晰区分开,并分别构建了SSR指纹图,为新品种的鉴定和登记利用提供了分子依据。
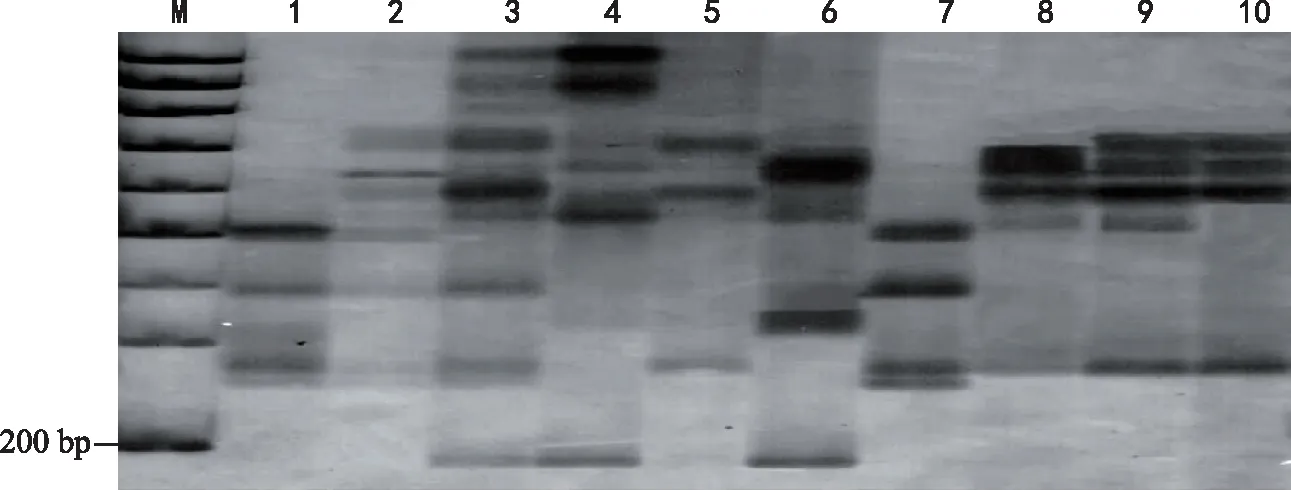
图2 引物S 189对10个马铃薯材料基因组DNA扩增的SSR指纹特征Fig.2 SSR fingerprint characteristics of genomic DNA amplification of 10 new varieties with primer S 189
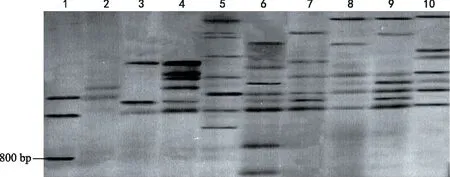
图3 引物S 25对10个马铃薯材料基因组DNA扩增的SSR指纹特征Fig.3 SSR fingerprint characteristics of genomic DNA amplification of 10 new varieties with primer S 25
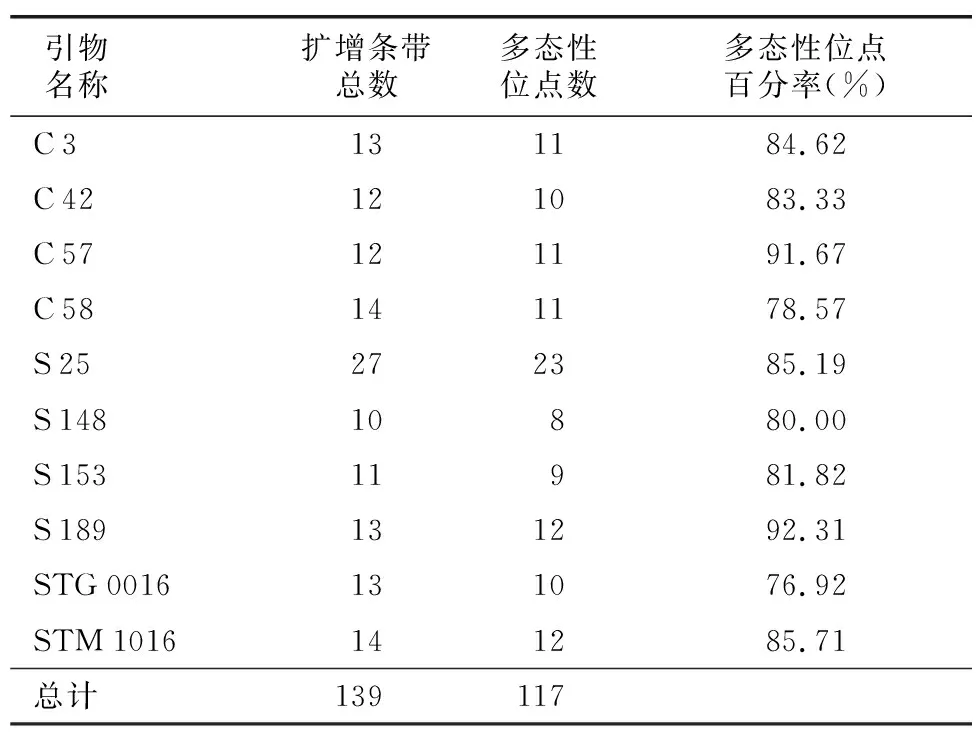
表2 各新品种的SSR扩增结果Table 2 SSR amplification results for each new variety
2.3 马铃薯新品种的遗传距离和聚类分析
由表3和图4可以看出,10个马铃薯新品种间的遗传距离(GD)变幅为0.126 4~0.714 3,平均遗传距离为0.555 4,材料NNS-4和ZC-1之间的遗传距离最大,为0.714 3,其亲缘关系较远;材料ZC-2和ZC-3之间的遗传距离最小,为0.126 4,说明这两个新品种间的亲缘关系较近。以GD值0.55为基准,将10个马铃薯品种分为四类:NNS-1、NNS-2和HC-5为第一类;NNS-3、NNS-4和NNS-5为第二类;ZC-1、ZC-2和ZC-3为第三类;NNS-6为第四类。

表3 各品种(材料)的遗传距离矩阵Table 3 Genetic distance matrix for each new varieties

图4 10个马铃薯材料的SSR聚类结果 Fig.4 SSR clustering results for 10 new potato varieties
3 讨论与结论
SSR分子标记也被称为微卫星DNA,即简单重复序列。SSR分子标记技术主要包含PCR扩增反应和聚丙烯酰胺凝胶电泳两个关键实验步骤,具有重复性好、操作简单,可在较短时间内获得大量的SSR多态性位点,明显提高了SSR引物的利用效率[2]。在SSR标记分析过程中,所选引物和供试材料是分子标记扩增位点多态性高低的关键因素,可明确显示出作物个体之间的遗传差异性,标记多态性越高,说明个体间的遗传差异越大[10]。近年来,SSR标记技术已广泛应用在作物的遗传多样性分析等研究中。李靓等[11]利用筛选出的10对SSR适宜引物,对7个马铃薯供试材料的基因组DNA进行扩增,经对试验结果统计分析,共获得多态性条带82个,多态性比率占78.10%;宋峥等[12]利用筛选的10对适宜性引物,对44份马铃薯种质材料进行PCR扩增,共获得108个SSR多态性位点,多态性比率88.5%。刘宇飞等[13]利用筛选出的10对SSR特异性引物,对5个马铃薯新品系基因组DNA进行PCR扩增,分析实验结果表明,其中SSR多态性位点有94个,多态性比率为72.23%。
本实验以杂交选育的10个马铃薯新品种内农薯1号、内农薯2号、内农薯3号、内农薯4号、内农薯5号、内农薯6号、红彩5号、紫彩1号、紫彩2号和紫彩3号的基因组DNA为材料,利用筛选出的10对SSR适宜引物对各供试材料基因组DNA进行扩增,经分析聚丙烯凝胶电泳数据显示,本实验共获取117个SSR多态性条带位点,多态性比率为84.17%,结果表明,在DNA分子水平上,10个马铃薯新品种之间存在显著差异,同时利用特异性引物S 189和S 25构建了2张能明确区分各供试材料的SSR指纹图。聚类分析显示,10个马铃薯新品种的GD值变幅为0.126 4~0.714 3,以GD值0.55为基准,将10个马铃薯试验材料划分为四类:第一类为NNS-1、NNS-2和HC-5;第二类为NNS-3、NNS-4和NNS-5;第三类为ZC-1、ZC-2和ZC-3; NNS-6为第四类。本研究再次证明SSR技术用于马铃薯品种间遗传差异分析是可行的。

