DNA条形码在海蜇食物组成鉴定中的应用
李玉龙,鲍相渤,高祥刚,段 妍,王 彬,董 婧,李云峰
(辽宁省海洋水产科学研究院,辽宁省分子生物学重点实验室,辽宁 大连 116023)
食性分析是生态学研究领域的重要内容之一,食性分析及食物网络的动态研究是探讨食物网结构特征和营养流动、评估物种生存状况和生态系统功能等热点问题的重要基础[1-2]。水母是一类海洋胶质类浮游动物,是海洋生态系统的重要组成部分,在海洋生态系统物质、能量传递过程中具有重要作用[3],有关水母类的摄食生态问题已成为新兴的研究热点[4-8]。在食性相关研究中,食性分析方法的准确性和精确性尤为重要,然而对水母这一海洋胶质类浮游动物而言,因其摄食范围广泛,食物组成包括桡足类,枝角类,一些无脊椎动物的浮游幼虫、鱼卵、仔稚鱼,甚至包括一些细菌、真菌、微藻和碎屑[7-12],食物在消化循环腔中被降解、消化后,通过传统镜检法很难简单判断其食物来源,使得传统镜检方法在解析水母食物组成方面存在着诸多局限性[6-8]。
为了对水母食物组成进行更快速、准确的鉴定,急需在传统镜检基础上建立并结合便捷准确的分子鉴定手段,DNA条形码提供了可信息化的分类标准和有效的分类学手段,已成为近年来物种分类与鉴定研究中重要的技术依托[13-18]。DNA条形码通常是指使用基因片段序列如线粒体COⅠ作为种类鉴定标签条码的技术[13-15],除了快速、准确,DNA条形码技术最大优点是无需待检测样品保存关键形态特征。随着DNA条形码技术的发展和物种数据库的完善,越来越多的学者开始尝试采用DNA条形码来解决一些诸如鱼类、头足类以及一些小型浮游动物等海洋动物的食性鉴定问题[16,18-22],说明DNA条形码在海洋动物食物种类组成鉴定中具有应用价值。
海蜇(Rhopilemaesculentum)属刺胞动物门钵水母纲根口水母目根口水母科海蜇属,主要分布于我国沿海近岸浅海海域,在日本西部、朝鲜半岛西部和俄罗斯远东地区也有少量分布,是食用价值与经济价值最高的一种大型食用水母,也是目前为数不多的得到系统性研究的大型水母种类[23-26]。迄今为止,关于海蜇食性方面的研究主要有基于镜检鉴定的消化腔内含物分析法[23,27]和稳定同位素分析法[6]对辽东湾自然海域分布的海蜇的食性进行的分析,尚未见利用分子生物学技术分析海蜇食性的相关报道。因此,笔者以辽东湾海域自然分布的海蜇为研究对象,利用通用引物同时扩增线粒体COⅠ基因片段和核基因ITS-5.8S rDNA这两种常用DNA条形码分子标记,比较两种分子标记作为海蜇食性鉴定条形码的适用性和潜力,并结合海蜇传统食性分析方法和稳定同位素分析方法的相关结果评估这两个DNA条形码用于检测海蜇食物组成的应用前景,以期为海蜇乃至水母的食性研究提供基础资料和技术支持。
1 材料与方法
1.1 样品采集及前处理
研究所用海蜇水母体样品共计30个,伞径40~570 mm,为2013年6—7月进行的辽东湾海蜇跟踪监测调查所得样品,调查站位见图1,随机取样。根据海蜇水母体以口腕和肩板上的吸口来摄食饵料生物的特性,采集样品时取口腕及肩板组织吸口分布丰富的区域以利于饵料生物的扩增,样品采集后于99%乙醇中保存备用。
1.2 DNA提取、扩增、测序及基因克隆
采用CTAB法提取基因组DNA,COⅠ序列以通用引物LCO-1490(5′-GGTCAACAAATCATAA
AGATATTGG-3′)和HCO-2198(5′-TAAACTTC AGGGTGACCAAAAAATCA-3′)[28]扩增,ITS-5.8S rDNA序列使用引物JF-18F1750(5′-AAAGTCGTA ACAAGGTTTCCG-3′)和JF-28R765(5′-TTGGTC CGTGTTTCAAGACG-3′)[29]进行扩增,PCR扩增均在Gene Amp PCR System 9700型PCR仪上进行,反应体系及反应条件见表1。
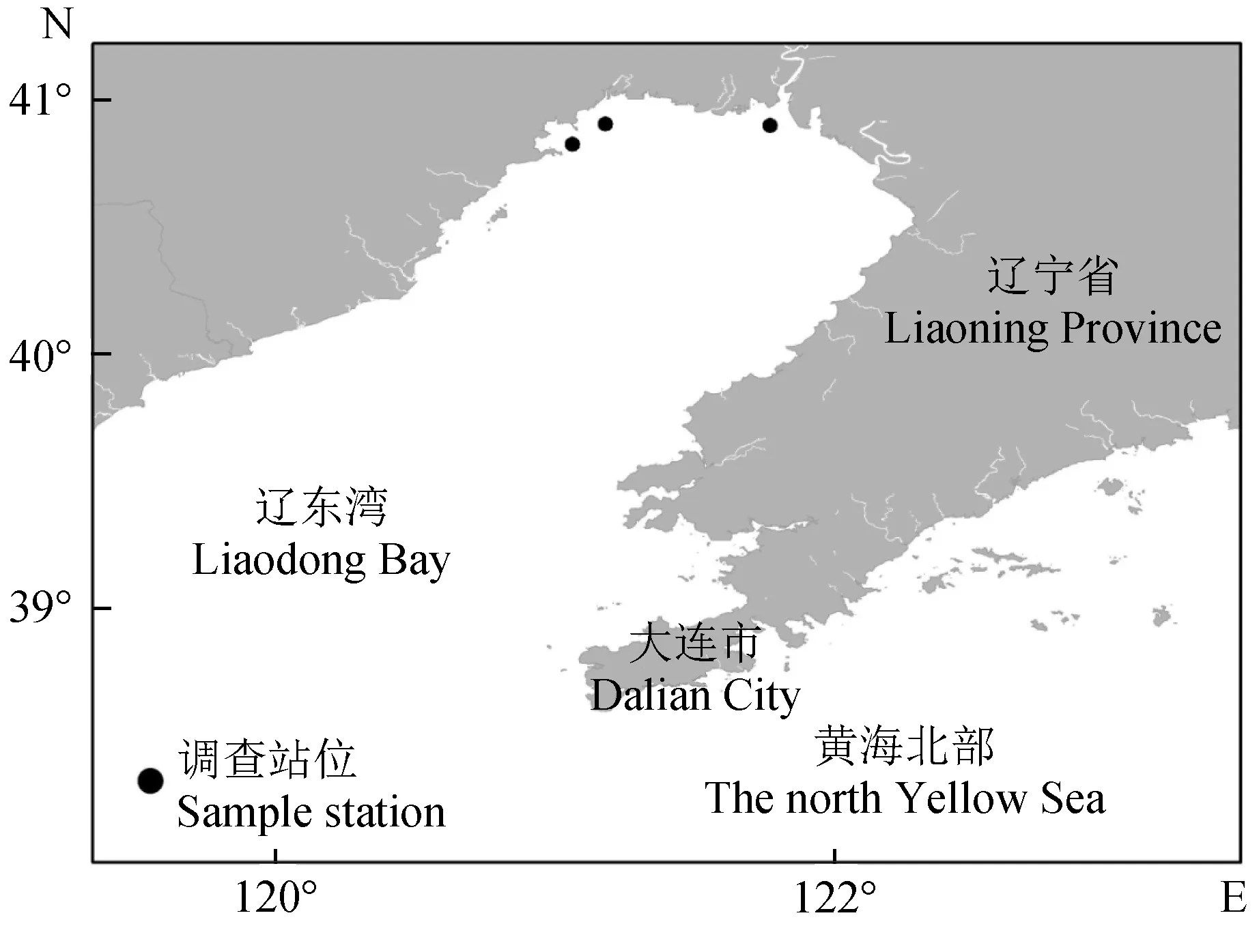
图1 辽东湾海蜇样品取样站位示意Fig.1 The sampling locations of jellyfish R. esculentum species

表1 COⅠ和ITS-5.8S rDNA序列PCR扩增信息
对每份样品的扩增产物使用1%琼脂糖凝胶电泳检测,纯化后直接进行双向测序进行分子物种鉴定,以剔除海蜇自身序列的样品。经分子物种鉴定检测后,选取测序双峰的样品经琼脂糖胶DNA纯化试剂盒Ver 2.0回收后与pMD18-T载体连接,采用蓝白斑筛选阳性克隆,经PCR 检测确认无误后每个样品随机挑取若干个单克隆进行测序,用于海蜇食性组成分析 (上海英潍捷基有限公司)。
1.3 数据分析
将克隆测序得到的COⅠ和ITS-5.8S rDNA序列进行拼接和人工校对,去掉两端引物后进行判读分析,用于比对潜在食物DNA测序结果,并剔除自身序列对结果的干扰。使用美国国家生物技术信息中心(https:∥www.ncbi.nlm.nih.gov)的BLAST程序检索测序片段在其GenBank数据库中的匹配序列。对序列进行筛选, 在相同的基因序列区域内,当覆盖度不低于95%、一致性超过98%时,且对应物种可能为海蜇的潜在摄食种类时认为检索结果有效,判定样品来自匹配序列对应的物种。若存在不止一种物种的匹配序列,则根据物种的分布特征及海蜇可能的食物种类排除不符合的物种,并推测可能的近缘物种或认为物种鉴定失败。
2 结 果
2.1 海蜇食性样品COⅠ序列的扩增
30个海蜇食性样品经COⅠ通用引物扩增后直接进行测序,其中26个样品经分子物种鉴定检测后为海蜇自身序列,剩余4个测序结果为双峰的样品经克隆构建文库后,随机挑选每个样品中的10个单克隆进行测序,共获取片段长度约660 bp的随机克隆序列共40条,剔除海蜇自身序列及污染的人类的COⅠ序列后,共得到36个有效序列,分属于脊椎动物亚门、子囊菌亚门及变形菌门3个门类共4个物种(表2)。其中:占比最高的是硬骨鱼纲的矛尾虾虎鱼(Chaeturichthysstigmatias),与GenBank同源序列相似度为99.41%,在全部序列中占比77.8%,全部在双台子河口外的3个海蜇食性样品中检测到;其次是硬骨鱼纲的黄鲫(Setipinnataty),与GenBank同源序列相似度为98%,在凌河口外采集的1个海蜇样品中检测到。此外在凌河口外的该海蜇样品中还检测到1条海洋真菌序列和1条光合细菌序列,由于与GenBank中同源序列相似度较低(86%~88%),故未进行最低分类阶元的推定。
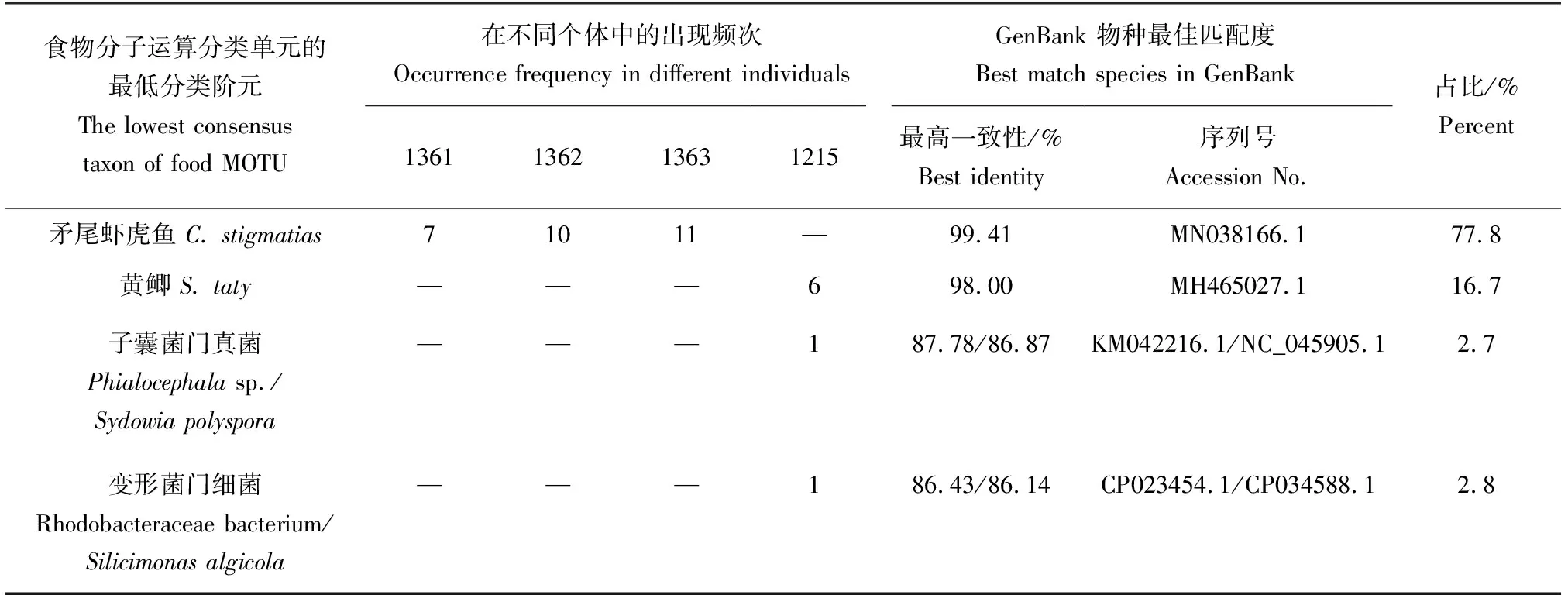
表2 基于COⅠ序列的海蜇食物分子运算分类单元分子鉴定结果
2.2 海蜇食性样品ITS-5.8S rDNA序列的扩增
样品经ITS-5.8S rDNA引物扩增后直接进行测序,其中仅4个样品测序结果为双峰,剩下26个样品经分子物种鉴定为海蜇自身序列。在4个测序结果为双峰的样品中随机挑选10~20个单克隆进行测序,共获取片段长度约1500 bp的ITS-5.8S rDNA序列共50条,剔除海蜇自身序列后,共得到30个有效物种序列,来源于10个物种(表3),为软体动物1种及子囊菌门真菌9种。其中:占比最高的是海绵共附生真菌Aspergilluspenicillioides或A.clavatophorus,与GenBank同源序列(KT390118.1、KY087703.1)的相似度分别为99.51%、99.81%,占比46.7%;其次为同属的薄曲霉A.gracilis,与GenBank同源序列(EF652045.1)的相似度分别为99.74%,占比16.7%;剩余红酵母属(Rhodotorula)、路德酵母属(Lodderomyces)、炭疽菌属(Colletotrichum)、短梗霉属(Aureobasidium)、节菱孢菌属(Sordariomycetes)、被孢霉科和爪甲团囊菌目真菌种类占比较少,仅为3.3%~6.7%;软体动物1种为扇贝科扇贝属的虾夷扇贝(Patinopectenyessoensis),与GenBank同源序列(DQ417586.1)的相似度为98.65%,在全部序列中占比6.7%。
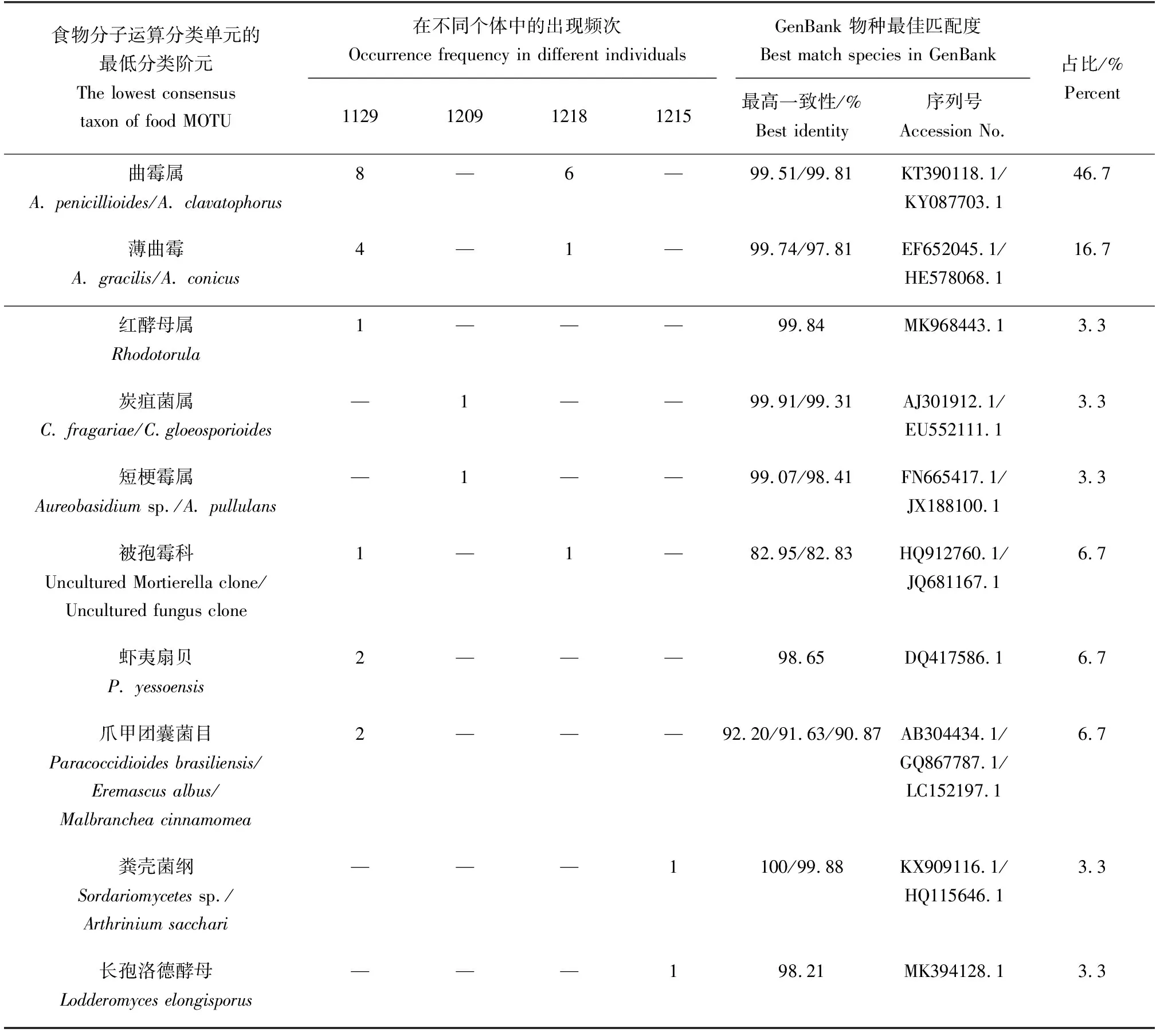
表3 基于ITS-5.8S rDNA序列的海蜇饵料生物运算分类单元分子鉴定结果
3 讨 论
3.1 DNA条形码技术在海蜇食性鉴定中的适用性
以往对海蜇食性的研究大多是在镜检分类、饲喂试验、育苗实践及资源调查中观察得出结论[23,27],此类方法不仅需要专门的形态学分类知识,还易受到诸如样品形态因素、环境条件以及样品采集者的经验和主观判断的影响,经验不足时存在着误判的可能性,所得数据的准确性及精确性无法保证。稳定同位素方法能够判断物种的主要食物来源,在食物网结构和能量流动方面具有优势,但取样可能造成损伤,且受限于取样条件的限制,不能准确提供摄食者在短期内的食物组成[30]。
利用DNA条形码技术对海蜇的食物组成进行分析,可以弥补传统技术手段和稳定同位素技术的缺陷,为深入查明海蜇主要饵料生物来源提供帮助。相较于传统技术手段,DNA条形码技术存在诸多优势:试验原理简单、操作简便,研究者不需具备专业的形态分类知识也可获得鉴定结果;所需样品量较少,不受样品形态完整性影响限制;物种鉴定适用范围广,运用DNA条形码如COⅠ基因片段能够对超过95%的物种进行准确鉴定[31-32]。席晓晴等[21]在马鞍列岛海域皮氏叫姑鱼 (Johniusbelengerii)胃含物分析中,比较了DNA条形码分子生物学手段与传统观测手段对皮氏叫姑鱼饵料生物的识别,发现传统观测方法仅获得口虾蛄(Oratosquillaoratoria)、日本鼓虾(Alpheusjaponicus)、沙蚕(Perinereisaibuhitensis)、日本蟳(Charybdisjaponica)幼体4种胃含物,尚存在大量不可辨认的虾类、蟹类,而通过COⅠ条形码分子标记对不可辨认食物糜的鉴定,识别出中华管鞭虾(Solenoceracrassicornis)、鲜明鼓虾(Alpheusdistinguendus)、长臂虾(Palaemongravieri)、中国毛虾(Aceteschinensis)、褐菖鲉(Sebastiscusmarmoratus)、日本鳀(Engraulisjaponius)等10多种胃含物种类,在较大程度上解决了对食物团中不可辨认成分鉴定的困惑;刘梦娜等[22]基于DNA条形码对中国枪乌贼(Loligochinensis)和鸢乌贼(Sthenoteuthisoualaniensis)的食物种类组成进行的研究也表明,DNA条形码技术可以将多数饵料物种准确鉴定到种,且在鉴定效率和准确性方面较传统形态学方法具有明显的优势。上述研究均已证实,基于传统测序的DNA条形码技术在胃含物饵料生物鉴定中具有应用价值。
本研究中,基于COⅠ基因通用引物扩增海蜇现场样品所构建的克隆文库共获取36个有效序列,来源于4个物种,分属于脊椎动物亚门、子囊菌亚门、变形菌门3个门类,其中在多个海蜇样品中检测到矛尾虾虎鱼和黄鲫的物种序列,占比较高且具有多种单倍型序列,推测其可能为两种鱼类的卵、仔稚鱼或成鱼碎屑等。基于ITS-5.8S rDNA条形码序列仅检测到虾夷扇贝幼虫这一种海蜇潜在食物种类,但检测到多种海洋真菌序列,如霉菌属、红酵母属、路德酵母属、炭疽菌属、短梗霉属、节菱孢菌属、被孢霉科和爪甲团囊菌目真菌种类。目前有学者认为,水母会选择性地摄食一些浮游植物、真菌和碎屑等[33-34]。张健等[7]基于脂肪酸标记法和稳定同位素技术对通州湾养殖水域海蜇进行食性分析发现,海蜇对细菌也存在摄食;徐盛楠等[8]基于高通量测序技术在对两种水螅水母现场食物进行研究时也发现,水母摄食组成中含有真菌,如枝孢菌(Cladosporium)、酵母菌和短梗霉菌(Aureobasidium)等,提示海蜇的摄食组成中可能也含有细菌、真菌等。但作为海蜇主要摄食种类的浮游动物类群,笔者采用基于两种基因片段的DNA条形码技术均未检出,说明基于传统测序的DNA条形码技术由于通量低等限制对海蜇这一广食性种类的食物谱进行分析时具有较大的局限性,但在进行动物饵料生物鉴定时仍具有较高的准确性,其在不易辨认饵料生物种类鉴定中的应用价值应得到重视。
3.2 传统测序方法的局限性及未来的发展方向
通过内含物形态鉴定、饲喂试验等传统技术手段[19,21]对海蜇摄食的研究表明,海蜇是广食性生物,对食物种类不加选择,而对其大小却有严格的限制,仅滤食其口器附近大小合适的食物颗粒(1 mm以下),其食物以小型的浮游生物为主,主要有桡足类、枝角类、介行类、涟虫类以及端足类等,也摄食鱼卵、仔稚鱼、轮虫、卤虫(Artemia)无节幼体、贝类浮游幼体、单细胞藻类和有机碎屑等,且食量很大。近期基于脂肪酸标记法和稳定同位素分析法对辽东湾和通州湾自然海域的海蜇水母体食性进行的分析也支持这一结果[6-7],≤1000 μm 浮游动物、1000~1500 μm浮游动物和>1500 μm浮游动物、底栖生物、悬浮物、浮游植物、鱼卵甚至一些海洋细菌等都是其食物类群,且≤1000 μm浮游动物是其主要摄食种类。本研究中,利用线粒体COⅠ和ITS-5.8S rDNA两种DNA条形码对辽东湾近岸采集的海蜇水母体食物组成进行分析,仅检测到鱼卵、仔稚鱼或成鱼碎屑、贝类浮游幼体、海洋真菌类、海洋细菌类等物种类别,且两种DNA条形码标记检测到的海蜇食物种类并不一致。借鉴上述传统技术手段和稳定同位素方法分析的相关结果,海蜇的潜在食物种类包括鱼卵、仔稚鱼或成鱼碎屑、贝类浮游幼体、细菌、真菌等,但检测到的海蜇潜在食物类群多样性较低,且缺乏海蜇饵料中最常见的浮游生物类群[6,23,27]。这一方面是因为样品量限制或者是通用引物在不同类群中的扩增效率差别很大,甚至无法成功扩增部分种类的目标序列[35-38],如:Zhan等[35]比较了COⅠ、16S rRNA和18S rRNA等分子标记基因在浮游动物条形码研究中的差别,发现其所用的COⅠ通用引物无法提供高质量的PCR产物;王敏晓等[36]也发现,COⅠ条形码通用引物在部分浮游生物种类中的扩增效率较低,从而造成测序结果的偏差而无法获取更多的物种信息。因此,在利用DNA条形码技术进行动物食性研究时设计通用性更好的引物序列有利于得到更多的物种信息。如Leray等[19]在研究天竺鲷科、金鳞鱼科等珊瑚礁鱼类的胃内容物时,对COⅠ基因设计了5组通用引物,结果显示,其中1组引物比传统通用引物(LCO1490/HCO2198)在后生动物类群中的扩增效率更高,能够获得更多的物种信息。此外,由于传统测序方法测序通量低,且需要结合克隆文库进行多次PCR反应并挑取有限数量的克隆序列,程序繁琐且随机性强,获取总有效食物序列数量有限,受限于工作量和研究成本,因此以传统测序为基础的DNA条形码技术会很大程度上低估海蜇这一广食性种类的食物组成,并影响海蜇食物组成物种分析的可靠性。
近年来,随着物种分子鉴定技术的发展、测序技术的进步和DNA条形码数据库的丰富完善,DNA分子追踪食物链和食物网正成为摄食生态学研究的主流方法[8,14-22]。基于高通量测序技术获得生物特异性基因识别DNA条形码序列的扩增子测序方法,称为DNA宏条形码技术。该技术可将整个混合样本的DNA片段扩增后再进行高通量测序,进而确定取样环境中生物的分布状况[17-18,35]。这一技术具有通量高、检测灵敏度高和信息量大等特点,弥补了传统测序技术的不足,目前已开始应用于包括水母类在内的海洋动物食性分析等领域[8,19,39-40],并展现出较大的应用潜能。如林先智等[39]以18S rDNA为条形码标记分别使用传统测序和高通量测序对金钱鱼(Scatophagusargus)稚鱼食物组成进行了分析,发现高通量测序方法在反映食物多样性和覆盖范围上更具优势,且灵敏度更高,检测出传统测序方法未发现的甲藻和褐藻种类,与传统测序技术相比,高通量测序技术在金钱鱼稚鱼摄食研究上优势明显,可以显著提升数据与结果的可信度。因此,未来基于高通量测序技术的DNA宏条形码方法将为海蜇乃至其他大型水母的食物组成分析提供有力工具。
4 结 论
基于DNA条形码技术,利用COⅠ基因和ITS-5.8S rDNA通用引物对辽东湾近海海蜇的现场食物组成进行分析,分别检测到海蜇的食物来源于4个和10个物种,其食物种类主要包括鱼卵、仔稚鱼或成鱼碎屑、贝类浮游幼体等,然而未检测到海蜇饵料中最常见的浮游生物类群。研究结果表明,以传统测序为基础的DNA条形码技术由于测序通量低,在研究海蜇这一广食性种类的食物谱时可能具有较大的局限性。本研究结果可为深入研究海蜇乃至水母的食物组成提供参考,未来可以利用基于高通量测序技术的DNA宏条形码方法并结合传统和稳定同位素技术对海蜇的食物谱进行研究,从而对海蜇的食性有一个更加全面准确的了解。

