番茄抗病品种吉美08 SlDFR基因的克隆与表达
梁思桃 高雁茹 刘广清 黄瑞基 刘强 任振新 乔清华





摘要:【目的】克隆番茄(Solanum lycopersicum)抗病品種吉美08二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase,DFR)基因,并分析枯萎病菌尖孢镰刀菌番茄专化型(Fol4287)诱导下其在番茄不同组织中的表达模式,为深入研究SlDFR基因在番茄抗病过程中的调控机制提供理论依据。【方法】采用同源克隆技术从番茄中克隆SlDFR基因cDNA全长序列,并进行生物信息学分析,采用实时荧光定量PCR(qRT-PCR)检测番茄根、茎、叶中SlDFR基因在枯萎病菌诱导下的表达模式。【结果】从番茄抗病栽培品种吉美08中克隆得到SlDFR基因,cDNA序列全长1659 bp,包含1个1149 bp的开放阅读框(ORF),编码379个氨基酸。SlDFR蛋白的相对分子质量为42.43 kD,等电点(pI)为6.08,是一个亲水的、稳定的酸性蛋白,亚细胞定位在高尔基体上,无信号肽和跨膜结构域,有38个磷酸化位点,其中,丝氨酸18个,苏氨酸15个,酪氨酸5个。SlDFR蛋白的二级结构中α-螺旋占37.73%,延伸链占13.98%,无规则卷曲占40.69%。系统发育进化树分析结果表明,SlDFR蛋白序列与同属茄科马铃薯(Solanum pennellii)亲缘关系较近,其次为黑果枸杞(Lycium ruthenicum)。qRT-PCR分析结果显示,SlDFR基因在番茄叶中表达量最高,在根中表达量最低。SlDFR基因在番茄根、茎、叶中表达量受番茄枯萎病菌诱导,均呈现不同程度上调,其中,在根中表达量变化最大,在叶中表达量变化较小,推测SlDFR基因的表达量与番茄枯萎病菌胁迫响应有关,且番茄根部类黄酮的合成为重要胁迫响应途径。【结论】SlDFR基因表达量受Fol4287的诱导,可能参与番茄枯萎病胁迫响应过程,且对番茄枯萎病菌的响应在番茄根部更强烈,并通过调控番茄根部黄酮类物质的合成提高根系分泌物的抑菌活性从而增强番茄对枯萎病的抗性。
关键词: 番茄;二氢黄酮醇4-还原酶(DFR);基因克隆;表达分析
中图分类号: S641.2 文献标志码: A 文章编号:2095-1191(2022)03-0813-08
Cloning and expression analysis of SlDFR gene in disease-resistant tomato varieties Jimei 08
LIANG Si-tao, GAO Yan-ru, LIU Guang-qing, HUANG Rui-ji, LIU Qiang,
REN Zhen-xin, QIAO Qing-hua
(College of Biology and Pharmacy, Yulin Normal University, Yulin, Guangxi 537000, China)
Abstract:【Objective】To clone the tomato dihydroflavonol 4-reductase(DFR) gene of the resistant varieties Jimei 08 and to analyze its expression in different tissues of tomato induced by Fusarium wilt, so as to provide theoretical reference for exploration of the regulation mechanism of SlDFR gene in the process of tomato disease resistance. 【Method】The full-length cDNA sequence of SlDFR gene from tomato was cloned through homologous cloning technology, and bioinformatics analysis was conducted, and real-time fluorescent quantitative PCR was used to detect the expression pattern of SlDFR gene in tomato roots, stems and leaves induced by Fusarium wilt. 【Result】The full-length cDNA of SlDFR was 1659 bp and cDNA contained an open reading frame (ORF) of 1149 bp that encoded 379 amino acids. SIDFR protein had a molecular weight of 42.43 kD and its theoretical isoelectric point(pI) was 6.08, indicating that it was a stable hydrophilic one. And its subcellular localization was on Golgi apparatus without signal peptide and transmembrane domain, with 38 phosphorylation sites, including 18 serine (Ser) phosphate sites, 15 threonine (Thr) phosphate sites and 5 tyrosine (Tyr) phosphate sites. In the secondary structure of SlDFR protein, α-helix accounted for 37.73%, extended chain accounted for 13.98%, and random coil accounted for 40.69%. Phylogenetic analysis showed that tomato DFR was closely related to Solanum pennelli followed by Lycium ruthenicum. The results of real-time fluorescence quantitative PCR showed that the expression of SlDFR gene was the highest in leaves, and lowest in roots. After SlDFR gene expression being induced by Fusarium wilt, the expression of SlDFR gene was up-regulated in varying degrees. The change of expression was the largest in roots and the smallest in leaves, indicating that SlDFR gene was related to the stress response of tomato Fusarium wilt, and the synthesis of flavonoids in roots was an important stress response pathway. 【Conclusion】After SlDFR gene expression being induced by Fol4287, SlDFR gene could participate in the stress response of tomato Fusarium wilt, and the response to Fusarium wilt is stronger in the root, and probably the gene regulates flavonoid synthesis in the root to enhance antibacterial activity of root exudates, thus increasing the resistance of tomato to Fusarium wilt.
Key words: tomato; dihydroflavonol 4-reductase (DFR); gene cloning; expression analysis
Foundation items: Guangxi Natural Science Foundation(2020GXNSFBA297036); Start Up Foundation for Scien-tific Research of High-level Talents of Yulin Normal University(G2020ZK06)
0 引言
【研究意义】番茄(Solanum lycopersicum)营养丰富,在全国各地均广泛种植。随着设施番茄种植面积逐年扩大及复种指数连年提高,番茄土传病害的发生与危害程度已呈逐年加重趋势(李焕玲,2014;杜建峰等,2020;祝海燕等,2020),其中,设施番茄枯萎病的危害尤为严重,一旦发生常给番茄生产造成严重损失。类黄酮是一种重要的植物保卫素,在植物抵御生物胁迫中发挥着重要作用,如一些黄酮类化合物可较好地抑制炭疽病菌、菜豆壳球孢菌、马铃薯坏疽病菌、立枯丝核菌、尖孢镰刀菌、疫霉病菌和大豆菌核菌等多种病原菌侵染(杨才琼等,2018)。二氢黄酮醇-4-还原酶(DFR)是类黄酮合成途径的一种关键酶,克隆番茄的SlDFR基因并分析其表达特性,探究其对致病菌的响应机制,对深入研究SlDFR基因在番茄抗病过程中的调控机制具有重要意义。【前人研究进展】DFR是烟酰胺腺嘌呤二核苷酸磷酸(NADPH)依赖的氧化还原酶,属于短链脱氢酶/还原酶(SDR)超家族,可选择性地催化3种二氢黄酮醇(二氢槲皮素、二氢山奈酚和二氢杨梅素)和2种黄烷酮(柚皮素和圣草酚)生成3种不同的无色花青素苷或黄烷-4-醇进而合成儿茶素、花青素苷和原花色素等(Li et al.,2012;左涛等,2016)。DFR的底物选择及催化效率是决定植物体内合成类黄酮成分和含量的关键因素。O’Reilly等(1985)采用转座子标签技术首次从玉米和金鱼草中分离DFR基因,其他学者从拟南芥、油菜、小麦、大豆、番茄和牵牛花等多种植物中也克隆到该基因(Kubasek et al.,1992;Inagaki et al.,1999;Gutierrez-Gonzalez et al.,2010;Cunmin et al.,2016;Zuo et al.,2019)。不同植物的DFR蛋白均存在保守的NADPH结合域和底物特异性结合域,但不同物种的DFR对底物结合的偏好性存在差异(于婷婷等,2018)。已有研究认为,DFR对底物结合的特异性通常是由第134位氨基酸残基决定(Johnson et al.,2001)。还有学者认为,虽然第134位氨基酸在底物选择中具有重要作用,但不能单独决定DFR的底物选择性,其催化效率还受其他氨基酸位阻效应影响(Yan et al.,2014;Chu et al.,2015)。DFR在植物花色形成、紫外线防护、抵抗病原体、花粉发育及激素运输等过程中发挥着重要作用(Adriana et al.,2019;Zhao et al.,2021)。DFR基因表达水平、底物结合特异性及催化效率是通过调控类黄酮合成影响植物抗逆性的重要途径。【本研究切入点】已有相关研究证实DFR基因参与植物胁迫响应,提高植物抗病性(李玉霞等,2020)。目前虽已克隆得到SlDFR基因,但对其结构和功能的研究,特别是对抗病性方面的相关研究仍有待补充。【拟解决的关键问题】采用同源克隆技术从番茄枯萎病抗性品种吉美08中克隆得到SlDFR基因,对其进行生物信息学分析,并研究其在番茄不同组织受番茄枯萎病菌尖孢镰刀菌番茄专化型(Fol4287)诱导后的表达模式,旨在为进一步研究SlDFR基因在番茄枯萎病抗性中的功能打下基础。
1 材料与方法
1. 1 试验材料
吉美08番茄种子购自山东寿光市洪亮种子有限公司。供试菌株为Fol4287,由福建农林大学闽台作物有害生物生态防控国家重点实验室云英子博士提供。RNA提取试剂盒购自北京华越洋生物科技有限公司。反轉录试剂盒(Prime ScriptTM Reverse Transcriptase)和pMD19-T Vector载体购自宝生物工程(大连)有限公司。DNA凝胶回收试剂盒为Axygen和AP-GX-250。PCR引物和qPCR引物由北京擎科生物科技有限公司合成。
1. 2 试验方法
1. 2. 1 引物设计与合成 根据数据库中已有的SlDFR基因片段保守区,使用Primer Premier 5.0设计该基因全长扩增引物(SlDFR-F:5'-GCTGACTCTT TTTTACCTTT-3',SlDFR-R:5'-ATTATGTATGGTTG GGTTTC-3')。根据SlDFR基因序列测序结果,使用Primer Premier 5.0设计该基因的荧光定量引物(qSlDFR-F:5'-ACTCTCCTCCGAAGACGACA-3',qSlDFR-R:5'-TCACCTTCTTCTGGTTCTCA-3')。以Actin为内参基因(Actin-F:5'-CTCAACCCCAAGG CTAACAG-3';Actin-R:5'-ACCTCAGGGCATCGG AAC-3')。
1. 2. 2 SlDFR基因克隆与测序 以番茄根、茎、叶提取总RNA逆转录的单链cDNA为模板,利用1.2.1中基因全长扩增引物进行PCR扩增。PCR扩增产物经克隆后送至北京擎科生物科技有限公司测序。
1. 2. 3 生物信息学分析 通过NCBI的开放阅读框(ORF)Finder工具和DNAman进行序列分析。使用在线软件Protparam(https://web.expasy.org/protparam/)进行DFR蛋白理化性质分析。以Signalp 5.0和TMHMM进行信号肽和跨膜结构域分析。使用NCBI的CDD(https://www.ncbi.nlm.nih.gov/cdd/)和SMART(http://smart.embl-heidelberg.de/)在线分析软件进行蛋白结构域分析。以Netphos 3.1 server分析潜在磷酸化位点。利用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)预测SlDFR蛋白的亚细胞定位。分别利用SOPMA和SWISS-MODEL对SlDFR蛋白的二级结构和三级结构进行预测。以MEGA 10.0进行序列比对和构建系统发育进化树。
1. 2. 4 SlDFR基因表达量测定及分析 以水培14 d的2叶1心期番茄幼苗为试验材料,以Fol4287进行不同时间处理后分别取根、茎和叶,提取总RNA,以反转录单链cDNA为荧光定量PCR(qRT-PCR)模板。以1.2.1中的荧光定量引物为PCR引物,以Actin为内参基因,使用Real Time PCR荧光定量仪(Eppendorf)进行检测。
1. 3 统计分析
试验数据采用Excel 2010进行统计分析,以2法计算基因相对表达量。
2 结果与分析
2. 1 SlDFR基因克隆结果
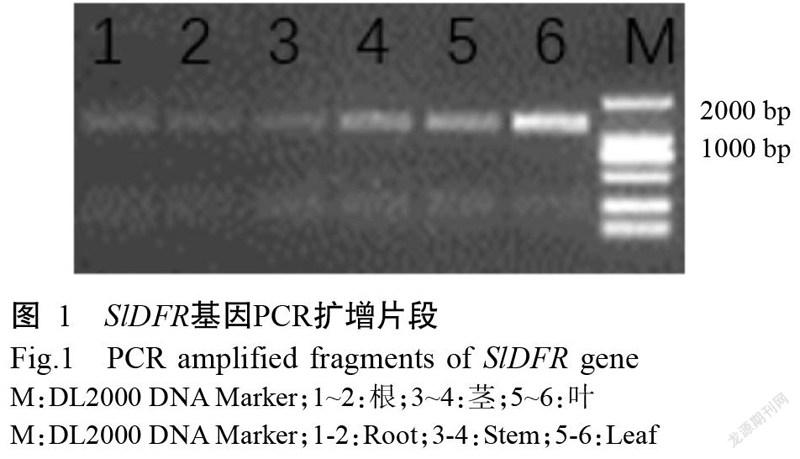
分别使用番茄根、茎、叶组织提取总RNA,使用反转录试剂盒反转录得到cDNA。cDNA作为模板直接或稀释后扩增基因全长,得到SlDFR基因cDNA序列(图1)。从图1可看出,扩增条带约1500 bp,对该cDNA序列测序结果显示SlDFR基因为1659 bp,说明电泳结果与目的片段大小一致。
2. 2 SlDFR蛋白序列及其理化性质分析
通过NCBI的ORF Finder对SlDFR基因全长(1659 bp)的ORF即编码区进行查找,结果发现SlDFR基因包含1个1149 bp的ORF(启动子为ATG,终止子为TAG),编码379个氨基酸。此外,在编码区的上游和下游分别含有一段93 bp的5'非翻译区和417 bp的3'非翻译区。
使用在线软件Protparam分析SlDFR蛋白的理化性质,发现SlDFR蛋白的分子式为C1914H2976N494O563S16,相对分子质量为42.43 kD,等电点(pI)为6.08,不稳定参数为32.84,蛋白性质稳定(通常认为蛋白的不稳定参数在40.00以下为稳定蛋白);SlDFR蛋白中含量相对较多的氨基酸为Lys(8.4%,32个)、Ala(8.2%,31个)和Leu(7.4%,28个);亲水性平均系数为-0.218,脂肪系数为82.88。说明该蛋白是亲水的、稳定的酸性蛋白。Signalp 5.0和TMHMM的分析结果表明,SlDFR蛋白无信号肽和跨膜结构域。
2. 3 SlDFR蛋白的保守结构域、磷酸化位点及亚细胞定位预测
使用NCBI的CDD和SMART蛋白结构域在线预测工具分析SlDFR蛋白的结构域,结果(图2)表明,SlDFR蛋白存在NADPH结合位点,烟酰胺腺嘌呤二核苷酸依赖的脱水酶结构域位于氨基酸序列的20~271位。以Netphos 3.1 server对SlDFR蛋白的潜在磷酸化位点进行分析,结果(图3)表明,该蛋白共有38个磷酸化位点,其中,丝氨酸18个,苏氨酸15个,酪氨酸5个。利用Cell-PLoc 2.0预测SlDFR蛋白的亚细胞定位,结果表明SlDFR蛋白可能定位在高尔基体上。
2. 4 SlDFR蛋白的二级和三级结构分析
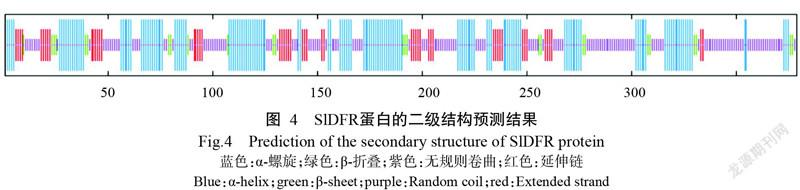
利用SOPMA預测SlDFR蛋白的二级结构,结果(图4)表明,该蛋白主要由α-螺旋、β-折叠、无规则卷曲和延伸链组成,其中,α-螺旋占37.73%,延伸链占13.98%,β-折叠占6.60%,无规则卷曲占40.69%。
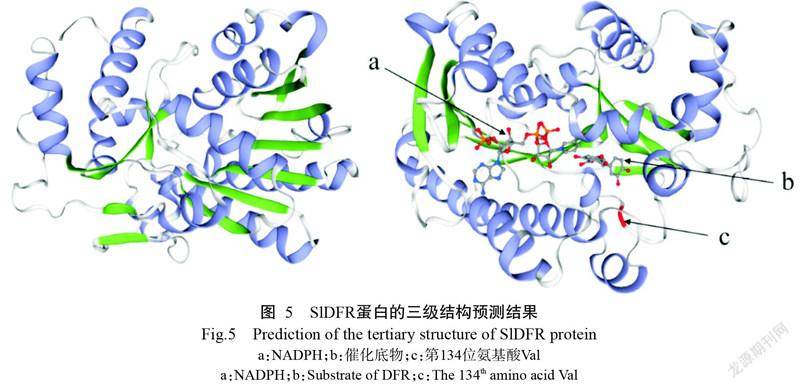
利用SWISS-MODEL在线软件同源建模得到SlDFR蛋白的三维模型(图5),其全球模型质量估计(GMQE)值为0.80,介于正常值0~1.00,表明该模型在空间结构上合理性较高;SlDFR蛋白主要是由无规则卷曲和α螺旋组成;SlDFR蛋白存在2个配体结合区(NADPH结合区和底物结合区),其第134位氨基酸为Val,属于非Asn/Asp型DFR蛋白。
2. 5 DFR蛋白序列系统发育进化树分析结果
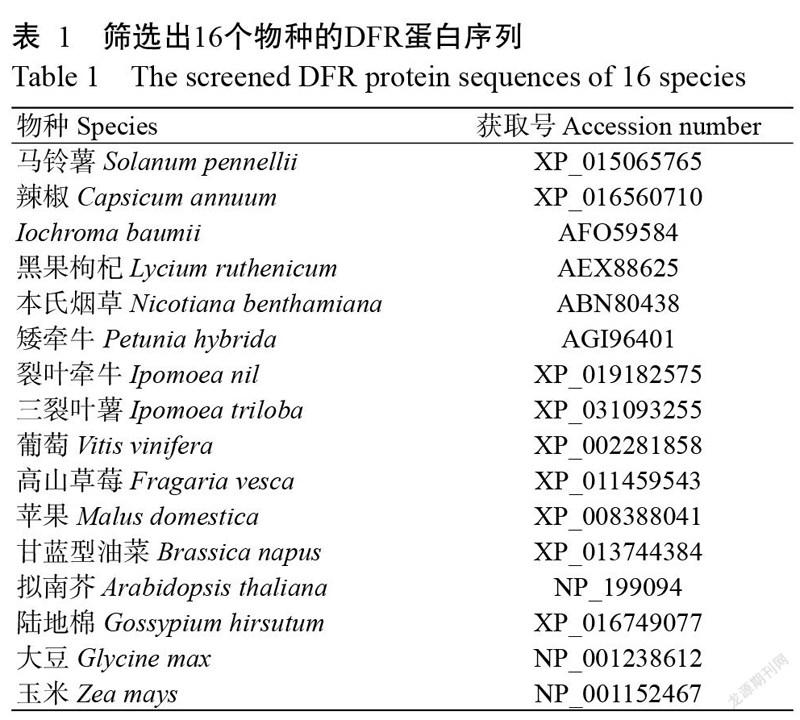
在NCBI中检索DFR蛋白氨基酸相似序列,筛选出16个物种(表1)的DFR蛋白序列,利用MEGA 10.0对番茄及16个物种的DFR蛋白序列进行比对,结果(图6)显示,SlDFR蛋白序列与茄科其他属物种的亲缘关系较近。其中,马铃薯(Solanum pennellii)与番茄的同源性最高,其次为黑果枸杞(Lycium ruthenicum)、Iochroma baumii和辣椒(Capsicum annuum),茄科植物中矮牵牛(Petunia hybrida)与番茄的亲缘关系较远;在非茄科植物中,旋花科的裂叶牵牛(Ipomoea nil)和三裂叶薯(Ipomoea triloba)与茄科植物的进化距离较近。
2. 6 SlDFR启动子顺式作用元件分析结果
为进一步了解SlDFR基因的调控功能,从NCBI数据库中找到番茄SlDFR基因CDS前2000 bp序列,利用启动子在线分析工具PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行启动子顺式作用元件分析,推测其可能参与的生理过程。结果(表2)表明,SlDFR启动子区域除含有大量光响应元件外还含有大量激素响应元件,例如茉莉酸响应元件、脱落酸响应元件和赤霉素响应元件等,此外还有1个胁迫响应元件和1个MYB结合位点。
2. 7 SlDFR基因响应Fol4287的表达情况分析
采用Fol4287处理水培番茄0~14 h,通过qRT-PCR分析不同处理时间下根、茎、叶SlDFR基因的表达情况。结果(图7)显示,未进行Fol4287处理的对照组中,SlDFR基因在叶中的表达量显著高于根(P<0.05,下同),为根中表达量的21.67倍(图7-A),而在茎中的表达量与根相比无显著差异(P>0.05)。Fol4287处理后SlDFR基因在番茄根、茎、叶中的表达量均表现不同程度上调,其中,在茎中的表达量呈先上升后下降变化趋势,处理10 h时表达量最高,为处理0 h的1.75倍,且显著高于除处理8 h外其他处理时间的表达量(图7-C);Fol4287处理后SlDFR基因在叶中的表达量快速升高,其中在处理6 h时出现最大值,为处理0 h的1.94倍,且显著高于其他处理时间的表达量(图7-D);而Fol4287处理后SlDFR基因在根的表达量出现2个峰值,其中,处理6 h时出现第一个峰值,随后表达量下降,处理12 h时出现最高值,表达量为对照组的6.31倍,且显著高于除处理14 h外其他处理时间的表达量(图7-B),与茎和叶的表达量相比变化最大,在叶中表达量变化较小。
3 讨论
DFR是类黄酮合成途径中的关键酶。1994年Bongue-Bartelsman等首次从番茄下胚轴中克隆得到SlDFR基因,并对其表达特性进行初步分析。本研究从番茄品种吉美08中克隆得到1个SlDFR基因,氨基酸序列分析结果表明,该基因编码379个氨基酸,与滇牡丹、威氏绿绒蒿和日本蛇根草等植物DFR基因编码的氨基酸数量相近(李云琴等,2020;冯猜等,2021;严朋飞等,2021);SlDFR蛋白是亲水的、稳定的酸性蛋白,且无信号肽和跨膜结构域,为非分泌性蛋白,定位在高尔基体上,而植物DFR蛋白多定位于细胞质中(肖向文等,2014;赖呈纯等,2016;王疆然等,2021),因此,SlDFR蛋白亚细胞定位预测结果是否准确有待进一步探究;启动子顺式作用元件分析结果表明,SlDFR启动子区域含有激素响应元件、胁迫响应元件和MYB结合位点,暗示SlDFR基因参与了番茄多种激素和逆境胁迫响应途径。
DFR底物结合的特异性控制类黄酮合成途径中生成的花色素苷、黄烷醇和原花青素支路通量,对植物体内具有抑菌活性的黄酮类物质合成和积累发挥着重要作用。左涛等(2016)研究表明,DFR基因通过调控儿茶素生物合成提高杨树的抗病性。Zhang等(2019)、Zhuang等(2019)研究指出,DFR基因对植物花青素的合成具有调节作用。花青素除了作为决定植物花色的主要色素外,在植物抵抗干旱、低温和盐胁迫等多种非生物胁迫及对火疫病菌、软腐病菌和黄萎病菌等多种致病菌的响应过程中也具有重要作用(Gould et al.,2018;王鸿雪等,2020)。DFR基因还可作为黄烷酮4-还原酶(FNR)催化柚皮素和圣草酚生成黄烷-4-醇(flavan-4-ols),进而在其他酶的作用下形成3-脱氧花青素(3-deoxyantho-cyanidin),3-脱氧花青素及其中间代谢产物五羟基黄酮(luteo-forol)可作为防御物质抵御真菌和细菌侵害(Li et al.,2012)。Johnson等(2001)通过对非洲菊的研究首次提出DFR底物结合区,并发现其中的第134位氨基酸种类是决定所催化底物的关键,其中,第一类的第134位氨基酸是天冬酰胺残基(Asn),与非洲菊相同,属于Asn型DFR;第二类的第134位氨基酸为天冬氨酸残基(Asp),属于Asp型DFR,可催化二氢栎皮黄酮(DHQ)和二氢杨梅黄酮(DHM),但不能有效催化二氢堪非醇(DHK);第三类的第134位氨基酸残基既不是天冬酰胺也不是天冬氨酸。此外,还有研究指出,虽然第134位氨基酸在底物选择中具有重要作用,但不能单独决定DFR底物的选择性,其催化效率还受其他氨基酸位阻效应影响(Yan et al.,2014;Chu et al.,2015)。本研究结果表明,番茄DFR氨基酸序列第134位氨基酸為缬氨酸(Val),为非Asn/Asp型DFR,但其作用底物的特异性及催化效率有待进一步探讨。
类黄酮参与植物防御的方式分为通过调节植物生理特性提高植物对病原菌的抵抗能力和直接抑制病原菌侵害2种类型。某些黄酮类物质不会在植物的根部累积,而是通过根系分泌到土壤中发挥作用(Akifumi and Yazaki,2014),此类抑菌物质可在病原菌与植物根系接触前分泌到土壤中从而抑制病原菌对根系的侵害。本研究结果表明,进行Fol4287处理后,番茄根、茎、叶中的DFR基因表达量均显著增加,说明其受Fol4287诱导参与了胁迫响应过程。此外,虽然DFR基因表达具有明显的组织特异性,在叶中的表达量显著高于根和茎,但在根中DFR基因受Fol4287的诱导表达量快速、大量升高,其响应的剧烈程度高于茎和叶,推测SlDFR基因可通过调控番茄根系类黄酮合成提高根系黄酮类物质的含量,从而调控番茄对枯萎病的抗性。但根部合成的黄酮类物质是作物信号物质参与番茄对病原菌的应答提高番茄根部对番茄枯萎病菌的抵抗能力,还是通过根系分泌物的形式提高根际环境中类黄酮的含量,抑制病原菌生长和定殖,抑或是二者兼有,目前还不清楚。因此,DFR基因对植物根部及根系分泌物中黄酮类物质的调控及对其抑菌活性的影响将是下一步的研究方向。
4 结论
SlDFR基因表达量受Fol4287的诱导,可能参与番茄枯萎病胁迫响应过程,且对番茄枯萎病菌的响应在番茄根部更强烈,并通过调控番茄根部黄酮类物质合成提高根部对病原菌的抵抗力或根系分泌物的抑菌活性从而增强番茄对枯萎病的抗性。
参考文献:
杜建峰,吴伟,张晓英,李洋,丁新华. 2020. 番茄颈腐根腐病的发生及其防治研究进展[J]. 生物技术通报,36(10):200-206. [Du J F,Wu W,Zhang X Y,Li Y,Ding X H. 2020. Research progress on the occurrence and control of Fusarium crown and root rot of tomato[J]. Biotechno-logy Bulletin,36(10):200-206.] doi:10.13560/j.cnki.biotech.bull.1985.2020-0260.
冯猜,周娜娜,孙世宇,周洁羽,孙威. 2021. 日本蛇根草DFR3基因的克隆分析及其编码蛋白的分离纯化[J]. 西北植物学报,41(6):926-932. [Feng C,Zhou N N,Sun S Y,Zhou J Y,Sun W. 2021. Cloning and analysis of DFR3 gene from Ophiorrhiza japonicus(OJDFR3) and isolation and purification of its coding protein[J]. Acta Bota-nica Boreali Occidentalia Sinica,41(6):926-932.] doi:10.7606/j.issn.1000-4025.2021.06.0926.
赖呈纯,黄贤贵,甘煌灿,潘红,范丽华. 2016. 刺葡萄DFR基因克隆及生物信息学分析[J]. 福建农业学报,31(7):683-689. [Lai C C,Huang X G,Gan H C,Pan H,Fan L H. 2016. Cloning and bioinformatics of DFR gene in Vitis davidii foex[J]. Journal of Fujian Agriculture,31(7):683-689.] doi:10.19303/j.issn.1008-0384.2016.07.003.
李焕玲. 2014. 番茄青枯病的发生与综合防治技术[J]. 北方园艺,(5):54-56. [Li H L. 2014. Occurrence and integra-ted control techniques of tomato bacterial wilt[J]. Nor-thern Horticulture,(5):54-56.]
李玉霞,曲延英,艾海提·艾合买提,王慧敏,黄启秀,陈琴,陈全家. 2020. 通过GbF3'H基因单独沉默及其与GbCHI和GbDFR基因共沉默研究其在海岛棉中抗枯萎病功能[J]. 棉花学报,32(1):1-10. [Li Y X,Qu Y Y,Aihaiti A H M T,Wang H M,Huang Q X,Chen Q,Chen Q J. 2020. Through single silencing GbF3'H gene and its co-silencing with GbCHI and GbDFR genes to study their function in resistance to Fusarium wilt in Gossypiu barbadense[J]. Cotton Science,32(1):1-10.]
李云琴,原晓龙,陈中华,王毅. 2020. 滇牡丹(Paeonia delavayi)二氢黄酮醇4-还原酶基因的鉴定及表达[J]. 分子植物育种,18(4):59-63. [Li Y Q,Yuan X L,Chen Z H,Wang Y. 2020. Identification and expression of dihydroflavonol 4-reductase gene in Paeonia delavayi[J]. Mole-cular Plant Breeding,18(4):59-63.] doi:10.13271/j.mpb. 018.001083.
王鸿雪,刘天宇,庄维兵,王忠,朱林,渠慎春,翟恒华. 2020. 花青素苷在植物逆境响应中的功能研究进展[J]. 农业生物技术学报,28(1):174-183. [Wang H X,Liu T Y,Zhuang W B,Wang Z,Zhu L,Qu S C,Zhai H H. 2020. Research advances in the function of anthocyanin in plant stress response[J]. Journal of Agricultural Biotechnology,28(1):174-183.] doi:10.3969/j.issn.1674-7968. 2020.01.018.
王疆然,王玉芬,王舒婷,张芳娟,牛颜冰,王德富. 2021. 不同花色黄芩中dfr基因的克隆及时空表达分析[J]. 生物工程学报,37(4):1312-1323. [Wang J R,Wang Y F,Wang S T,Zhang F J,Niu Y B,Wang D F. 2021. Cloning and temporal-spatial expression analysis of dfr gene from Scutellaria baicalensis with different colors[J]. Journal of Bioengineering,37(4):1312-1323.] doi:10.13345/j.cjb.200 466.
肖向文,朱奇朗,刘海峰,王俊铎,罗城,曾闻,梁亚军,龚兆龙,李晓波. 2014. 棕色棉DFR基因的克隆与生物信息学分析[J]. 生物技术通报,30(5):88-95. [Xiao X W,Zhu Q L,Liu H F,Wang J D,Luo C,Zeng W,Liang Y J,Gong Z L,Li X B. 2014. Cloning and bioinformatics analysis of DFR gene from brown cotton[J]. Biotechno-logy Bulletin,(5):88-95.] doi:10.13560/j.cnki.biotech.bull.1985.2014.05.021.
严朋飞,张莹欣,贾维嘉,刘建,郭小雪,江元祺,屈燕. 2021. 威氏绿绒蒿二氢黄酮醇4-还原酶DFR基因克隆与表达分析[J]. 分子植物育种,19(24):8064-8070. [Yan P F,Zhang Y X,Jia W J,Liu J,Guo X X,Jiang Y Q,Qu Y. 2021. Cloning and expression analysis of dihydroflavonol 4-reductase gene from Meconopsis wilsonii[J]. Molecular Plant Breeding,19(24):8064-8070] doi:10.13271/ j.mpb.019.008064.
楊才琼,杨文钰,刘江. 2018. 植物类黄酮的化学生态学意义[J]. 天然产物研究与开发,30(11):2009-2016. [Yang C Q,Yang W Y,Liu J. 2018. Advances on chemical ecology of plant flavonoids[J]. Natural Product Research and Development,30(11):2009-2016.] doi:10.16333/j.1001-6880.2018.11.025.
于婷婷,倪秀珍,高立宏,韓国军,朱长甫,盛彦敏. 2018. 高等植物二氢黄酮醇4-还原酶基因研究进展[J]. 植物研究,38(4):632-640. [Yu T T,Ni X Z,Gao L H,Han G J,Zhu C F,Sheng Y M. 2018. Advances in study of dihydroflavonol 4-reductase(DFR) genes of higher plants[J]. Bulletin of Botanical Research,38(4):632-640.] doi:10. 7525/j.issn.1673-5102.2018.04.020.
祝海燕,李婷婷. 2020. 过量施肥对设施番茄影响的调查分析[J]. 中国瓜菜,33(1):55-58. [Zhu H Y,Li T T. 2020. Effect of excessive fertilization on facilities tomato[J]. Chinese Cucurbits and Vegetables,33(1):55-58.] doi:10.16861/j.cnki.zggc.2020.0011.
左涛,赵树堂,卢孟柱,孙爱东,王延伟,贺伟. 2016. 杨树二氢黄酮醇-4-还原酶基因(DFR)的克隆及反义表达对儿茶素合成的影响[J]. 东北林业大学学报,44(10):49-55. [Zuo T,Zhao S T,Lu M Z,Sun A D,Wang Y W,He W. 2016. Cloning dihydroflavonol-4-reductase gene(DFR) of Poplar and its antisense expression effects on catechin[J]. Journal of Northeast Forestry University,44(10):49-55.] doi:10.13759/j.cnki.dlxb.2016.10.010.
Adriana G F,Armando V M D,Elvira O R,Luis F J. 2019. Photoprotective and antigenotoxic effects of the flavonoids apigenin,naringenin and pinocembrin[J]. Photochemistry and Photobiology,95(4):1010-1018. doi:10. 1111/php.13085.
Akifumi S,Yazaki K. 2014. Flavonoids in plant rhizospheres:Secretion,fate and their effects on biological communication[J]. Plant Biotechnology,31(5):431-443. doi:10.5511/ plantbiotechnology.14.0917a.
Bongue-Bartelsman M,O'Neill S,Tong Y,Yoder J. 1994. Characterization of the gene encoding dihydroflavonol 4-reductase in tomato[J]. Gene,138(1-2):153-157. doi:10. 1016/0378-1119(94)90799-4.
Chu Y X,Chen H R,Wu A Z,Cai R,Pan J S,Research M. 2015. Expression analysis of dihydroflavonol 4-reductase genes in Petunia hybrida[J]. Genetics and Molecular Research,14(2):5010-5021. doi:10.4238/2015.May.12.4.
Cunmin Q,Zhao H Y,Fu F Y,Wang Z,Zhang K,Zhou Y,Wang X,Wang R,Xu X F,Tang Z L,Lu K,Li J N. 2016. Genome-wide survey of flavonoid biosynthesis genes and gene expression analysis between black- and yellow-seeded Brassica napus[J]. Frontiers in Plant Scien-ce,7:1755. doi:10.3389/fpls.2016.01755.
Gould K S,Jay-Allemand C,Logan B A,Baissac Y,Bide L P R. 2018. When are foliar anthocyanins useful to plants? Re-evaluation of the photoprotection hypothesis using Arabidopsis thaliana mutants that differ in anthocyanin accumulation[J]. Environmental and Experimental Botany,154:11-22. doi:10.1016/j.envexpbot.2018.02.006.
Gutierrez-Gonzalez J,Guttikonda S,Tran L,Aldrich D,Zhong R,Yu O,Nguyen H,Sleper D. 2010. Differential expression of isoflavone biosynthetic genes in soybean during water deficits[J]. Plant Cell Physiology,51(6):936-948. doi:10.1093/pcp/pcq065.
Hong M,Kim D,Ahn J,Kang S,Seo Y,Kim J. 2018. Comparison of radiosensitivity response to acute and chronic gamma irradiation in colored wheat[J]. Genetics and Molecular Biology,41(3):611-623. doi:10.1590/1678-4685-GMB-2017-0189.
Inagaki Y,Johzuka-Hisatomi Y,Mori T,Takahashi S,Hayakawa Y,Peyachoknagul S,Ozeki Y,Iida S. 1999. Genomic organization of the genes encoding dihydroflavonol 4-reductase for flower pigmentation in the Japanese and common morning glories[J]. Gene,226(2):181-188. doi:10. 1016/s0378-1119(98)00571-x.
Johnson E T,Ryu S,Yi H,Shin B,Cheong H,Choi G. 2001. Alteration of a single amino acid changes the substrate specificity of dihydroflavonol 4-reductase[J]. The Plant Journal,25(3):325-333. doi:10.1046/j.1365-313x.2001. 00962.x.
Kubasek W,Shirley B,McKillop A,Goodman H,Briggs W,Ausubel F. 1992. Regulation of flavonoid biosynthetic genes in germinating arabidopsis seedlings[J]. Plant Cell,4(10):1229-1236. doi:10.1105/tpc.4.10.1229.
Li H,Qiu J,Chen F,Lü X,Fu C,Zhao D,Hua X,Zhao Q. 2012. Molecular characterization and expression analysis of dihydroflavonol-4-reductase(DFR) gene in Saussurea medusa[J]. Molecular Biology Reports,39(3):2991-2999. doi:10.1007/s11033-011-1061-2.
O'reilly C,Shepherd N S,Pereira A,Schwarz-Sommer Z,Bertram I,Robertson D S,Peterson P A,Saedler H. 1985. Molecular cloning of the a1 locus of Zea mays using the transposable elements En and Mu1[J]. The EMBO Journal,4(4):877-882.
Yan F,Di S,Rodas F,Torrico T,Murai Y,Iwashina T,Anai T,Takahashi R. 2014. Allelic variation of soybean flower color gene W4 encoding dihydroflavonol 4-reductase 2[J]. BMC Plant Biology,14(1):58. doi:10.1186/1471-2229-14-58.
Zhang L,Sun X,Wilson I W,Shao F,Qiu D. 2019. Identification of the genes involved in anthocyanin biosynthesis and accumulation in Taxus chinensis[J]. Genes,10(12):982. doi:10.3390/genes10120982.
Zhao Y, Zhou W J, Chen Y, Li Z J, Song X Q, Wang J, Tian D K, Niu J. 2021. Metabolite analysis in Nymphaea ‘Blue bird’ petals reveal the roles of flavonoids in color formation,stress amelioration,and bee orientation[J]. Plant Science,312:111025.
Zhuang H,Lou Q,Liu H,Han H,Wang Q,Tang Z,Ma Y,Wang H. 2019. Differential regulation of anthocyanins in green and purple turnips revealed by combined de novo transcriptome and metabolome analysis[J]. International Journal of Molecular Sciences,20(18):4387. doi:10.3390/ ijms20184387.
Zuo L,Trisha L,Moira G,Shirley J,Tom E,Jeffrey P. 2019. Assessing anthocyanin biosynthesis in Solanaceae as a model pathway for Secondary metabolism[J]. Genes(Basel),10(8):559. doi:10.3390/genes10080559.
(責任编辑 思利华)

