基于高光谱成像技术的鲜莲直链淀粉含量检测*
高 胜,黄 亮,邹金平,李佳育,魏 萱,2,3
(1.福建农林大学机电工程学院,福建 福州 350100;2.福建农林大学食品科学学院,福建 福州 350002;3.福建省农业信息感知技术重点实验室,福建 福州 350100)
0 引言
随着生活水平的提高,人们对莲子的口味和营养方面的要求也越来越高。莲子作为药材的同时也是一种滋补品,其直链淀粉的含量直接影响着莲子的品质和口感。作为一种高淀粉含量的食品,莲子的直链淀粉含量会影响莲子的吸水性、膨胀性、固体物质溶解性、颜色、光泽、黏性和柔软性[1]。不同品种莲子的直链淀粉含量差别较大,因此检测莲子的直链淀粉含量对后续加工有重要意义。传统的直链淀粉检测一般是利用碘比色法、碘亲和力滴定法以及横切侵染法,这些方法费时费力,且容易受实验条件的影响[2]。
高光谱成像技术是一种能获取丰富光谱和图像信息的无损检测技术,相较于化学检测方法,具有省时、省力、环境友好的优点[2]。现已有很多基于高光谱成像技术的农产品内部品质的研究。在直链淀粉含量研究方面,利用反射光谱来预测谷类籽粒的粗蛋白和直链淀粉含量的可行性得到了初步验证[3],如李亨[4]建立了基于高光谱成像技术的马铃薯淀粉含量定量检测模型,相关系数(rp)达到0.925,效果比较好。王丽平[5]建立了基于近红外光谱技术的水稻直链淀粉含量检测模型,其中对非糯性水稻的预测结果比较好。如刘芸等[6]比较了直链淀粉、粗淀粉、粗蛋白三者光谱间的差异性,并分析其高光谱特征,分别建立了相关预测模型(r>0.7)。结果表明,基于高光谱成像技术可以很好地预测小麦、大米内部的直链淀粉的含量,但是目前还缺乏基于高光谱成像技术的鲜莲内部直链淀粉含量检测的研究。
课题组采用高光谱成像技术对鲜莲直链淀粉进行检测研究,并采用波长优选方法筛选对直链淀粉敏感的特征波段,从而建立鲜莲直链淀粉检测模型,以期为鲜莲加工过程中的直链淀粉含量在线检测提供解决方案。
1 材料和方法
1.1 试验材料
试验样本来源于福建省三明县莲科所,选用宣莲、广昌莲、建选36号、满天星、太空莲和湘莲等品种。在成熟期采摘后,将新鲜莲蓬置于液氮中保存,运输至福建农林大学实验室,在温度为4 ℃冰箱中冷藏保存12个小时。
1.2 高光谱图像采集与校正
高光谱成像系统主要包含的部件有高光谱成像仪(ImSpector V10E, SPECIM, Spectral Imaging Ltd.Oulu, Finland)、光源、载物台、黑箱以及高光谱数据采集软件等。整套系统由台湾五铃光学提供,可采集光谱范围为400 nm~1 000 nm,光谱分辨率为2.8 nm。高光谱成像系统如图1所示,载物平台的移动速度设置为3.5 mm/s,曝光时间为30 ms。镜头距离移动平台40 cm,且垂直向下。调节高光谱仪相机的焦距对系统进行黑白校正。校正公式如下:
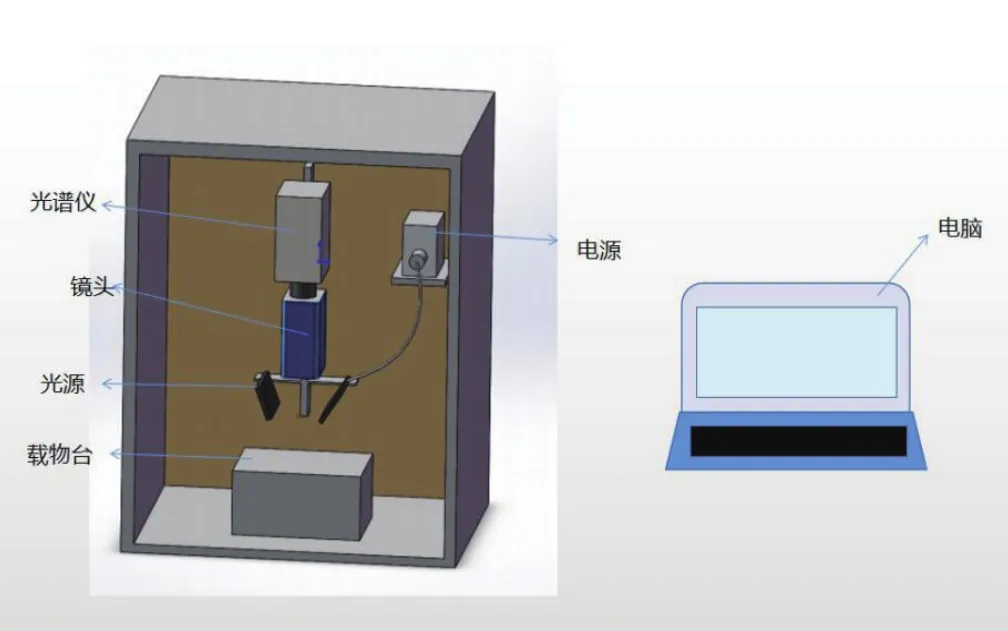
图1 高光谱成像系统示意图

其中,M——校正后的高光谱图像数据;M0——原始高光谱图像数据;A——黑暗背景下高光谱图像数据;N——标准反射白板的高光谱数据。
1.3 莲子直链淀粉含量的测定
采集高光谱图像后,采用直链淀粉含量测试盒(苏州科铭生物技术有限公司生产)测试直链淀粉含量。称取0.01 g的烘干样本于研钵中研碎,经过匀浆、提取、离心等步骤,取20 μL上清液置于96孔板中依次加入显色剂,测定620 nm处吸光值,再通过96孔板的计算公式测定莲子直链淀粉的含量。多次测量取平均值,利用吸光值测定直链淀粉的公式如下:

1.4 数据处理
采用ENVI 4.7(ITT Visual Information Solution,America)、Matlab 2017(MathWorks, America)和The Unscrambler 10.1(CAMO, America)等分析软件处理数据。
1.4.1 光谱预处理
采用ENVI 4.7对莲子的光谱图像进行感兴趣区域(ROI)的平均光谱提取,为了消除噪声、外部杂光等的影响,对比一阶导数、二阶导数、SG平滑(S-G smoothing)、多元散射校正(MSC)、标准正态变量转换(standard normal variate transformation, SNV)等预处理方法建模效果[7],选取最佳的预处理方法。
1.4.2 样本集划分
建立光谱和化学值之间的分析模型,采集大量样本的光谱数据和化学值,在这大量的样本数据中挑选出具有该样本特性的数据,并将获得的样本集划分为校正集和预测集,选择具有强样本特性的校正集样本可以使得建模时计算量减少,并且还可以提高预测模型的准确度[8]。本试验采用的是SPXY法(sample set partitioning based on joint x-y distances)。SPXY法的优点:使用SPXY法对样本集进行划分时,可以在考虑样本集光谱变量的同时兼顾到化学值[9],SPXY法的具体计算公式如下:
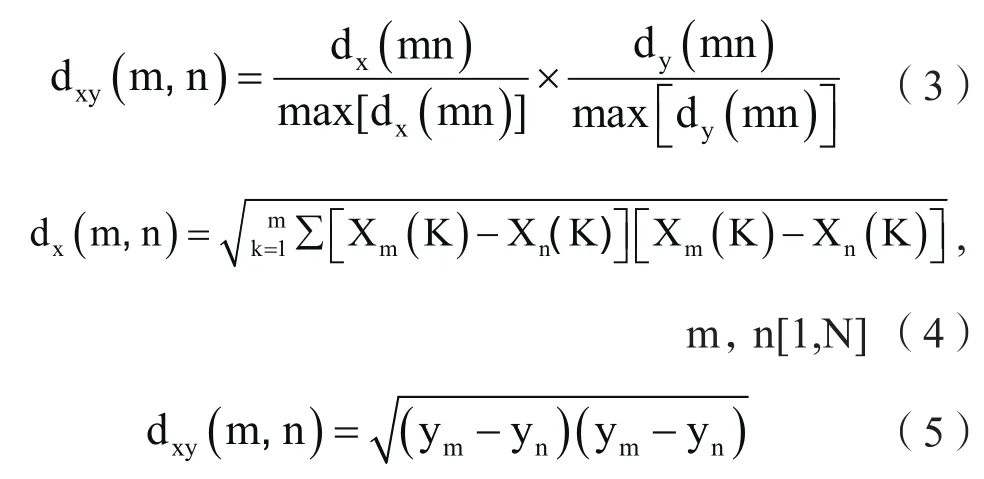
计算公式中,dxy(m,n)指的是计算样本m和样本n之间的欧式距离,dx(m,n)指的是基于样本中光谱变量x来计算样本中的欧式距离,dy(m,n)指的是以化学值来计算样本之间的欧氏距离。n指的是总的样本集个数,m指的是样本集光谱曲线波长的数目,为了使获得的样本集在两个不同的空间上有相同大小的权重,所以对样本集的光谱数据和化学值分别进行标准化处理。
为了使样本在两个空间具有相同的权重,分别除以各个空间的最大值进行标准化处理。
1.4.3 特征波长提取方法
连续投影算法(SPA)是一种去除样本集光谱数据之间线性影响的算法[10]。SPA算法的原理是在所有样本集的光谱数据之间进行循环投影计算,以便于找到冗余数据信息变量最少的组合,这样使得各个样本集数据之间共线性最小,使得建模不仅快而且还拥有更好的建模精度[11]。算法原理如下:

令xj为校正集光谱第j列,令xj=pxj,再令n=n+1,如此循环进行计算。
回归系数法(RC)是通过因变量和若干个自变量之间的定量表达式获得的,即回归方程通过控制可控变量的数值[12],从影响变量变化的自变量中区分出重要因素和次要因素,表达式如式(7)所示:

其中A0,A1分别为回归常数和回归系数,M为随机误差。
1.4.4 建模方法
本文采用偏最小二乘回归(PLSR)方法建立直链淀粉检测模型,PLSR同时具有主成分分析和多元线性回归分析的优点[13]。在建模过程中,通过计算自变量和因变量之间的方差来提高模型的预测精度,所以建立PLSR模型的关键是确立主因子数目或者是隐含变量(LV)的数目。假如主因子数目量太少,则无法表达出被测样本光谱数据的变化。如果主因子数目过多,则会使得一些噪声的数据加入到模型中,导致建立的模型预测能力变差[14]。
1.4.5 模型评价
以相关系数(R)、校正集均方根误差(RMSEC)、预测集均方根误差(RMSEP)作为衡量直链淀粉预测效果的主要指标,并另外选用相对分析误差(RPD)对预测精度再次进行评价[15],通过式(8)获得。如果RPD≥2,则表明模型预测效果良好,可进一步用于实际检测研究;如果1.4≤RPD<2,则表明模型可进行定量分析,由于RPD值较小,所以预测精度还有待提高[16];如果RPD<1.4,则表明模型预测能力较差,难以进行定量分析[17]。RPD的表达式如下:

式中,SD为标准差,SEP为标准预测误差。
2 结果与分析
2.1 感兴趣区域平均光谱
本文将单个样本感兴趣区域(region of interest,ROI)中每个像素点的光谱曲线用于后续处理,去除首尾噪声后(400 nm~971 nm)的平均光谱图如图2所示。从图中可以看出不同样本的光谱数值变化趋势比较一致,波段在460 nm~570 nm有明显的上升偏移,可能是由于水分波段发生偏移所致,波段在500 nm~920 nm处存在较为明显的吸收,可能与直链淀粉分子中的C-H基团的四级倍频、O-H二级倍频、O-H一级倍频有关。

图2 感兴趣区域平均光谱图
2.2 莲子的直链淀粉含量
采用SPXY法所划分的校正集和预测集直链淀粉含量结果如表1所示。从表中可以看出鲜莲的直链淀粉含量差异较大,校正集莲子直链淀粉的含量最大值为227.90 mg/g,最小值为100.82 mg/g,标准差为44.73 mg/g。预测样本的直链淀粉含量在校正集样本的范围内,故样本划分合理。
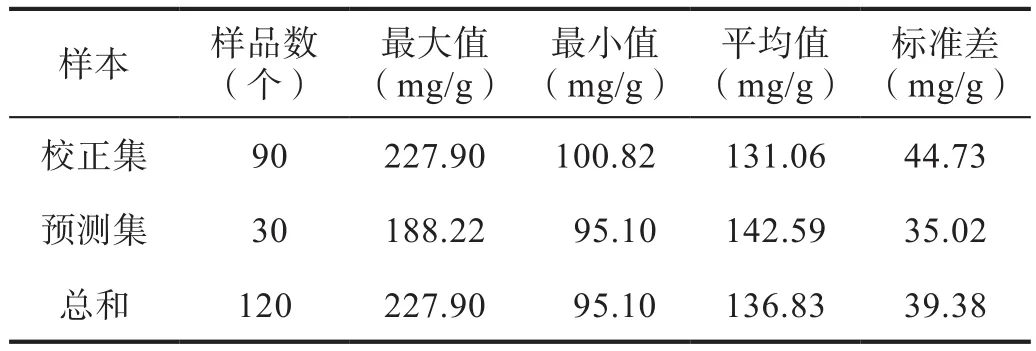
表1 莲子直链淀粉含量化学值统计值
2.3 光谱分析与预处理
在高光谱图像的采集过程中可能会受到暗电流、外部杂光和外部环境噪声等设备环境的影响,而这些无关的信息会影响光谱数据的准确性,故需要对光谱数据进行预处理。将不同预处理的光谱作为模型输入,建立直链淀粉含量检测PLSR回归预测模型,结果如表2所示。从表中可以看出,经过MSC和一阶导数的复合预处理方法最优,所建PLSR模型的校正集相关系数(Rc)为0.803,校正集均方根误差(RMSEC)为1.855,预测集相关系数(Rp)为0.836,预测集均方根误差(RMSEP)为2.072,相对分析误差(RPD)为1.623,结果表明使用该方法进行莲子直链淀粉含量检测具有一定可行性。
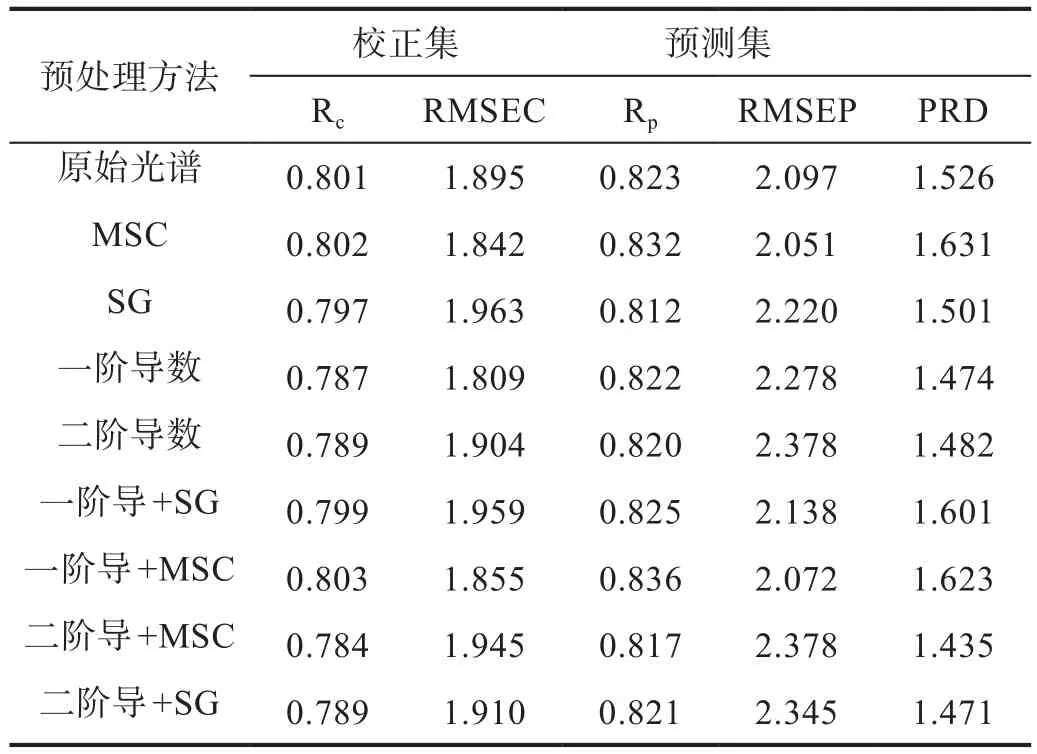
表2 采用不同预处理光谱建立的鲜莲直链淀粉含量检测PLSR模型结果
2.4 特征波长的提取
采用回归系数法(regression coefficient, RC)和连续投影算法(SPA)提取预处理后的光谱特征波长,然后建立PLSR预测模型。在RC方法中,选择如图3中所示的波峰波谷位置所对应的波段作为特征波段,提取的特征波段有418.5 nm、425.7 nm、463.4 nm、509.0 nm、543.8 nm、584.0 nm、683.4 nm、795.8 nm、879.1 nm、919.5 nm和974.2 nm共计11个波段。

图3 基于回归系数法特征波段提取图
图4为SPA选取的波长数对应的交叉验证均方根误差。SPA方法选择特征波长变量时,根据RMSEC确定最佳的特征波长数,最佳的特征波长数对应于最小的RMSEC。由图4可知,当特征提取的波长数为9时,建立的PLSR模型有最小的RMSEC值,其值为1.802。因此,SPA选择了449.5 nm、464.1 nm、500.6 nm、582.7 nm、644.3 nm、756.1 nm、812.4 nm、872.3 nm、977.6 nm共9个特征波段,特征波段数约占全光谱的2%,极大地减少光谱波长变量,加快建模速度。

图4 变量筛选图
由表3可知,经过连续投影算法(SPA)提取特征波长后所建立的模型效果最好,偏最小二乘模型的校正集的相关系数均在0.835左右。如图5和图6所示,利用SPA提取的特征波段建立的PLSR模型定量评价指标都有提升,通过直线拟合可以发现校正集和预测集的直链淀粉测量值分别与相应的预测值有良好的线性关系。

图5 校正集的预测值和真实值相关系数图
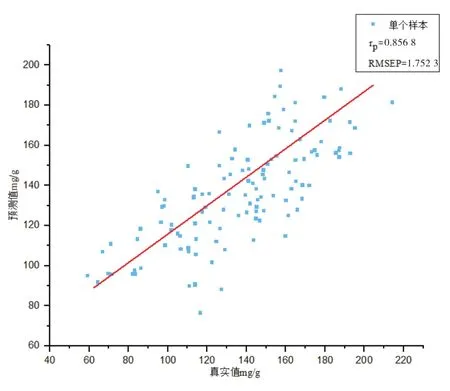
图6 预测集的预测值和真实值相关系数图

表3 基于不同特征波长的直链淀粉检测PLSR模型
SPA-PLSR模型的相对分析误差(RPD)为1.944。这说明SPA能够有效地提取特征波段,提高建模效率,但RPD值略小于2,说明所建立的检测模型精度仍然有待提高。在试验过程中,可能由于模型的单一性以及样本数量少等原因导致模型的精度不高,后期可通过增加试验样品数量和建立多种预测模型对比以获得更高检测精度的模型。
3 结论
本文采用高光谱成像技术对直链淀粉含量进行快速检测。结果表明,采用一阶导数和多元散射校正(MSC)预处理后的建模效果最好。然后采用SPA提取了9个特征波段,所建PLSR预测模型的校正集相关系数(Rc)为0.835,校正集均方根误差(RMSEC)为1.802,预测集相关系数(Rp)为0.856,预测集均方根误差(RMSEP)为1.752,相对分析误差(RPD)为1.944。经过RC法所建立的PLSR预测模型的预测集相关系数(Rp)为0.838,预测集均方根误差(RMSEP)为1.897,相对分析误差(RPD)为1.761。本研究为进一步开发直链淀粉含量在线检测仪器提供了思路,并奠定了良好的基础。

