羊肚菌转录因子分类和生物信息学分析
任瑞芳 张绍鹏 蒋 鹏 张 瑞 张 祚 郑涵予 史梦华 马晓龙
羊肚菌转录因子分类和生物信息学分析
任瑞芳1张绍鹏2蒋 鹏1张 瑞1张 祚1郑涵予1史梦华1马晓龙3*
(1. 武汉轻工大学 生命科学与技术学院,湖北 武汉 430023;2. 武汉轻工大学硒科学与工程现代产业学院,湖北 武汉 430023;3. 武汉市农业科学院蔬菜研究所,湖北 武汉 430345)
首次根据Genbank中已知的7237条羊肚菌序列,对所有编码转录因子的序列进行了功能分类,并重点对羊肚菌中的锌指蛋白(ZFP)编码序列进行了生物信息学分析。结果表明,Genbank中所有7237条羊肚菌来源的核苷酸序列,有167条是编码转录因子的序列,其中88条序列编码了锌指蛋白。这些序列都含有相应的保守基序,编码蛋白质的二级结构以α-螺旋为主,三级结构极为相似,功能上以亲水性蛋白为主。
羊肚菌;转录因子;锌指蛋白;生物信息学
羊肚菌()又名美味羊肚菌,俗称羊雀菌、包谷菌,是子囊菌中有名的食用真菌,属于子囊菌亚门()、盘菌纲()、盘菌目(),马鞍菌科()、羊肚菌属()[1]。因其菌盖表面生有许多小凹坑,外观极似羊肚而得名。羊肚菌性平,味甘寒,无毒;有益肠胃、助消化、化痰理气、壮肾补阳、补脑提神、强身健体、预防感冒和增强人体免疫力等功效[2]。羊肚菌在全世界都有自然分布,以在法国、德国、美国、印度及中国分布较为广泛。在我国,羊肚菌分布北至东北三省,南至广东、福建、台湾,东至山东,西至新疆、西藏、宁夏、贵州,共28个省、市、自治区,但每个地区羊肚菌种类资源丰富度不同且有很大差异,其中以甘肃和四川地区的种类资源较为丰富[3]。
据报道,羊肚菌的营养成分很丰富,含有多糖、酶类、氨基酸、吡喃酮抗生素、脂肪酸等。羊肚菌的菌丝体含有丰富的氨基酸及其他营养元素,总氨基酸含量大大高于一般食用菌,必需氨基酸含量占比较高,占氨基酸总量的49%。其中决定鲜味的谷氨酸、赖氨酸、天冬氨酸含量尤为突出,是优质的蛋白源。羊肚菌含有20种以上无机元素,其中微量元素有Zn、Mn、Cu、Co、Cr、Fe、Ni、B、Sr、V等10种,并含有人体必需的Ca、Mg、P等3种常量元素[1]。
转录因子(Transcription Factor,TF)是一组独特的DNA结合蛋白,也称反式作用因子,其可直接或间接与基因启动子区域中顺式作用元件特异性结合,对基因转录进行调控。其主要功能是激活或抑制基因的转录效应。转录因子在基因表达调控中起着核心作用,能够协调很多生物过程,包括分化、发育、新陈代谢、凋亡、自噬等。在真菌响应外界氮源、生长发育、次级代谢和抗逆过程中发挥了重要作用。根据不同的DNA结合基序,转录因子主要分为经典的锌指结构、同源结构域和基本螺旋-环-螺旋三大类。其中,锌指蛋白(ZFP)是最大的具有序列特异性的DNA结合蛋白家族[4]。锌指在结构上是多样的,并且存在于在各种细胞活动中执行多种功能的蛋白质中,例如复制和修复,转录和翻译,代谢和信号传导,细胞增殖和凋亡[5]。
1 材料与方法
1.1 数据来源
羊肚菌目前在Genbank数据库中已知的7237条核酸序列条目中找出所有能编码具有转录因子活性的序列。将这些序列下载下来进行结构域分析,并鉴定候选序列的结构域[6]。
1.2 结构域分析
利用NCBI提供的在线软件Conserved Domain Search进行结构域分析,然后以结构域类型为依据将转录因子进行分类[7]。
1.3 羊肚菌ZFP系统发生树的构建
运用MEGA X内本身具有的Clustal W对羊肚菌ZFP序列进行多序列比对。将比对结果保存,然后基于多序列比对结果,利用MEGA X软件中ML(maximum likelihood)方法构建生物进化树。
1.4 羊肚菌ZFP保守基序鉴定
运用Meme在线平台对羊肚菌锌指结构蛋白中的保守基序(motif)进行在线分析,将最大的motif检索数值定为12[8]。
1.5 羊肚菌ZFP的结构预测
(1)一级结构预测。采用ExPASy提供的在线软件Protparam对锌指蛋白进行一级结构特性分析。主要包括氨基酸的种类和含量、理化性质及相对分子质量等。
(2)二级结构预测。利用SOPMA程序对羊肚菌锌指结构转录因子的蛋白质二级结构进行在线预测,预测分析二级结构的组成和含量。
(3)三级结构预测。采用Phyre2.0对三级结构进行建模。也可以采用SWISS-MODEL进行三级结构预测[9]。
2 结果与分析
2.1 羊肚菌转录因子家族的鉴定
在Genbank数据库中,属于羊肚菌的核酸序列共有7237条,有72条scafford长序列(大于1000 bp)中包含有能够编码羊肚菌转录因子的编码序列,这72条scafford长序列中,70条序列来自于尖顶羊肚菌(),另外2条来自于梯棱羊肚菌()。在这些scafford序列中,能够编码具有转录因子活性的编码序列(CDS)共有169条。
2.2 羊肚菌转录因子家族的结构域分析
将169条蛋白候选序列进行结构域分析,有2条没有找到结构域,其余167条转录因子按照结构域可以分为28类:①Zn2Cys6,②GATA,③HLH,④Homeobox,⑤HMG-box,⑥ZIP,⑦Forkhead,⑧HSF,⑨Copperfirst,⑩RFX,TAF,⑪TFⅡ,⑫zf,⑬RING-finger,⑭MADS,⑮TEA,⑯SH2,⑰WD40,⑱YL1,⑲PC4,⑳CCAAT-binding,㉑Zn ribbon,㉒p53,㉓Tfb,㉔kila-N,㉕TBP,㉖spt,㉗Elongin_A,㉘Fungal_trans_2(编辑注:受版面限制,详表未予刊载)。最大的一组转录因子是Zn2Cys6占40%,第二大类是基本转录因子(TFⅡ)占11%(图1)。广义上来说,含Zn2Cys6,Zn ribbon,zf,RING-finger,GATA[10]的蛋白均属于锌指蛋白,共88条[4,5,11]。
2.3 羊肚菌锌指蛋白序列的系统发育树的构建
为了评估羊肚菌中的锌指蛋白之间的进化关系,利用MEGA对羊肚菌88条含锌指结构的转录因子编码序列做进化分析,构建蛋白系统发育树(图2)。将系统发育树分为11类,第7类最多。以第1类为例,除RPB15307.1、RPB14569.1、RPB11370.1外,均为含有Zn2Cys6的锌指蛋白,其中RPB14569.1、RPB11370.1含有GATA且属于同一小分支。含有Zn2Cys6的锌指蛋白大多同属一个分支且分支较短,说明它们的亲缘关系较近,如RPB14600.1和RPB07552.1,RPB13300.1和RPB15722.1;而它们又属于不同的大分支,因此在进化过程中可能发生了变异,变异的程度也存在差别。而含ZIP的锌指蛋白RPB15307.1与含Zn2Cys6的锌指蛋白RPB17815.1同属一个分支,它们之间的亲缘关系较近,同源性高。结果表明,羊肚菌锌指结构转录因子在进化上具有不同程度的分化。
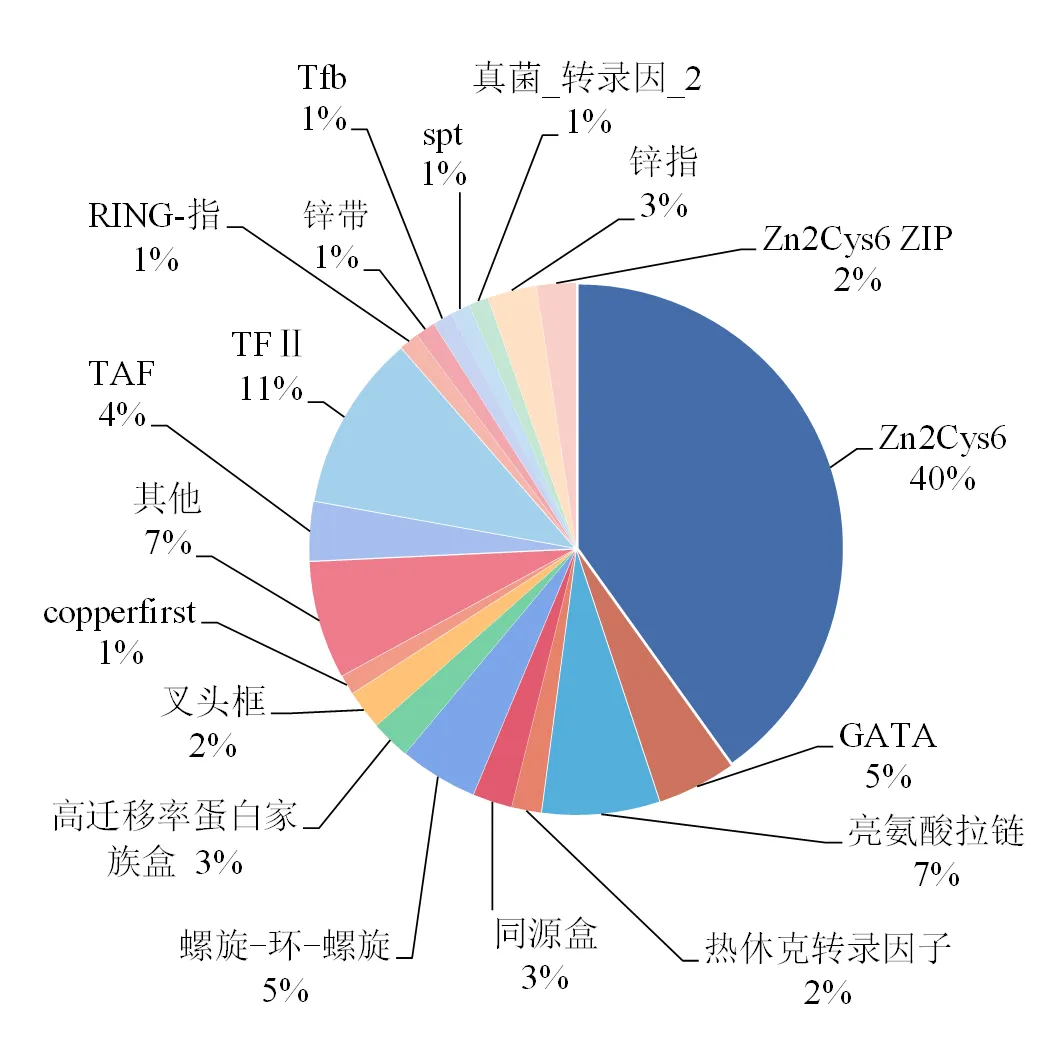
图1 羊肚菌各类型转录因子的分布
2.4 羊肚菌锌指结构转录因子保守基序分析
通过MEME在线分析得出,羊肚菌锌指蛋白具有11个保守基序。基序1为RACDRCRRRK IKCDG;基序2为PVERELRRRVFWALYVLD;基序3为LVDLYFQNIHPLFPILHRPTF;基序4为KPPCSRCVKLGLECV;基序5为LGLAVRMAQD LGLHRE;基序6为QQQQQQQQQQQQQQH;基序7为LGRPVLINDEDIDVELP;基序8为VPRGY VEELEARLKALEKLL;基序9为QQLHPHQQQQ QQHQQHQQQQQQAQHQQ;基序11为WLLLHY LFNAGLVLL;基序12为LHLSYYATLILLHRP。基序1、基序4比较保守,大部分锌指包合蛋白N端都有分布。基序2,基序5,基序6,基序7也比较保守,大部分蛋白都有分布。这些保守基序是重要的结构单位,可以用来分析进化关系,拥有的相同序列越多,表明亲缘关系越近。RPB15301.1,RPB14688.1,RPB08201.1和RPB11254.1只含有一个基序,与其他蛋白的进化关系较远。剩余的锌指蛋白所含基序较多,进化关系较近[12](图3)。
2.5 羊肚菌锌指蛋白一级结构预测
蛋白质一级结构是指肽链中氨基酸排列顺序和连接方式,是氨基酸分子结构的基础,包含了决定蛋白质分子所有结构层次构象的全部信息。分析结果显示:不同的羊肚菌锌指蛋白在氨基酸的组成、等电点、蛋白质分子量和理化性质等方面存在着一定的差异。共含有20种基本氨基酸,以丝氨酸、脯氨酸、甘氨酸和亮氨酸为主。pH在5.5~10之间,蛋白质分子量为3.1~11.5 kDa,氨基酸数目最少为259个,最多为1339个。

图2 羊肚菌锌指蛋白序列的系统发育树(部分图)
疏水性是决定蛋白质三级结构构象的重要因素之一,许多转录因子都是利用其疏水性区域与其他蛋白质相互作用。疏水性负值越大表示越亲水,正值越大表示越疏水,两性蛋白的疏水性值在0.5~−0.5之间。羊肚菌锌指蛋白疏水性值均为负值,在−0.06~− 1.3之间,说明它们主要是以亲水性蛋白为主[13]。
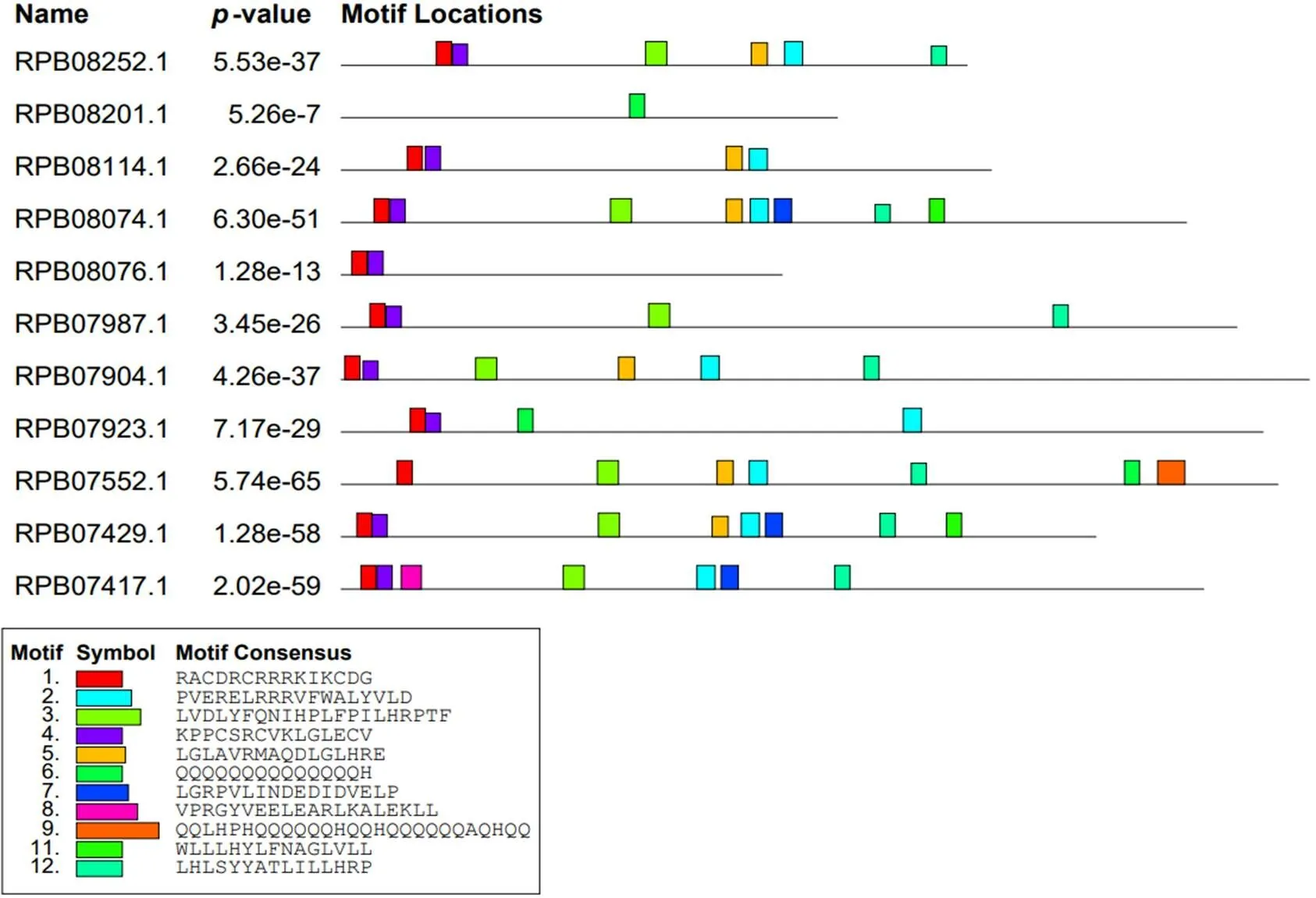
图3 羊肚菌锌指结构转录因子保守基序(部分图)
2.6 羊肚菌锌指蛋白二级结构预测
蛋白质二级结构的形成,主要是其多肽链的主链骨架上所含有的羰基和亚氨基,在主链骨架盘曲折叠时可以形成氢键,依靠这种氢键的维持固定,多肽链主链上的若干肽段可以形成有规律的空间排布,与R侧链构象无关。因此,对于羊肚菌锌指结合蛋白的二级结构预测很有必要,利用SOPMA程序进行预测和分析,结果表明羊肚菌锌指蛋白家族二级结构是以α-螺旋、延伸链、β-折叠、β-转角、无规卷曲为构成原件,以α-螺旋、无规卷曲为主。二级结构聚集在一起就能形成超二级结构,参与三级结构的形成,是蛋白质发挥作用的单位[14]。
2.7 羊肚菌锌指蛋白质三级结构预测
蛋白质的三级结构是建立在二级结构、超二级结构,乃至结构域的基础上的,与侧链的相互作用有关。蛋白质的三级结构即一个蛋白质的立体结构,或称三维结构,包括多肽链中的一切原子的空间排列方式。三级结构稳定的主要作用力是非共价键,是疏水相互作用、氢键、范德华力和静电作用来维持的特定空间结构。蛋白质只有在折叠成特定的结构之后方能行使特定的生物学功能,因此了解蛋白质结构对于认识其功能有着重要意义。
利用Swiss-model对88条羊肚菌锌指包合蛋白的三级结构进行预测。结果表明,大部分锌指蛋白三级结构组成相似,但又存在差异。图4是16个有代表性的羊肚菌锌指蛋白的的三级结构预测图。它们都是以α-螺旋为主要构件,但α-螺旋的数目存在差异。虽然有些锌指蛋白的α-螺旋数目相等,但是蛋白质的三级结构是由α-螺旋、β-转角、β折叠,延伸链和无规卷曲共同组成的,氨基酸数目存在差异,肽链长度存在差异,导致三级结构必然存在着差异。包含锌指结构的蛋白,它们能结合的锌离子,根据结构域数目和种类的不同,结合1到4个锌离子不等,以维持依赖锌离子的蛋白质三级结构的稳定[15]。
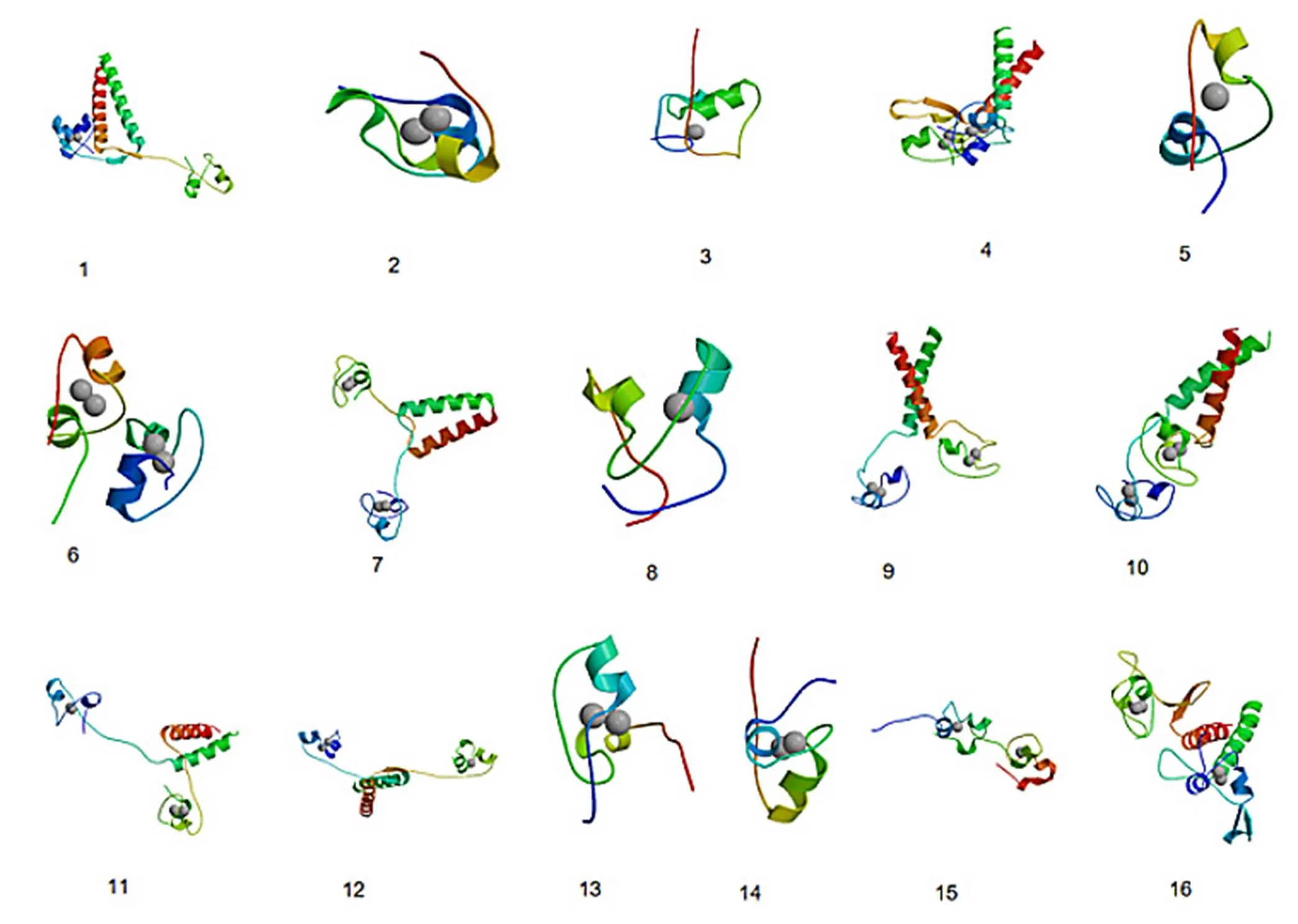
图4 16个典型的羊肚菌锌指蛋白质三级结构
3 讨 论
羊肚菌锌指蛋白作为羊肚菌转录因子中最大的一类,对其研究具有重要意义。本实验对羊肚菌转录因子蛋白家族进行分类,将167条编码的具有转录因子活性的蛋白分成了28类。对羊肚菌编码锌指蛋白基因家族进行分析得出,编码的88个羊肚菌锌指蛋白以亲水性蛋白为主,其二级结构以α-螺旋为主。它们朝着不同方向进行着不同程度的进化,能够结合不同数目的锌离子,维持结构的稳定。
锌在植物生长过程中发挥着不可或缺的作用。如果使用锌离子螯合剂脱锌或用Fe、Al、Cu、Ag、Mn等其他金属离子代替锌离子,锌指蛋白与DNA或RNA等结合特异性就会被明显抑制,不能形成折叠结构,同时稳定的蛋白也会失去支撑力,影响基因表达及胁迫响应,使其丧失大部分功能[16]。锌指序列主要存在于转录因子中,越来越多的锌指蛋白已被证实与生物体内的各种RNA代谢有关,能够提高植物遭受盐、冷冻、干旱、热、渗透的耐受性以及病菌感染的生物抗性[17]。
羊肚菌编码锌指蛋白基因的研究基础薄弱,功能研究几乎是空白,本实验对羊肚菌中所有编码锌指结构的转录因子序列做了分类和生物信息学分析,为羊肚菌转录因子的挖掘和功能调控研究提供了理论依据。
[1] 辛青霞, 芦永昌, 赵英, 等. 羊肚菌多糖的提取及含量测定[J]. 四川师范大学学报(自然科学版), 2020, 43(5):671-676.
[2] 任廷远, 安玉红. 羊肚菌活性成分及营养保健功能的研究现状(综述)[J]. 浙江食用菌, 2010, 18(1): 21-23.
[3] 任怡莲, 冯锐, 朱金霞, 等. 羊肚菌国内外研究现状及产业发展建议[J]. 宁夏农林科技, 2020, 61(8): 50-53.
[4] Jen J, Wang Y C. Zinc finger proteins in cancer progression[J]. Journal of Biomedical Science. 2016, 23(1): 53.
[5] Krishna S S, Majumdar I, Grishin N V. Structural classification of zinc fingers: survey and summary[J]. Nucleic Acids Research. 2003, 31(2): 532-550.
[6] 方亦圆, 严维, 吴建新, 等. 花生MYB转录因子的鉴定与生物信息学分析[J]. 生物信息学, 2021, 19(2): 1-15.
[7] 安秀红, 徐锴, 厉恩茂, 等. 苹果抗性相关的谷胱甘肽转移酶基因MdGSTU1的生物信息学和表达分析[J]. 中国农业科学, 2014, 47(24): 4868-4877.
[8] 谷彦冰, 冀志蕊, 迟福梅, 等. 苹果WRKY基因家族生物信息学及表达分析[J]. 中国农业科学, 2015, 48(16):3221-3238.
[9] 孙雪婧, 杜晓华, 杨孝朴, 等. 牦牛CYGB基因CDS区克隆与生物信息学分析[J]. 中国农业科学, 2014, 47(13): 2690-2698.
[10] Gao J, Chen Y H, Peterson L C. GATA family transcriptional factors: emerging suspects in hematologic disorders[J]. Experimental Hematology and Oncology. 2015, 4(1): 28.
[11] Carrillo A J, Schacht P, Cabrera I E, et al. Functional profiling of transcription factor Genes in[J]. G3: Genes, Genomes, Genetics. 2017, 7(9): 2945-2956.
[12] 何欣, 叶伟, 高晓霞, 等. 白木香倍半萜合成酶As-SesTPS1基因的克隆、生物信息学和表达分析[J]. 中草药,2015, 46(5): 733-739.
[13] 董庆龙, 冀志蕊, 迟福梅, 等. 苹果MADS-box转录因子的生物信息学及其在不同组织中的表达[J]. 中国农业科学, 2014, 47(6): 1151-1161.
[14] 李思楠, 苏安玉, 于以成, 等. 大豆再生相关基因GmARF的生物信息学及表达分析[J]. 基因组学与应用生物学, 2015, 34(10): 2187-2193.
[15] 黎帮勇, 胡尚连, 曹颖, 等. 毛竹NAC转录因子家族生物信息学分析[J]. 基因组学与应用生物学, 2015, 34(8): 1769-1777.
[16] 李琳, 丁峰, 潘介春, 等. 植物锌指蛋白转录因子家族研究进展[J]. 热带农业科学, 2020, 40(2): 65-75.
[17] 赵丽娟, 易小娅, 曾幼玲. 植物逆境相关C2H2型锌指蛋白的研究进展[J]. 分子植物育种, 2016, 14(3): 578-585.
Classification of transcription factors inand bioinformatics analysis
Ren Ruifang1Zhang Shaopeng2Jiang Peng1Zhang Rui1Zhang Zuo1Zheng Hanyu1Shi Menghua1Ma Xiaolong3*
(1. School of life Science and Techonogy, Wuhan Polytechnic University, Wuhan, Hubei 430023, China; 2. School of Modern Industry for Selenium Science and Engineering, Wuhan Polytechnic University, Wuhan, Hubei 430023, China; 3. Vegetable Research Institute of Wuhan Academy of Agricultural Sciences, Wuhan, Hubei 430345, China)
In this study, all sequences encoding transcription factors were functionally classified according to 7237sequences known in Genbank for the first time, and the coding sequence of zinc finger protein (ZNF) fromwas analyzed by bioinformatics. The results showed that 167 of the 7237 nucleotide sequences fromin GenBank were sequences encoding transcription factors, of which 88 sequences encoded zinc finger proteins, these sequences contain corresponding conserved motifs. Encoding the secondary structure of proteins is mainly α-helix, its tertiary structure is very similar. It is mainly hydrophilic protein in function. The results of this study will lay a theoretical foundation for further study of the regulatory role of transcription factors in the growth and development of.
; transcription factors; Zinc finger protein; bioinformatics
S646
A
2095-0934(2022)02-127-06
武汉市应用基础前沿专项(2020020601012297);财政部和农业农村部国家现代农业产业技术体系(CARS-20)
任瑞芳(1997—),女,硕士,主要从事中药材的分子生物学研究。E-mail:2893468536@qq.com。
马晓龙(1982—),男,高级农艺师,博士,主要从事食药用菌的栽培育种及生理药理学研究。E-mail:22843560@qq.com。

