郏县红牛POLB基因克隆及生物信息学分析
李玉龙,吕世杰,翟亚莹,张志杰,朱肖亭,张子敬,陈付英,施巧婷,王二耀
(1.河南农业大学动物科技学院,郑州 450002;2.河南省农业科学院畜牧兽医研究所,河南省畜禽繁育与营养调控重点实验室,郑州 450002)
郏县红牛虽然具有耐粗饲等特点,但其与国外专门化的肉牛品种相比,其生长发育速度缓慢、肉用性能低[1]。随着消费者对牛肉需求量的增加,提高中国本土良种肉牛的生产力迫在眉睫。因此,为提高郏县红牛的生长潜能,吕世杰等[2-3]利用选择性清除方法发现,DNA聚合酶β(DNA polymerase beta,POLB)基因位于郏县红牛和安格斯牛两品种间差异的基因组区域中,并位于牛日增重相关QTL(QTL 69373)内[4-5],推测POLB基因可能与牛肌肉发育相关。
POLB基因是1971年Weissbach等[6]首次从牛胸腺中分离出的一种DNA聚合酶,该基因是真核细胞中最小的DNA多聚酶,其蛋白分子质量仅为39 ku[7]。POLB属于DNA聚合酶X家族中的一员,这些DNA聚合酶的家族成员在DNA复制、DNA损伤修复、细胞周期调控中起重要作用[8]。研究发现,降低食管癌细胞(EC9706)中POLB基因的表达量,会导致该细胞生物学特性发生改变,从而抑制细胞增殖、促进细胞凋亡[9]。采用RNA干扰技术抑制乳癌细胞(MCF-7)中POLB基因的表达发现,细胞的增殖也受到显著抑制[10]。沈亮[11]研究发现,POLB基因可能通过PI3K/Akt信号通路对MCF-7的增殖和迁移进行调控。朱艳艳[12]利用甲基苄基亚硝胺(N-nitrosomethylbenzylamine,NMBA)分别诱导POLB基因敲除的小鼠食管上皮组织,经mRNA转录组芯片及差异表达基因GO功能富集分析发现,有33个差异表达的基因与细胞增殖调控相关,有27个差异表达的基因与细胞周期调控相关,其中涉及到PI3K-Akt、MAPK-ERK、PLK1/CENP-A[13-14]等信号通路。以上报道表明,POLB基因表达量的变化与细胞的增殖、凋亡、周期调控等活动关系密切。因此,本研究通过克隆牛POLB基因并进行生物信息学分析,以期为进一步探究POLB基因生物学功能与调控机理提供参考。
1 材料与方法
1.1 材料
1.1.1 试验动物及样品采集 5头18月龄郏县红牛来源于河南省平顶山市郏县红牛良种繁育中心。在屠宰场处死后,采集其背最长肌组织,立即放入液氮中冷冻,带回实验室,-80 ℃保存备用。
1.1.2 载体和感受态细胞 pMD18-T载体购自TaKaRa公司;大肠杆菌DH5α感受态细胞购自生工生物工程(上海)股份有限公司。
1.1.3 主要试剂及仪器 TRIzol试剂和Trans2K®DNA Marker均购自北京全式金生物技术有限公司;反转录试剂盒购自南京诺唯赞生物科技有限公司;KOD OneTMPCR Master Mix-Blue购自东洋纺(上海)生物科技有限公司。PCR扩增仪(6325ZO810735)和微量移液枪均购自Eppendorf公司;微量紫外分光度计(NanoDrop 2000)购自Thermo Fisher Scientific公司;电泳仪(DYY-6C)购自北京六一生物科技有限公司。
1.2 方法
1.2.1 引物设计、合成及反转录 参考GenBank中公布的牛(Bostaurus)POLB基因序列(登录号:NC_037354.1),利用Oligo 7.0设计普通PCR引物。上游引物序列:5′-ATGAGCAAACGG-AAGGCGCCGCAGGAGA-3′;下游引物序列:5′-GTGAGCCCAAGGACCGAAGCGAG-3′。引物由生工生物工程(上海)股份有限公司合成。用TRIzol提取郏县红牛背最长肌组织总RNA,利用微量紫外分光光度计检测RNA质量,要求A260 nm/A280 nm值介于1.8~2.0、A260 nm/A230 nm值介于2.0~2.3,-80 ℃保存备用。利用反转录试剂盒将RNA反转录为cDNA,-20 ℃保存备用。
1.2.2 PCR扩增及克隆 以郏县红牛背最长肌cDNA作为模板进行PCR扩增,PCR反应体系50 μL:上、下游引物各1.5 μL,KOD OneTMPCR Master Mix-Blue 25 μL,cDNA 1 μL,ddH2O 21 μL。PCR反应程序:94 ℃预变性5 min;94 ℃变性15 s,61 ℃退火30 s,72 ℃延伸45 s,共35个循环;72 ℃延伸10 min;4 ℃保存。反应结束后将PCR产物进行1.5%琼脂糖凝胶电泳,胶回收目的片段后连接至pMD18-T载体,转化大肠杆菌DH5α感受态细胞,在LB固体培养基上涂板,37 ℃恒温培养箱中过夜培养(12 h),挑选阳性克隆送生工生物工程(上海)股份有限公司进行测序。
1.2.3 生物信息学分析 通过NCBI在线数据库查找基因序列和氨基酸序列信息;利用Mega 7.0软件和BLAST(http:∥blast.ncbi.nlm.nih.gov/Blast.cgi)程序进行遗传距离分析和系统进化树构建,各物种POLB蛋白具体登录信息见表1;利用ExPASy(http:∥web.expasy.org/tools)在线软件进行蛋白理化性质和疏水性分析;利用NetPhos 3.1 Server(http:∥www.cbs.dtu.dk/services/NetPhos)在线软件预测POLB蛋白磷酸化位点;利用TMHMM(http:∥www.cbs.dtu.dk/services/TMHMM)在线软件预测POLB蛋白跨膜结构域;利用SignaIP(http:∥www.cbs.dtu.dk/services/SignalP)在线软件分析POLB蛋白信号肽;利用SOPMA(http:∥npsa-prabi.ibcp.fr/NPSA/npsa_sopma.html)在线软件预测POLB蛋白二级结构;利用SWISS-MODE(swissmodel.expasy.org)在线软件预测POLB蛋白三级结构;利用SMART(smart.embl-heidelberg.de)在线软件分析POLB蛋白功能结构域;利用STRING(version11.string-db.org)在线软件分析POLB蛋白的互作蛋白(设定最小所需相互作用评分>0.4)。

表1 13个不同物种POLB蛋白登录信息
2 结 果
2.1 POLB基因克隆结果
以郏县红牛背最长肌cDNA作为模板进行PCR扩增,PCR产物经1.5%琼脂糖凝胶电泳检测结果显示,产物大小在1 008 bp左右(图1),与预期片段大小相符。单克隆抗体菌液目的条带单一且清晰,克隆测序得到的郏县红牛POLB基因CDS区长度为1 008 bp,测序结果与NCBI上POLB基因的序列相似性为100%。
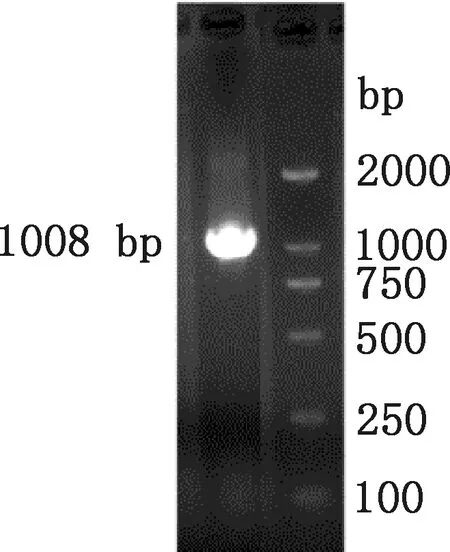
图1 郏县红牛POLB基因CDS区PCR扩增产物Fig.1 PCR products of CDS region of POLB gene in Jiaxian Red cattle
2.2 遗传距离分析及系统进化树构建
将郏县红牛POLB氨基酸序列与绵羊、中国水牛等12个物种的氨基酸序列进行遗传距离分析和系统进化树构建。结果显示,郏县红牛与绵羊等反刍动物的遗传距离最近,与其他哺乳类动物和禽类次之,与斑马鱼最远(表2);系统进化树分析结果显示,郏县红牛与绵羊等反刍动物的亲缘关系最近,与其他哺乳类动物和禽类次之,与斑马鱼(鱼类)最远(图2)。

表2 POLB基因氨基酸序列遗传距离分析
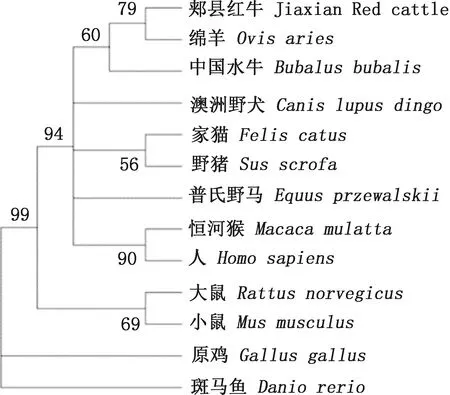
图2 POLB氨基酸序列系统进化树Fig.2 Phylogenetic tree of POLB amino acid sequences
2.3 理化性质分析
利用ProtParam在线软件预测郏县红牛POLB基因CDS区发现,其编码335个氨基酸,由20种氨基酸组成,其中赖氨酸(Lys)所占比例最大,为10.7%,色氨酸(Trp)所占比例最小为0.3%,不含吡咯赖氨酸(Ply)和硒半胱氨酸(Sec)(表3),负电荷残基总数天冬氨酸(Asp)+谷氨酸(Glu)为49,正电荷残基总数精氨酸(Arg)+赖氨酸(Lys)为57。 郏县红牛POLB蛋白分子式为C1699H2721N475O509S10,分子质量为38.26 ku,理论等电点(pI)为9.10,半衰期为30 h;肽链N-端为甲硫氨酸(Met),不稳定系数为31.06(<40),属于稳定蛋白,脂溶指数为78.30(<80),为非脂溶性蛋白。
2.4 疏水性预测
利用ProtScale在线软件对郏县红牛POLB蛋白进行疏水性分析发现,在第177位氨基酸处出现最高值1.400,在第403位氨基酸处出现最低值-3.278,总平均亲水系数为-0.659,为亲水性蛋白(图3)。
2.5 磷酸化位点预测
利用NetPhos 3.1 Server在线软件预测郏县红牛POLB蛋白磷酸化位点发现,POLB蛋白含17个丝氨酸(Ser)磷酸化位点、12个苏氨酸(Thr)磷酸化位点和4个酪氨酸(Tyr)磷酸化位点(图4)。
2.6 POLB蛋白跨膜区及信号肽预测
利用TMHMM在线软件对郏县红牛POLB蛋白跨膜区进行预测发现,POLB蛋白不存在跨膜螺旋结构(图5);采用SignalP在线软件预测信号肽发现,该蛋白没有信号肽(图6)。 表明郏县红牛POLB蛋白不属于跨膜蛋白和分泌型蛋白。
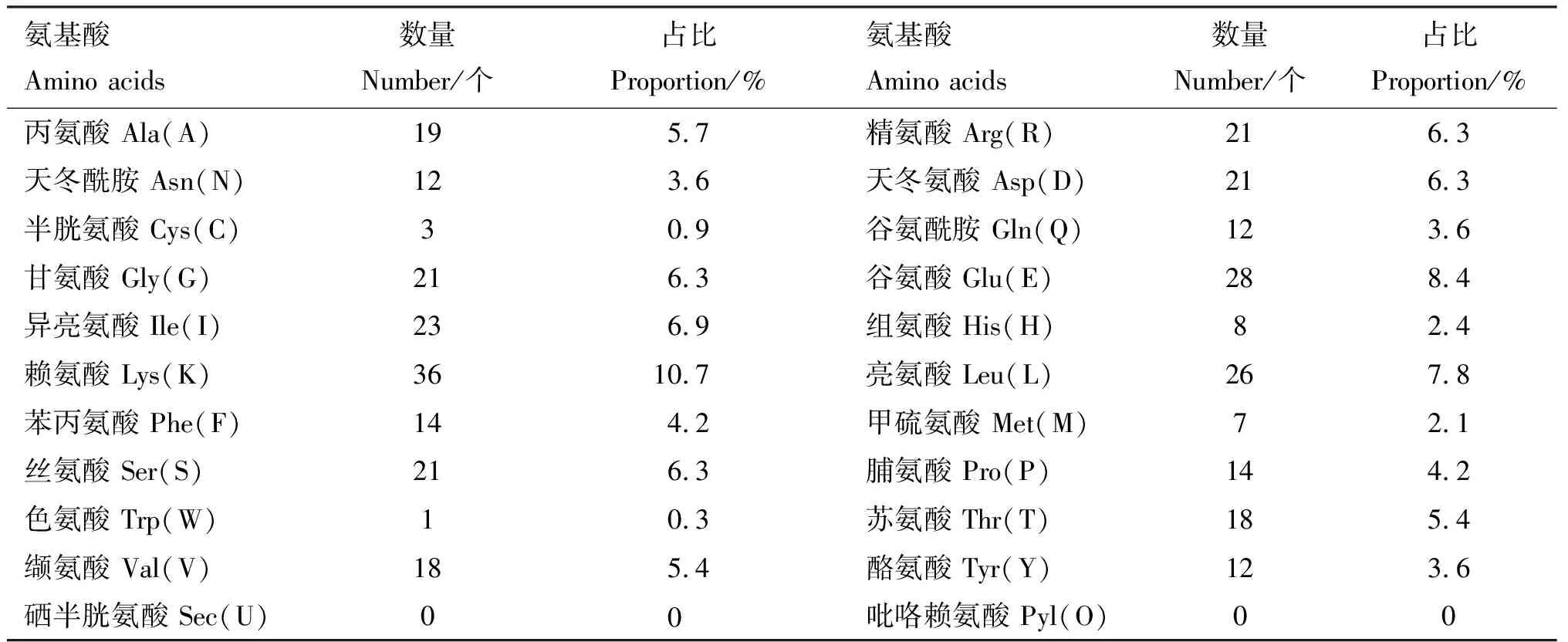
表3 郏县红牛POLB蛋白氨基酸组成
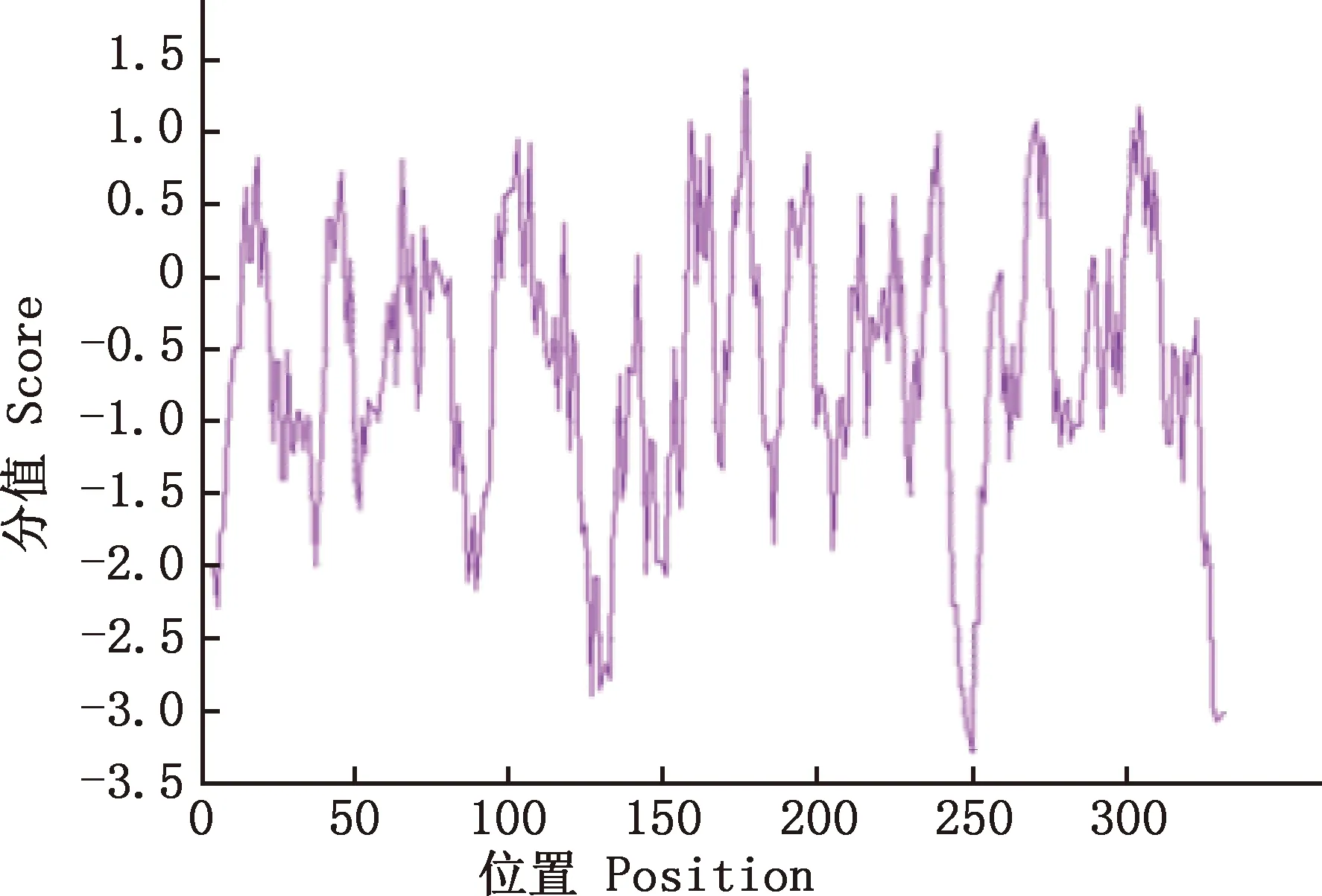
图3 郏县红牛POLB蛋白疏水性分析Fig.3 Hydrophobicity analysis of POLB protein in Jiaxian Red cattle
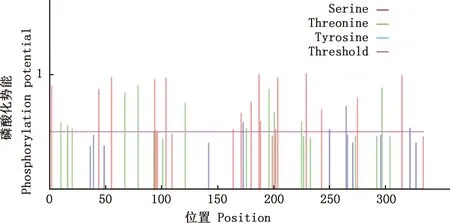
图4 郏县红牛POLB蛋白磷酸化位点预测Fig.4 Phosphorylation site prediction of POLB protein in Jiaxian Red cattle
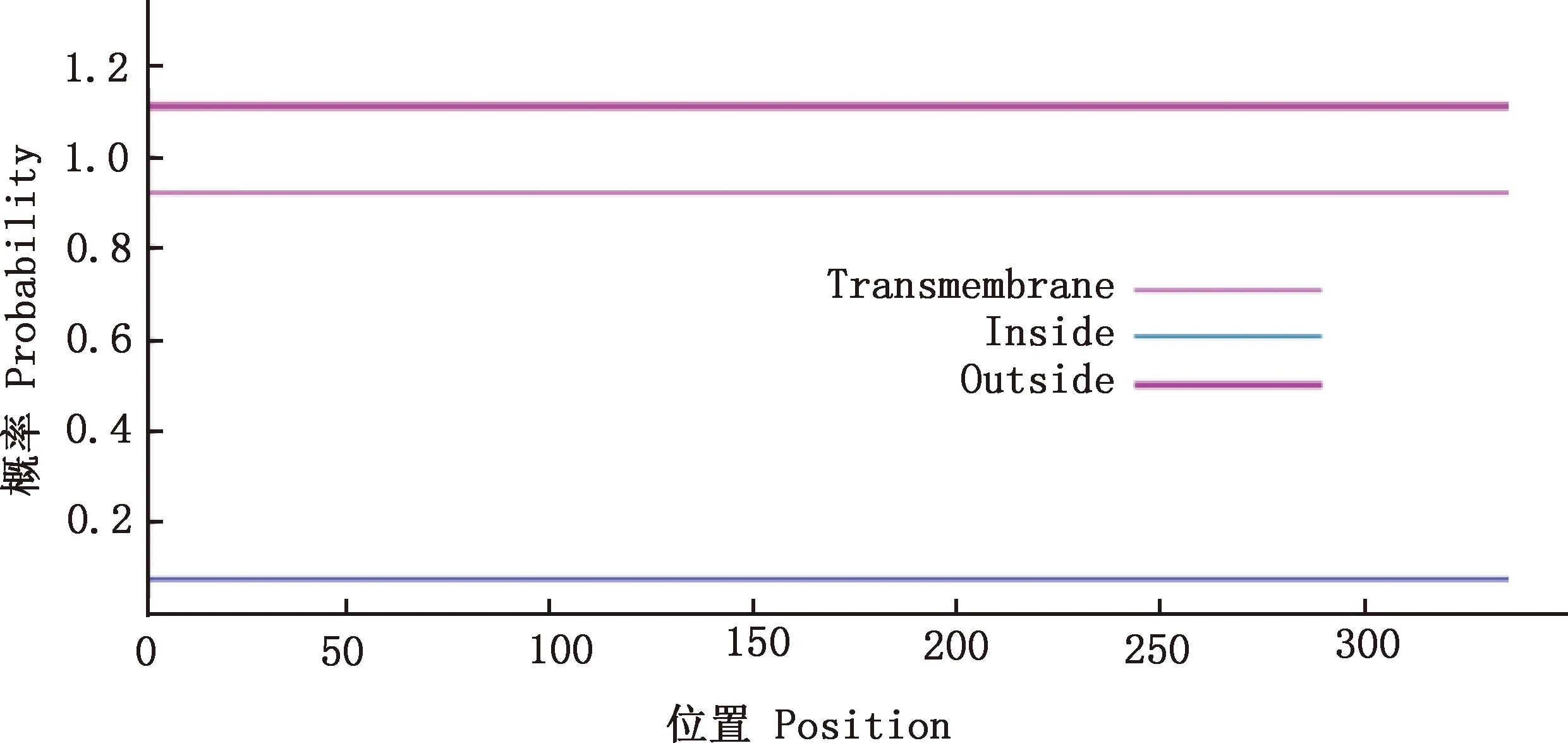
图5 郏县红牛POLB蛋白跨膜区预测Fig.5 Transmembrane structure prediction of POLB protein in Jiaxian Red cattle
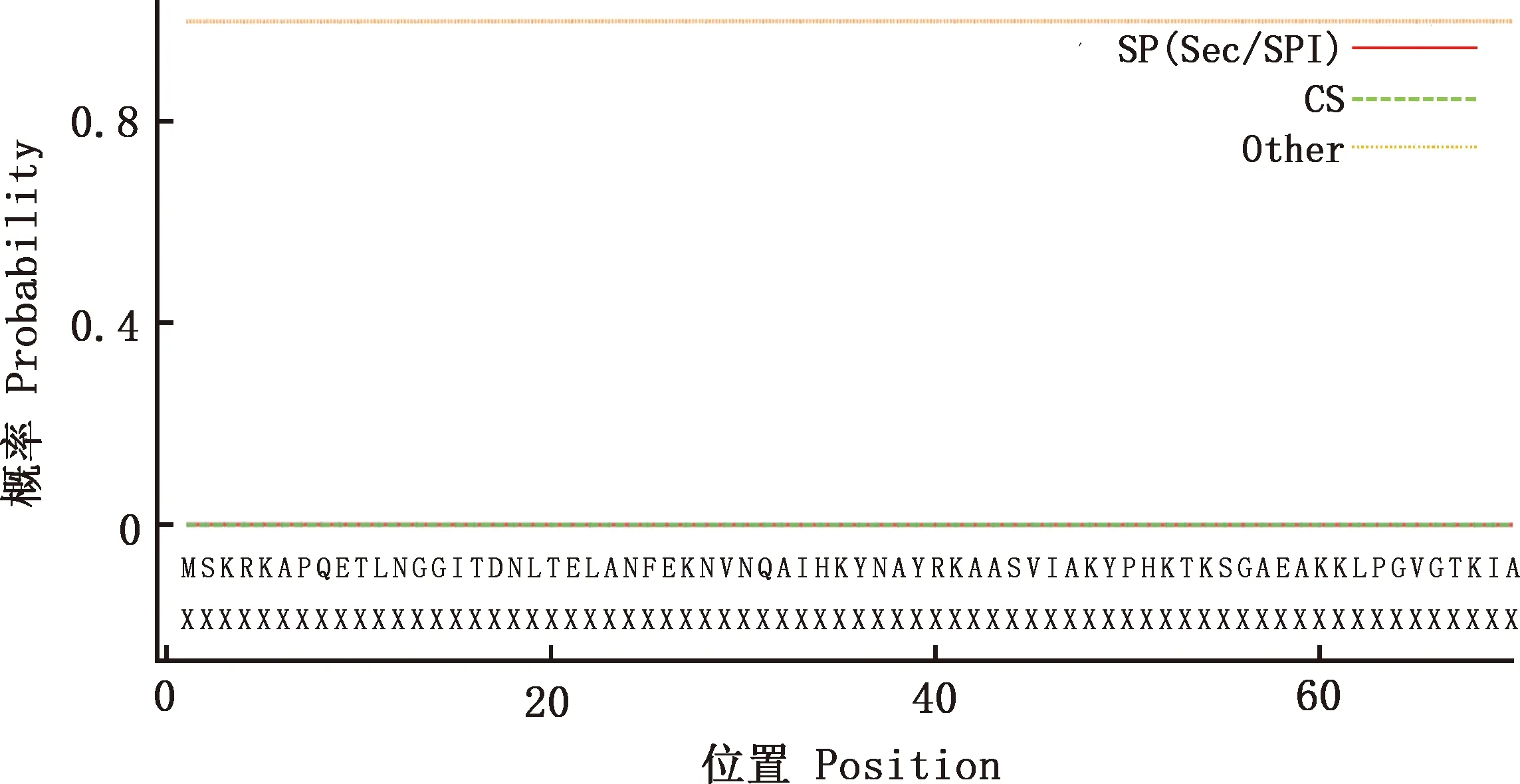
图6 郏县红牛POLB蛋白信号肽预测Fig.6 Signal peptide prediction of POLB protein in Jiaxian Red cattle
2.7 二级结构和三级结构预测
运用SOPMA在线软件对郏县红牛POLB蛋白二级结构进行预测,结果显示,POLB蛋白主要包含α-螺旋、β-转角、延伸链和无规则卷曲4种结构,分别占48.96%、2.99%、12.84%和35.22%(图7)。通过SWISS-MODE在线软件对郏县红牛POLB蛋白三级结构进行预测,结果显示,其与人POLB蛋白序列的一致性高达98.21%(>30%),预测结果可用;GMQE值为0.92,QMEAN4值为1.07,模型准确度可信(图8)。

①h,α-螺旋;t,β-转角;c,无规则卷曲;e,延伸链。②线条按照从长到短依次代表α-螺旋、延伸链、β-转角和无规则卷曲① h,Alpha helix;t,Beta turn;c,Random coil;e,Extended chain.②The lines represent alpha helix,extended chain,beta turn and random coil by the length,respectively图7 郏县红牛POLB蛋白二级结构预测Fig.7 Secondary structure prediction of POLB protein in Jiaxian Red cattle
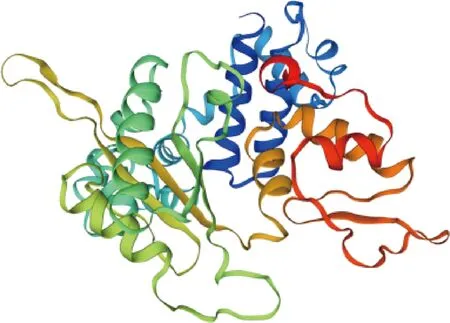
图8 郏县红牛POLB蛋白三级结预测Fig.8 Tertiary structure prediction of POLB protein in Jiaxian Red cattle
2.8 功能结构域预测及蛋白互作分析
运用SMART在线软件对郏县红牛POLB进行蛋白功能结构域预测分析,结果显示,POLB蛋白由335个氨基酸构成,包括2个螺旋-发卡-螺旋(helix-hairpin-helix,HhH)重复结构基序(图9)。利用STRING在线软件进行互作蛋白分析结果表明,X-射线修复交联-补充基因1(X-rayrepair cross-complementing gene 1,XRCC1)、瓣状核酸内切酶1(FEN1)、多聚ADP-核糖聚合酶1(poly ADP-ribose polymerase 1,PARP1)和DNA连接酶Ⅲ(LIG3)等与POLB蛋白具有强相关性(评分>0.900),其中与XRCC1蛋白互作程度最高,评分为0.995(图10)。

图9 郏县红牛POLB蛋白功能结构域预测Fig.9 Functional domain prediction of POLB protein in Jiaxian Red cattle
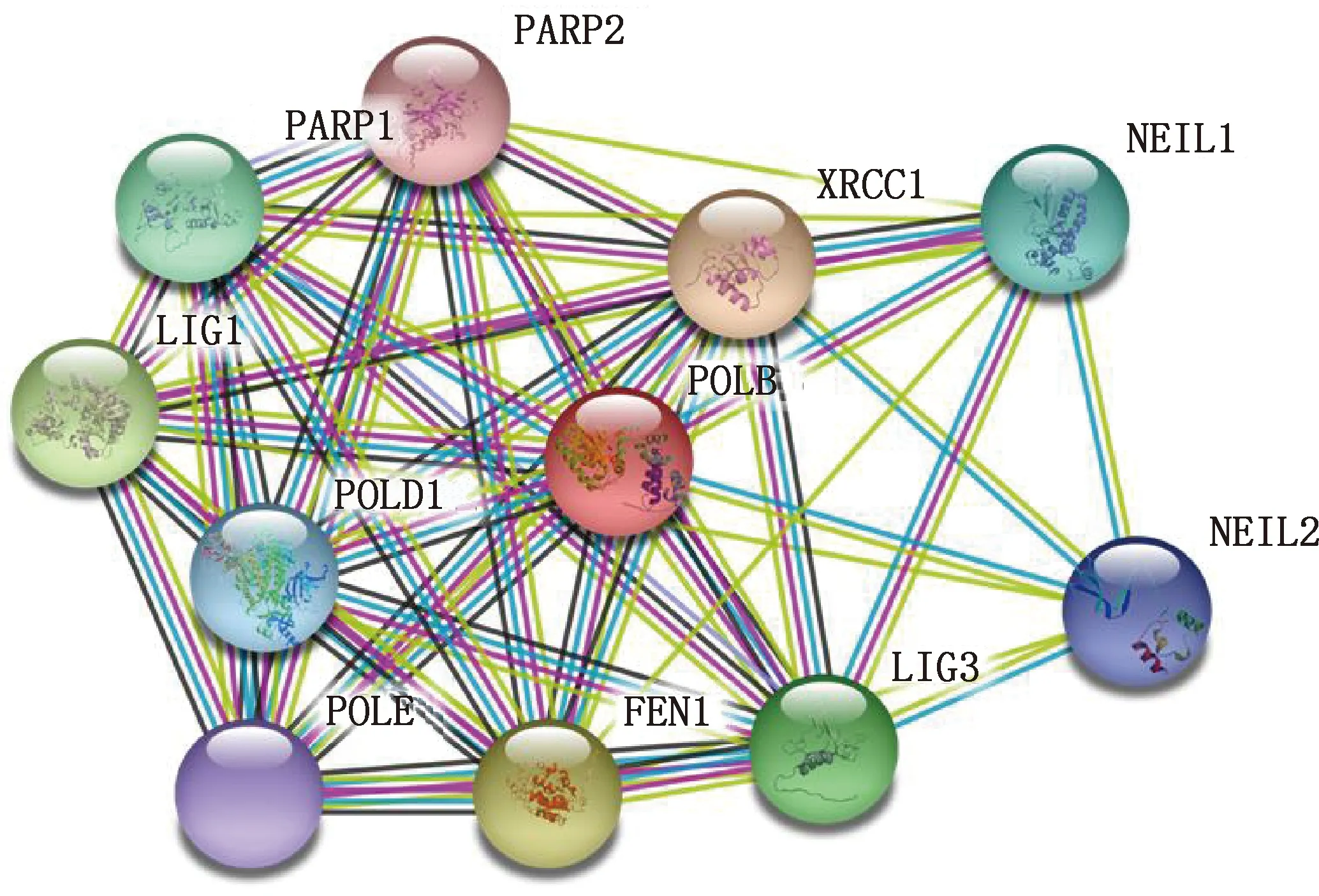
图10 与郏县红牛POLB蛋白互作的蛋白预测Fig.10 Protein prediction of interaction with POLB protein in Jiaxian Red cattle
3 讨 论
POLB基因所在的DNA修复基因与癌基因、抑癌基因、细胞周期调节基因以及细胞凋亡基因共同维持着细胞基因组的稳定性和完整性。POLB主要功能是通过参与碱基切除修复(base excision repair,BER)系统对DNA损伤进行修复。DNA损伤在细胞中如不能得到有效修复,易导致细胞的衰老、凋亡和癌变[15]。在小鼠食管癌细胞和人口腔鳞状细胞癌细胞中,沉默或降低POLB基因的表达可以调节细胞周期,导致有丝分裂异常,促进细胞增殖[16-17]。Baguma-Nibasheka等[18]研究发现,POLB基因在成肌调节因子(MyoD)基因缺失小鼠肌肉中比在正常小鼠显著高表达;POLB蛋白R137Q位点突变导致POLB结构变化并引起蛋白功能受损,可造成小鼠胎儿在15.5、17.5和19.5 d的体重显著降低[19],表明POLB基因可能参与肌肉的发育过程。
本研究克隆了郏县红牛POLB基因CDS区,结果显示,POLB基因蛋白编码区碱基序列长1 008 bp,共编码335个氨基酸,与人的POLB蛋白氨基酸组成一致[20]。系统进化树分析结果显示,郏县红牛与绵羊的亲缘关系最近,与哺乳动物的亲缘关系较鸡和斑马鱼更近,推测POLB基因在哺乳动物中可能具有相似的功能。Chang等[21]也研究发现POLB在哺乳类动物中的同源性以及在结构进化过程中相当保守,说明其在哺乳类动物中功能相似。理化性质预测结果显示,POLB蛋白不稳定系数为31.06,脂溶指数为78.30,亲水系数为―0.659,属于稳定的亲水性蛋白。其蛋白分子质量为38.26 ku,与人POLB蛋白的分子质量(39 ku)接近[7]。磷酸化位点预测结果表明,郏县红牛POLB蛋白具有33个磷酸化位点,其中17个为丝氨酸磷酸化位点和12个苏氨酸磷酸化位点。Tokui等[22]研究发现,POLB被蛋白激酶C磷酸化会造成POLB活性丧失。蛋白激酶C通过对蛋白质的苏氨酸和丝氨酸羟基进行磷酸化来调控蛋白活性[23],其是否影响牛POLB蛋白磷酸化需要进一步验证研究。对郏县红牛POLB蛋白信号肽的预测表明,POLB蛋白不是分泌蛋白。在细胞内定位研究发现,POLB蛋白主要在细胞核中发挥BER功能[24-25],这与本研究预测结果是一致的。
郏县红牛POLB蛋白结构预测结果显示,蛋白二级结构中α-螺旋和无规则卷曲所占比例最高,说明该结构为POLB蛋白的重要构件。蛋白三级结构预测显示,郏县红牛POLB蛋白与人POLB蛋白三级结构的相似度高达98.21%。Tanabe等[26]从多种哺乳类动物细胞中提取POLB蛋白并测定其分子结构,发现POLB蛋白的分子结构大致相同,表明POLB蛋白在哺乳动物中发挥可能类似生物学功能。郏县红牛POLB蛋白结构域预测结果发现,其包括2个螺旋-发卡-螺旋重复结构基序。Beard[27]结果表明,在人体中POLB蛋白的螺旋-发卡-螺旋结构是参与DNA作用的重要区域,表明POLB蛋白在哺乳动物中发挥类似的生物学功能。
有研究表明,在BER途径中XRCC1和PARP1这2种蛋白都可以与POLB蛋白结合[21]。POLB和XRCC1蛋白结合过程中,XRCC1还可与PARP1形成二聚体包裹DNA损伤的断裂位点,进而防止核酸酶消化,增强细胞抗凋亡的能力[28]。此外,在小鼠中敲除POLB和XRCC1基因均会导致胚胎死亡[29-30]。本研究对郏县红牛POLB蛋白的互作蛋白预测结果显示,XRCC1、FEN1、PARP1和LIG3与POLB蛋白具有强相关性,其中与XRCC1蛋白互作程度最高。因此,在深入研究牛POLB蛋白功能时应重视XRCC1蛋白的影响。
4 结 论
本研究成功克隆了郏县红牛POLB基因,其全长为1 008 bp,编码335个氨基酸,其氨基酸序列与绵羊等反刍动物的亲缘关系最近,与其他哺乳类动物和禽类次之,与斑马鱼(鱼类)最远;郏县红牛POLB蛋白稳定、不存在跨膜螺旋结构、没有信号肽;与XRCC1蛋白互作程度最高,结果可为进一步研究牛POLB基因的生物学功能与调控机理提供参考。

