格氏束腰蟹(Somanniathelphusa grayi)的线粒体基因组序列测定和基因顺序进化研究
杜诗雨,张康琴,潘 达,孙红英
(南京师范大学生命科学学院,江苏省生物多样性与生物技术重点实验室,江苏 南京 210023)
格氏束腰蟹(Somanniathelphusagrayi(Alcock,1909))(=Parathelphusa(Parathelphusa)chongiWu,1935)隶属于拟地蟹总科(Gecarcinudoidea Rathbun,1904),拟地蟹科(Gecarcinucidae Rathbun,1904),束腰蟹属(SomanniathelphusaBott,1968),喜生活在泥洞中[1-2]. 其最显著的特征为雄性腹部第五、六节呈束腰状[1].
近年来,高通量测序技术结合生物信息学注释分析的方法已广泛应用于动物线粒体基因组的测定[3-7]. 由于线粒体基因组具有基因组小、大多为母系遗传、进化速率快以及缺少内含子等特点,被广泛地应用于种群遗传、物种鉴定、分子进化和不同分类水平上的系统发生关系等研究[8]. 此外,由于线粒体基因功能上受约束,在DNA序列进化上相对保守,因此在基因的排列和DNA序列进化两个层面上都可以为动物的系统进化研究提供丰富的信息[9].
迄今为止,已知的拟地蟹科淡水蟹类的线粒体基因组仅束腰蟹属两种:波阳束腰蟹(SomanniathelphusaboyangensisDai,Peng & Zhou,1994[17])和坝王束腰蟹(SomanniathelphusabawangensisDai & Xing,1994[14]),这两种线粒体基因组排列呈现出了相同的基因排列顺序[14]. 但是,现有线粒体基因组数据的匮乏,阻碍了拟地蟹科的分子进化和系统分类研究. 本文以格氏束腰蟹为研究对象,利用高通量测序技术首次测定了其线粒体基因组近全长序列,并通过序列比对和比较研究,解析了格氏束腰蟹线粒体基因组的结构特征和基因排列顺序. 该研究结果为束腰蟹属近缘物种的系统分类和扩散提供了新的分子依据.
1 材料与方法
1.1 样本采集和保存
格氏束腰蟹标本在2015年10月11日采于云南省玉溪市华宁县盘溪镇(103.0631°E,24.2019°N). 标本在采集并记录生境后立即保存于95%乙醇中. 根据《中国动物志》(束腹蟹科,溪蟹科)[1]在体式显微镜(Nikon SMZ645)下对标本进行分类鉴定. 标本保存在南京师范大学生命科学学院,江苏省生物多样性与生物技术重点实验室.
1.2 DNA提取,扩增及质量检测
将存放于95%乙醇中的样本取出,用已灭菌的镊子在无菌环境下取适量的鳃组织样本,浸泡于生理盐水中,每30 min更换一次生理盐水,进行脱醇处理. 使用动物基因组DNA提取试剂盒(通用型)(Generay Biotech)进行DNA的提取(具体操作步骤参照试剂盒说明书),获得总DNA样品. 使用琼脂糖凝胶电泳检测其完整性,并使用微量分光光度计检测其浓度及质量. 将质量达标后的DNA样品送至北京诺禾致源科技股份有限公司进行高通量测序.
1.3 序列组装和基因组注释
本文使用MitoZ v1.04[21]将测序结果(clean data)进行序列组装和注释. 随后根据目前NCBI已知的 2种束腰蟹(波阳束腰蟹和坝王束腰蟹)线粒体基因组注释结果进行手动校对,从而获得格氏束腰蟹的线粒体基因组全序列. 并将注释后的线粒体基因组序列使用MITOS Web Server(http://mitos.bioinf.uni-leipzig.de/index.py)进行再次识别确认.
1.4 线粒体基因组组成和结构特征分析
用MEGA v7.0[22]进行碱基组成、密码子使用情况和蛋白编码基因同义密码子相对使用率(Relative Synonymous Condon Usage,RSCU)的计算. 利用通用公式:AT-skew=(A-T)/(A+T),GC-skew=(G-C)/(G+C)进行碱基偏斜率的计算[23]. 使用在线MITOS Web Server对所有物种的tRNA基因二级结构进行预测. 使用VARNA v3-93[24]和Rnaviz 2.0.3[25]对预测的tRNA基因二级结构进行绘制,并使用Adobe Photoshop CS 6.0进行最终的修饰与美化.

图1 格氏束腰蟹线粒体基因组结构图Fig.1 Mitogenomic map of Somanniathelphusa grayi
2 结果与讨论
2.1 线粒体基因组结构特征
格氏束腰蟹线粒体基因组为长度17 654 bp的未闭合环状DNA(图1),其序列已提交至GenBank,检索号为OM214524. 该线粒体基因组共有37个基因:13个蛋白质编码基因(PCGs),22个转运RNA基因(tRNA gene)和2个核糖体RNA基因(rRNA gene). 由于主非编码区(main non-coding region,mNCR)序列有着冗长的AT重复,对该区段DNA序列的拼接造成干扰. 这种情况在节肢动物线粒体基因组中比较常见. 本文该种的mNCR所在片段区域在测定时同样遇到此类情况,导致未能被完整测序. 有23个基因由正链编码:9个PCGs(atp6,atp8,cox1,cox2,cox3,cob,nad2,nad3和nad6)和14个tRNA基因(trnI,trnM,trnW,trnL2,trnD,trnK,trnG,trnA,trnR,trnN,trnS1,trnE,trnT和trnS2);其余14个基因由反链编码:4个PCGs(nad1,nad4,nad4L和nad5),8个tRNA基因(trnQ,trnC,trnY,trnF,trnH,trnP,trnL1和trnV)和2个rRNA基因(rrnL和rrnS)(表1),这与泛甲壳类和短尾下目线粒体基因组祖先型的编码方向一致[19,26]. 该线粒体基因组碱基组成AT含量高达72.8%(35.1% A,18.4% C,8.8% G,37.7% T,表2),其AT偏斜率为-0.0357,GC偏斜率为-0.3529(表2).
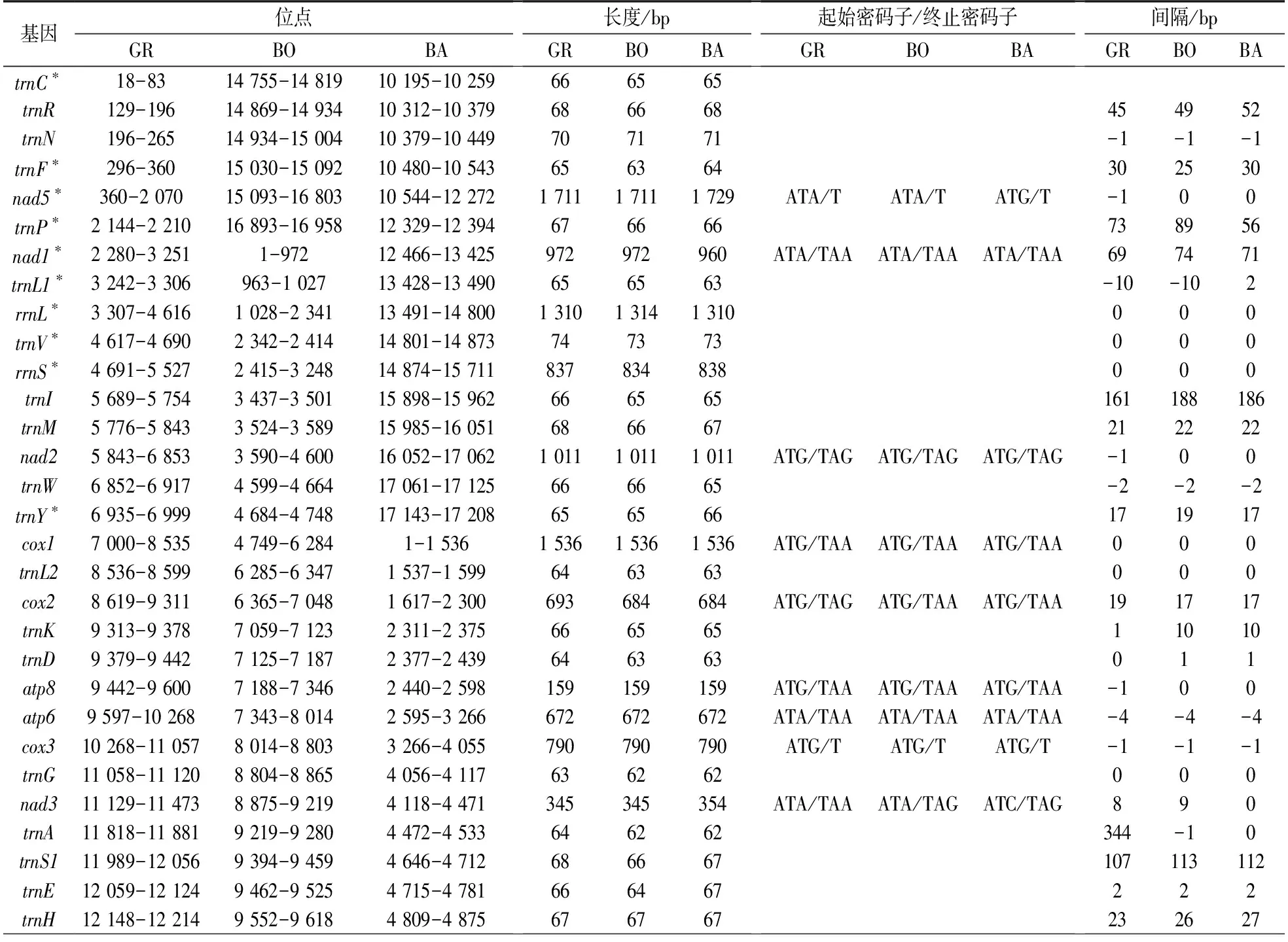
表1 格氏束腰蟹线粒体基因组特征及波阳束腰蟹和坝王束腰蟹线粒体基因组特征Table 1 Mitogenomic features of Somanniathelphusa grayi,S.boyangensis and S.bawangensis
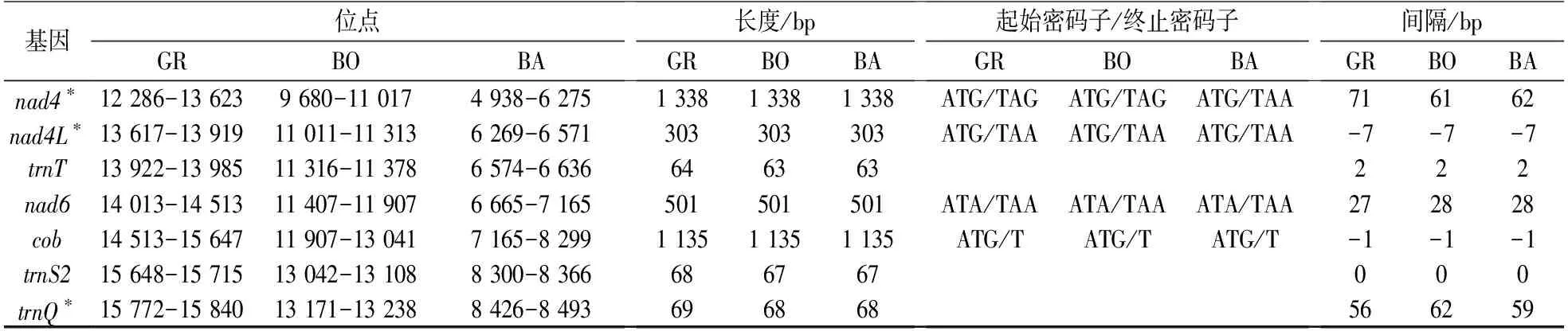
续表1 Table 1 continued
在格氏束腰蟹线粒体基因组中,共存在11处重叠区,其长度范围在1 bp~10 bp(表1);共存在18处间隔区,其长度范围在1 bp~344 bp,其中最长的间隔区位于nad3和trnA之间(表1),而在已公布的波阳束腰蟹和坝王束腰蟹线粒体基因组中,其最长间隔区均位于rrnS和trnI之间,长度分别为188 bp和 186 bp(表1). 由于每个线粒体基因组序列具有不等的基因间隔非编码序列(intergenic noncoding sequences,IGNs),在十足目线粒体基因组中也经常发现基因重叠和非编码区[27-29],淡水蟹即是如此. 一些重叠区在淡水蟹类线粒体基因组中是相对保守的,这些重叠区也存在于格氏束腰蟹线粒体基因组中,例如,atp8和atp6之间的重叠序列为ATAA;nad4和nad4L之间的重叠序列为ATGTTAA[7,10-11,14,20].
2.2 蛋白基因
在格氏束腰蟹的13个蛋白质编码基因中,所有蛋白质编码基因均为典型的ATN起始密码子,其中,cox1,cox2,cox3,atp8,nad2,nad4,nad4L和cob的起始密码子为ATG;atp6,nad1,nad3,nad5和nad6的起始密码子为ATA. 与已有的两种束腰蟹线粒体基因组序列相比,除坝王束腰蟹的nad3为ATC,nad5为ATG外,其余基因起始密码子均相同(表1). 而终止密码子中,除nad5和cox3的终止密码子为不完整的TA,cob的终止密码子为不完整的T外,其余10个蛋白质编码基因均为完整的终止密码子(TAA或TAG),和已知的2条束腰蟹线粒体情况相同(表1). 蛋白质编码基因的起始密码子通常是ATN,但终止密码子主要是TAA,而TAG、TA和T很少,这种现象也发生在其他短尾下目物种中[7,14,17,28-30].
在13个蛋白质编码基因中,atp8的A+T含量最高,为76.9%;cox1的A+T含量最低,为65.2%(表2). 位于正链上的蛋白质编码基因发生了T偏斜(AT偏斜率为-0.157 0)和C偏斜(GC偏斜率为-0.279 7);而位于反链上的蛋白质编码基因发生了T偏斜(AT偏斜率为-0.199 4)和较明显的G偏斜(GC偏斜率为0.412 2)(表2).
RFID防伪平台负责对商品上的RFID电子标签进行防伪溯源信息(生产日期、物流信息等)的写入并上传至溯源信息管理平台。为了实现溯源这项功能, 采集成功的RFID标签数据还需要进行相应的处理和数据存储。防伪平台的硬件结构如图4所示:
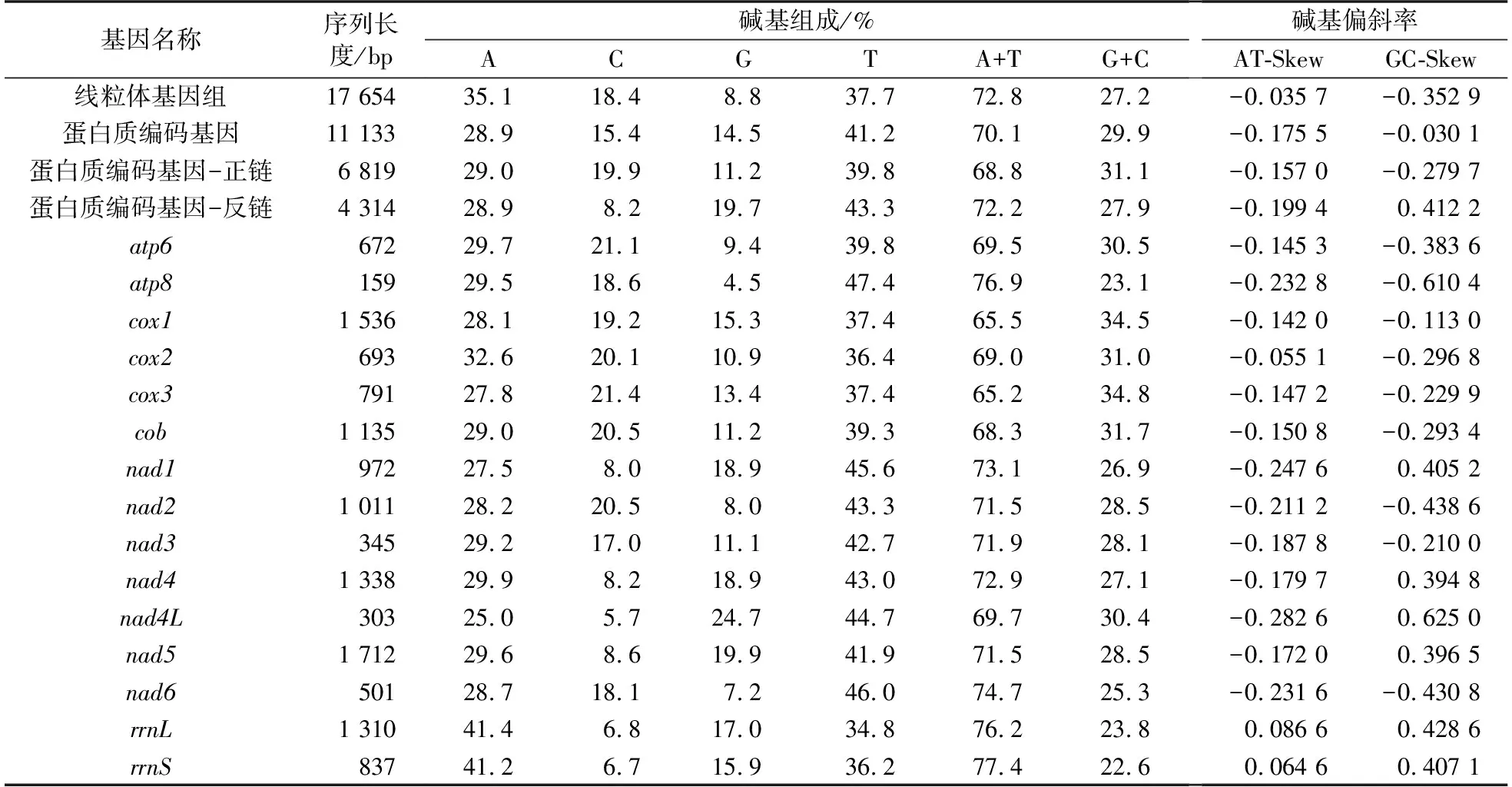
表2 格氏束腰蟹线粒体基因组碱基组成Table 2 Nucleotide composition of mitochondrial genome of Somanniathelphusa grayi
格氏束腰蟹的密码子使用总数为3 711次,其中使用频率最高的密码子是亮氨酸(UUA-Leu)(共被使用351次,RSCU=3.53). 蛋白编码基因的同义密码子相对使用率情况如图2所示. 其中,亮氨酸Leu(595次)、苯丙氨酸Phe(355次)和异亮氨酸Ile(336次)是使用最频繁的3个氨基酸(图2),约占34.65%.

图2 格氏束腰蟹、波阳束腰蟹和坝王束腰蟹线粒体基因组蛋白质编码基因同义密码子相对使用率Fig.2 The relative synonymous codon usage(RSCU)of 13 PCGs in Somanniathelphusa grayi,S.boyangensis and S.bawangensis
2.3 tRNA基因和rRNA基因
格氏束腰蟹的22个tRNA基因的二级结构图如图3所示. 除了trnS1-AGN缺少一个稳固的双氢尿嘧啶(DHU)臂,其余所有tRNA基因的二级结构均为典型的三叶草结构,即拥有4个恒定臂. 其中trnS1-AGN二级结构DHU臂的缺失在大部分后生动物中均有报道[31-32],且在很多短尾下目物种中也发现了这种情况[7,17-18,20].
22个tRNA基因的长度范围为63 bp~74 bp(表1),由于各可变环长度有所不同,以及DHU臂和TΨC臂的长度变化使得各tRNA基因的长度有所差异[33]. 其中,15种tRNA基因(trnA,trnR,trnD,trnC,trnQ,trnE,trnG,trnH,trnK,trnF,trnP,trnS1,trnS2,trnY,trnV)共存在30对GU碱基错配(图3),多数发生在DHU臂,氨基酸接受(AA)臂和反密码子(AC)臂,较少数发生在TΨC臂,trnC和trnQ中的错配数最多,均为 4对(图3).
2个rRNA基因,即rrnL和rrnS,分别位于trnL1和trnV,trnV和trnI之间(表1).rrnL和rrnS的长度分别为1 310 bp和837 bp,其AT含量分别为76.2%和77.4%(表1).

图3 格氏束腰蟹22个tRNA基因二级结构图Fig.3 Secondary structures of 22 tRNA genes from Somanniathelphusa grayi
2.4 线粒体基因组重排分析
与短尾下目祖先型线粒体排列顺序(brachyuran ground-pattern mitochondrial gene order,BMGO)相比较发现,格氏束腰蟹的线粒体排列顺序发生了较明显的变化. Jia等[17]在研究波阳束腰蟹的线粒体基因组排列顺序时,认为mNCR位于rrnS与trnI之间,长度为188 bp(表1),AT含量为87.8%. 而目前已知的海洋类短尾下目物种的mNCR长度范围在332 bp~2 140 bp[14]. 因此,Zhang等在分析坝王束腰蟹和波阳束腰蟹的线粒体基因组后,通过序列长度,AT含量和串联重复元件等综合比较发现,认为坝王束腰蟹和波阳束腰蟹的mNCR均应位于trnC与trnQ之间[14],修正了波阳束腰蟹的线粒体基因排列顺序,认为波阳束腰蟹与坝王束腰蟹拥有相同的基因排列顺序[14]. 而本文所研究的格氏束腰蟹线粒体基因组排列顺序与波阳束腰蟹和坝王束腰蟹的线粒体基因排列顺序均相同.
迄今为止,约有55.2%的已知的短尾下目物种发生线粒体基因组重排情况. 格氏束腰蟹与此前所报道的2种束腰蟹的基因排列顺序一致,即拟地蟹线粒体基因组排列类型I(gecarcinucid mitochondrial gene order pattern I,GMGO1)(图4),该重排类型涉及6个tRNA基因(trnR,trnN,trnF,trnP,trnQ和trnC),1个PCG(nad5)和1个mNCR的排列顺序变化(图4). 这些基因的重排可以用置换[34]和复制/随机丢失(TDRL)[35]模型进行解释. 本文通过以下3个假设步骤解释了GMGO1的形成过程:首先,mNCR从[rrnS-trnI]基因簇中移出(图5a),然后移入[trnW-trnC]基因簇中(图5 b);其次,基因簇[trnR-trnN-trnS1-trnE-trnH-trnF-nad5-nad4-nad4L-trnT-trnP-nad6-cob-trnS2-nad1-trnL1-rrnL-trnV-rrnS-mNCR-trnI-trnQ-trnM-nad2-trnW-trnC](图5c)被串联复制并生成两组相同的基因簇[trnR-trnN-trnS1-trnE-trnH-trnF-nad5-nad4-nad4L-trnT-trnP-nad6-cob-trnS2-nad1-trnL1-rrnL-trnV-rrnS-mNCR-trnI-trnQ-trnM-nad2-trnW-trnC-trnR-trnN-trnS1-trnE-trnH-trnF-nad5-nad4-nad4L-trnT-trnP-nad6-cob-trnS2-nad1-trnL1-rrnL-trnV-rrnS-mNCR-trnI-trnQ-trnM-nad2-trnW-trnC](图5d);最后,串联复制后生成的基因簇在随机丢失26个基因之后,形成了目前已知的线粒体基因重排类型,GMGO1.

BMGO:短尾下目祖先型线粒体基因排列顺序;GMGO1:拟地蟹线粒体基因组排列顺序;箭头显示重新排列的基因或基因块图4 BMGO和GMGO1两种基因排列顺序的比较Fig.4 Comparison of gene arrangements of BMGO and GMGO1
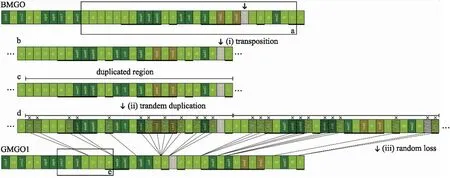
(a)BMGO基因排列顺序;(b)mNCR的异位;(c)重复基因区域;(d)26个基因的串联重复(16个tRNA基因,7个PCGs,2个rRNA基因和1和mNCR):trnR/trnN/trnF/nad5/trnP/nad1/trnL1/rrnL/trnV/rrnS/trnI/trnM/nad2/trnW/trnS1/trnE/trnH/nad4/nad4L/trnT/nad6/cob/trnS2/trnQ/mNCR/trnC;且复制的26个基因随机丢失形成新的基因顺序;(e)GMGO1基因顺序图5 串联重复/随机丢失和重复/反密码子突变假设过程Fig.5 The hypothetical process of gene rearrangement in the model of tandem duplication/random loss and duplication/anticodon mutation
2.5 线粒体基因顺序重排的进化含义
对十足目动物的线粒体基因组研究发现,线粒体基因组的排列顺序可能与生态环境转变之间有着重要的联系[12,36]. 线粒体基因组的排列顺序可能会在适应新环境时发生变化. 例如柯氏绒铠虾(ShinkaiacrosnieriBaba & Williams,1998)和汤花深白蟹(Gandalfusyunohana)(Takeda,Hashimoto & Ohta,2000)这两类适应极高温环境的十足类,其线粒体基因排列顺序发生了变化[37-38];一种适应从水生过渡到洞栖陆生生活的一种滑鳌虾(Cheraxdestructor(Clark,1936)),其线粒体排列顺序也发生了变化[3];中华绒螯蟹(EriocheirjaponicasinensisMilne-Edwards,1853)的线粒体基因组基因的加速重排可能提示适应非海洋生活过程中的快速辐射进化[28]. 本文发现3种束腰蟹类的线粒体基因组重排变化较大,共有1个蛋白质编码基因和6个tRNA基因发生重排,这是否与它们对淡水环境的适应性进化有关,还需要通过更多相关类群的线粒体基因组数据进行分子进化研究.
另外,在溪蟹科淡水蟹类发现了线粒体基因组的高度重排现象[14],但是在拟地蟹科中,是否也会出现与这些已知的重排类型类似或者新的重排方式,这些都还有待进一步扩大拟地蟹科类群的取样,获得必要的线粒体基因组数据,从而挖掘蕴含在线粒体基因组中的进化信息,并探索线粒体基因重排在拟地蟹科类群中的进化含义.
2.6 束腰蟹线粒体基因顺序在其分类和分布的意义
格氏束腰蟹的主要鉴别特征为背部颈沟较为明显,且雄性腹部呈明显的“沙漏形”(hourglass-shaped)[39]. Alcock[39]首次描述了格氏束腹蟹(Parathelphusa(Parathelphusa)grayi),模式标本采自印度Moung Sal. 伍献文[40]描述了常氏束腹蟹(Parathelphusa(Parathelphusa)chongiWu,1935),模式标本采自中国云南,该物种系首个由中国学者发现和描述的中国淡水蟹类物种[1,41]. 随后,这两个物种均被厘定至束腰蟹属(SomanniathelphusaBott,1968[42]),即格氏束腰蟹(Somanniathelphusagrayi(Alcock,1909))和常氏束腰蟹(Somanniathelphusachongi(Wu,1935)). 戴爱云等[43]描述了中华束腰蟹常氏亚种(SomanniathelphusasinensischongiDai,Feng,Chen,Song,1984),模式标本采自中国云南. 随后,戴爱云认为中华束腰蟹常氏亚种(S.sinensischongi)是常氏束腰蟹(S.changi)的同物异名[1],并将该种归并于常氏束腰蟹S.chongi. Ng等梳理了世界短尾类分类系统,认为常氏束腹蟹P.(P.)chongi(即常氏束腰蟹S.chongi)是格氏束腰蟹S.grayi的同物异名[2],并将该种归并于格氏束腰蟹S.grayi.值得注意的是,常氏束腰蟹分布在印度MoungSai,而格氏束腰蟹S.grayi分布在我国云南,地理相隔较远,很可能不是同一物种.束腰蟹S.Chongi属种间雄性腹肢整体形态极为相似,形态特征差异不明显,分类鉴定较为困难,加上缺乏对格氏束腰蟹S.grayi正模的详细形态描述[39],因此常氏束腹蟹P.(P.)chongi和格氏束腰蟹S.grayi的分类厘定还有待进一步的研究.
线粒体基因排列顺序可以作为特定谱系和分类群的共衍生性状,并为系统发生和进化关系提供了重要的分子证据[44].trnH基因从nad5-nad4基因块易位到nad3-nad5之间被认为是短尾下目所共享的特征[28],本文所研究的格氏束腰蟹和已知的两种束腰蟹均存在trnH基因的易位现象[14,17]. Zhang等在对溪蟹科淡水蟹的线粒体基因排列顺序的进化历程分析时发现,小华溪蟹(SinopotamonparvumDai,Song,Li,Chen,Wang & Hu,1985)与小石蟹属(TenuilapotamonDai,Song,Li,Chen,Wang & Hu,1984)物种共享一致的重排类型,且亲缘关系较近,认为小华溪蟹的分类地位应重新厘订[14]. 已知动物线粒体基因组的基因排列顺序相对保守[44-46],因此不同的类群共享相同或相似的线粒体基因排列顺序可能是来源于共同祖先的结果[14]. 本研究发现,目前已有线粒体基因组序列的三种束腰蟹分别分布在我国云南(普文、芒掌、思茅、芒洪、景洪、勐洪、小勐养、曼尾、勐罕、勐养、勐海、麻栗坪)、江西(波阳、都昌、沿山)和海南岛(坝王岭、儋县、黎母山、白沙)[1]. 虽然这3个物种呈异域分布,其分布区之间相隔较远,但线粒体基因排列顺序相一致,提示它们是由共同的祖先进化而来. 对于常氏束腰蟹与格氏束腰蟹的分类关系,线粒体基因组信息(包括DNA序列和基因排列顺序)或可以提供新的证据. 因此,亟需获取分布于印度格氏束腰蟹的线粒体基因组数据做进一步验证.
3 结论
本文首次测定了格氏束腰蟹的线粒体基因组近全长序列,确定其为长度17 654 bp的未闭合环状DNA. 与短尾类祖先类型线粒体基因组比较,格氏束腰蟹线粒体基因组基因顺序与已公布的两种束腰蟹线粒体基因顺序变化一致,共涉及7个基因的重排(1个蛋白质编码基因和6个tRNA基因). 三种在我国云南、江西和海南岛异域分布的束腰蟹种类所共享的线粒体基因排列顺序是该类群重要的共近裔特征之一. 该研究为束腰蟹线粒体基因顺序在其分类和分布方面提供了借鉴,也为拟地蟹科淡水蟹类线粒体基因组信息提供了新的分子证据.

