耐盐/嗜盐菌对PAHs的降解及分子机制研究综述
范韦华, 王重阳
(河南大学迈阿密学院, 河南 开封 475004)
0 引言
多环芳烃 (Polycyclic Aromatic Hydrocarbons,PAHs)是指分子中含有2 个或2 个以上并环苯环结构的持久性有机污染物。由于其在环境中广泛分布,难以降解,并具有致癌、致畸、致突变的“三致”特性,严重威胁着生态环境的稳定性和人类的生命健康安全[1],已引起各方面的广泛关注。2005年4月至2013年12月《全国土壤污染状况调查公报》数据表明,我国受PAHs 污染土壤的点位超标率为1.4%,PAHs污染广泛分布于耕地、工业废弃地、工业园区、采油区、采矿区、污水灌溉区、干线公路两侧等地。
目前对PAHs 的降解方法主要有化学氧化法、光催化氧化法、化学淋洗法、微生物修复技术、热修复技术、电动修复技术、植物修复技术等[2]。由于微生物修复技术具有经济且环境友好等特点, 因此在处理有机污染物方面具有明显的优势[3]。 然而,盐环境自身具有以下的特殊性:①渗透压增加,容易破坏细胞膜;②降低溶解氧含量;③盐析作用使得PAHs 的溶解性降低,继而进一步降低生物可利用性;④盐析作用容易导致酶,尤其是胞外酶的变性[4]。 这些性质使得具有良好PAHs 降解能力的普通微生物无法在盐环境中发挥功能。 耐盐/嗜盐菌具有较好的盐度耐受性,在盐环境中可发挥良好的降解作用,从而达到去除PAHs 的目的,因此,对此类微生物资源的深入研究将为盐环境中PAHs 的降解起到技术支撑。
1 嗜盐PAHs 降解菌
近代工业的发展使石油得到了越来越广泛的应用,PAHs 是石油中重要的持久性有机污染物之一。 因此, 随着石油的开采、运输和使用,大量的PAHs 被排放进入环境中,造成了严重的危害[5]。 生物降解为去除环境中的PAHs 提供了一种重要的手段,早在1950年,有研究[6]发现菌属Pseudomonas中的细菌可在好氧条件下降解PAHs。 1976年KIYOHARA H 等[7]在土壤中分离得到可降解菲的菌属Aeromonas。 1989年HEITKAMP M A 等[8]在油田的岩石中分离出可降解芘的菌属Mycobacterium。 截至2018年, 共发现了100 余属的细菌具有降解PAHs 的功能, 其中最典型的为Pseudomonas,Mycobacterium,Rhodoccus 等[9]。绝大多数PAHs 降解菌均为普通菌,缺乏在盐环境中降解PAHs 的能力。嗜盐菌可耐受较为广泛的盐度变化, 且具有营养需求简单、代谢能力强等特点,被认为是盐环境中污染物修复的重要微生物资源。 但由于其筛选条件的苛刻,此类微生物资源一直较少。 近年来,随着对各种PAHs 降解菌的深入研究,分离出的嗜盐PAHs 降解菌株也越来越多。 如今分离筛选出可降解多环芳烃的嗜盐菌主要来自于菌属Cycloclasticus,Micrococcus,Pseudomonas putida,Alcaligenes,Haloarcula 等。典型的耐盐/嗜盐PAHs 降解菌见表1。
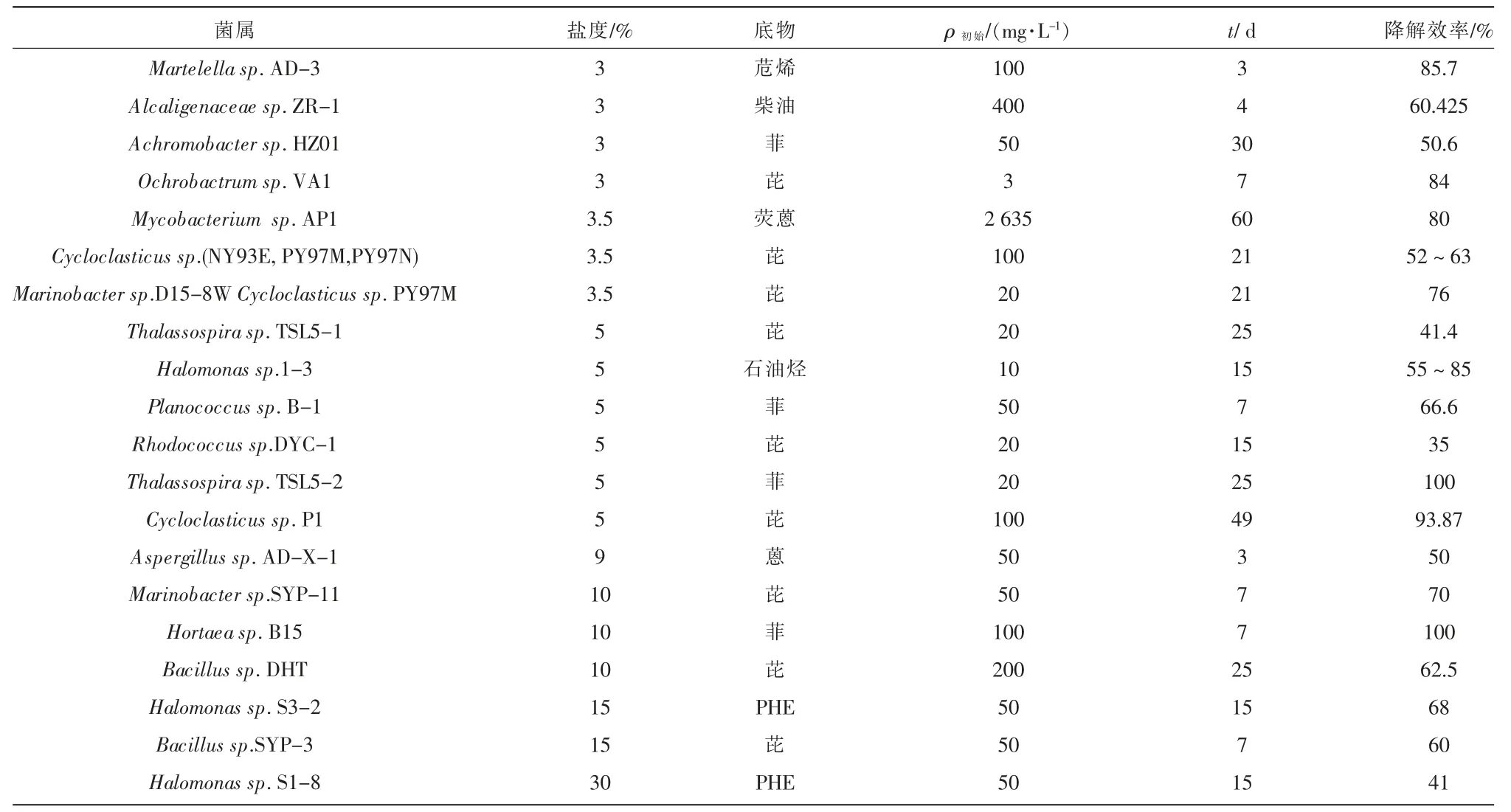
表1 典型的耐盐/嗜盐PAHs 降解菌
2 嗜盐PAHs 降解菌的代谢途径
目前, 对部分嗜盐菌中PAHs 降解途径的研究主要集中在真菌(Aspergillus,Penicillium 等)和细菌(Marinobacter,Cycloclasticus 等) 对PAHs 的好氧降解及少数嗜盐菌对PAHs 的厌氧降解。
2.1 好氧代谢途径
好氧降解指微生物在有氧条件下, 将氧气作为最终电子受体对污染物进行降解。 在PAHs 降解过程中, 真菌一般通过木质素酶系(木质素过氧化物酶、锰过氧化物酶和漆酶)和细胞色素P450 酶系(细胞色素P450、细胞色素b5、NADPH- 细胞色素P450还原酶、NADH-细胞色素b5 还原酶和磷脂) 降解PAHs[10]。 田晶[11]研究发现中度嗜盐菌Aspergillus flavus sp. AD-X-1 和Penicillium chrysogenum sp.APD-X-1 通过木质素酶系降解PAHs, 木质素酶系通过开环作用把PAHs 代谢成为水溶性较强的醌类中间产物后再由其进行后续降解。细胞色素P450 单加氧酶氧化PAHs 形成环氧化物后水解形成反式二氢二醇类衍生物参与接下来的降解反应, 或形成中间体与其他物质相结合,进行下一步的降解[12]。
由于降解PAHs 细菌种类更多, 故在降解多环芳烃的应用比真菌更为广泛。王重阳[9]研究发现嗜盐菌Marinobacter sp.N4 和Halomonas sp.G29 通过水杨酸途径实现对菲的联合降解。 于瑶瑶等[13]通过萃取中度嗜盐菌Demequina salsinemorus sp. BJ1 降解蒽的中间代谢产物进行质谱分析, 发现主要产物为9,10-蒽醌和邻苯二甲酸等, 说明其通过邻苯二甲酸途径降解蒽。 余志伟[14]采用Cycloclasticus sp.P1 通过2 次双加氧和开环生成1-羟基-2-萘甲酸后,经水杨酸途径降解芘。 嗜盐微生物对PAHs 的降解也可以同时存在水杨酸和邻苯二甲酸途径。周海燕[15]通过GC-MS 分析嗜盐菌Halassospira sp.TSL5-1 对芘的降解过程中发现同时存在水杨酸途径和邻苯二甲酸代谢途径。嗜盐菌对菲的降解途径及其中间产物,详见图1。
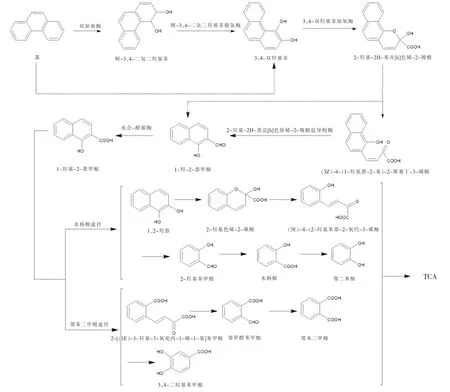
图1 嗜盐菌对菲的降解途径及其中间产物
2.2 厌氧代谢途径
大多数微生物需在有氧的条件下降解PAHs,但在江河湖泊的底泥和海底沉积物中, 研究发现了兼性厌氧PAHs 降解菌和严格的PAHs 降解菌。 如NADEZHDA F 等[16]从污水处理厂的污泥中分离出可在厌氧条件下降解PAHs 的菌属Bacillus cereus,SELESI 等[17]从河道底泥中筛选分离出可降解2-甲基萘的厌氧菌群Desulfobacterium sp.N47。厌氧降解途径主要利用无机分子作为最终电子受体降解转化PAHs,其主要反应涉及羟基化、甲基化、羧化反应、延胡索酸盐结合反应等[18]。 孙明明等[19]研究发现硫酸盐还原细菌降解菲的途径是通过菲分解成对甲酚后进行甲基羟基化, 生成对羟基苯甲醇或对羟基苯甲醛后再进一步转化为对羟基苯甲酸和酚, 最后发生脱羧反应后将菲彻底厌氧降解。
好氧降解不仅效率高而且速率快, 是高盐环境下治理PAHs 污染最有效的途径之一。 而厌氧降解因其中间代谢产物结构较为简单、毒性较小,也逐渐成为多环芳烃污染治理的有效途径之一。
3 耐盐/嗜盐PAHs 降解菌的降解机制
3.1 耐盐/嗜盐PAHs 降解菌降解机制
目前研究发现降解PAHs 的功能基因簇包括nah,phd,nag,phn,nar,nid 等6 类[20],大部分可降解多环芳烃的微生物均含有与它们具有很高同源性的基因簇,而在高盐环境下,部分基因簇控制的酶系活性较低,对于嗜盐菌而言,其降解PAHs 的功能基因簇主要为nah 基因簇和其他基因簇, 嗜盐PAHs 降解菌nah 降解基因簇排列顺序,见图2。
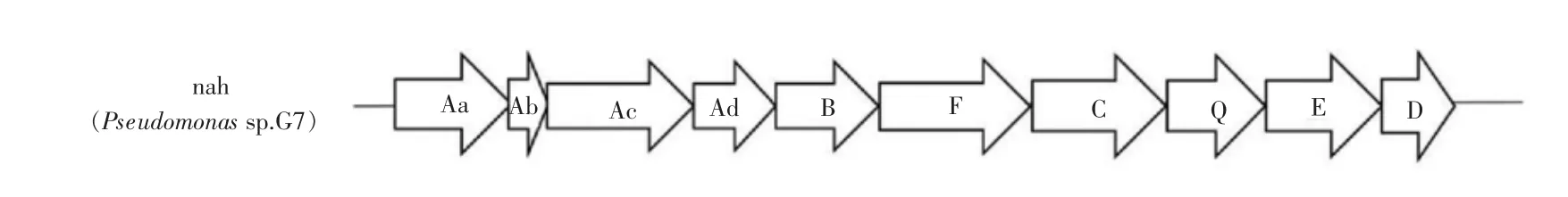
图2 嗜盐PAHs 降解菌nah 降解基因簇排列顺序
nah 基因簇是一大类降解基因簇, 目前可分离培养的降解菌株主要以nah 基因簇或经典的“nahlike”基因簇为主,如dox,pah,pdo 等。 nah/nah-like基因簇排列顺序为nahAaAbAcAdBFCQED, 大部分能降解PAHs 的基因排列顺序与组成与其相似。nah降解基因一般由3 个操纵子组成,nah1 编码的酶将萘转化成水杨酸,nah2 编码的酶通过间位裂解将水杨酸降解为中间代谢产物进入TCA 循环, 在nah1和nah2 之间,还存在一个调节基因nah3,该基因表达后的蛋白会促进nah1 和nah2 的表达。 如嗜盐菌群Sphingomonas CHY-1 中基因簇nahAaAbAcAdBFCED 将菲降解成为1,2-二羟基萘,再由基因簇nahGTHINLOMKJ 进一步进行降解,同时在nahR 调控基因下,菲完全降解[21]。 何芬等[22]通过分析嗜盐PAHs 降解菌群的6 种萘双加氧酶(ndo)基因型,发现其中3 种主要基因型(占总克隆子92.7%)与经典nah-like 基因的相似度为89%。 张心平等[23]通过对Pseudomonas sp. ND6 的降解质粒pND6 进行DNA杂交实验, 发现pND6 质粒含有与Pseudomonas sp.G7 菌株NAH7 质粒同源基因nahY。 王重阳对嗜盐菌Marinobacter sp. N4 中PAHs 上游降解基因簇Hpah1 的排列方式与Pseudomonas sp. G7 中PAHs降解基因簇nah1 进行相似性分析, 相似性达80%,其推测认为Marinobacter sp.N4 中的Hpah 与Burkholderiales 中的nag 降解基因可能存在水平转移,并由nag 降解基因在海洋盐环境中进化为耐盐的Hpah 降解基因簇[9]。
目前, 虽然对嗜盐PAHs 降解基因簇大部分由普通PAHs 降解基因进化而来, 但还存在其他降解基因簇与其差异性较大。 如Rhodococcus sp.OA1 降解基因簇pcaJIGHBARC,与传统基因簇调控的邻苯二酚和水杨酸降解途径不同, 其可通过原儿茶酸途径降解PAHs[24]。 耐盐菌Cycloclasticus sp.A5 功能基因簇phnA1bA1aA2CA4A3D 与普通PAHs 降解菌Burkholderia sp. RP007 中基因簇排列顺序phnRSFECDAcAdB 完全不同[25]。据此推测,嗜盐菌或具有与普通环境中PAHs 降解菌类似的降解基因簇,也可能具有与普通降解菌差异较大的基因簇,关于嗜盐PAHs 降解菌中的降解基因及系统发育关系,有待进一步研究。
3.2 耐盐/嗜盐PAHs 降解菌的耐盐机制
嗜盐菌的耐盐机制主要有3 个方面: ①吸钾排钠;②胞内积累小分子相容性溶质;③嗜盐酶的氨基酸组成特性[26],这3 个机制确保了嗜盐PAHs 降解菌在高盐环境有较好的盐度耐受性, 可高效降解PAHs。 其中吸钾排钠机制主要由nha H 基因、trk 类基因和Kdp 类基因调控。nha H 基因通过增强Na+/H+逆向转运蛋白的活性而减少嗜盐菌细胞体内的Na+浓度,trk 类基因和kdp 类基因通过调节K+/Cl-单向转运蛋白来提高细胞内K+浓度,从而维持在高盐环境下细胞内外的渗透压平衡。 高盐压力下嗜盐菌在胞内会积累小分子相容性溶质如糖类、氨基酸等,这些小分子物质既不影响细胞代谢还可提高胞内水活度,从而使内外渗透压达到平衡。 同时,嗜盐菌的蛋白质肽链发生折叠,形成独特的功能结构,保证酶活性正常发挥[27]。
从分子水平来探索其降解途径和降解机制, 分析菌株在高盐环境下对PAHs 的降解特性, 深入研究各类相关功能基因, 对于研究微生物高效处理高盐环境中有机污染物方面具有一定的指导意义,可极大促进对PAHs 污染修复机理的了解, 更好地解释环境中PAHs 的去除过程。 同时可结合已获得PAHs 降解基因簇以及与耐高盐相关基因,探究两者的协同作用, 利用调控基因和嗜盐酶的氨基酸组成特点等将传统的PAHs 降解菌改造为耐盐/嗜盐PAHs 降解高效基因工程菌,可更好地将其应用于微生物修复。 但目前对于耐盐/嗜盐菌的研究还不够深入,何芬等[22]的研究发现,ndo 基因的相对表达量在盐度为10%下较在盐度为20%下高, 证明了盐度可抑制嗜盐菌群功能基因的表达, 从而影响其降解速率。 但确切的盐度对PAHs 生物降解的影响机制仍有待深入研究。
4 结论
多环芳烃的生物降解是目前国内外研究的一大热点,无论是降解特性、降解途径、降解机制还是实际应用等均有深入的研究。 但是如今对于一些极端条件下,PAHs 降解菌属不够丰富,对其降解机理的认知仍然不足。 随着高盐环境中的多环芳烃污染日益严重, 嗜盐微生物的筛选和分子机制等方面的研究仍将是今后研究的热点。 目前嗜盐PAHs 的降解菌属约有100 多种, 根据对PAHs 的降解途径一般分为好氧降解和厌氧降解。 好氧降解主要通过邻苯二甲酸和水杨酸代谢途径降解, 厌氧降解中间代谢产物简单,主要涉及羟基化、甲基化、羧化反应、延胡索酸盐结合反应等。 嗜盐菌降解PAHs 的功能基因簇主要来源于普通的PAHs 降解基因簇的进化或其他差异较大的基因簇。 嗜盐菌的耐盐机制包括吸钾排钠、 积累小分子相容性溶质和嗜盐酶的氨基酸组成特性3 方面。 关于嗜盐PAHs 降解菌的研究可为在高盐碱环境下多环芳烃污染修复工程的实际应用提供科学依据。 如可利用微生物技术去富集筛选降解PAHs 的嗜盐菌纯菌或核心小菌群并对其降解途径和降解特性进行研究, 对相关功能基因进行筛选扩增,通过基因重组、原生质体融合等各种构建技术等分子生物学手段去构建高效基因工程菌以达到高效的生物修复。

