两株娄彻氏链霉菌基因组分析
王雪竹,罗晓霞,张利莉*
(1塔里木大学生命科学与技术学院,新疆 阿拉尔 843300)
(2塔里木盆地生物资源保护利用兵团重点实验室,新疆 阿拉尔 843300)
学者们从放线菌、其他细菌和真菌中发现的天然产物对人类医学、动物健康和植物保护有着重要的贡献[1],然而使用传统挖掘抗生素的方法已经无法满足日益增长的药物需求,因此学者们将注意力转向了两个具有吸引力的天然产物发现来源:极端环境中未开发的放线菌的生物合成途径以及放线菌基因组中隐藏的生物合成途径[2]。生物基因组数据包含了其主要遗传信息,全基因组测序有助于加深对微生物生命活动的认知。近年来,随着高通量测序技术的不断发展,微生物基因组测序的成本不断降低,这使得大部分科研工作者利用基因组学的方法对微生物进行功能预测、进化分析及代谢产物挖掘等方面的研究[3]。
娄彻氏链霉菌是一种极为重要的微生物资源,可以产生诸多结构新颖、生物活性好的次级代谢产物,应用于工业、农业、医药及环境污染治理等多个方面,具有进一步深入研究和开发的潜力。本研究以一株分离自塔克拉玛干沙漠骆驼蓬的娄彻氏链霉菌TRM65215和一株分离自胡杨湿地的娄彻氏链霉菌TRM68008为研究对象,采用平板对峙的方法发现两株菌均对多种动植物病原菌具有拮抗活性,且两株菌的拮抗病原菌种类及拮抗活性存在差异,分析可知具有潜在的研究价值,因此通过对两株菌进行基因组测序,分析其代谢潜能及差异。
1 材料与方法
1.1 菌株来源
菌株TRM65215分离自塔克拉玛干沙漠骆驼蓬,菌株TRM68008分离自胡杨湿地,菌株于实验室−80℃冰箱甘油管中保存。
1.2 实验方法
1.2.1 全基因组测序、组装
菌株测序由上海派森诺生物科技有限公司采用全基因组鸟枪法完成,采用A5-MiSeq和SPAdes对去除接头序列的测序数据进行从头拼装得到contig和scaffold。
1.2.2 系统发育分析
采用San Prep柱式质粒DNA抽提试剂盒提取菌株DNA,使用引物27F和1492R扩增菌株16S rRNA基因片段并进行测序。利用SeqMan软件对序列进行拼接,EzBioCloud[4](https://www.ezbiocloud.net/identify)对16S rRNA基因序列进行多序列比对及计算序列相似性水平。使用MEGA 7[5]软件邻接法(neighbor-joining,NJ)构建16S rRNA基因系统发育进化树及全基因组系统发育进化树。
1.2.3 基因组功能注释
使用 GO[6]、COG[7]和 KEGG[8]数据库对菌株蛋白编码基因功能进行注释。
1.2.4 次级代谢产物预测
采用antiSMASH 5.0[9]软件预测菌株次级代谢产物生物合成基因簇,并对次级代谢产物生物合成基因簇进行分析。
1.2.5 基因组同源分析
通过 NCBI网站(http://www.ncbi.nlm.nin.gov)下载3株娄彻氏链霉菌全基因组序列,Streptomyces rochei7434AN4(NZAP018517.1/AP018517.1)、StreptomycesrocheiNS1(JAJIRV000000000.1)和Streptomyces rocheiSID8161(JAAGMZ000000000.1)。将S.rochei7434AN4、S.rocheiNS1、S.rocheiSID8161和TRM65215、TRM68008全基因组序列上传至 OrthoVenn2[10]数据库进行直系同源基因分析,通过交互式维恩图显示所选物种之间集群的分离和交叉的功能注释和总结。
2 结果与分析
2.1 基因组分析
TRM65215及TRM68008基因组特征如表1所示。TRM65215的基因组大小为6 844 901 bp,DNA(G+C)mol%值为72.67%,共编码5 947个基因,通过序列拼接获得54个contings,非编码RNA中含有1个 5S rRNA、1个 16S rRNA、1个 23S rRNA、79个tRNA以及1个tmRNA。
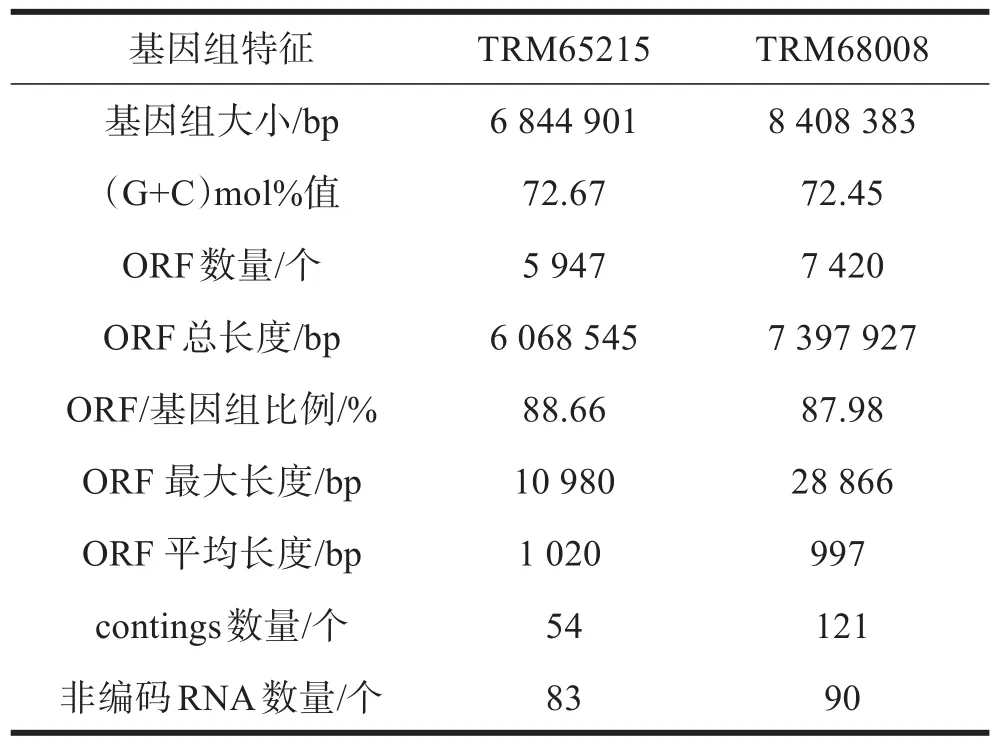
表1 基因组特征描述
TRM68008的基因组全长为8 408 383 bp,DNA(G+C)mol%值为72.45%,共编码7 420个基因,通过序列拼接获得121个contings,非编码RNA中含有1个 5S rRNA、1个16S rRNA、1个23S rRNA、86个tRNA以及1个tmRNA。
2.2 TRM65215和TRM68008系统发育分析
通过对TRM65215和TRM68008进行16S rRNA基因鉴定,两株菌均与Streptomyces rocheiNRRL B-2410为同种。使用邻接法对TRM65215和TRM68008的16S rRNA基因序列构建系统发育进化树,如图1所示;对TRM65215和TRM68008的全基因组构建系统发育进化树,如图2所示。
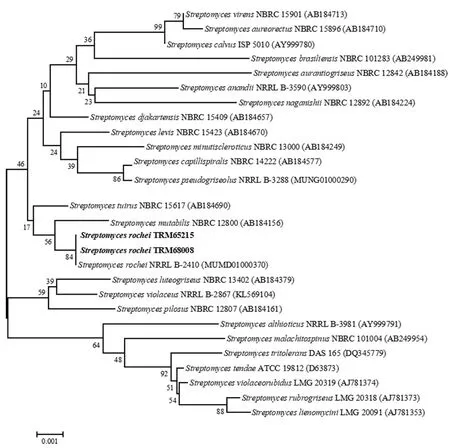
图1 基于16S rRNA基因邻接法系统发育进化树
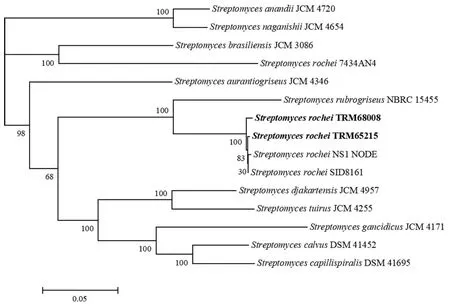
图2 基于全基因组邻接法系统发育进化树
2.3 TRM65215和TRM68008全基因组蛋白编码基因功能注释
2.3.1 TRM65215基因注释
使用GO、COG和KEGG数据库对菌株TRM65215蛋白编码基因功能进行注释,注释统计结果如表2所示。

表2 TRM 65215基因组蛋白编码基因功能的数据统计
菌株TRM65215的全基因组序列的GO注释如图3所示。在菌株TRM65215的全基因组序列的GO注释中,与生物进程(biological process)相关的GO注释结果最多,有3 987个,占GO注释总数的50.23%;与分子功能(molecular function)相关的GO注释结果有3 262个,占GO注释总数的41.09%;其中与细胞组件(cellular component)相关的GO注释结果有689个,占总体的8.68%。
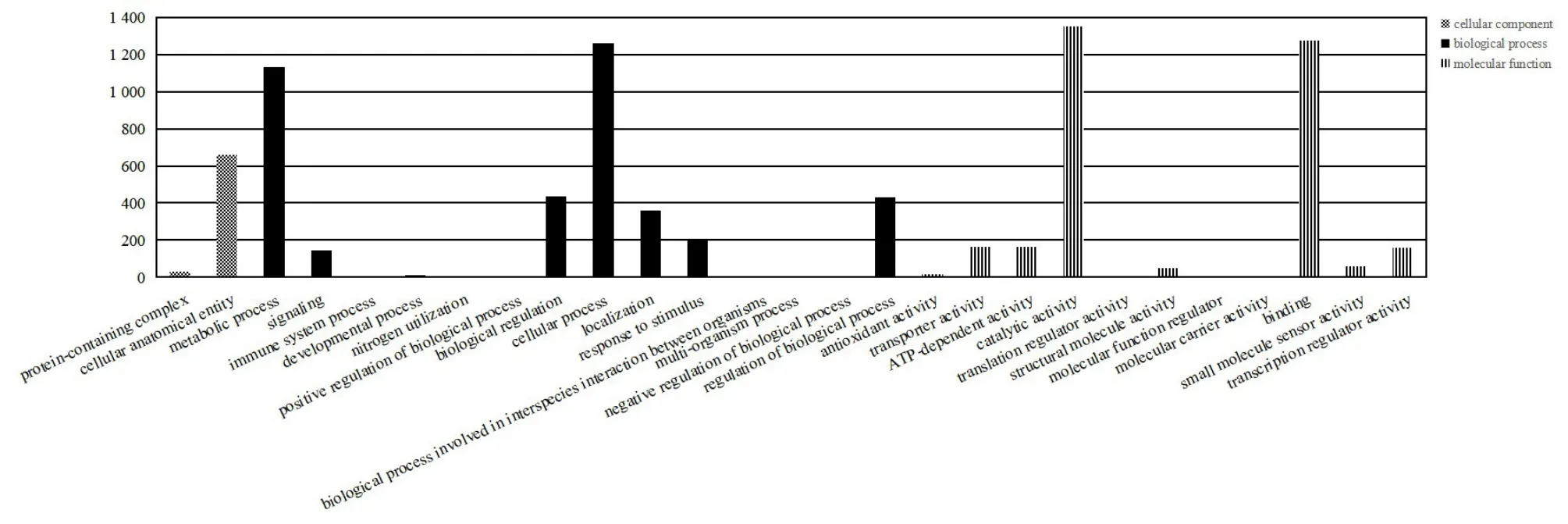
图3 TRM65215的GO功能注释分类图
菌株TRM65215的全基因组序列的COG注释如图4所示。在TRM65215的全基因组COG注释中,COG数据库将编码的蛋白分为22类,与转录(transcription)相关的蛋白编码基因最多,有503个,占COG注释基因总数的11.45%;其次为碳水化合物的运输和代谢(carbohydrate transport and metabolism)相关的蛋白编码基因,有475个,占COG注释基因总数的10.81%;与氨基酸转运与代谢(amino acid transport and metabolism)相关蛋白编码基因有386条,占COG注释基因总数的8.78%。
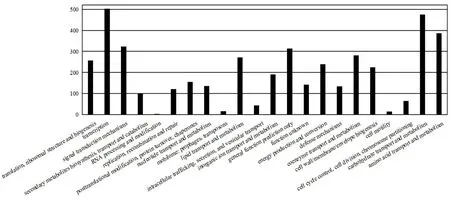
图4 TRM65215的COG功能注释分类图
菌株TRM65215的全基因组序列的KEGG注释如图5所示。TRM65215的KEGG注释共包括39种,大致可分为6大类:与代谢(metabolism)相关的基因有1 896个,占KEGG注释基因的67.67%;与环境信息处理(environmental information processing)相关的基因有256个,占KEGG注释基因的9.14%;与遗传信息处理(genetic information processing)相关的基因有245个,占KEGG注释基因的8.74%;与细胞过程(cellular processes)相关的基因有171个,占KEGG注释基因的6.10%;与生物系统(organismal systems)相关的基因有123个,占KEGG注释基因的4.39%;与人类疾病(human diseases)相关的基因有111个,占KEGG注释基因的3.96%。
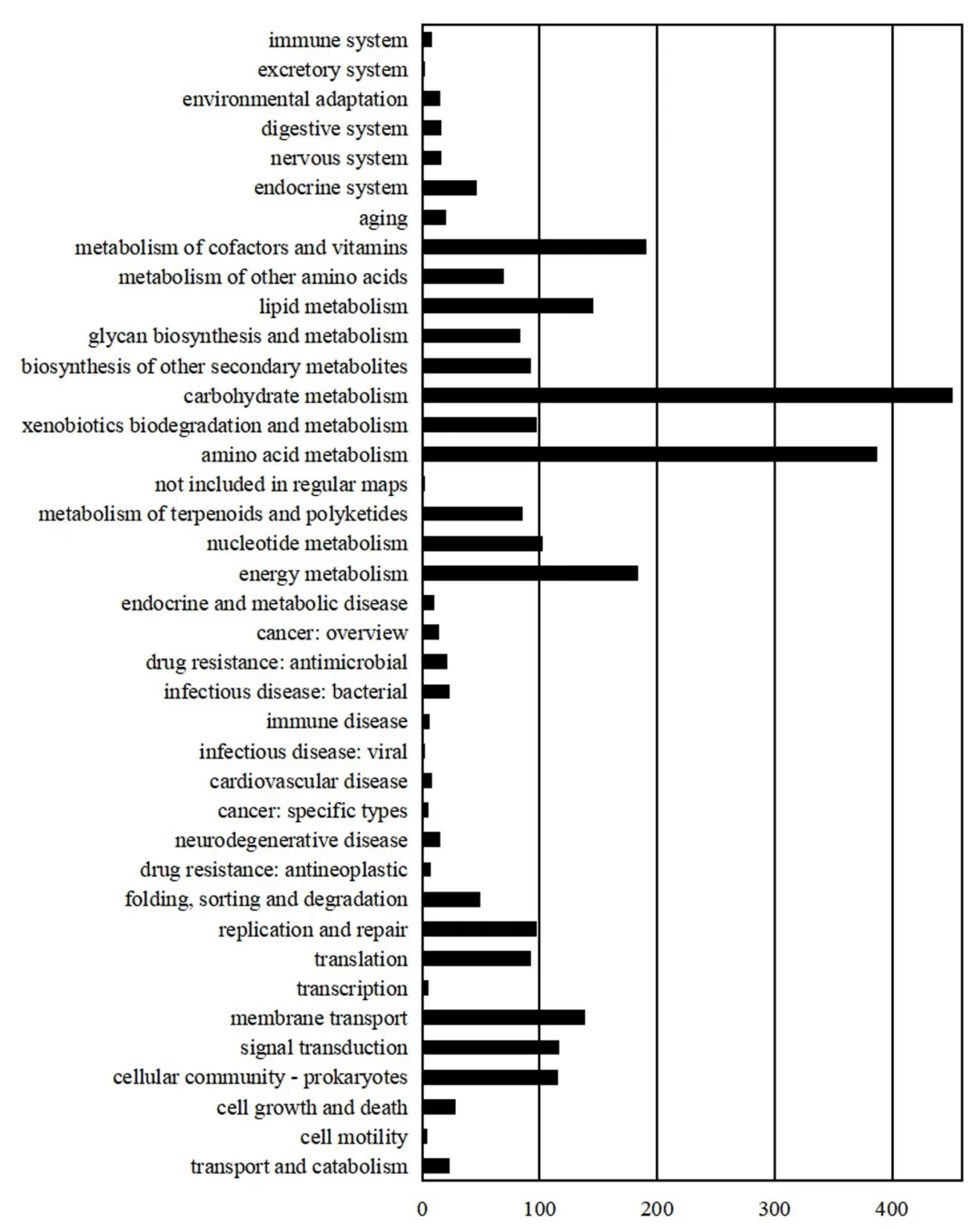
图5 TRM65215的KEGG功能注释分类图
2.3.2 TRM68008基因注释
使用GO、COG和KEGG数据库对菌株TRM68008蛋白编码基因功能进行注释,注释统计结果如表3所示。

表3 TRM68008基因组蛋白编码基因功能的数据统计
菌株TRM68008的全基因组序列的GO注释如图6所示。在菌株TRM68008的全基因组GO注释结果中,与生物进程(biological process)相关的GO注释结果最多,有4 689个,占GO注释总数的50.56%;与分子功能(molecular function)相关的GO注释结果有3 816个,占GO注释总数的41.15%;其中与细胞组件(cellular component)相关的GO注释结果有769个,约占总体的8.29%。

图6 TRM68008的GO功能注释分类图
菌株TRM68008的全基因组序列的COG注释如图7所示。在TRM68008的全基因组COG注释结果中,COG数据库将编码的蛋白分为22类,与转录(transcription)相关蛋白编码基因最多,有607个,占COG注释基因总数的11.65%;其次为碳水化合物的运输和代谢(carbohydrate transport and metabolism)相关蛋白编码基因,有539个,占COG注释基因总数的10.35%;与氨基酸转运与代谢(amino acid transport and metabolism)相关蛋白编码基因有423条,占COG注释基因总数的8.12%。

图7 TRM68008的COG功能注释分类图
菌株TRM68008的全基因组序列的KEGG注释如图8所示。在TRM68008的全基因组序列中,TRM68008的KEGG注释共包括40种,大致可分为6大类:与代谢(metabolism)相关的基因有2 149个,占KEGG注释基因的68.44%;与环境信息处理(environmental information processing)相关的基因有272个,占KEGG注释基因的8.66%;与遗传信息处理(genetic Iinformation processing)相关的基因有256个,占KEGG注释基因的8.15%;与细胞过程(cellular processes)相关的基因有187个,占KEGG注释基因的5.96%;与生物系统(organismal systems)相关的基因有145个,占KEGG注释基因的4.62%;与人类疾病(human diseases)相关的基因有131个,占KEGG注释基因的4.17%。
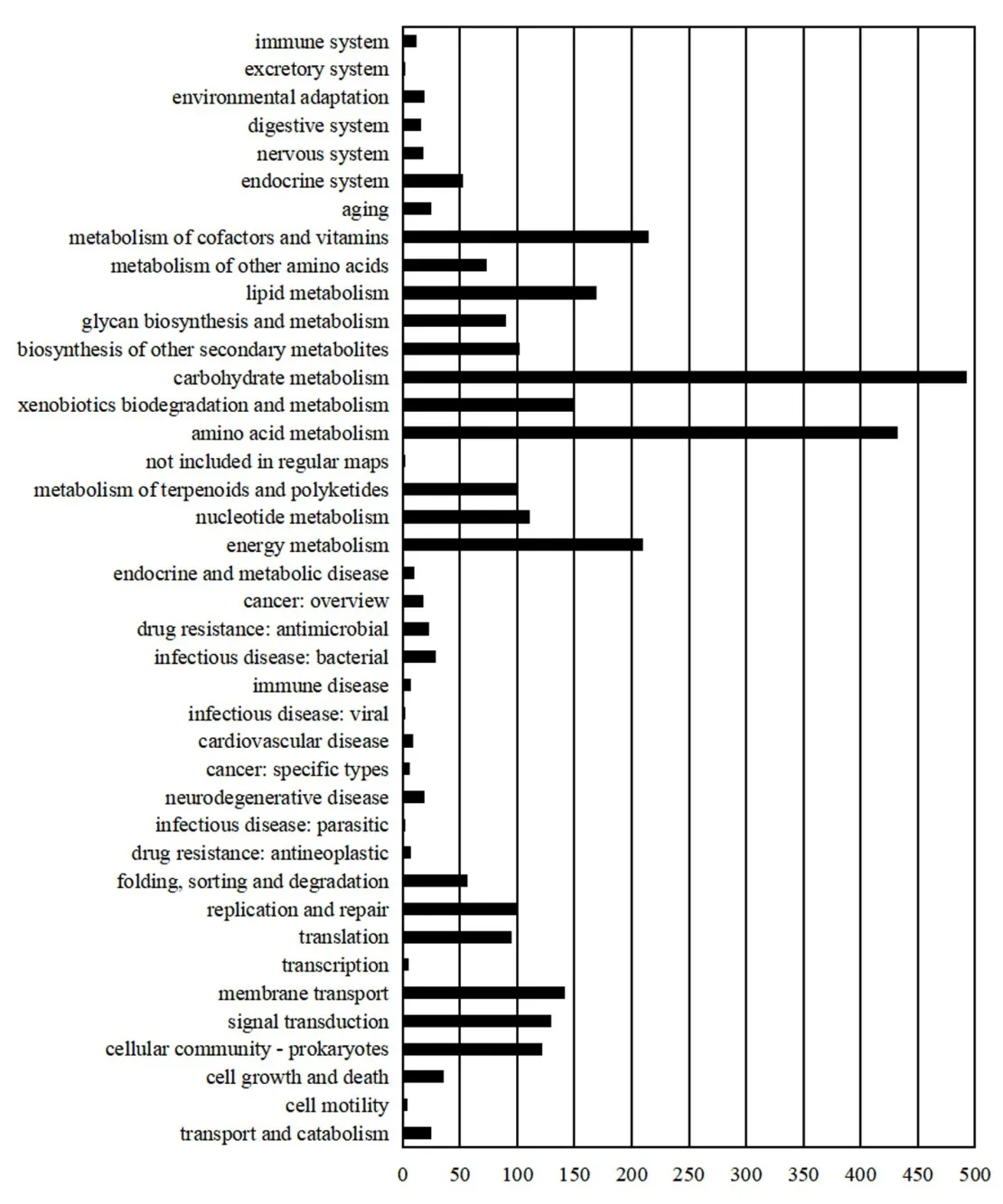
图8 TRM68008的KEGG功能注释分类图
2.4 次级代谢产物分析
通过antiSMASH对TRM65215次级代谢潜能分析,TRM65215基因组中含有30个潜在天然产物生物合成基因簇。TRM65215合成基因簇包括6个非核糖体肽合成酶(non-ribosomal peptide synthetase,NRPS)基因簇,3个Ⅰ型聚酮合酶(polyketide synthase,PKSⅠ)基因簇,2个Ⅱ型聚酮合酶(PKSⅡ)基因簇,1个Ⅲ型聚酮合酶(PKSⅢ)基因簇,4个萜烯(terpene)基因簇,1个Ⅰ型羊毛硫肽(lanthipeptideclassⅠ)基因簇,2个Ⅲ型套羊毛硫肽(lanthipeptideclassⅢ)基因簇,1个Ⅴ型羊毛硫肽(lanthipeptideclassⅤ)基因簇。TRM65215的7个基因簇(cluster 1、cluster 5、cluster 7、cluster 8、cluster 15、cluster 22、cluster 24)与已知基因簇(albaflavenone、SapB、coelibactin、coelichelin、geosmin、hopene、ectoine)的相似度为100%,表明TRM65215具有完整的合成基因簇,具有产生这7种代谢产物的能力。TRM65215中的4个基因簇(cluster 6、cluster 15、cluster 16、cluster 19)未比对到已知的基因簇,具有产生结构新颖的次级代谢产物的潜能。
通过antiSMASH对TRM68008次级代谢潜能分析,TRM68008基因组中含有36个潜在天然产物生物合成基因簇。TRM68008合成基因簇包括6个非核糖体肽合成酶基因簇,3个Ⅰ型聚酮合酶基因簇,1个Ⅱ型聚酮合酶(PKSⅡ)基因簇,1个Ⅲ型聚酮合酶(PKSⅢ)基因簇,6个萜烯(terpene)基因簇,2个Ⅰ型羊毛硫肽基因簇,2个Ⅲ型套羊毛硫肽基因簇,1个Ⅴ型羊毛硫肽基因簇。TRM68008的 8个基因簇(cluster 1、cluster 5、cluster 9、cluster 15、cluster 21、cluster 23、cluster 34、cluster 35)与已知基因簇(ectoine、hopene、coelichelin、geosmin、streptothricin、albaflavenone、7-prenylisatin)的相似度为100%,表明TRM68008具有完整的合成基因簇,具有产生这8种代谢产物的能力。TRM68008中的7个基因簇(cluster 2、cluster 7、cluster 10、cluster 12、cluster 16、cluster 17、cluster 21)未比对到已知的基因簇,可能会产生结构新颖的次级代谢产物。
2.5 直系同源基因聚类分析
直系同源基因聚类分析结果如图9所示。使用OrthoVenn2比较和注释5株娄彻氏链霉菌之间的直系同源基因簇,该物种形成7 135个簇,3 274个直系同源簇(至少包含两个物种)和3 861个单拷贝基因簇。TRM65215和TRM68008共有5 686个直系同源簇,TRM65215有2个特有的基因簇,TRM68008有19个特有的基因簇。

图9 直系同源基因聚类分析
3 讨论
本研究通过16S rRNA基因比对发现TRM65215、TRM68008与Streptomyces rocheiNRRL B-2410 16S rRNA基因序列相似度均为100%,然而通过对TRM65215和TRM68008全基因组测序及系统发育进化分析可知,TRM65215和TRM68008基因组仍存在较大差异。通过比较TRM65215及TRM68008全基因组测序及注释信息可知,TRM65215和TRM68008作为放线菌均具有非常高的DNA(G+C)mol%含量,TRM68008的全基因组大小、编码蛋白数量及非编码RNA数量均比TRM65215多。
两株菌全基因组测序后经COG预测发现其蛋白功能主要集中在转录、碳水化合物和氨基酸转运和代谢方面,KEGG预测表明代谢通路中与代谢相关的蛋白占比例较高,GO预测与生物进程相关的蛋白最多。在TRM68008基因组蛋白编码基因功能注释数量均比TRM65215基因组注释数量多,GO注释比其结果多551个蛋白编码基因,COG注释比其结果多816个蛋白编码基因,KEGG注释比其结果多338个蛋白编码基因。TRM65215和TRM68008共有5 686个直系同源簇,TRM65215有2个特有的同源基因簇,TRM68008有19个特有的同源基因簇。
TRM65215与TRM68008有20个相似的基因簇,在这20个相似基因簇中有12个基因簇的基因是完全相同的,除了相似的基因簇,TRM65215还具有合成锌离子载体coelibactin(cluster 7)、versipelostatin(cluster 10)、克拉维烷类化合物alanylclavam/2-hydroxymethylclavam/2-formyloxymethylclavam(cluster 17)及大环内酰胺类抗生素vicenistatin(cluster 25)的潜力,TRM68008则具有5-异戊烯吲哚-3-羧酸β-D-糖苷酯 5-isoprenylindole-3-carboxylate β-D-glycosylester(cluster 19)、异胡萝卜素isorenieratene(cluster 20)、大环内酯类抗生素阿扎霉素azalomycinF3a(cluster 25)、溶血脂质lysolipin I(cluster 26)、30元环多醇大环内酯 aculeximycin(cluster 28)、moomysin(cluster 30)、抗霉素antimycin(cluster 32)、异海绵烯isorenieratene(cluster 33)及异戊烯吲哚类化合物7-prenylisatin(cluster 34)的潜能。
TRM65215和TRM68008虽然在16S rRNA基因序列分析中与Streptomyces rocheiNRRL B-2410相似度为100%,但通过对两株菌的基因组测序分析,TRM68008与TRM65215在基因组大小、非编码RNA数量、编码蛋白数量及次级代谢产物合成基因簇等多个方面均存在差异,TRM68008基因组较大,编码了更多的蛋白,能够产生与TRM65215不同的生物特性。
由于基因组测序技术的快速发展,大规模的基因组测序不断揭示着微生物中蕴藏的丰富天然产物资源,微生物的生物合成潜力被大大低估,使得微生物天然产物挖掘进入“基因组时代”[11]。基因组挖掘更善于合理利用生物信息学分析工具对次级代谢基因簇进行预测从而实现产物的定向挖掘,目前是药物发现工作的重要组成部分[12],结合基因组挖掘方法可以有效提高天然产物的成功率,从而加快药物的开发。同时,菌株的基因组信息对探究菌株的进化、反应及环境的适应机理有着重要意义[13]。
传统Sanger测序技术由于耗时较长且费用较高,因此研究人员在对未知菌种鉴定时通常采用更加高效且经济的16S rRNA基因序列测序,然而这种方法是以牺牲分类分辨率为代价的,通过保守的16S rRNA基因引物来检测细菌,仅能检测出菌株与微生物群落中的其他成员(如真菌、病毒和原生动物)是没有同源性的,对于同种间的差异却难以鉴定[14],通过16S rRNA基因对菌株进行鉴定只能初步判断菌株种属,存在一定的局限性,因此对于菌株的鉴定仍需要更多的鉴定方法及手段[15]。
4 总结
本研究对两株娄彻氏链霉菌TRM65215及TRM68008进行了全基因组测序、组装及功能基因注释,TRM65215基因组大小为6 844 901 bp,GO功能注释蛋白编码基因2 993个,COG功能注释蛋白编码基因4 394个,KEGG功能注释蛋白编码基因2 802个;TRM68008基因组大小为8 408 383 bp,GO功能注释蛋白编码基因3 544个,COG功能注释蛋白编码基因5 210个,KEGG功能注释蛋白编码基因3 140个,研究表明两株16S rRNA基因序列完全相同的菌株在基因功能等多方面仍存在差异。预测两株娄彻氏链霉菌次级代谢产物合成基因簇,TRM65215基因组中含有30个潜在天然产物生物合成基因簇,TRM68008基因组中含有36个潜在天然产物生物合成基因簇,TRM65215及TRM68008均具有产生新型抗生素的潜能且均含有对方不具备的天然产物生物合成基因簇,进一步验证了两株菌的代谢差异,并为后续挖掘菌株次级代谢产物提供了理论依据。

