海南部分地区黄牛无浆体分子流行病学调查及遗传多样性分析①
黄良圆 周飒 廖慧钰 王金花
(海南大学 海南 海口 570228)
无浆体是一类无固定形态的微生物,专性寄生于脊椎动物血细胞中。根据无浆体代谢特征和超微结构,将其归为立克次体目(Rickettsiales)无浆体科(Anaplasmataceae)无浆体属(Anaplasma)。主要种类有:嗜吞噬细胞无浆体(Anaplas-ma pagocytophilum),绵羊无浆体(Anaplasma ovis)、牛无浆体(Anaplasma bovis)、边缘无浆体(Anaplasma marginale)、中央无浆体(Anaplasma centrale)、扁平无浆体(Anaplasma platys)、山羊无浆体(Anaplasma capra)等[1]。其中,A.ovis、A.pagocytophilum对羊 危害 较 大,A.marginale对牛危害比较大,A.pagocytophilum已经被公认是一种引起人畜共患的病原。
无浆体病(Anaplasmosis)是由几种无浆体病原经蜱媒传播引起的一种立克氏体疾病,主要在热带和亚热带地区流行,主要表现为发烧、贫血、黄疸、流产等症状,严重时会引起死亡,给畜牧养殖业造成严重的经济损失[2],还威胁到人的健康。该病已被世界动物卫生组织(OIE)列为B类动物传染病,也被我国农业农村部列为进出口动物检疫项目之一[3]。目前,我国多个地区均有无浆体病报道,海南地区尚未发现相关报道。
1 材料与方法
1.1 样品
对海南地区176头黄牛颈静脉采血(见图1),其中,定安县161头、海口市4头、琼海县5头、儋州县6头。记录动物的饲养模式、性别、年龄和品种,将血液于‐20℃保存。

图1 样品来源分布图
1.2 提取血液基因组DNA
将冷冻EDTA血液样品于室温下解冻并涡旋。吸取200μL全血,使用血液基因组DNA抽提试剂盒(Sangon Biotech,ShangHai,China)进 行DNA提取,使用Nanodrop ND1000分光光度计(Thermo Scientific©,美国)测DNA浓度和纯度。丢弃小于5 ng/μLDNA样品,并重新提取全血DNA,于‐20°C保存。
1.3 巢式PCR和单PCR扩增
表1为本次研究使用引物序列。对A.phagocytophilum和A.bovis使用基于16Sr RNA基因对巢式PCR扩增,第一轮引物为EE1和EE2,可能存在的所有无浆体物种的16S rRNA基因,第二轮引物使用SSAP2 f/r和AB1f/r进行巢式PCR扩增[4];A.marginale使用基于MSP4基因设计的特异性引物[5]。以上所有引物均由上海生工生物工程股份有限公司合成。第一轮PCR反应在25μL的最终体积中进行,其中含有13μL Mix酶(中国南京诺维赞公司)、双蒸水10μL、左右引物各0.5、1.0μL DNA。热循环体系如[6]。A.phagocytophilum和A.bovis的第二轮PCR反应最终体积为50μL,其中Mix酶(中国南京诺维赞公司)25μL、双蒸水21μL、引物1μL、第一轮PCR反应的产物2μL。A.ovis、A.marginale反应的最终体系为25μL。热循环反应条件如表2所示。
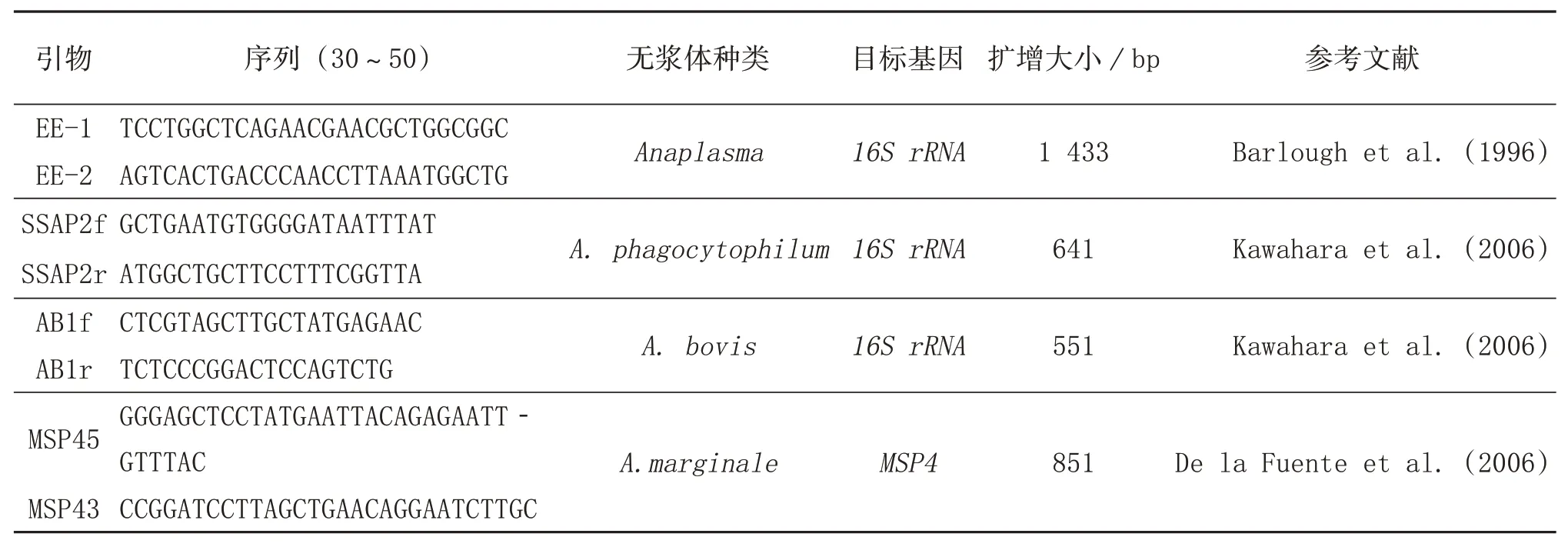
表1 无浆体引物序列

表2 无浆体PCR扩增条件
1.4 琼脂糖凝胶电泳
用1×TAE电泳缓冲液和1.5%琼脂糖配置凝胶液,稍冷后加入DNA Green染料(每100 mL琼脂糖凝胶溶液加1μL DNA Green染料)混匀制胶,冷却20 min,将巢式PCR第二轮产物和常规PCR产物进行琼脂糖凝胶电泳。取8μL PCR扩增产物加入凝胶孔,120 V恒压电泳。电泳结束后凝胶放置在紫外凝胶成像仪观察拍照。
1.5 系统进化树分析
使用16S rRNA、Gro EL(热休克蛋白)和MSP4(主要表面蛋白4)基因序列构建系统发育树。首先从GenBank中获取世界各地菌株并筛选(即丢弃重复测序,不完整的序列,并在非常相似的序列子集中选择一个序列)。通过MegAlign软件(DNAStar,Madison,WI)中Clustal W方法比对。使用Mega X软件,Neighbor‐joining(NJ)算法中Kimura‐2‐parameter参数模型,Bootstrop值选择1 000进行系统发育树构建。最后,选用Figtree软件将Mega X导出进行原树染色编辑。
2 结果与分析
2.1 PCR扩增
黄牛中检测到A.bovis、A.phagocytophilum、A.marginale MSP4,其目的条带大小分别为551、641、851 bp,如图2所示。
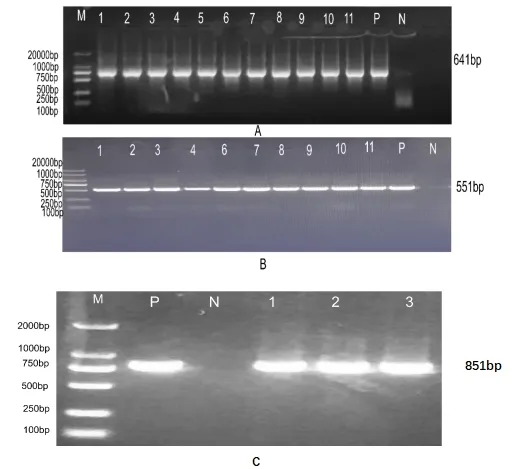
图2 部分黄牛样品PCR结果
2.2 黄牛无浆体感染
2.2.1 不同地区黄牛无浆体感染情况分析
4个市县牛均检测出无浆体感染,其中海口市、琼海县和儋州3个地区无浆体感染率高达100.0%、80.0%、50.0%,但并不能说明无浆体在这3个地区的流行情况。Yan等[7]报道在新疆塔里木河上游493份奶牛血液样本中,无浆体的感染率为3.2%。Li等[8]报道在196份牛血液样品中无浆体感染率为5.1%。Yang等[9]发现我国10省557头牛中有31.6%的牛检出A.marginale。El‐Dakhly等[10]报道在埃及,牛检出A.marginale感染率为10.6%(16/150)。Fernandes等[11]报道在莫桑比克,牛中检出A.marginale、A.phagocytophilum感染率分别为97.3、2.7%(6/219)。Sisson等[12]报道在南非克鲁格国家公园饲养的非洲水牛747份血液样本中,A.marginale、A.centale感染率别为17.3%、13.1%,总感染率为15.1%。本研究中黄牛的A.phagocytophilum感染率为6.3%(11/176),A.bovis感染率为11.4%(20/176),A.marginale感染率为5.7%(10/176),海南地区牛无浆体的感染率最高。Yan等报道了A.bovis、A.ovis、A.phagocytophilum和A.platys感染率分别为0.2%(1/493)、0.4%(2/493)、2.4%(12/493)、2.4%(12/493)。Rjeibi等[14]报道在阿尔及利亚的牛中,A.centrale、A.marginale、A.bovis三种无浆体的感染率为30.5%(55/180)、1.66%(3/180)、0(0/180)。本研究A.bovis感染率为11.4%(20/176),未检出牛无浆体。
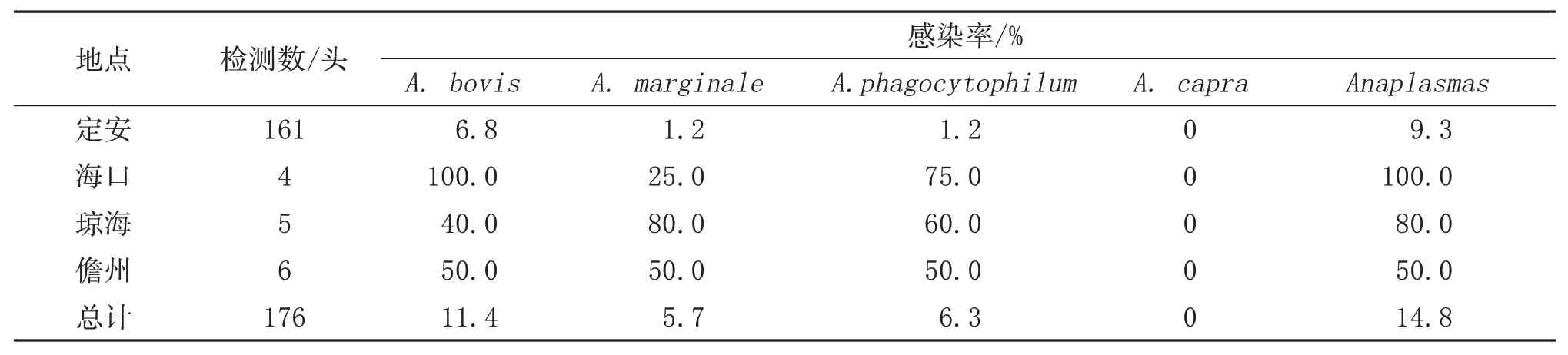
表3 不同地区黄牛无浆体感染情况分析
2.2.2 无浆体混合感染情况分析
研究发现,A.bovis+A.phagocytophilum感染率为2.0%(3/176),A.bovis+A.marginale的感染率为1.1%(2/176),A.phagocytophilum+A.marginale的感染率为0.6%(1/176),A.bovis+A.phagocytophilum+A.marginale感染率为3.4%(6/176)。海南地区牛A.bovis+A.phago-cytophilum+A.marginale混合感染情况比较普遍。Rjeib等[14]报道在阿尔及利亚的牛中A.centrale+A.marginale感染率为5.5%(10/180),A.marginale+A.bovis感染率为1.1%(2/180),A.centrale+A.marginale+A.bovis感染率为2.8%(5/180),其中A.centrale+A.marginale混合感染情况比较普遍,该结论与本研究结果不一致。

表4 牛混合感染情况统计表
2.2.3 黄牛不同年龄、性别和饲养模式无浆体感染情况
调查不同性别牛无浆体感染情况发现,感染无浆体公牛有3头,感染率为14.3(3/21),母牛23头,感染率为14.8%(23/155),公牛和母牛感染无浆体的差异不明显。调查不同年龄牛无浆体的感染情况发现,小于3岁的牛感染无浆体有9头,感染率为14.3%(9/63),3~6岁牛感染无浆体有13头,感染率为15.1%(13/86),大于6岁牛感染无浆体有3头,感染率为11.1%(3/27),显示不同年龄段牛感染率差异不明显。研究发现散养模式下牛无浆体的感染率为73.3%,圈养模式下无浆体感染率为9.3%,可见牛在散养模式下无浆体感染率明显高于圈养模式,该结论与张晶报道一致[15]。这可能与规模化农场养殖环境管理更科学,蜱虫叮咬少,而自由放牧条件下,蜱虫叮咬高,导致无浆体感染率高。
2.3 系统发育树分析
2.3.1A.phagocytophilum16S rRNA基因序列系统进化树分析

表5 不同年龄、性别、饲养模式无浆体感染率
本研究获得的5株A.phagocytophilum 16S rRNA基因部序列与A.phagocytophilum16S rRNA基 因 序 列(AB196720、AB19672、GQ175174、JN558812、JN558816、JN990105、KC246018、KF569915、KJ782381、KJ782386、KP062963、KP276588、KR002114、KT944029、KU321298、KU870667、KX083402、KX236051、KX236051、KX450278、LC060986、LC060987、MF992253、MG002405、MN097858、MN216240、NR044762)以及Rickettsia(JX885456)、A.ovis(KJ459342)、A.platys(KU500900、KU500914、MH255941)、A.bovis(MH255928、MH255938)、A.centrale(MH588232、MH588233)和A.capra(MH762071、MT798602、MT052418)6种16SrRNA基因序列作为外群构建系统发育树。A.phagocytophilum系统发育树分析结果表明,本研究获得黄牛4株序列(XYAP2、RYAP3、RYAP4、RYAP6)独立聚为一 类群,与(KT944029,KT782381)参照序列比较相近;NDAAP2序列独立在不同的分支与来自巴基斯坦牛(MN216240)分离参照株比较相近。如图3所示。

图3 A.phagocytophilum16S rRNA基因序列(641 bp)系统发育树
2.3.2A.bovis16S rRNA基因序列系统进化树分析
本研中获得的7株A.bovis 16S rRNA部分基因序列与A.bovis 16SrRNA基因序列(KU509992、KU509996、KX450273、KY242455、MH255927、MH255934、MH255935、MH255936、MH255939、MH255940、MH255941、MK028572、MK028574、MK446832、MN044717、MN309842、MN309843、MT036513),以及Rickettsia(JX885456)、A.ovis(KC484562、MN795156)、A.platys(KU500900、KU500914)和A.capra(LC432092、MH762071,MT052418、MT798602)4种16S rRNA基因序列作为外群构建系统发育树。A.bovis系统发育树分析表明,海南地区A.bovis序列与其他地区(包括:马来西亚,俄罗斯,韩国)的分离株聚集为一类群,得到了95.5%个节点支持,其中RYAB9的遗传距离比较远。如图4所示。

图4 A.bovis16S rRNA基因序列(551 bp)系统发育树
2.3.3A.marginale MSP4基因序列系统进化树分析
本研究获得的10株A.marginale MSP4基因部序列与A.marginale MSP4基因序列(AF428082、
AF428086、AY283190、AY456002、AY665997、AY665999、AY851150、EF053264、EU283844、EU677383、JN564646、KX989516、KX989512、KX989513、MG676453、MG676455、MG676459、MH026093、MH172467、MH373246,MH939155、MK809379、MK809381、MK809384、MK809386、MK809387、MT268094)以及A.ovis(HM063433、KU497703)、A.centrale(KY305601、KY305604、KY305621)和A.phagocytophilum(EU008082、EU180058、MF974857)的MSP4基因序列作为外群构建系统发育树。A.marginale基于MSP4基因系统进化树分析显示,A.marginale、A.phagocytophilum、A.centrale和A.ovis的MSP4基因序列分别独立聚为不同分支。本研究获得的10株A.marginale序列与其他地区A.marginale参照序列同聚为一类群,分支节点支持率高达100%,其中DAAM9株遗传距离较远。如图5所示。
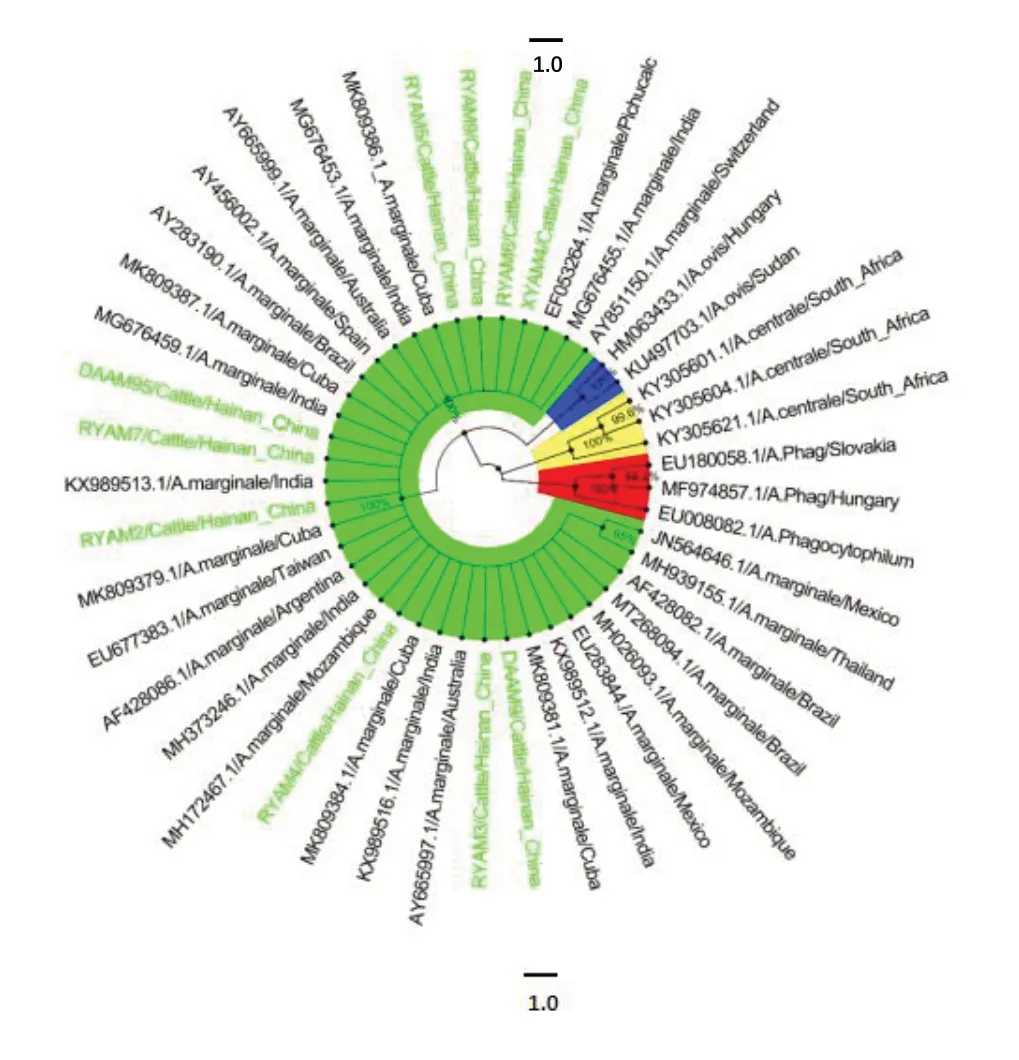
图5 A.marginale MSP4基因序列(851 bp)系统发育树
3 结论
本次研究使用PCR方法对海南地区黄牛进行了无浆体分子流行病学调查。海南地区4个市县黄牛中无浆体感染率为14.8%,并在黄牛中检测出A.bovis、A.pagocytophilum、A.marginale,其感染率分别为11.4%、6.3%、5.7%,混合感染率为11.8%,A.bovis为优势种,且A.bovis+A.pagocytophilum+A.marginale共同感染比较常见,散养比圈养更容易感染无浆体。本研究通过16S rRNA/MSP4基因构建系统进化树分析发现海南地区黄牛中,A.pagocytophilum、A.bovis、和A.marginale序列与来不同地区的无浆体参照序列之间未发现明显地理隔离分化特征,而在A.pagocytophilum系统进化树中发现不同宿主存在明显分支,可能存在宿主分化特征。

