黄颡鱼溶酶体酸性脂肪酶基因的cDNA序列、启动子及其转录调控分析
谌 芳 仲崇超 陈姝为 张电光 吕武宏 谭肖英
(华中农业大学水产学院, 农业农村部淡水生物繁育重点实验室, 武汉 430070)
目前, 养殖鱼类普遍存在脂肪肝、内脏脂质过多和相关代谢紊乱的问题, 导致成活率下降、生长性能下降和抗病性下降等[1]。在脂肪分解中除了中性脂肪酶介导的脂解外, 溶酶体酸性脂肪酶(Lysosmal acid lipase, LAL)承担着重要脂解功能。lal作为溶酶体中的酸性脂肪酶, 由LIPA基因编码, 主要作用是将胆固醇酯(Cholesteryl esters, CEs)和甘油三酯(Triglycerides, TGs)水解成游离的胆固醇(Free cholesterols, FCs)和脂肪酸(Free fatty acids,FFAs)[2]。而lal在脂解反应中, 除了水解由低密度脂蛋白(Low density lipoprotein, LDL)内吞进入胞质内的CEs、TGs和REs (Retinyl esters, REs)以外[3],还通过自噬分解脂滴[4]。人的LAL由372个氨基酸组成含有27个残基的疏水信号肽[5]、6个N-糖基化位点[6], 并且通过与高尔基体中甘露糖-6-磷酸受体结合, 靶向溶酶体发挥其生理作用[7]。目前有关LAL的研究多集中于哺乳动物的分子结构[5]、LIPA基因突变或缺失导致脂肪沉积、肝脏病变[8]、免疫机能下降[9]和冠状动脉疾病[10]等, 而有关lal在鱼类生长发育和生理营养过程中所起到的作用尚不清楚。
黄颡鱼(Pelteobagrus fulvidraco)肉质细嫩、耐低温, 是我国重要的淡水经济养殖鱼类, 其养殖产量逐年增加[11]。鉴于lal对脂质代谢的重要作用, 为深入解析lal功能, 本实验首先克隆黄颡鱼lal基因全长的cDNA序列, 进行生物信息学分析并探讨其组织表达模式, 然后从启动子结构及活性分析研究黄颡鱼lal的转录调控方式, 以期为深入解析黄颡鱼lal基因的功能奠定基础。
1 材料与方法
1.1 试验鱼及样品采集
本实验的黄颡鱼分为2组, 购自武汉市农贸市场。黄颡鱼样品采集参照本实验室的方法[12]。第一组黄颡鱼, 取肝脏和卵巢组织用于lal基因cDNA全长序列的克隆, 取肝脏、脾脏、肾脏、肠道、肠系膜脂肪、肌肉、大脑、心脏和性腺组织用于组织表达水平的测定。第二组黄颡鱼用于lal基因的启动子克隆, 取样参照本实验室的方法[13], 随机选取黄颡鱼3尾, 麻醉后冰浴上迅速剪取尾鳍进行总DNA提取。
1.2 实验材料
凝胶回收试剂盒购自Omega公司; TRIzol总RNA提取试剂盒、胰蛋白酶、DNA提取试剂盒、Lipo2000转染试剂盒、DMEM培养基和新生胎牛血清(FBS)等购自Invitrogen公司; 其他分子试剂均购自TaKaRa公司; 化学试剂均为分析纯, 购自上海国药; 胰蛋白胨、酵母提取物、琼脂粉、大肠杆菌DH5α感受态细胞和氨苄青霉素等购自武汉迪跃创新生物有限公司。HEK293T细胞株来自华中农业大学水产学院细胞资源中心。
1.3 黄颡鱼lal基因cDNA序列的克隆及测序
根据本实验室已有的方法[12], 参照Invitrogen的TRIzol说明书进行总RNA的提取。1%的琼脂糖凝胶电泳和Nanodrop ND-2000分光光度计检测总RNA的质量和纯度。用TaKaRa的反转录试剂盒(PrimeScriptTMII 1st Strand cDNA Synthesis Kit)进行反转录。
根据GenBank数据库中已报道的黄颡鱼lal基因序列, 用Premier 5.0分别设计3′和5′ RACE特异性引物(表 1), 通过巢式PCR反应进行末端的扩增, 第一轮PCR反应参数: 95℃ 5min; 然后95℃ 30s, 55℃30s, 72℃ 1min, 25个循环; 72℃ 5min。第二轮PCR反应参数: 95℃ 3min; 95℃ 30s, 57℃ 30s, 72℃1min, 30个循环; 最后72℃ 5min。
1.4 序列分析
用Seqman软件将扩增得到的核心片段、3′ 和5′ 末端序列拼接, 从而获得基因cDNA全长。利用NCBI进行BLAST, 以确定该序列对应的基因亚型(http://blast.ncbi.nlm.nih.gov/)。同时, 利用NCBI中ORF finder软件(https://www.ncbi.nlm.nih.gov/orffinder/)找出开放阅读框(ORF)并翻译成氨基酸序列。信号肽用Signal 5.0在线软性预测(http://www.cbs.dtu.dk/services/SignalP/), 通过在线软件Expasy(https://web.expasy.org/compute_pi/)预测蛋白分子量大小及等电点, NetNGlyc 1.0 Server(http://www.cbs.dtu.dk/services/NetNGlyc/)分析N-糖基化位点, 使用Clustal-W软件进行序列比对和氨基酸同源性分析。用MEGA 5.0软件采用邻接法(NJ) 构建进化树[14],选择的最适进化模型为JTT+G[15], 每个节点的可信值进行1000次重复计算。
1.5 黄颡鱼LAL三维结构模型分析
利用SWISS-MODEL同源蛋白建模构建黄颡鱼LAL蛋白质的三维模型, 选择狗的胃脂肪酶(PDB code 1k8q.1)[16]作为合适的结构模板, 其中两者序列一致度达到56.84%。用VMD 1.9.2(https://www.ks.uiuc.edu/Research/vmd/)显示和分析得到蛋白单体的理论模型。
1.6 黄颡鱼lal基因的组织表达分析
参照文献[12], 利用实时荧光定量PCR(q-PCR)方法检测lal基因在黄颡鱼不同组织中的表达。q-PCR反应参数: 95℃ 30s; 95℃ 5s, 57℃ 30s, 72℃30s, 40个循环。选用β-actin和ubce作为内参基因,相对表达水平采用2-ΔΔCt方法计算[17]。荧光定量引物见表 1。
1.7 黄颡鱼lal基因启动子的克隆及质粒构建
利用RNA连接酶介导的5′ cDNA末端快速扩增(RLM-5′ RACE)方法鉴定黄颡鱼lal的5′ cDNA序列和转录起始位点(Transcription start sites, TSS)。启动子克隆是基于上一步得到的5′末端的DNA序列及已发表的黄颡鱼[18]的基因组, 实验过程参照本实验室已有的方法[13]。利用Omega试剂盒从黄颡鱼尾鳍中提取基因组DNA, 并设计具有酶切位点的特异性引物(表 2), 将PCR产物和pGL3-Basic质粒纯化并使用相应的内切酶(Hind Ⅲ和SacⅠ)消化, 重组连接按照ClonExpressTMⅡ One Step Cloning Kit说明书操作。简要操作步骤如下: 第一轮进行RTPCR反应: 95℃ 3min; 95℃ 15s, 57℃ 15s, 72℃ 50s,35个循环; 72℃ 3min。以第一轮PCR产物为模板,加入含有酶切位点的引物进行第二轮PCR: 95℃3min; 95℃ 15s, 55℃ 30s, 72℃ 2min, 35个循环;72℃ 3min, 获得对应的目的片段。将目的片段与pGL-3载体连接30min后, 转化至DH5α感受态细胞中, 氨苄抗性平板筛选出阳性克隆, 送北京擎科生物科技有限公司测序。将测序后返还的质粒重新转化后, 进行扩大培养和质粒抽提。质粒抽提方法参照说明书进行。本实验共构建4个黄颡鱼lal启动子缺失载体, 分别命名为pGL3-554/+51、pGL3-1016/+51、pGL3-1567/+51和pGL3-2052/+51。

表 1 黄颡鱼lal基因cDNA全长序列的克隆及荧光定量所用的引物Tab. 1 Primers used for lal cDNA cloning and PCR

表 2 黄颡鱼lal cDNA启动子克隆所用到的引物Tab. 2 Primers used for lal promoter cloning of P. fulvidraco
利用JASPAR数据库(http://jaspar.genereg.net/)预测黄颡鱼lal启动子可能的转录因子结合位点。
1.8 细胞转染及双荧光素酶活性检测
HEK293T细胞在10% FBS-DMEM培养基中生长, 并置于37℃、5% CO2的环境中培养。瞬时转染前, 将HEK293T细胞以1.2×105的密度接种于24孔细胞培养板中, 培养24h, 需达到70%—80%的密度。使用LipofectamineTM2000将450 ng构建好的质粒和20 ng pRL-TK(内参质粒)共转染到HEK293T细胞中, 阴性对照为pGL3-Basic质粒。4h后, 更换含有10% FBS-DMEM培养基。24h后收集细胞,进行双荧光素酶活性检测, 检测步骤参照说明书进行。
1.9 数据统计与分析
采用平均值±标准误(mean±SE)表示结果。统计分析之前, 采用Kolmogorov-Smirnov检验所有数据的正态分布性。利用SPSS 19.0软件采用单因素方差分析和Duncan’s多重比较评估组织分布数据,采用Student’sttest评估双荧光活性数据中相邻两组之间的差异。显著性水平为P<0.05, 极显著性水平为P<0.01。
2 结果
2.1 黄颡鱼lal cDNA的序列特征及进化分析
本研究通过RT-PCR和RACE方法获取lal基因的cDNA全长序列, 长度为1802 bp, 序列分析显示它们的5′非翻译区长度为131 bp, 3′非翻译区长度为474 bp, cDNA序列ORF长度为1197 bp, 编码398个氨基酸, 理论蛋白分子量为45.42 kD, 等电点为7.70。黄颡鱼LAL蛋白序列含有一段具有23个氨基酸残基的信号肽、α/β水解酶折叠类结构域, 5个N-糖基化位点(Asn35-Ile36-Ser37、Asn101-Thr102-Ser103、Asn273-Met274-Thr275、Asn320-Gln321-Ser322和Lys161-Thr162-Thr163)、2个氧阴离子孔(Leu89和Gln175)、1个“帽子”结构域(Thr205-Val329)和“盖子”结构域(Phe233-Leu272), 及催化三元体(Ser174、Asp344和His373)和三个半胱氨酸残基(Cys257、Cys265和Cys284)。根据以上主要位点绘制出黄颡鱼LAL的氨基酸结构示意图(图 1)。
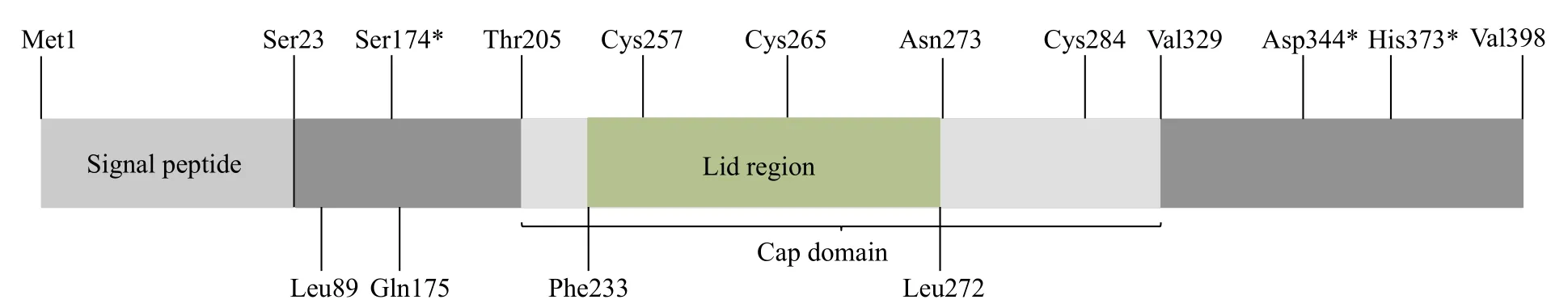
图 1 黄颡鱼LAL氨基酸结构示意图Fig. 1 Structural diagram of LAL amino acids from P. fulvidraco
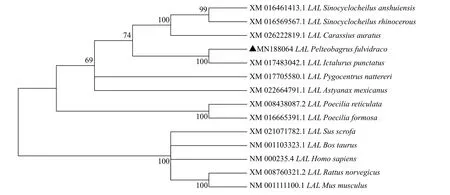
图 2 基于NJ法构建的脊椎动物LAL氨基酸序列的系统进化树Fig. 2 The neighbor-joining phylogenetic tree based on the amino acid sequences from P. fulvidraco (▲) and other vertebrate species,using MEGA 5.0 with 1000 bootstrap replicates
多重序列比显示, 黄颡鱼LAL氨基酸序列与斑点叉尾鮰相应氨基酸序列的相似性为85.64%, 与哺乳类的相似性为57.66%—62.77%, 其中与人的相似性为60.83%, 与大鼠的相似性为57.66%。
在进化树中, 哺乳类和两栖类聚为一支, 硬骨鱼独立聚为另一支(图 2)。同时在硬骨鱼类中, 黄颡鱼lal多肽与斑点叉尾鮰(Ictalurus punctatus)亲缘关系最近, 且这两2个物种与水金线鲃(Sinocyclocheilus anshuiensis)、犀角金线魮(Sinocyclocheilus rhinocerous)和金鱼(Carassius auratus)聚为一支, 其他硬骨鱼类聚为另一支。
2.2 黄颡鱼LAL蛋白三级结构分析
LAL的三维结构主要由3个领域组成, 包括α螺旋、β折叠及转角, 3个半胱氨酸残基Cys257、Cys265和Cys284的结合位点在LAL中相互接近(图 3)。
2.3 黄颡鱼lal mRNA组织表达分析
黄颡鱼lal基因在多个组织中(心脏、肝脏、脑、脾脏、肾脏、肌肉、肠系膜脂肪、肠道和性腺)均有表达, 其中脾脏、肠道和精巢中表达最高,表达量最低的是心脏和大脑(图 4)。
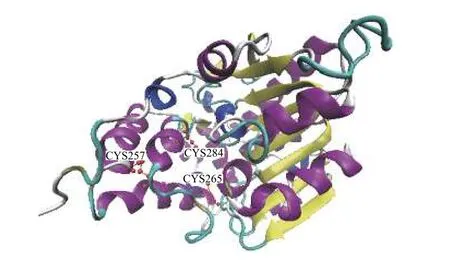
图 3 黄颡鱼LAL蛋白三级预测Fig. 3 The three-dimensional structure prediction of LAL protein from P. fulvidraco
2.4 黄颡鱼lal启动子序列及活性分析
本研究克隆得到2103 bp的黄颡鱼lal序列, 并将其提交到在线转录因子数据库(JASPAR)进行序列分析。lal的5′ cDNA序列的第一个碱基为基因的转录起始位点, 其位置定义为+1。利用JASPAR(http://jaspar.genereg.net/)软件预测转录因子结合位点, 发现lal启动区存在Sp1、PPARα、FOXO1、PPARγ、HNF4α和TFEB等转录因子结合位点, 同时核心启动子结构含有TATA盒和CCAAT盒。
通过5′缺失载体构建、细胞转染及双荧光素酶活性测定, 结果显示lal的-1507/-1016区域负调控启动子活性, 而-1016 bp/+51 bp区域正调控启动子活性(图 5)。
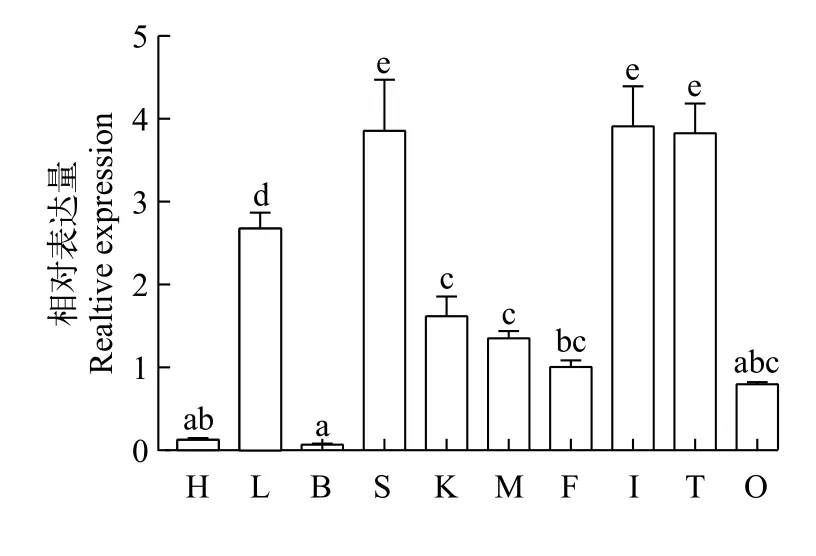
图 4 黄颡鱼lal mRNA的组织表达Fig. 4 The relative mRNA levels of P. fulvidraco lal in different tissues
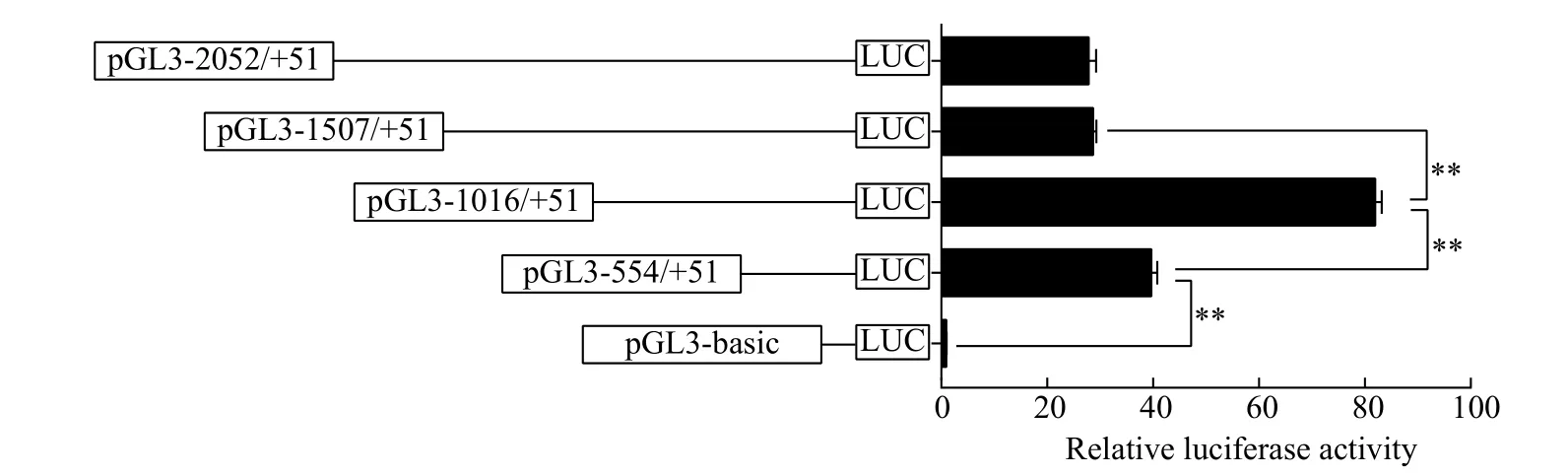
图 5 黄颡鱼5′ lal启动子区启动子活性Fig. 5 5′ unidirectional deletion analysis of the lal promoter region for P. fulvidraco
3 讨论
3.1 lal序列的分子特征及进化分析
本研究获得cDNA全长为1802 bp的黄颡鱼lal基因序列, 通过对其蛋白序列分析发现, LAL蛋白质结构拥有保守的α/β水解酶折叠类结构域, 一个活性位点(Gly172-His173-Ser174-Gln175-Gly176), 1个“帽子”结构域(Thr205-Val329)和“盖子”结构域(Phe233-Leu272), 2个氧阴离子孔(Leu89、Gln175), 催化三元体(Ser174、Asp344、His373)和3个半胱氨酸残基(Cys257、Cys265、Cys284), 及5个潜在的N-糖基化位点(Asn35-Ile36-Ser37、Asn101-Thr102-Ser103、Asn273-Met274-Thr275、Asn320-Gln321-Ser322、Lys161-Thr162-Thr163)。除此之外, LAL具有23个残基的信号肽, 能够将该蛋白输送到内质网中进行信号肽裂解和N-键糖基化[5,19]。在本研究中, 黄颡鱼具有5个N-糖基化位点, 其中除第三个Lys161-Thr162-Thr163糖基化位点以外, 其余4个在进化上都非常保守[20]。蛋白质的N-糖基化是一种新生肽链的共翻译或翻译后修饰方式, 对膜和分泌蛋白的折叠和稳定性调节具有重要意义[21]。其中, 由Asn-X-Thr/Ser组成的N-糖基化位点[22], 这3种氨基酸与高尔基体中甘露糖-6-磷酸结合, 能够识别出溶酶体表面的特定甘露糖受体[23]。当N-糖基化位点中Asn273发生突变时, LAL活性完全丧失[19], 这表明糖基化位点对LAL的功能、定位及活性至关重要。负责酶活性的催化三元体(Ser174、Asp344和His373)有2个位于蛋白质的C-末端区域, 当2个等位基因都存在时, 几乎所有的无义突变都会导致溶酶体酸性脂肪酶缺乏(Lysosomal acid lipase deficiency, LALD)[24]。在人的LAL蛋白质序列中, 半胱氨酸残基有助于提高LAL对底物的选择活性[25], 而人与黄颡鱼半胱氨酸数目不同, 可能由于物种的差异性。此外, 我们还发现黄颡鱼LAL氨基酸序列上存在一个“帽子”结构域和“盖子”结构域, Rajamohan等[26]认为“盖子”结构域打开, LAL活性被激活, 同时移动“盖子”, 使底物接近催化三元体Ser174[16]。Holmes等[27]认为来自灵长类等近亲物种的LAL半胱氨酸残基显示出高度的序列一致性,在我们的进化树分析中, 黄颡鱼lal与其他硬骨鱼类聚为聚为一只, 哺乳动物聚为另一大分支。
3.2 LAL蛋白三级结构分析
利用已知的脂肪酶结构模型来预测LAL的三级结构, 通过三级同源模型揭示了LAL结构的保守性[23,28], 我们的研究也显示LAL和dGL之间具有较高的氨基酸序列相似度(56.84%), 这使得基于dGL结构构建LAL的三级结构模型具有一定的现实意义。同时, Ataya[23]预测LAL的三级结构中带负电荷的Asp344与His373和Ser174形成氢键网络, 激活丝氨酸羟基。该结构隐藏在由Gln175和Leu89主链的NH基团组成的氧阴离子孔中, 它们通过形成2个短氢键为催化反应的过渡态提供了稳定的环境[29]。
3.3 lal序列的组织表达模式
了解基因的生理功能可以通过组织分布模式分析。本研究发现, 黄颡鱼lal基因在各组织中均有表达, 但在脾脏、肠道和精巢中高表达, 心脏和大脑表达量最低, 这与其他研究报道相同[30]。另外Ataya认为[23]lal在精巢中高表达的原因, 可能是由于精巢组织分裂活跃, 精子细胞膜脂双层的产生和降解需要持续进行。溶酶体酸性脂肪酶能为这一过程提供能量。
3.4 lal启动子序列及活性分析
RNA聚合酶能够识别真核生物的核心启动子,并与其结合后, 可调控基因的转录起始和效率, 保证基因转录的准确性和精确性[31]。通常基因的TATA盒位于转录起始位点的上游-50 bp至-100 bp区域[32], 并且除虹鳟(Oncorhynchus mykiss)和斑马鱼(Danio rerio)外, 类似TATA的基序(TTTAAA)在所有鱼类中都比较保守[33]。因此识别这些核心启动子是转录起始的一般机制的第一步[34]。在本研究中,lal核心启动子区存在CCAAT和TATA盒,分别位于-8 bp至-19 bp, -22 bp至-27 bp。黄颡鱼lal的核心启动子区存在下游核心元件(Downstream core element, DCE)、转录起始元件(Initiator, INR),从而保证基因转录的正常启动[31]。
基因的转录也受转录因子结合位点(Transcription factor binding site, TFBS)的调控作用[35]。根据在线软件预测, 黄颡鱼lal基因的启动子上存在Sp1(-87 bp/-101 bp)、STAT3(-233 bp/-243 bp)和PPARα(-286 bp/-313 bp)结合位点。本研究结果显示5′缺失突变分析结果显示lal的-1016 bp/+51 bp启动子区域正调控启动子活性, 这表明Sp1、STAT3和PPARα可能是lal基因启动子可能存在的正调控转录因子。Sp1属于C2H2型锌指蛋白家族, 它与富含GC的基序结合调节基因的表达[36], Sp1能够协同AP-2正调控lal转录表达[37]。STAT3作为一种转录因子, 正调控CPT Iα1b启动子活性[38]。PPARα是草鱼脂肪组织甘油三酯脂肪酶(Adipose triglyceride lipase, ATGL)基因启动子活性的顺式作用元件[39]。而LAL作为一种酸性脂肪酶, 将胆固醇酯(CE)和甘油三酯(TG)水解成游离的胆固醇(FC)和脂肪酸(FFA)[2], 我们可以推测STAT3和PPARα也是lal基因启动子活性的顺式作用元件。
本研究结果显示-1507/-1016区域负调控lal启动子活性。在这个区域中, 存在2个FOXO1结合位点(-1066 bp/-1076 bp、-1333 bp/-1343 bp)。FOXO1在调节代谢、应激反应和细胞凋亡中发挥中心作用, 研究表明FOXO1正调控lal的转录[4]。在本研究中, FOXO1结合位点处于黄颡鱼lal启动子的负调控区域, FOXO1可能是黄颡鱼lal基因潜在的负调控转录因子, 但需要在往后的点突变实验进行论证。另外, 在-2052 bp/-1507 bp区域存在1个PPARγ(-1755 bp/1774 bp)和1个HNF4α(-1758 bp/-1772 bp)位点, 值得注意的是P P A R γ结合位点正包含HNF4α结合位点, 这与You等[36]报道PPARα启动子上 也 存 在P P A R位 点 包 含H N F 4 α结 果 相 似。PPARγ作为脂质代谢过程中的一种重要转录因子,对脂肪细胞的分化和脂质储存具有重要作用[40], 同样HNF4α是一种核受体转录因子, 在肝脏特异性基因表达中起关键作用[41]。本研究也发现lal在肝脏的表达量比较高, 这提示我们PPARγ和HNF4α能够调节lal的转录表达, 但具体的调节方式有待深入探讨。
4 结论
本研究克隆得到了lal基因的cDNA全长和5′上游启动子序列, 为深入了解该基因的功能提供了基础。组织表达分析结果表明,lal在心、肝脏、脑、脾脏、肾脏、肌肉、肠系膜脂肪、肠及性腺等组织中都有表达, 这表明lal基因对黄颡鱼体内的代谢调控发挥重要的作用。启动子序列及活性分析, 发现lal核心启动区含TATA和CCAAT盒, 存在多个转录因子, 确定了lal启动子的-1507 bp/-1016 bp区域负调控启动子活性, -1016 bp/+51 bp区域正调控启动子活性, 表明不同的转录因子对lal的转录表达有不同作用。此研究为日后深入了解鱼类lal基因在脂质代谢及相关转录水平调控机制等研究提供科学依据。

