绣球染色体制片技术优化及rDNA物理定位*
郭瑞红 邱 帅 刘光欣 高 凯 魏建芬 席梦利
(1.南京林业大学 林木遗传与生物技术教育部重点实验室 南京 210037; 2.杭州市园林绿化股份有限公司 杭州 310020)
绣球(Hydrangeamacrophylla)隶属于虎耳草科(Saxifragaceae)绣球属(Hydrangea)。绣球属植物原产于日本和我国长江流域,共约73种,我国是世界绣球属植物的分布中心,拥有47个种和11个变种(海风等,2014;乔谦等,2020)。该属植物具有硕大饱满的聚伞花序、丰富多变的花色、适应性强、易扦插繁殖等特征,现已广泛用于鲜切花生产、园林造景和盆花栽培(吴可鹏等,2019)。目前,国内市场的主导商品栽培种多引自欧洲、美国及日本等国,我国虽然有丰富的绣球属种质资源,但这些资源尚未被充分开发利用,市场上仍缺乏具有自主产权的优良品种(乔谦等,2020)。绣球属新品种选育的主要方法仍然是传统的杂交育种,但其杂交技术复杂,种子微小,种子的收获及播种比较困难(曾奕等,2018),加之对绣球属植物的分类、系统进化和远缘杂交技术等基础研究工作开展较少,现有成果难以对育种实践起到辅助作用,导致我国的绣球属育种严重落后于欧美等发达国家。绣球属植物分子细胞遗传学研究可以揭示其染色体组成及特征,探究其系统进化关系,为绣球属杂交育种的亲本选配提供依据,从而指导绣球属植物的杂交育种。
荧光原位杂交(fluorescenceinsituhybridization,FISH)是20世纪80年代发展起来的一项分子细胞遗传学技术,该技术利用特异的DNA序列为探针,通过原位杂交方式将其杂交到特定染色体上,从而达到标定并识别不同染色体的目的。染色体特异序列探针FISH技术为染色体图谱的构建提供了高效方法(辛昊阳等,2016;Xinetal.,2020),已广泛应用于多种植物的分子细胞遗传学研究(Jiangetal.,2006;Lanetal.,2018),但绣球属物种的细胞学研究报道较少。已有研究结果显示,绣球属物种染色体较小,中期染色体平均长度1~2 μm,基因组2C值1.95~5.00 pg(Cerbahetal.,2001),部分物种中存在B染色体(Mortreauetal.,2010),二倍体及多倍体均有报道(Laereetal.,2010;Mortreauetal.,2010)。而基于FISH技术的研究仅查阅到1篇以45S rDNA为探针及1篇以45S rDNA和5S rDNA为探针的报道,上述研究揭示了绣球属物种间45S rDNA和5S rDNA在染色体上的分布具有多态性(Laereetal.,2010;Mortreauetal.,2010)。本研究以绣球水培根尖为材料,探讨不同预处理试剂、预处理时间和酶解时间对染色体形态及制片效果的影响,比较用45S rDNA和5S rDNA质粒间接标记的长探针与直接合成的45S rDNA和5S rDNA短探针FISH的信号差异,以期为绣球属植物分子细胞遗传学研究奠定基础。
1 材料与方法
1.1 植物材料
植物材料来源于南京林业大学校园内种植的绣球‘无尽夏’(Hydrangeamacrophylla‘Endless Summer’),2020年9—10月间,选取当年生半木质化枝条,置于光照培养箱内水培,培养条件为每天光照10 h、黑暗14 h,温度26 ℃,每天换水1次。
1.2 根尖取材及预处理
选择晴天上午9:00—10:00,收集水培绣球枝条上长0.5~1 cm的根尖。将根尖分别采用以下3种方法进行不同时间梯度预处理:1)根尖用1 MPa一氧化二氮(N2O)室温处理0.5、1、2 h;2)根尖置于0.7 mmol·L-1环己酰胺水溶液中室温处理1、3、5 h;3)根尖置于2 mmol·L-18-羟基喹啉水溶液中避光室温处理2、4、6 h。根尖经过预处理后,立刻用去离子水洗净,然后转入新鲜配制的卡诺固定液中(V无水乙醇︰V冰乙酸=3∶1)保存,次日重新换1次卡诺固定液,-20 ℃保存备用。
1.3 染色体制片及荧光原位杂交
1.3.1 染色体制片 染色体制片参照兰月(2018)的方法,并进行部分调整。根尖从固定液中取出后放入去离子水中,洗去卡诺固定液。切取根尖分生区组织,置于4%纤维素酶和2%果胶酶混合酶液中,在37 ℃恒温箱内分别酶解0.5、1、2 h。酶解后的分生组织用移液枪转移到水中终止酶解,再从水中转移至载玻片上。用镊子轻轻挤压出分生组织细胞,除去多余杂质,滴加20 μL 60%的乙酸,用解剖针混合搅匀组织。将载玻片置于温度为55 ℃的烘片机(Leica HI 1220)上,用解剖针涂抹细胞悬液,制片干燥前用新鲜配制的卡诺固定液冲洗后气干。相差显微镜下初步检片,选择染色体形态较好且分散的制片,-20 ℃保存备用。
1.3.2 染色体制片固定 为了改善DAPI染色效果,制片用4%多聚甲醛固定10 min,然后用2×SSC室温洗3次,每次5 min,再用70%、90%、100%乙醇进行逐级脱水各5 min。
1.3.3 探针制备 本研究用rDNA探针分为2类:一类为美国密西根州立大学蒋继明教授实验室惠赠的水稻(Oryzasativa)5S rDNA质粒和45S rDNA质粒,采用缺刻平移法分别用地高辛和生物素对其进行标记,为了描述方便,这类探针本文称为长探针;另一类为本团队依据拟南芥(Arabidopsisthaliana)5S rDNA和45S rDNA编码区序列合成的寡核苷酸(Oligo)探针,5S rDNA用FAM(6-carboxyfluorescein)修饰,45S rDNA用TAMRA(rhodamine)修饰,这类探针本文称为短探针(席梦利等,2018;Lanetal.,2018)。
1.3.4 杂交液制备及变性 每张染色体制片配制20 μL杂交液,杂交液成分包括约40 ng的探针DNA、50%去离子甲酰胺、2×SSC和10%硫酸葡聚糖。包含长探针的杂交液于98 ℃变性10 min后立即放入冰中冷却5 min以上;短探针是合成的单链DNA,故包含短探针的杂交液不需要变性。
1.3.5 染色体制片变性与杂交 染色体制片在85 ℃、70%去离子甲酰胺中变性2 min后,立即转移到70%、90%、100%冰乙醇中进行逐级脱水各5 min,制片气干。每张染色体制片滴加20 μL杂交液,用Rubber Cement封片。制片放置于湿盒中,37 ℃恒温箱内杂交过夜。
1.3.6 制片漂洗及抗体检测 长探针制片从湿盒中取出后,揭掉Rubber Cement,2×SSC室温洗5 min,42 ℃热洗20 min,1×TNT洗5 min。地高辛和生物素标记的探针分别用anti-digoxigenin rhodamine (Anti-Digoxigenin-Rhodamine Fab fragments,Roche,11207750910)和anti-biotin FITC(Fluorescein Anti-Biotin,Vector Laboratories,SP-3040)进行抗体检测。每张制片加约100 μL抗体孵育液(取anti-digoxigenin rhodamine和anti-biotin FITC各1 μL加入到100 μL的1×TNT中混匀),37 ℃恒温箱内孵育1 h。然后进行第2次洗脱,1×TNT洗3次,每次5 min,1×PBS洗5 min,晾干制片。每张制片用20 μL含有DAPI的封片胶(Vector Laboratories,H-1200)封片。制片在Olympus BX51型荧光显微镜下观察拍照,所有拍摄图片用Photoshop CS6与Image J软件进行后期处理与图片合成。
短探针带有荧光基团,杂交完成后可直接在荧光显微镜下检测拍照。短探针杂交完成后,揭掉Rubber Cement,用2×SSC室温洗5 min,洗掉盖玻片,然后用1×TNT漂洗3次,每次5 min,用1×PBS洗5 min,晾干制片。每张制片用20 μL含有DAPI的封片胶封片。制片在Olympus BX51型荧光显微镜下观察拍照,所有拍摄图片用Photoshop CS6与Image J软件进行后期处理与图片合成。
1.4 数据统计与分析
在研究不同预处理对染色体形态的影响时,为了避免根尖生理状态对试验的影响,每种处理制备10张染色体制片,每张制片用2个根尖,选择其中3张分裂相较多的制片进行数据统计,不同处理统计的分裂相数有30~64个。为了减少误差,每张染色体制片在显微镜下进行3次重复观察,分别统计出分裂相总数和染色体形态良好的分裂相数,采用Excel软件进行计算分析,得出平均值和标准差。形态良好的中期分裂相占比(%)=形态良好的中期分裂相数/总分裂相数×100%,形态良好的中期分裂相是染色体数目足够且染色体浓缩适当的分裂相。平均值±标准偏差(%)是3张染色体制片中形态良好的中期分裂相占比的平均值和标准偏差。
2 结果与分析
2.1 不同预处理对染色体形态的影响
采用1 MPa N2O、0.7 mmol·L-1环己酰胺和2 mmol·L-18-羟基喹啉3种方法对绣球根尖进行不同时间梯度预处理,观察统计发现:1 MPa N2O预处理0.5 h,大部分中期分裂相染色体较长,浓缩不充分(图1A),形态良好的中期分裂相比例为46.67%(表1);预处理1 h,染色体浓缩适当,大部分染色体的着丝粒位置可辨(图1B),形态良好的中期分裂相比例达58.8%(表1);预处理2 h,染色体浓缩过度,呈点状(图1C),形态良好的中期分裂相比例为50%(表1)。0.7 mmol·L-1环己酰胺预处理1 h和3 h,大部分中期分裂相染色体较长,浓缩不充分(图1 D、E),形态良好的中期分裂相比例分别为31.25%和52.39%(表1);预处理5 h,染色体浓缩适当,但大部分染色体的着丝粒位置不可辨(图1F),形态良好的中期分裂相比例为26.37%(表1)。2 mmol·L-18-羟基喹啉预处理2、4、6 h,形态良好的中期分裂相比例分别为32.5%、45.24%和34.92%,3个预处理时间染色体浓缩适当,但大部分染色体的着丝粒位置不可辨(图1G、H、I),处理2 h的染色体可以观察到异染色质(图1G)。综合考虑染色体长度、着丝粒位置及形态良好的中期分裂相比例,认为1 MPa N2O处理1 h是绣球根尖预处理的最适条件。
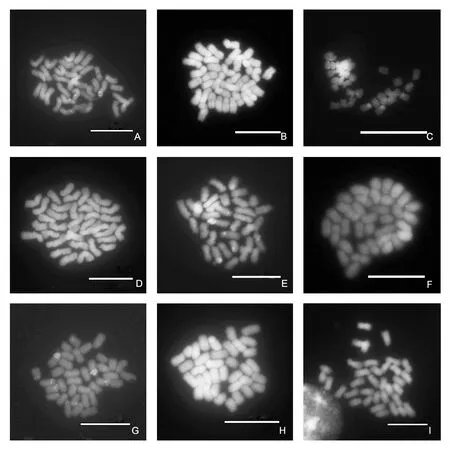
图1 不同预处理对染色体形态的影响Fig.1 Effect of different pretreatment methods on chromosome morphology标尺 Scale bars:10 μm.A-C:N2O分别预处理0.5、1、2 h;D-F:环己酰胺分别预处理1、3、5 h;G-I:8-羟基喹啉分别预处理2、4、6 h。A-C:N2O pretreated for 0.5,1 and 2 h,respectively;D-F:Cycloheximide pretreated for 1,3 and 5 h,respectively;G-I:8-hydroxyquinoline pretreated for 2,4 and 6 h,respectively.
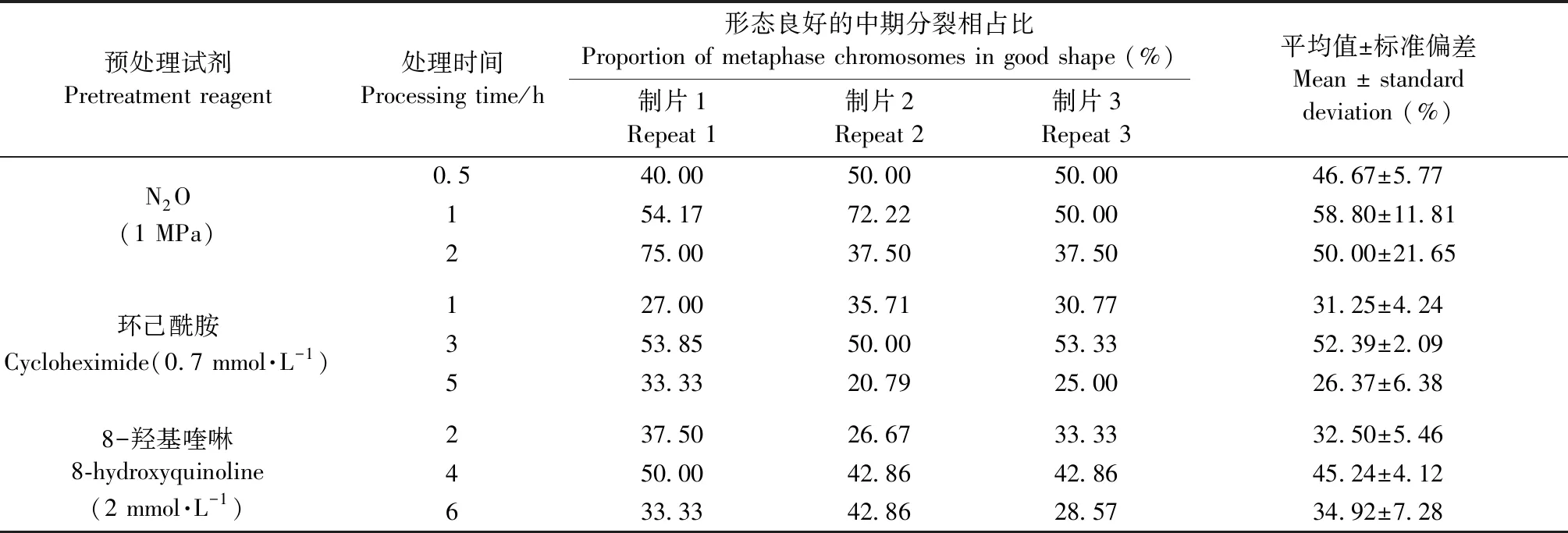
表1 预处理对根尖细胞染色体制片效果的影响Tab.1 Effect of pretreatment on chromosome preparation of root tip cells
2.2 不同酶解时间对染色体制片的影响
选用1 MPa N2O预处理1 h的根尖,将根尖分生组织用4%纤维素酶和2%果胶酶混合酶液于37 ℃分别酶解0.5、1、2 h后涂片,制片用卡宝品红染色后,在Olympus BX41显微镜下观察拍照。观察发现:酶解0.5 h获得的制片,中期分裂相和细胞核周围都可观察到明显的细胞质(图2A、B),这些细胞质的存在使染色体不能充分外露,在后续的FISH试验中容易产生背景信号,从而影响FISH信号的捕获;酶解1 h获得的制片,中期分裂相背景干净、染色体分散程度好(图2C);酶解2 h获得的制片,染色体容易丢失,观察到很多不完整分裂相(图2D)。对比后发现,酶解1 h所获得的染色体制片质量明显高于酶解0.5 h和2 h,因此,绣球根尖分生组织在4%纤维素酶和2%果胶酶混合酶液中37 ℃酶解1 h比较合适。
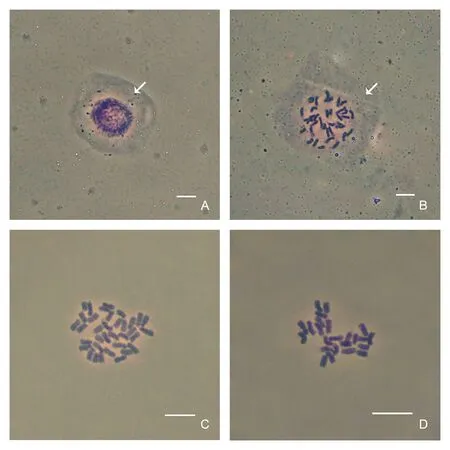
图2 不同酶解时间对染色体制备效果的影响Fig.2 Effect of different enzymatic hydrolysis time on chromosome preparation标尺 Scale bars:10 μm.箭头所指代表细胞质。The arrow points to the cytoplasm.A&B:0.5 h;C:1 h;D:2 h.
2.3 长探针和短探针rDNA对FISH结果的影响
选用1 MPa N2O处理1 h的根尖,酶解1 h后用涂片法制备的高质量中期染色体制片进行FISH对比试验。观察30个中期分裂相后发现:绣球染色体数目为36,染色体基数为18,是二倍体(2n=2x=36)。45S rDNA的长探针和短探针均可产生良好的杂交信号,2种探针产生的杂交信号强度差异不明显,几乎每个分裂相都可检测到2个很强的杂交信号。这说明45S rDNA的长探针和短探针均可用于绣球45S rDNA在染色体上的物理定位研究。多个分裂相对比确定,45S rDNA位于1对近端着丝粒染色体的短臂末端(图3)。5S rDNA的长探针和短探针产生的杂交信号均很弱,长探针在50%的中期分裂相上可以检测到2个清晰的信号,而短探针仅在37%的中期分裂相上可以检测到2个稍弱的信号,且多数分裂相上都呈现明显的背景信号。多个分裂相对比确定,5S rDNA位于1对端着丝粒染色体的长臂末端(图4)。
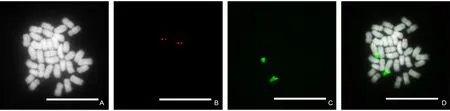
图3 长探针FISH结果Fig.3 The FISH results of long probes标尺 Scale bars:10 μm.A:中期染色体灰度图像;B:5S rDNA信号;C:45S rDNA信号;D:染色体与rDNA信号合成图。A:Grayscale image of metaphase chromosomes;B:Signals of 5S rDNA;C:Signals of 45S rDNA;D:Merged picture from A,B and C.
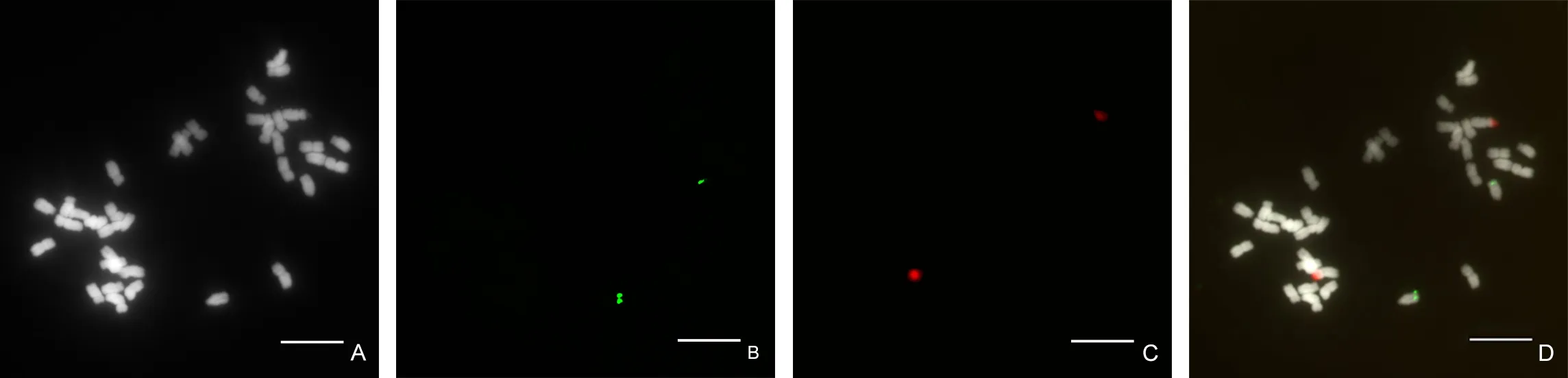
图4 短探针FISH结果Fig.4 The FISH results of short probes标尺 Scale bars:10 μm.A:中期染色体灰度图像;B:5S rDNA信号;C:45S rDNA信号;D:染色体与rDNA信号合成图。A:Grayscale image of metaphase chromosomes;B:Signals of 5S rDNA;C:Signals of 45S rDNA;D:Merged picture from A,B and C.
3 讨论
获得高质量染色体制片是开展分子细胞遗传学研究的基础,高质量染色体制片不仅要求染色体浓缩适当、形态良好、着丝粒位置易辨,而且要求分裂相没有细胞质背景,染色体分散性好,以便进行后续的测量分析或FISH试验。植物材料的预处理及制片方法对染色体制片质量影响显著,在开展细胞学研究时需要针对不同的植物材料,探索预处理及制片方法对染色体制片质量的影响。根尖分生组织比其他分生组织细胞分裂更活跃、材料获取更容易,因此,根尖是细胞学研究的常用材料(林秀琴等,2011;杨亚飞等,2020),加之绣球扦插易生根,故本研究选用根尖作为试验材料,探索不同预处理方式、预处理时间及酶解时间对染色体形态及制片效果的影响。细胞学研究中富集中期染色体的常用预处理有物理方法和化学方法。N2O预处理属于物理方法,主要通过高压使染色体凝缩,该处理方法已在马铃薯(Solanumtuberosum)、玉米(Zeamays)、小麦(Triticumaestivum)、柑橘(Citrus)等植物中成功获得了形态良好的中期染色体(Brazetal.,2018;Martinsetal.,2019;Songetal.,2020;Heetal.,2020)。本研究采用1 MPa N2O预处理绣球根尖1 h,也获得了形态良好的中期分裂相占比高、着丝粒位置可辨的染色体制片。环己酰胺和8-羟基喹啉预处理属于化学方法,通过抑制或破坏纺锤丝的形成,使染色体处于中期。本文研究团队采用环己酰胺预处理曾在百合(Lilium)、郁金香(Tulipa)、杨树(Populus)等植物中取得了很好的效果(Liuetal.,2017;Lanetal.,2018;Xinetal.,2020),但预处理绣球根尖后,制片中大部分染色体的着丝粒位置不可辨。尽管已有报道认为8-羟基喹啉可使染色体的缢痕和次缢痕清晰可见(刘丹等,2015;官锦燕等,2020;叶天文等,2020),但8-羟基喹啉预处理绣球根尖后,其大部分中期染色体的缢痕不可辨,这可能是由于不同植物根尖对不同预处理方式的敏感程度不同所致。比较分析发现:1 MPa N2O预处理1 h的绣球根尖,用4%纤维素酶和2%果胶酶混合酶液于37 ℃酶解1 h后涂片,是获得高质量绣球根尖染色体制片的最佳试验流程。
rDNA(45S rDNA和5S rDNA)为探针的FISH技术,是进行植物染色体识别、植物分类及植物系统进化等领域研究的强有力工具。传统的rDNA-FISH技术,多将模式植物拟南芥、水稻或小麦的45S rDNA和5S rDNA分别构建在质粒载体中,通过大肠杆菌(Escherichiacoli)繁殖克隆质粒DNA、质粒DNA提取、缺刻平移法标记探针等步骤获得可用于FISH的rDNA探针。该方法获得的rDNA探针为长度100~300 bp的双链DNA(即长探针)。近年来,随着DNA合成技术的迅猛发展,依据rDNA的序列特征,开发出了长度20~59 bp的rDNA单链寡核苷酸(Oligo)探针(即短探针),与传统的长探针构建过程相比,短探针具有纯度高、合成成本低、探针长度一致、使用简便、杂交效率高等特点(Tangetal.,2014)。为了对2种探针的杂交效果进行比较,本研究采用长、短探针分别在绣球根尖中期染色体上进行原位杂交试验,结果发现45S rDNA长、短探针的杂交效果相似,5S rDNA长探针的检出率高于短探针,但短探针也可以达到试验目的。鉴于短探针具有试验便捷、成本低廉的特点,因此,推荐选用短探针进行绣球rDNA在染色体上的物理定位研究。
绣球属植物的染色体基数、倍性及rDNA杂交信号在染色体上的分布变化较大。多数绣球属植物染色体基数为18,二倍体36条染色体(2n=2x=36),栽培品种存在大量的三倍体(2n=3x=54)和四倍体(2n=4x=72),但长叶绣球(Hydrangeainvolucrata)染色体基数为15(2n=2x=30),马桑绣球(H.aspera)染色体基数为17(2n=2x=34)(Mortreauetal.,2010)。45S rDNA在绣球属植物均位于染色体的短臂末端,圆锥绣球‘Unique ’(H.paniculata‘Unique’)分布在2对染色体上,栎叶绣球‘Sikes Dwarf’(H.quercifolia‘Sikes Dwarf’)和马桑绣球分布在2对染色体上,而绣球‘Fasan’(H.macrophylla‘Fasan’)、长叶绣球及本研究的绣球‘无尽夏’均位于1对染色体上(Laereetal.,2010;Mortreauetal.,2010)。5S rDNA在绣球属植物均分布在1对染色体上,长叶绣球和粗枝绣球(H.robusta)位于近着丝粒的位置,紫彩绣球(H.sargentiana)及本研究的绣球‘无尽夏’位于长臂末端(Laereetal.,2010;Mortreauetal.,2010)。这就意味着rDNA在绣球属植物中的分布具有广泛的多态性,可以对绣球属植物的分类提供参考。
国内外学者在绣球属植物的系统发育方面已开展了一些研究工作,但绣球属植物种类繁多、形态变异复杂,基于形态学和不同分子标记揭示的种间亲缘关系仍有不少分歧(张梅等,2021)。rDNA在染色体上的分布特征是揭示物种间亲缘关系的有效手段,其在染色体上分布的位点数量及分布位置越相似,说明其亲缘关系越近(翁天均,2011)。因此,未来应在多种绣球属植物中开展rDNA物理定位研究,通过比较分析rDNA在染色体上的分布特点、染色体基数及倍性,揭示绣球属物种间的亲缘关系,以辅助绣球属植物的杂交育种实践。
4 结论
绣球根尖用1 MPa N2O预处理1 h,根尖分生组织置于4%纤维素酶和2%果胶酶混合酶液中在37 ℃酶解1 h后用涂片法制片,获得的中期分裂相染色体浓缩适当,背景干净,染色体分散程度好,大部分染色体的着丝粒位置可辨,该方法可以获得高质量染色体制片。45S rDNA长、短探针的FISH效果相似,5S rDNA长探针的检出率高于短探针。但由于短探针具有试验便捷、成本低廉的特点,因此,推荐选用短探针进行绣球rDNA在染色体上的物理定位研究。本研究建立的绣球根尖高质量中期染色体制备及rDNA物理定位技术体系可高效分析鉴定绣球属植物的染色体数目及rDNA的分布,为绣球属杂交育种的亲本选配提供依据,从而指导绣球属植物的杂交育种工作。

