精子变形过程中组蛋白–鱼精蛋白替换调控机制
袁露,葛婷婷,牛长敏,徐文华,郑英
精子变形过程中组蛋白–鱼精蛋白替换调控机制
袁露1,2,葛婷婷1,2,牛长敏1,2,徐文华1,2,郑英1,2
1. 扬州大学医学院组织学与胚胎学教研室,扬州 225009 2. 江苏省非编码RNA基础与临床转化重点实验室,扬州 225009
精子形成是精子发生的最后阶段,圆形精子细胞经历了一系列的形态变化和染色质凝聚,形成了具有物种特异性的成熟精子。组蛋白–鱼精蛋白替换是精子形成过程中的重要事件。在组蛋白–鱼精蛋白替换过程中,组蛋白首先被睾丸特异性的组蛋白变体所替代,随后过渡蛋白整合到细胞核中,最后过渡蛋白被鱼精蛋白取代。组蛋白–鱼精蛋白替换缺陷可能导致无精子症、少精子症或畸精症,从而导致男性不育。本文系统总结了组蛋白–鱼精蛋白替换过程中的调控机制研究进展,以期为男性不育症的诊断和治疗提供理论基础。
精子发生;组蛋白–鱼精蛋白替换;翻译后修饰
在精子发生过程中,精原干细胞(spermatogonial stem cells, SSC)经历自我更新并分化为精原细胞,精原细胞通过有丝分裂产生初级精母细胞,后者再经过两次连续的减数分裂产生单倍体精细胞,再经过精子变态产生成熟的精子。在精子形成过程中,一个极为明显的形态学特点是精子头部圆球形的细胞核逐渐转变为卵圆形或镰刀状的细胞核。精子头部塑形是精子变形过程中的核心事件,而组蛋白–鱼精蛋白替换是其中的重要步骤之一。在这个过程中,大多数组蛋白首先被睾丸特异的组蛋白变体取代,随后过渡蛋白(transition proteins, TP)被整合到精子细胞的细胞核中,鱼精蛋白(protamines, PRM)进一步取代晚期精子细胞中的TP,将基因组包装到高度浓缩的精子核中[1]。目前已经发现许多基因参与组蛋白–鱼精蛋白替换调控,这些基因的缺失或突变可能会导致组蛋白–鱼精蛋白替换缺陷,从而导致精子形成异常,雄性生育力下降甚至不育(表1)。目前,新的研究发现对过去的一些结论也提出了挑战与质疑,一些调控因子在组蛋白–鱼精蛋白替换过程中的作用还有待进一步深入探索。本文系统总结了组蛋白–鱼精蛋白替换过程中的调控机制研究进展,以期为男性不育症的诊断和治疗提供理论基础。
1 组蛋白的掺入、解聚与降解
1.1 组蛋白变体的掺入产生高度不稳定的核小体
在真核生物中,核小体是DNA的基本结构单位,包含4种核心组蛋白(H2A、H2B、H3和H4)和连接组蛋白H1。在精子发生的组蛋白–鱼精蛋白替换过程中,多种组蛋白变体呈时空特异性表达,在多种翻译后修饰(post-translational modification, PTM)的参与下产生高度不稳定的核小体(图1)。
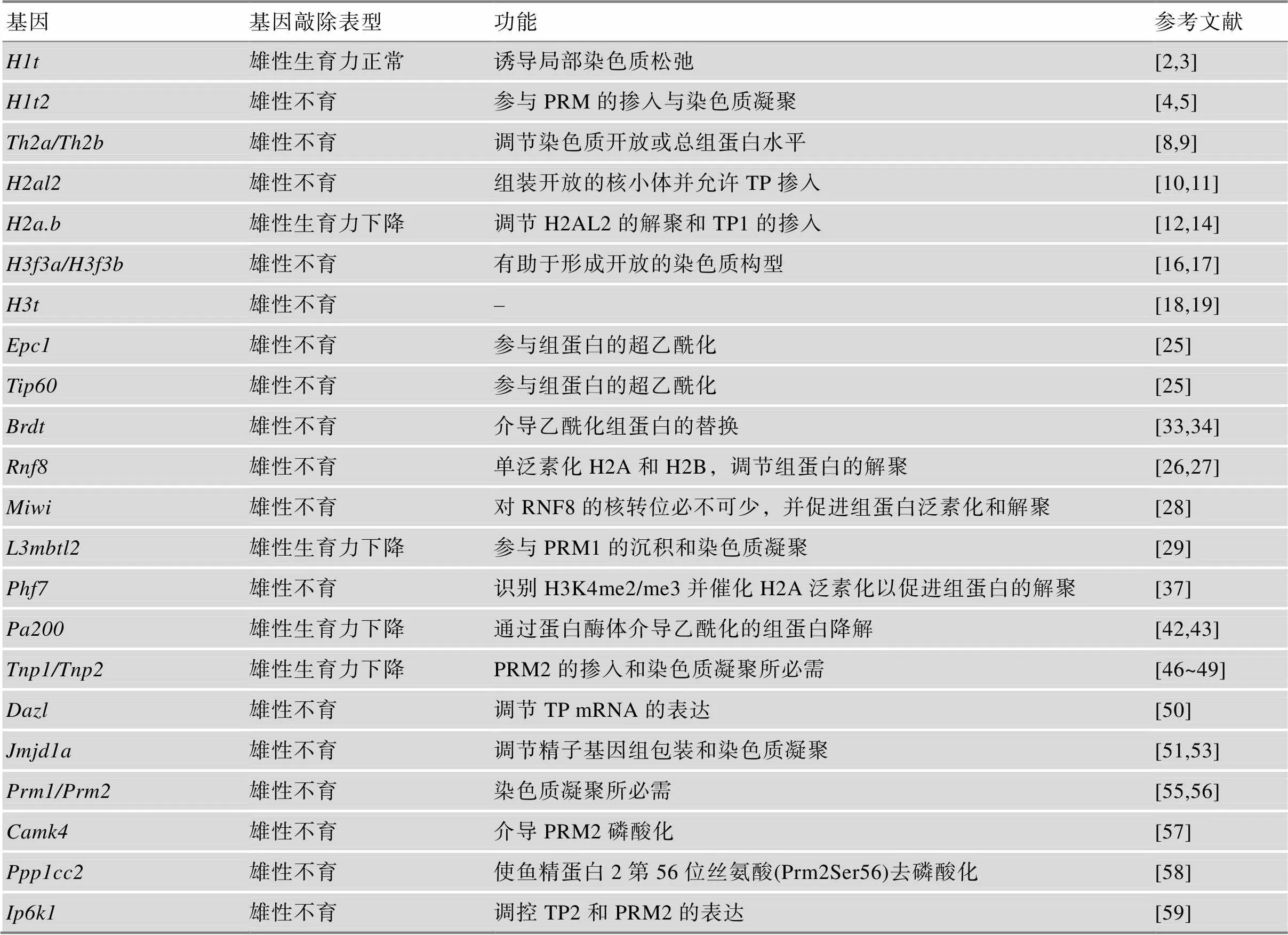
表1 组蛋白–鱼精蛋白替换调控相关基因
1.1.1 H1变体
在哺乳动物中,大约有11种不同的组蛋白H1亚型,包括体细胞和睾丸特异性亚型。其中H1T、H1T2和HILS1是睾丸特异表达的H1变体。在小鼠()睾丸中,H1T在中晚期的粗线期精母细胞至早期单倍体细胞中表达,但基因敲除小鼠组蛋白–鱼精蛋白替换正常,精子发生过程不受影响,小鼠具有正常的生育能力。但其他H1亚型的表达有所升高,可能是对基因缺失的完全代偿[2]。最近的研究表明,H1T可能诱导局部染色质松弛,以募集抑制异染色质和转座因子(transposable element, TE)所必需的蛋白质因子,最终的生物学效应是形成封闭的染色质抑制结构[3]。
H1T2是另一种睾丸特异性的组蛋白H1变体,定位于圆形精子细胞核顶极。H1T2首先在IV期的圆形精子细胞中检测到弱表达,在V-VIII期表达明显增加。H1T2在细胞核内的定位高度极性,集中表达于核膜内周的帽状结构中,显示了精子细胞核内固有的极性。但H1T2极性定位机制尚不清楚[4]。与H1T不同,H1T2对精子发生至关重要。纯合突变雄性小鼠由于精子细胞的核凝聚异常和伸长异常而不育,在H1T2缺失的精子中,鱼精蛋白水平显著降低,说明H1T2在鱼精蛋白取代组蛋白的过程中起着关键作用[4,5]。序列分析表明,大鼠()H1T2蛋白的C-末端结构域中存在Walker基序、SR结构域和卷曲螺旋结构域。免疫沉淀和质谱分析表明,H1T2相关的染色质结构域存在H4乙酰化等其他组蛋白的翻译后修饰(post-translational modification, PTM),其中H4乙酰化是启动组蛋白解聚的关键,表明H1T2参与精子发生中染色质重塑的启动,并进一步促进组蛋白–鱼精蛋白替换。H1T2相关染色质的相互作用蛋白主要为核骨架成分、RNA结合蛋白及其伴侣蛋白。这些发现为后续研究精子发生过程中染色质重塑的分子机制奠定了基础[6]。
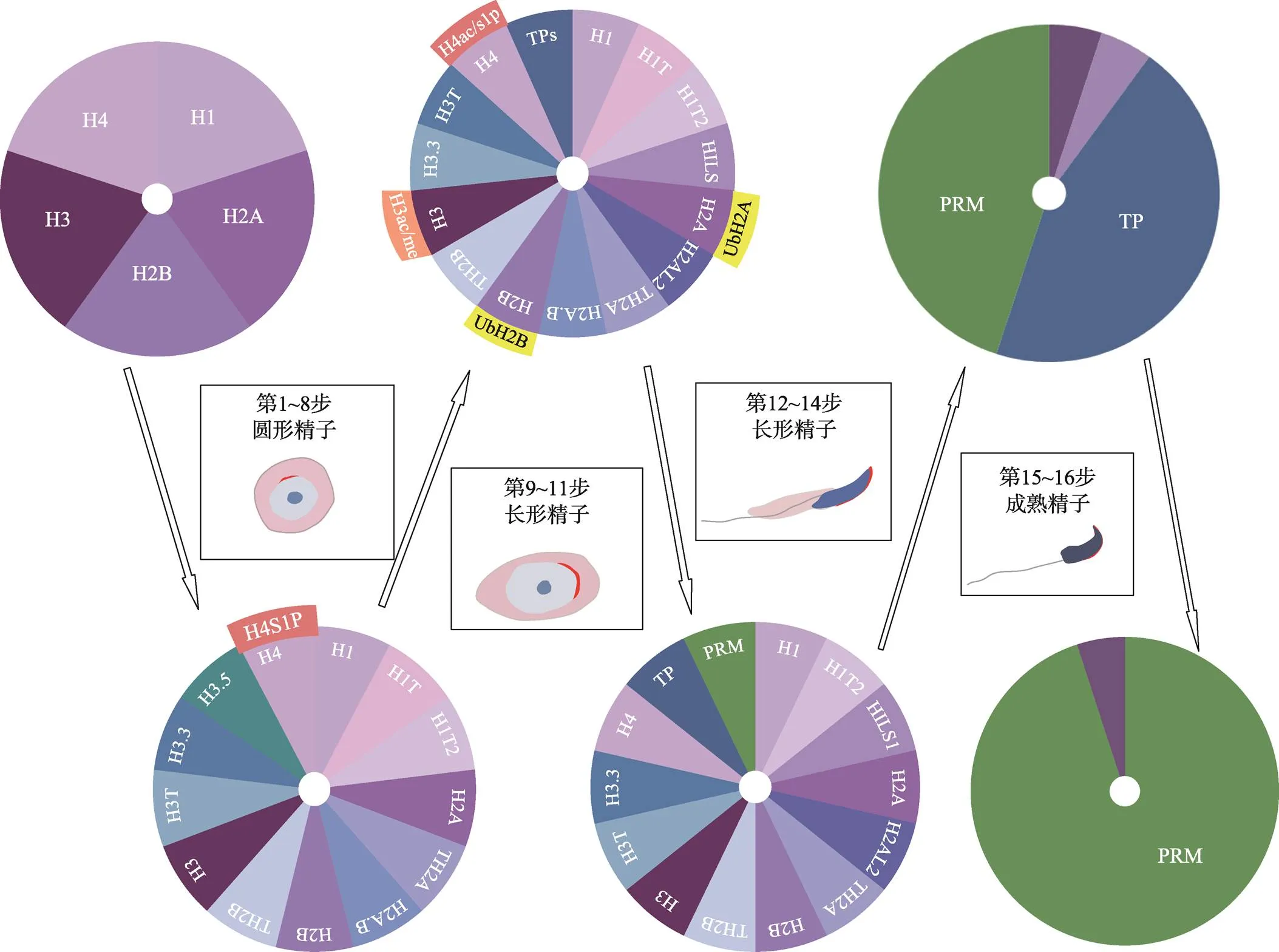
图1 组蛋白–鱼精蛋白替换的关键时间点及翻译后修饰
HILS1 (spermatid-specific linker histone H1-like protein, HILS1)是保守程度最低的H1变体,在组蛋白变体H1T消失的同时,精子细胞染色质中出现了HILS1。与其他组蛋白H1变体(如H1T和H1D)相比,HILS1具有显著较低的α-螺旋度,对DNA和染色质的凝集力较差。在哺乳动物精子发生的组蛋白替代过程中,HILS1可能在特定的位点为精子细胞提供了一个开放的染色质结构,用于其他碱性蛋白的募集和沉积[7]。生物信息学分析发现:小鼠基因由1个外显子组成,无内含子。目前尚未见其基因敲除小鼠的报道。
1.1.2 H2A和H2B变体
已经在哺乳动物中发现了多种睾丸特异的H2A和H2B组蛋白变体,包括TH2A、TH2B、H2AL1、H2AL2、H2AL3和H2A.B等。
TH2A和TH2B分别是组蛋白H2A和H2B的睾丸特异性变体,Shinagawa等[8]研究发现,在和双基因敲除小鼠中,TP2的染色质掺入受损,H2B表达水平升高,这表明TH2A和TH2B可能具有调节染色质开放或总组蛋白水平的功能,以促进精子形成期间的组蛋白替换。由于基因敲除可以通过组蛋白H2B表达增加和相应的翻译后修饰增加实现代偿,其基因敲除的雄性小鼠精子发生和生育力均正常[9],和双敲除雄性小鼠中的组蛋白替换缺陷可能是TH2A耗尽或它们的协同作用缺陷所致。
组蛋白H2A的变体H2AL2在减数分裂后的长形精子细胞中特异表达,且与TP的表达相关。Barral等[10]通过比较基因敲除的小鼠和野生型小鼠,证明H2AL2对于组蛋白–鱼精蛋白替换过程中核小体上TP的装载和PRM的高效掺入是必需的。后续研究发现在组蛋白解聚后,一小部分H2AL2在长形精子细胞核以及附睾中的精子中有保留。H2AL2具有靶向定位于染色质凝聚区域的特性,其定位受其N端RNA结合基序控制,这种特殊结构可能对受精后男性异染色质的结构和转录有重要影响,但其特异性保留在成熟精子染色质凝聚区域的具体机制仍未阐明[11]。
H2A.B在粗线期精母细胞到圆形精子细胞中均有表达[12]。体外研究表明,H2A.B能够破坏染色质的稳定性,并对染色质具有去折叠特性,说明H2A.B可能促进TP置换组蛋白[13]。Anuar等[14]用TALEN技术构建了基因敲除FVB/NJArc小鼠模型,发现由于精子形成异常和生精小管堵塞,基因敲除的雄性小鼠生育力下降。在H2A.B缺失的长形精子细胞中,在着丝粒周围的异染色质中无法检测到H2AL2,鱼精蛋白取代TP1的过程推迟。这些结果表明,H2A.B可能通过调节H2AL2和TP1染色质的掺入和解聚来参与组蛋白–鱼精蛋白替换。而在另一项研究中,基因敲除的C57BL6/J小鼠并未观察到H2A.B缺失对精子发生、精子或睾丸形态的影响;另外,研究还发现H2A.B的主要生物学作用似乎是在受精后的发育中,在完全缺乏H2A.B的情况下,胚胎在移植后的存活率会降低[15]。
1.1.3 H3变体
组蛋白H3具有多种变体,例如H3.1、H3.2、H3.3、H3T、CENP-A、H3.X、H3.Y和H3.5,除组蛋白变体H3.1和H3.2以外,H3.3、H3T和H3.5均在哺乳动物中表达,且在男性生殖中起重要作用[1]。
哺乳动物和两个基因均可编码H3.3,或基因敲除可导致雄性不育。基因敲除会产生异常精子,而基因敲除会导致小鼠生长缺陷和出生后死亡,存活的基因敲除雄性表现出完全不育[16]。H3f3b缺失的生殖细胞染色质重组异常,鱼精蛋白掺入减少,H3.3有助于形成开放的染色质构型,是染色质重组和组蛋白–鱼精蛋白替换所必需[17]。
Tachiwana等[18]研究表明,含有H3T的核小体可以形成更开放的构型,说明H3T可能在组蛋白–鱼精蛋白替换中起到开放染色质的作用。后续的研究发现,H3T仅在精母细胞中表达,在长形精子细胞中表达减弱。由于缺失小鼠睾丸中没有精母细胞和精子细胞,敲除可导致无精子症。因此,H3T在精子形成中的具体功能仍有待进一步研究[19]。
H3.5是组蛋白H3的变体之一,人H3.5表达于睾丸精原细胞和初级精母细胞中。体外研究表明,H3.5可降低核心组蛋白H4的疏水作用[20]。另有研究发现,H3.5在非梗阻性无精子症(non-obstructive azoospermia, NOA)患者中显著降低[21],但H3.5在精子发生中的确切作用目前仍不清楚。
1.2 组蛋白的解聚
组蛋白翻译后修饰普遍存在于精子发生染色质重塑过程中,组蛋白H4超乙酰化在促进染色质解聚和开放过程中起着至关重要的作用[22](图2)。H4乙酰化在精子发生过程中呈阶段性,H4K5ac、H4K8ac和H4K12ac在精原细胞和细线前期精母细胞中出现,在细线期到粗线期精母细胞中消失,在长形精细胞中重新出现,最后在成熟精子中消失,而H4K16ac只能在长形精子细胞中检测到[23,24]。
EPC1 (enhancer of polycomb homolog 1)/TIP60 (Tat-interactive protein, 60 kDa)等核小体乙酰化转移酶复合物参与组蛋白超乙酰化。EPC1和TIP60是哺乳动物H4的核小体乙酰基转移酶(nucleosome acetyltransferase of H4,NuA4)复合物的两个组成部分,共同定位于圆形精子细胞和长形精子细胞顶体附近的核外围,或的缺失扰乱了组蛋白的超乙酰化,特别是H4乙酰化,并影响了精子发生过程中的组蛋白替换[25]。
泛素–蛋白连接酶RNF8 (ring finger protein 8)在组蛋白–鱼精蛋白替换过程中有重要的调控作用。在基因敲除小鼠的成熟精子中可以检测到常规组蛋白,睾丸中泛素化的H2A和H2B降低,H4K16ac也显著减少[26]。进一步研究表明,RNF8主要在H2A和H2B的单泛素化中起作用,与组蛋白乙酰化酶 MOF (males absent of the first)协同诱导H4K16的乙酰化,从而调控组蛋白的解聚[27]。
RNF8的活性同样受到上游蛋白的调节。MIWI通过不依赖于piRNAs的方式与早期精子细胞胞浆中的RNF8结合,而泛素连接酶APC/C (anaphase promoting complex/cyclosome)介导的MIWI在晚期精子细胞中的降解是RNF8核转位的关键。D-box的杂合突变会导致MIWI泛素化修饰障碍从而无法降解,使得后期MIWI异常积累在胞质中,而泛素–蛋白连接酶RNF8由于与MIWI蛋白相互作用,也被滞留在精子细胞胞质中,无法进入胞核启动组蛋白H2A和H2B的泛素化修饰和后期组蛋白–鱼精蛋白的替换[28]。L3MBTL2 (lethal (3) malignant brain tumor like 2)也可与RNF8相互作用。L3MBTL2是与染色质凝聚相关的MBT结构域蛋白之一,L3MBTL2条件性基因敲除小鼠畸形精子数量增多、精子数进行性下降,睾丸早衰。L3MBTL2缺乏还导致长形精子细胞中RNF8和组蛋白泛素化水平降低,从而进一步影响精子发生过程中PRM1的掺入和染色质凝聚[29]。
值得注意的是,RNF8在组蛋白–鱼精蛋白替换过程中的作用仍存在争议。有研究认为,RNF8主要在减数分裂过程中性染色体的泛素化和减数分裂后精子细胞中性染色体相关基因的激活中具有重要功能[30]。最近也有研究报道:RNF8既与组蛋白解聚的启动信号H4K16乙酰化无关,也不影响鱼精蛋白的掺入,RNF8并不是组蛋白–鱼精蛋白替换所必需[31]。RNF8在组蛋白–鱼精蛋白替换过程中的作用还需进一步验证。
组蛋白乙酰化可能被一些染色质重构体识别以传递下游信号。BET (bromodomain and extra-terminal)蛋白家族是BRD (bromodomain proteins)家族的一个亚类,包括广泛表达的BRD2、BRD3、BRD4及正常情况下仅表达于睾丸组织的BRDT。BRDT能特异性识别乙酰化赖氨酸残基,是可以与乙酰化组蛋白结合并调节精子发生中染色质结构和组织变化的关键表观遗传阅读器[32]。BRDT与超乙酰化组蛋白H4尾部结合,并与乙酰化的H4共同定位在长形精子细胞中[33]。在敲除的BD1结构域小鼠的长形精子细胞中,TP和PRM保留在细胞质中,没有发生组蛋白替换,这表明BRDT介导乙酰化组蛋白的替换是实现组蛋白到鱼精蛋白的替换所必需的[34]。此外,有证据表明BRDT可与SMARCE1 (SWI/SNF- related matrix-associated actin-dependent regulator of chromatin subfamily E member 1)的N端结合,SMARCE1是ATP依赖的染色质重塑复合物SWI/ SNF家族的成员,BRDT可能与SMARCE1共同作用,启动组蛋白解聚和被TP取代的过程[35]。
PHF7 (PhD finger protein 7)是小鼠睾丸中一种H2A泛素化连接酶。PHF7特异性地定位于长形精子细胞核中,基因敲除会导致精子数量的减少和异常精子比例的增加,雄性小鼠不育。PHF7可通过其PHD结构域识别H3K4me3/me2,并通过其环区催化H2A泛素化。在PHF7缺失的精子细胞中,H2A泛素化显著降低,导致组蛋白保留和鱼精蛋白替换缺陷[37]。还有研究发现,基因敲除会导致早期精子细胞中BRDT的表达降低,精子细胞中组蛋白滞留;通过进一步实验发现PHF7介导组蛋白泛素化有助于稳定BRDT,从而促进精子细胞中组蛋白的解聚[38]。
综上所述,由EPC1/Tip60或其他一些核小体乙酰化转移酶复合物介导组蛋白超乙酰化,然后关键赖氨酸的乙酰化进一步破坏了核小体的稳定,组蛋白H4尾部的超乙酰化为BRDT的招募提供了一个平台。BRDT与SWI/SNF家族蛋白相互作用,启动组蛋白解聚和被TP取代的过程(图2)。
1.3 组蛋白解聚后的降解
泛素(ubiquitin, Ub)是由76个氨基酸组成的蛋白质,通过附着在靶蛋白上,调节蛋白质降解、自噬、DNA损伤反应等细胞过程。泛素-蛋白酶体途径(ubiquitin–proteasome pathway, UPP)主要由泛素、泛素激活酶(ubiquitin-activating enzyme, UBA, E1)、泛素结合酶(ubiquitin-conjugating enzyme, UBC, E2)、泛素–蛋白质连接酶(ubiquitin-protein ligase, E3)及蛋白酶体组成,在整个生精过程中起着至关重要的作用[39]。泛素结合酶UBC4是E2酶的睾丸特异性亚型,在圆形精子细胞和早期长形精子细胞表达。UBC4与睾丸特异的泛素连接酶LASU1在精原细胞至早期精母细胞促进组蛋白泛素化,但可能与靶向组蛋白通过蛋白酶体降解无关[40]。在大鼠长形精子细胞中组蛋白多泛素化的位点也与蛋白酶体在细胞核的定位不一致[41]。这样,多泛素化可能并不能介导组蛋白通过蛋白酶体降解。

图2 组蛋白-鱼精蛋白替换的调控机制
此外,PA200是20S蛋白酶体的激活剂,在睾丸中高表达,基因敲除导致严重的精子发生缺陷,雄性小鼠生育力大幅下降[42]。在精子发生过程中,PA200能显著促进乙酰化组蛋白的非ATP依赖性蛋白酶体降解,但不能促进多泛素化蛋白的降解[43]。因此,PA200特异性地识别乙酰化组蛋白,并在精子发生过程中通过蛋白酶体介导乙酰化的组蛋白降解。睾丸特异性蛋白酶体亚基α4s定位于联会复合体的中心区,可能促进PA200与核心颗粒的组装,其缺失会导致PA200显著降低[44]。
2 过渡蛋白与鱼精蛋白促进染色质凝聚
2.1 过渡蛋白在组蛋白–鱼精蛋白替换中的作用及调控机制
以前的研究普遍认为:精子细胞核中组蛋白解聚和鱼精蛋白掺入过程中,90%的组蛋白被TP1和TP2取代,随后鱼精蛋白取代了过渡蛋白[45]。但最近的研究报道了 TP有助于浓缩精子DNA的另一种机制。组蛋白变体H2AL2被结合到核小体中,并通过开放核小体来启动转换过程,随后TP与H2AL2及精子DNA结合,以促进鱼精蛋白的结合和加工[10]。
TP1和TP2分别由和编码,功能相似,但并不完全相同。TP碱性区域在体外与裸露的DNA结合并浓缩,但TP2对DNA的亲和力强于TP1。在小鼠中,TP1的表达量是TP2的2倍左右。或的单一敲除都会导致雄性小鼠生育力下降和轻微的精子形态变化,在缺失和缺失的精子细胞中分别观察到TP2和TP1蛋白升高[46,47]。而和双基因敲除小鼠表现出精子尾部形态异常,活力普遍下降,雄性不育。同时可以检测到严重的组蛋白保留,染色质凝聚受到干扰[48]。这可能是由于TP1和TP2可以相互代偿,因而单基因敲除或不会影响精子形成。在组蛋白–鱼精蛋白替换过程中,TP在染色质凝聚、鱼精蛋白掺入等过程中发挥重要作用。
随着染色质的浓缩,染色质的转录机制逐渐沉默。TP在精子发生后期短暂表达,这种表达必须从生殖细胞发育的早期转录和储存的mRNA中产生,TP表达的时机对生育能力至关重要。有研究发现TP2 mRNA在圆形精子细胞中提前翻译会导致雄性小鼠不育,相关的翻译调控因子对TP在精子发生过程中产生重要作用[49]。Phillips等[50]利用蛋白质组学方法,确定了6种与TP mRNA相关的蛋白质:CSDE1、ELAVL1、IGF2BP3、LRPPRC、MATR3和DAZL。通过免疫荧光发现这些候选的TP mRNA调控因子在表达TP的mRNA的生殖细胞中表达,说明这些mRNA调控因子可能对TP的表达产生重要的调控作用。由于大多数RNA调节蛋白控制多种不同的RNA,RNA调节蛋白作用于TP mRNA的分子机制还有待进一步研究。
和基因的表达受JMJD1A (Jumonji domain-containing 1A, JMJD1A)调控,JMJD1A是一种组蛋白去甲基化酶,可特异性地使H3K9me1/me2去甲基化,而不会使H3K9me3去甲基化。有研究认为JMJD1A可直接与和基因启动子上甲基化的H3K9相结合,从而进一步调节精子的染色质凝聚[51]。基因敲除小鼠出现生殖细胞广泛凋亡,严重的少精子症、小睾丸,雄性不育。生殖细胞中组蛋白乙酰化水平降低,cAMP反应元件调节器(cAMP response element modulator, CREM)的募集减少,CREM共激活因子及其靶基因、、和的表达减少,而这些蛋白是精子染色质凝聚所必需的[52]。这些结果表明,Jmjd1a控制的组蛋白修饰对于CREM调控的基因表达和精子发生是至关重要的。Eelaminejad等[53]研究发现,JMJD1A在患有圆形精子细胞成熟停滞(round spermatid maturation arrest, SMA)的人睾丸中低表达,提示JMJD1A可能在人中有类似的作用,其表达不足可能是SMA的原因之一。
2.2 鱼精蛋白替代过渡蛋白并压缩染色质
鱼精蛋白是在晚期精子细胞中取代TP的基本蛋白质[54]。鱼精蛋白通过一个中心富含精氨酸的DNA结合域与DNA相互作用,高水平的精氨酸使其与DNA强烈结合,而半胱氨酸促进鱼精蛋白间和鱼精蛋白内二硫键的形成,促进染色质凝聚[45]。
与基因不同,或基因的缺失会导致小鼠雄性不育[55]。精蛋白有多个PTM位点,目前在小鼠精子精蛋白上共发现了11个PTM,包括乙酰化、磷酸化和甲基化[56]。其中一个位点是CAMK4 (Ca2+/calmodulin-dependent protein kinase IV, CAMK4),基因敲除的雄性小鼠不育,PRM2置换过渡蛋白被干扰,CAMK4缺失的精子细胞中PRM2特异性丢失和TP2保留[57]。在体外,PRM2可以被CAMK4磷酸化,这意味着在精子发生过程中鱼精蛋白的掺入需要CAMK4介导的PRM2磷酸化。Ppp1cc磷酸酶(Ppp1cc phosphatase)可使鱼精蛋白2第56位丝氨酸(Prm2Ser56)去磷酸化,其基因敲除后表现为小鼠雄性不育和精子头部畸形,说明PRM2第56位丝氨酸去磷酸化是小鼠精子成熟的关键[58]。
肌醇六磷酸激酶(inositol hexakisphosphate kinases, IP6Ks)是一种合成肌醇焦磷酸5-二磷酸肌醇五磷酸(inositol pyrophosphate 5-diphosphoinositol pentakisphosphate, 5-IP7)酶,已知其调节多种生理过程。在小鼠睾丸中,IP6K1在粗线期晚期精母细胞和圆形精细胞中高水平表达。基因敲除小鼠的精子细胞中由于翻译去阻遏而表现出TP2和PRM2的过早表达。TP2和PRM2等这些精子特异性染色质成分的定位异常,常规组蛋白的持续存在,导致这些小鼠的精子细胞伸长异常、精子细胞分化不完全和无精子症[59]。这些数据表明IP6K1在圆形精子细胞中对和的转录后调控中发挥重要作用。
3 结语与展望
本文以精子变形的顺序阐述了组蛋白–鱼精蛋白替换的动态变化过程,总结了该过程中每一步骤可能的调控机制。尽管在过去的研究中已经有许多有价值的发现,但仍有很多问题未能得到解答,例如组蛋白变体的掺入是如何启动的?过渡蛋白掺入以及解聚的确切机制也鲜有研究,鱼精蛋白诱导精子DNA构象改变和染色质凝聚的机制也不清楚。这些问题都需要在未来进一步的深入探索。
总之,组蛋白–鱼精蛋白替换是精子发生过程中的关键步骤,已有许多研究表明组蛋白–鱼精蛋白替换环节的许多缺陷都会导致无精症或少、弱、畸精子症,从而导致男性不育。系统、全面地研究其动态变化过程和调控机制将为人们了解男性不育的致病机制提供重要的理论支撑,也为男性不育症的诊断和治疗提供了潜在靶标。
[1] Hao SL, Ni FD, Yang WX. The dynamics and regulation of chromatin remodeling during spermiogenesis., 2019, 706: 201–210.
[2] Lin Q, Sirotkin A, Skoultchi AI. Normal spermatogenesis in mice lacking the testis-specific linker histone H1t., 2000, 20(6): 2122–2128.
[3] Mahadevan IA, Kumar S, Rao MRS. Linker histone variant H1t is closely associated with repressed repeat- element chromatin domains in pachytene spermatocytes.,2020, 13(1): 9.
[4] Martianov I, Brancorsini S, Catena R, Gansmuller A, Kotaja N, Parvinen M, Sassone-Corsi P, Davidson I. Polar nuclear localization of H1T2, a histone H1 variant, required for spermatid elongation and DNA condensation during spermiogenesis., 2005, 102(8): 2808–2813.
[5] Tanaka H, Iguchi N, Isotani A, Kitamura K, Toyama Y, Matsuoka Y, Onishi M, Masai K, Maekawa M, Toshimori K, Okabe M, Nishimune Y. HANP1/H1T2, a novel histone H1-like protein involved in nuclear formation and sperm fertility., 2005, 25(16): 7107–7119.
[6] Shalini V, Bhaduri U, Ravikkumar AC, Rengarajan A, Satyanarayana RMR. Genome-wide occupancy reveals the localization of H1T2 (H1fnt) to repeat regions and a subset of transcriptionally active chromatin domains in rat spermatids., 2021, 14(1): 3.
[7] Mishra LN, Shalini V, Gupta N, Ghosh K, Suthar N, Bhaduri U, Rao MRS. Spermatid-specific linker histone HILS1 is a poor condenser of DNA and chromatin and preferentially associates with LINE-1 elements., 2018, 11(1): 43.
[8] Shinagawa T, Huynh LM, Takagi T, Tsukamoto D, Tomaru C, Kwak HG, Dohmae N, Noguchi J, Ishii S. Disruption of Th2a and Th2b genes causes defects in spermatogenesis.,2015, 142(7): 1287–1292.
[9] Montellier E, Boussouar F, Rousseaux S, Zhang K, Buchou T, Fenaille F, Shiota H, Debernardi A, Héry P, Curtet S, Jamshidikia M, Barral S, Holota H, Bergon A, Lopez F, Guardiola P, Pernet K, Imbert J, Petosa C, Tan MJ, Zhao YM, Gérard M, Khochbin S. Chromatin-to- nucleoprotamine transition is controlled by the histone H2B variant TH2B., 2013, 27(15): 1680–1692.
[10] Barral S, Morozumi Y, Tanaka H, Montellier E, Govin J, de Dieuleveult M, Charbonnier G, Couté Y, Puthier D, Buchou T, Boussouar F, Urahama T, Fenaille F, Curtet S, Héry P, Fernandez-Nunez N, Shiota H, Gérard M, Rousseaux S, Kurumizaka H, Khochbin S. Histone variant H2A.L.2 guides transition protein-dependent protamine assembly in male germ cells.,2017, 66(1): 89–101.e8.
[11] Hoghoughi N, Barral S, Curtet S, Chuffart F, Charbonnier G, Puthier D, Buchou T, Rousseaux S, Khochbin S. RNA-guided genomic localization of H2A.L.2 histone variant.,2020, 9(2): 474.
[12] Soboleva TA, Nekrasov M, Pahwa A, Williams R, Huttley GA, Tremethick DJ. A unique H2A histone variant occupies the transcriptional start site of active genes., 2011, 19(1): 25–30.
[13] Soboleva TA, Parker BJ, Nekrasov M, Hart-Smith G, Tay YJ, Tng WQ, Wilkins M, Ryan D, Tremethick DJ. A new link between transcriptional initiation and pre-mRNA splicing: the RNA binding histone variant H2A.B.,2017, 13(2): e1006633.
[14] Anuar ND, Kurscheid S, Field M, Zhang L, Rebar E, Gregory P, Buchou T, Bowles J, Koopman P, Tremethick DJ, Soboleva TA. Gene editing of the multi-copy H2A.B gene and its importance for fertility.,2019, 20(1): 23.
[15] Molaro A, Wood AJ, Janssens D, Kindelay SM, Eickbush MT, Wu S, Singh P, Muller CH, Henikoff S, Malik HS. Biparental contributions of the H2A.B histone variant control embryonic development in mice.,2020, 18(12): e3001001.
[16] Tang MCW, Jacobs SA, Mattiske DM, Soh YM, Graham AN, Tran A, Lim SL, Hudson DF, Kalitsis P, O'Bryan MK, Wong LH, Mann JR. Contribution of the two genes encoding histone variant H3.3 to viability and fertility in mice.,2015, 11(2): e1004964.
[17] Yuen BTK, Bush KM, Barrilleaux BL, Cotterman R, Knoepfler PS. Histone H3.3 regulates dynamic chromatin states during spermatogenesis.,2014, 141(18): 3483–3494.
[18] Tachiwana H, Kagawa W, Osakabe A, Kawaguchi K, Shiga T, Hayashi-Takanaka Y, Kimura H, Kurumizaka H. Structural basis of instability of the nucleosome containing a testis-specific histone variant, human H3T.,2010, 107(23): 10454–10459.
[19] Ueda J, Harada A, Urahama T, Machida S, Maehara K, Hada M, Makino Y, Nogami J, Horikoshi N, Osakabe A, Taguchi H, Tanaka H, Tachiwana H, Yao T, Yamada M, Iwamoto T, Isotani A, Ikawa M, Tachibana T, Okada Y, Kimura H, Ohkawa Y, Kurumizaka H, Yamagata K. Testis-specific histone variant H3t gene is essential for entry into spermatogenesis.,2017, 18(3): 593– 600.
[20] Urahama T, Harada A, Maehara K, Horikoshi N, Sato K, Sato Y, Shiraishi K, Sugino N, Osakabe A, Tachiwana H, Kagawa W, Kimura H, Ohkawa Y, Kurumizaka H. Histone H3.5 forms an unstable nucleosome and accumulates around transcription start sites in human testis.,2016, 9: 2.
[21] Shiraishi K, Shindo A, Harada A, Kurumizaka H, Kimura H, Ohkawa Y, Matsuyama H. Roles of histone H3.5 in human spermatogenesis and spermatogenic disorders.,2018, 6(1): 158–165.
[22] Luense LJ, Wang XS, Schon SB, Weller AH, Lin Shiao E, Bryant JM, Bartolomei MS, Coutifaris C, Garcia BA, Berger SL. Comprehensive analysis of histone post- translational modifications in mouse and human male germ cells., 2016, 9: 24.
[23] Bao JQ, Bedford MT. Epigenetic regulation of the histone to protamine transition during spermiogenesis., 2016, 151(5): R55–R70.
[24] Ketchum CC, Larsen CD, McNeil A, Meyer-Ficca ML, Meyer RG. Early histone H4 acetylation during chromatin remodeling in equine spermatogenesis.,2018, 98(1): 115–129.
[25] Dong YX, Isono KI, Ohbo K, Endo TA, Ohara O, Maekawa M, Toyama Y, Ito C, Toshimori K, Helin K, Ogonuki N, Inoue K, Ogura A, Yamagata K, Kitabayashi I, Koseki H. EPC1/TIP60-mediated histone acetylation facilitates spermiogenesis in mice.,2017, 37(19): e00082–e00117.
[26] Lu LY, Wu JX, Ye L, Gavrilina GB, Saunders TL, Yu XC. RNF8-dependent histone modifications regulate nucleosome removal during spermatogenesis.. 2010, 18(3): 371–384.
[27] Guo YL, Song YF, Guo Z, Hu MJ, Liu B, Duan HY, Wang L, Yuan TX, Wang DG. Function of RAD6B and RNF8 in spermatogenesis., 2018, 17(2): 162–173.
[28] Gou LT, Kang JY, Dai P, Wang X, Li F, Zhao S, Zhang M, Hua MM, Lu Y, Zhu Y, Li Z, Chen H, Wu LG, Li DS, Fu XD, Li JS, Shi HJ, Liu MF. Ubiquitination-deficient mutations in human Piwi cause male infertility by impairing histone-to-protamine exchange during spermiogenesis.,2017, 169(6): 1090–1104.e13.
[29] Meng CL, Liao JY, Zhao DF, Huang HH, Qin JZ, Lee TL, Chen DG, Chan WY, Xia Y. L3MBTL2 regulates chromatin remodeling during spermatogenesis.,2019, 26(11): 2194–2207.
[30] Sin HS, Barski A, Zhang F, Kartashov AV, Nussenzweig A, Chen JJ, Andreassen PR, Namekawa SH. RNF8 regulates active epigenetic modifications and escape gene activation from inactive sex chromosomes in post-meiotic spermatids., 2012, 26(24): 2737–2748.
[31] Abe H, Meduri R, Li ZW, Andreassen PR, Namekawa SH. RNF8 is not required for histone-to-protamine exchange in spermiogenesis., 2021, ioab132.
[32] Berkovits BD, Wolgemuth DJ. The role of the double bromodomain-containing BET genes during mammalian spermatogenesis.,2013, 102: 293–326.
[33] Miller TCR, Simon B, Rybin V, Grötsch H, Curtet S, Khochbin S, Carlomagno T, Müller CW. A bromodomain- DNA interaction facilitates acetylation-dependent bivalent nucleosome recognition by the BET protein BRDT.,2016, 7: 13855.
[34] Gaucher J, Boussouar F, Montellier E, Curtet S, Buchou T, Bertrand S, Hery P, Jounier S, Depaux A, Vitte AL, Guardiola P, Pernet K, Debernardi A, Lopez F, Holota H, Imbert J, Wolgemuth DJ, Gérard M, Rousseaux S, Khochbin S. Bromodomain-dependent stage-specific male genome programming by Brdt., 2012, 31(19): 3809–3820.
[35] Dhar S, Thota A, Rao MRS. Insights into role of bromodomain, testis-specific (Brdt) in acetylated histone H4-dependent chromatin remodeling in mammalian spermiogenesis., 2012, 287(9): 6387–6405.
[36] Guo YL, Song YF, Guo Z, Hu MJ, Liu B, Duan HY, Wang L, Yuan TX, Wang DG. Function of RAD6B and RNF8 in spermatogenesis.,2018, 17(2): 162–173.
[37] Wang XK, Kang JY, Wei LX, Yang XG, Sun HD, Yang SM, Lu L, Yan M, Bai MZ, Chen YY, Long JJ, Li N, Li DS, Huang J, Lei M, Shao Z, Yuan W, Zuo EW, Lu KH, Liu MF, Li JS. PHF7 is a novel histone H2A E3 ligase prior to histone-to-protamine exchange during spermiogenesis.,2019, 146(13): dev175547.
[38] Kim CR, Noda T, Kim H, Kim G, Park S, Na Y, Oura S, Shimada K, Bang I, Ahn JY, Kim YR, Oh SK, Choi HJ, Kim JS, Jung I, Lee H, Okada Y, Ikawa M, Baek SH. PHF7 modulates BRDT stability and histone-to-protamine exchange during spermiogenesis., 2020, 32(4): 107950.
[39] Berruti G. Destruction or reconstruction: a subtle liaison between the proteolytic and signaling role of protein ubiquitination in spermatogenesis., 2021, 1288: 215–240.
[40] Liu ZQ, Miao DS, Xia QW, Hermo L, Wing SS. Regulated expression of the ubiquitin protein ligase, E3(histone)/ LASU1/Mule/ARF-BP1/HUWE1, during spermatogenesis., 2007, 236(10): 2889–2898.
[41] Haraguchi CM, Mabuchi T, Hirata S, Shoda T, Tokumoto T, Hoshi K, Yokota S. Possible function of caudal nuclear pocket: degradation of nucleoproteins by ubiquitin- proteasome system in rat spermatids and human sperm., 2007, 55(6): 585–595.
[42] Khor B, Bredemeyer AL, Huang CY, Turnbull IR, Evans R, Maggi LB, White JM, Walker LM, Carnes K, Hess RA, Sleckman BP. Proteasome activator PA200 is required for normal spermatogenesis., 2006, 26(8): 2999–3007.
[43] Qian MX, Pang Y, Liu CH, Haratake K, Du BY, Ji DY, Wang GF, Zhu QQ, Song W, Yu YD, Zhang XX, Huang HT, Miao SY, Chen LB, Zhang ZH, Liang YN, Liu S, Cha H, Yang D, Zhai YG, Komatsu T, Tsuruta F, Li HT, Cao C, Li W, Li GH, Cheng YF, Chiba T, Wang LF, Goldberg AL, Shen Y, Qiu XB. Acetylation-mediated proteasomal degradation of core histones during DNA repair and spermatogenesis.,2013, 153(5): 1012–1024.
[44] Zhang ZH, Jiang TX, Chen LB, Zhou WH, Liu YX, Gao F, Qiu XB. Proteasome subunit α4s is essential for formation of spermatoproteasomes and histone degradation during meiotic DNA repair in spermatocytes., 2021, 296: 100130.
[45] Rathke C, Baarends WM, Awe S, Renkawitz-Pohl R. Chromatin dynamics during spermiogenesis.,2014, 1839(3): 155–168.
[46] Yu YE, Zhang Y, Unni E, Shirley CR, Deng JM, Russell LD, Weil MM, Behringer RR, Meistrich ML. Abnormal spermatogenesis and reduced fertility in transition nuclear protein 1-deficient mice., 2000, 97(9): 4683–4688.
[47] Zhao M, Shirley CR, Yu YE, Mohapatra B, Zhang Y, Unni E, Deng JM, Arango NA, Terry NH, Weil MM, Russell LD, Behringer RR, Meistrich ML. Targeted disruption of the transition protein 2 gene affects sperm chromatin structure and reduces fertility in mice., 2001, 21(21): 7243–7255.
[48] Shirley CR, Hayashi S, Mounsey S, Yanagimachi R, Meistrich ML. Abnormalities and reduced reproductive potential of sperm from Tnp1- and Tnp2-null double mutant mice., 2004, 71(4): 1220–1229.
[49] Tseden K, Topaloglu O, Meinhardt A, Dev A, Adham I, Müller C, Wolf S, Böhm D, Schlüter G, Engel W, Nayernia K. Premature translation of transition protein 2 mRNA causes sperm abnormalities and male infertility., 2007, 74(3): 273–279.
[50] Phillips BT, Williams JG, Atchley DT, Xu XJ, Li JL, Adams AL, Johnson KL, Hall TMT. Mass spectrometric identification of candidate RNA-binding proteins associated with transition nuclear protein mRNA in the mouse testis.,2019, 9(1): 13618.
[51] Okada Y, Scott G, Ray MK, Mishina Y, Zhang Y. Histone demethylase JHDM2A is critical for Tnp1 and Prm1 transcription and spermatogenesis., 2007, 450(7166): 119–123.
[52] Liu ZL, Zhou SL, Liao L, Chen X, Meistrich M, Xu JM. Jmjd1a demethylase-regulated histone modification is essential for cAMP-response element modulator-regulated gene expression and spermatogenesis., 2010, 285(4): 2758–2770.
[53] Eelaminejad Z, Favaedi R, Sodeifi N, Sadighi Gilani MA, Shahhoseini M. Deficient expression of JMJD1A histone demethylase in patients with round spermatid maturation arrest.,2017, 34(1): 82–89.
[54] Bao JQ, Bedford MT. Epigenetic regulation of the histone-to-protamine transition during spermiogenesis., 2016, 151(5): R55–R70.
[55] Cho C, Willis WD, Goulding EH, Jung-Ha H, Choi YC, Hecht NB, Eddy EM. Haploinsufficiency of protamine-1 or -2 causes infertility in mice., 2001, 28(1): 82–86.
[56] Brunner AM, Nanni P, Mansuy IM. Epigenetic marking of sperm by post-translational modification of histones and protamines.,2014, 7(1): 2.
[57] Wu JY, Ribar TJ, Cummings DE, Burton KA, McKnight GS, Means AR. Spermiogenesis and exchange of basic nuclear proteins are impaired in male germ cells lacking Camk4., 2000, 25(4): 448–452.
[58] Itoh K, Kondoh G, Miyachi H, Sugai M, Kaneko Y, Kitano S, Watanabe H, Maeda R, Imura A, Liu Y, Ito C, Itohara S, Toshimori K, Fujita J. Dephosphorylation of protamine 2 at serine 56 is crucial for murine sperm maturation.,2019, 12(574): eaao7232.
[59] Malla AB, Bhandari R. IP6K1 is essential for chromatoid body formation and temporal regulation ofandexpression in mouse spermatids., 2017, 130(17): 2854–2866.
Regulation of histone-to-protamine transition during spermiogenesis
Lu Yuan1,2, Tingting Ge1,2, Changmin Niu1,2, Wenhua Xu1,2, Ying Zheng1,2
Spermiogenesis is the final stage of spermatogenesis, in which round spermatids differentiate into mature sperms through a complex process including morphological changes and chromatin condensation. Histone-to-protamine transition during spermiogenesis is a critical part of this biological process. Histones are initially replaced by testis-specific histone variants, then transition proteins integrate into the nucleus, and are in turn replaced by protamine. Impaired histone-to-protamine transition may cause azoospermia, oligospermia or teratospermia, which lead to male infertility. In this review, we summarize the research progress of regulatory mechanisms of the histone-to-protamine transition, thereby providing the theoretical basis for the diagnosis and treatment of male infertility.
spermiogenesis; histone-to-protamine transition; post-translational modification
2021-06-10;
2021-10-27
国家自然科学基金项目(编号:82071696,82101674),江苏省高校自然科学研究重大项目(编号:20KJA310002)和扬州大学研究生科研创新计划项目(编号:XKYCX20_35)资助[Supported by the National Natural Science Foundation of China (Nos. 82071696, 82101674), the Key Scientific Project for Jiangsu Provincial Universities (No. 20KJA310002) and the Postgraduate Scientific Research Innovation Program of Yangzhou University (No. XKYCX20_35)]
袁露,在读硕士研究生,专业方向:生殖医学。E-mail: 2920054914@qq.com
郑英,教授,博士生导师,研究方向:生殖医学。E-mail: yzzkl@163.com
10.16288/j.yczz.21-205
2021/11/12 09:59:20
URI: https://kns.cnki.net/kcms/detail/11.1913.r.20211110.1349.002.html
(责任编委: 史庆华)

