基于多元统计分析的牛排掺假定量判别及差异分析
张颖颖,王守伟,康超娣,张明悦,李莹莹
(北京食品科学研究院,中国肉类食品综合研究中心,北京 100068)
近年来,基于质谱技术测定靶标多肽的方法已被广泛应用于食品真伪鉴定中[1-3],该类方法具有高热稳定性、高灵敏度、高通量等优点,应用于多类食品,如牛奶[4]、肉制品[5-6]、动物副产品[7]。方法的基本流程[8]包括蛋白提取、胰蛋白酶酶解、高分辨质谱采集数据、专业软件搜索数据库进行蛋白及多肽鉴定、统计学分析。其中肉的物种特异性多肽主要来源于肌红蛋白[9]、肌球蛋白[10]、血红蛋白[11]、乳酸脱氢酶和肌酸激酶蛋白[12],大豆的特异性多肽主要来源于大豆球蛋白[13],牛奶的特异性多肽主要来源于酪蛋白[14]和乳球蛋白[15]。
目前,绝大多数靶标多肽方法主要应用于物种的定性鉴别实验中,然而,食品掺假不仅涉及原料种类掺假,即用低价原料代替高价原料,还包括食品掺假程度的问题,如在肉制品中加入较大量的非肉蛋白(植物蛋白)替代肉的含量冒充等级高的产品,而我国针对肉制品等级的判定,如GB/T 20712—2006《火腿肠》[16]、SB/T 10610—2011《肉丸》[17],仅能通过一些常规的参数检测,如蛋白总量、淀粉等,而无法测定其中真正肉的含量,因此在肉制品定量检测方面存在方法盲区。目前的定量研究主要集中于2 个方向的探讨:一是相对定量法,即测定食品中不同物种蛋白含量的比例。Camerini等[18]分析意大利乳清干酪的掺假现象,从β-乳球蛋白和α-乳清蛋白中选择了特定的肽段,以鉴定牛、水牛、绵羊、山羊源性成分,并且在1%~50%的范围内进行曲线拟合,线性相关系数高于0.99,方法的检出限可测定0.5%的牛乳清成分。Watson等[9]建立了一种液相色谱-串联质谱方法判定肉制品的掺假,用各个物种特异性多肽的峰面积之比进行物种相对定量分析,分别对5%、10%、20%、50%、80%和90%的马肉掺假比例进行线性拟合分析,其相关系数大于0.99,该方法的检出限可达1%。另一研究方向是绝对定量法,可直接测定食品中蛋白质的含量。Montowska等[19]提出了一种通过同位素内标法定量鸡、鸭、鹅、猪、牛、大豆、牛奶和蛋清的专属性多肽折算其中的蛋白含量。上述定量方法的原理均是物种含量与其含有的蛋白及多肽呈正相关,通过测定其中多肽含量推算其中物种的含量,因此,筛选用于物种定量分析的多肽是整个实验的关键因素。由于高分辨质谱分辨率高,质量数可精确至小数点后四位,可用于分析鉴别物种蛋白及多肽,但同时得到的谱图具有高纬度、高噪声、高复杂度等特征,且每个物种得到的多肽数量众多,因此需要有相关的专业技术方法从复杂数据中提取出有价值的信息,便于后期的实验验证。
近年来,多元数据统计方法已广泛用于代谢组学和蛋白质组学分析[20],包括主成分分析(principal component analysis,PCA)、偏最小二乘判别分析(partial least squares-discrimination analysis,PLS-DA)和正交偏最小二乘判别分析(orthogonal partial least squares-discrimination analysis,OPLS-DA)统计方法,这些方法可通过减少数据维数,有效地解决样本少、自变量多、相关性高的数据分析。Yang Jinhui等[4]使用PCA方法分析了不同比例的掺假牛奶,通过建立模型,可以准确地将样品结果与模型进行匹配。Yuswan等[21]使用PCA方法建立了猪、牛、鸡的模型,能够准确区分这3 个物种,并使用OPLS-DA用于区分和增强模型的解释性。Du Lijuan等[22]分别使用PCA、PLS-DA和支持向量机3种统计方法区别掺假牛奶与纯牛奶。
本研究采用多元统计分析建立一种可用于筛选物种定量分析多肽的研究方法,将牛排作为研究对象,用高分辨率质谱分析不同比例的掺假牛排,并建立多变量统计分析模型,用于筛选猪、牛差异显著性的定量多肽,根据定量多肽含量区分掺假牛排中的牛肉和猪肉含量,旨在为掺假定量分析提供新的研究思路。
1 材料与方法
1.1 材料与试剂
生牛肉、猪肉购于屠宰场;市购商业牛排样品于-18 ℃冷冻保存。
胰蛋白酶(测序级)、二硫苏糖醇(dithiothreitol,DTT)(生化级) 美国Promega公司;甲酸(formic acid,FA)、乙酸、乙腈(均为色谱纯) 德国Merck公司;碘乙酰胺(iodoacetamide,IAA)、三氟乙酸(trifluoroacetic acid,TFA)(均为生化级) 美国Sigma公司;尿素、硫脲、盐酸(均为分析纯) 上海国药集团化学试剂有限公司。
1.2 仪器与设备
Q Exactive HF-X液相色谱四极杆静电场轨道阱高分辨质谱系统(配有电喷雾离子源) 美国Thermo公司;HENGAO T&D固相萃取装置 美国Agilent公司;CR21N高速冷冻离心机 日本Hitachi公司;HGC-24A氮吹仪 天津市恒奥科技发展有限公司;HLB固相萃取柱 美国Waters公司。
1.3 方法
1.3.1 样品制备
生牛肉、猪肉样品分别剔除脂肪、筋膜后搅碎均匀,按照市场中冷冻牛排的制作工艺,制备10种含不同比例牛肉、猪肉的牛排制品,并加入大豆蛋白粉、盐、水、磷酸盐等其他添加剂,委托工厂生产模拟牛排,其中,猪肉和牛肉的比例如表1所示。
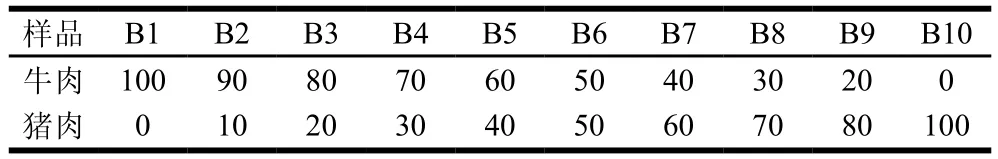
表1 模拟牛排中牛肉和猪肉的混合比例Table 1 Mixing ratio between beef and pork content in simulated steak%
1.3.2 样品前处理
采用的样品前处理过程与相关文献[23]方法相似。准确称取2 g样品,加入20 mL提取溶液(7 mol/L尿素、2 mol/L硫脲、50 mmol/L Tris-HCl(pH 8.0))在冰水浴条件下采用均质方法提取蛋白质,12 000 r/min离心20 min后,准确移取200 μL上清液于小离心管中,加入30 μL DTT(50 mmol/L)溶液在56 ℃反应1 h,待冷却至室温后,加入30 μL IAA(100 mmol/L)溶液暗处室温条件下反应0.5 h,再加入1.8 mL Tris-HCl(25 mmol/L,pH 8.0)、60 μL胰蛋白酶溶液(2 mg/mL)于37 ℃水浴中酶解过夜。冷却至室温后,用10% TFA调节pH值小于2.0以终止酶解反应,然后使用HLB固相萃取柱进行净化,HLB萃取柱先用乙腈、50%乙腈-水、0.1% TFA进行活化,上样,再分别用0.1% TFA、0.5%乙酸淋洗,最后用2 mL乙腈-0.5%乙酸(60∶40,V/V)洗脱,收集洗脱液,混匀后过0.22 μm滤膜,准备上机测定。每个样品进行4 次平行实验。
1.3.3 数据采集
利用高分辨质谱进行多肽鉴定及定量分析,色谱柱为Hypersil GOLD C18色谱柱(2.1 mm×100 mm,1.9 μm),流速为0.2 mL/min,流动相A为0.1%甲酸-H2O,流动相B为0.1%甲酸-乙腈,梯度条件如下:0~0.2 min,97%~90% A,3%~10% B;15.8 min,60% A,40% B;16.8 min,20% A,80% B;17.3 min,20% A,80% B;18.3 min,20% A,80% B,18.5 min,97% A,3% B;25 min,97% A,3% B。进样体积为5 μL。
质谱参数如下:喷雾电压3 400 V,毛细管温度320 ℃,鞘气40 arb,辅助气15 arb,全扫描分辨率120 000,质量扫描范围m/z350~2 000,自动增益值为3×106,注入时间为100 ms,二级扫描,topN为10,分辨率为30 000,自动增益值为2×105,注入时间为50 ms,碰撞能量为30 V。
1.4 数据处理
高分辨质谱对数据进行采集后,利用Proteome Discoverer(PD)软件进行数据处理,通过搜索Uniprot数据库,对物种蛋白及多肽进行鉴定分析。PD软件分析包括2 个工作流程:Processing Step和Consensus Step(图1)。Processing Step涉及物种多肽的筛选及定量分析,需要从Sequest HT模块中加入猪、牛的蛋白质数据库,数据库是从UniProt数据库(https://www.uniprot.org/)中下载fasta文件,导入至PD软件后进行搜索,其他参数设置为默认值即可。Consensus Step是通过增强多肽和蛋白质注释对高可信度多肽进行过滤,经过该步骤分析后,数据可进行可视化结果展示。通过使用悟空云平台(https://www.omicsolution.com/wkomics/main/)在线工具对PD软件分析的数据进行PCA、PLS-DA、OPLS-DA统计分析[24]。

图1 PD软件分析流程图Fig.1 Workflow of PD analysis
2 结果与分析
2.1 基于多肽组成确定牛排比例的可行性分析
2.1.1 构建PCA模型
PCA可通过正交变换将一组可能存在相关性的变量转换为一组线性不相关的变量[25]。当样品B10的牛肉多肽丰度设为0时,将样品B1~B9中不同比例的牛肉多肽信息用以构建PCA模型;同样,样品B1的猪肉多肽丰度设为0,将样品B2~B10中不同比例的猪肉多肽用以构建PCA模型。
如图2a所示,具有不同牛肉含量比例的牛排样品通过PCA可以得到有效分离,并且按照牛肉含量由大至小进行从左至右排列,牛肉含量较高的样品主要分布在第3、4象限,而牛肉含量较低的样品主要分布在第1、2象限。其中PC1的方差贡献率为82.6%,表明该成分可以较好地反映原始样品的信息;且PC1和PC2的累计方差贡献率高达86.5%,因此该模型有望用于分析不同比例的牛肉样品。相较而言,图2b中不同比例猪肉含量样品的PCA模型效果稍差。PCA图的聚拢程度反映了样品的相似性,即不同含量比例样品的聚类程度。除样品B7外,B2~B10均实现了完全分离,而样品B7的数据之间存在较大的距离,表明此比例下的数据差异较大,聚类效果不佳。
2.1.2 构建PLS-DA模型
PLS-DA[26]是在已知样本的分组关系时,用PLS回归方法,在对数据降维的同时建立回归模型,并对回归结果进行判别分析。目前,PLS-DA已广泛应用于蛋白质组学及代谢组学中,用以分析样本量小、变量之间独立性强及相关性高的样品数据[27]。
同上述构建PCA模型的过程,分别进行不同比例猪肉和牛肉的PLS-DA。在建立的PLS-DA模型中,从图3a(R2Y=0.124,Q2=0.092 5)和图3b(R2Y=0.571,Q2=0.338)可以看出,各自的样品组都得到了明显区分,说明使用该模型分类效果显著,组间差异明显。尤其是图3b中不同猪肉含量的样品数据模型,相较于PCA,使用PLS-DA实现了更好分离。图3中均按照样品含量比例递减的规律从左到右排列,并且2 个模型中,除B9外的其他组样本均比较集中,说明组内差异较小。而B9样品椭圆范围最宽,样品点分散,组内差异较大,原因可能是采用PLS-DA模型分析时,不同批次之间数据差异比较大。
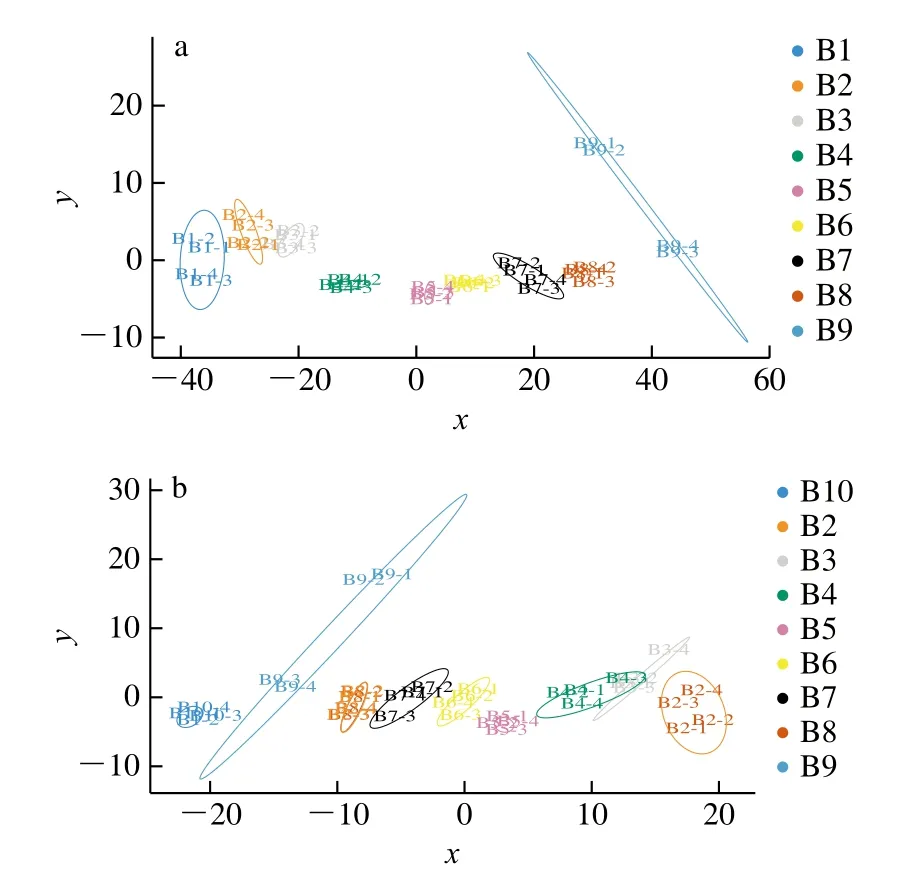
图3 不同比例的牛肉和猪肉的PLS-DA模型Fig.3 PLS-DA score plot of beef and pork mixtures in different proportions
2.1.3 构建OPLS-DA模型
OPLS-DA是在PLS-DA基础上发展而来的算法,相较于PLS-DA,将X变量中的系统变异分解为2 部分,即同Y线性相关的部分和同Y正交的部分。随着正交变异组分的增加,将提供更多的解释性,并减少结果的误差。Trygg等[28]认为,该方法可有效降低模型的复杂性并增强模型的解释能力,但并不会降低模型的预测能力。对上述猪、牛数据进行OPLS-DA模型分析,猪的模型中的R2Y为0.401、Q2为0.139;牛模型中的R2Y为0.479、Q2为0.295。由图4可知,猪、牛的每个混合比例的样品组都得到了明显区分,与PLS-DA建立的模型对比,组间间距更大,可以看出OPLS-DA模型分类效果更为显著;并且组内之间的数据点间距较小,说明组内差异较小。表明利用高分辨鉴别的多肽中可以筛选到差异显著性的变量以实现不同含量样本的区分。

图4 不同比例的牛肉和猪肉的OPLS-DA模型Fig.4 OPLS-DA score plot of beef and pork mixtures in different proportions
2.1.4 模型评价
由上述3种可视化模型图可以得出,采用基于物种多肽响应值进行多元统计分析的方法对物种掺假量化判别可行。由上述模型的判别效果看,OPLS-DA模型的判别效果最好,因此后续数据的筛选将在OPLS-DA模型的基础上进行。
在进行模型判别的过程中,需要关注模型的评估指标,即模型的拟合程度(R2Y)和模型的预测能力(Q2Y),从而可以识别出差异显著性的肽段。R2Y指标的数值越接近1,表明OPLS-DA模型的数据拟合度越好[29]。
变量重要度投影(variable importance for the projection,VIP)是OPLS-DA模型中评价变量贡献率最常用的参数,一般认为VIP值大于1的变量能够反映统计模型中一些重要特征[30]。因此设置OPLS-DA模型中VIP值大于1,相关系数P值小于0.05,筛选出模型中差异显著的物种多肽,其中筛选出牛多肽为257 条,猪多肽为288 条。对筛选出的多肽重新进行OPLS-DA模型拟合,如图5所示。

图5 筛选VIP值大于1的多肽数据绘制OPLS-DA模型Fig.5 OPLS-DA score plot of beef and pork mixtures with selected peptides based on VIP
通过删除VIP值较小的多肽数据,新生成的OPLS-DA模型均得到了显著改善。牛模型的R2Y为0.96,Q2Y为0.721,猪模型的R2Y为0.479,Q2Y为0.582,进一步说明该模型具有较高的拟合度和预测能力。由图5a所示,不同比例的样品组均能够得到良好区分,除B3样品较为分散外,其他样品组均聚拢良好。相对而言,图5b中所有样品组均有良好的聚拢和区分。表明经过筛选后的多肽建立模型的预测能力更好,为后续筛选物种定量多肽打下基础。
2.2 定量测定
2.2.1 差异显著性多肽的筛选
由2.1.4节中牛OPLS-DA模型绘制S图,图中相关系数p1值和pcorr1值均大于0时,相应的多肽响应值与物种的含量比例呈正相关,并且值越大,其相关性越高。按数值降序的顺序选择前10%的多肽,详细信息如表2所示。表2中选择的25 条牛肉多肽主要来源于肌联蛋白、肌球蛋白结合蛋白C、α-1,4-葡聚糖磷酸化酶、L-乳酸脱氢酶A链、肌球蛋白。同样,也选择了猪的25 条差异显著性多肽,信息列于表3。这些多肽主要来自肌球蛋白、血清白蛋白、肌球蛋白结合蛋白C、α-1,4-葡聚糖磷酸化酶和钙转运ATP酶。这些蛋白大多数是高丰度蛋白质,具有较多数量的特异性多肽,如牛的肌联蛋白、肌球蛋白、肌球蛋白结合蛋白C,猪的肌球蛋白结合蛋白C、α-1,4-葡聚糖磷酸化酶。

表2 牛显著性多肽信息Table 2 Selected significantly differential beef peptides

表3 猪显著性多肽信息Table 3 Selected significantly differential pork peptides
目前,已有采用上述来源蛋白中的多肽进行物种定性鉴别的研究,如采用血清白蛋白和L-乳酸脱氢酶A中的多肽进行物种鉴别[12,23]。Ruiz等[31]从肌红蛋白、肌球蛋白1和肌球蛋白2中筛选物种特异性多肽用以掺假鉴别。由此也进一步证实通过OPLS-DA模型筛选的物种多肽及蛋白可靠且有效。
2.2.2 差异显著性多肽的线性方程分析及应用
对上述筛选的猪、牛的差异显著性多肽进行线性拟合分析,以肉的含量为横坐标,多肽的响应值为纵坐标,绘制标准曲线。其中牛多肽中有10 条多肽呈现良好的线性关系即R2大于0.99,猪源多肽中有6 条呈现良好的线性关系,如表4所示。

表4 筛选多肽的线性拟合信息Table 4 Linear equations based on the selected peptides
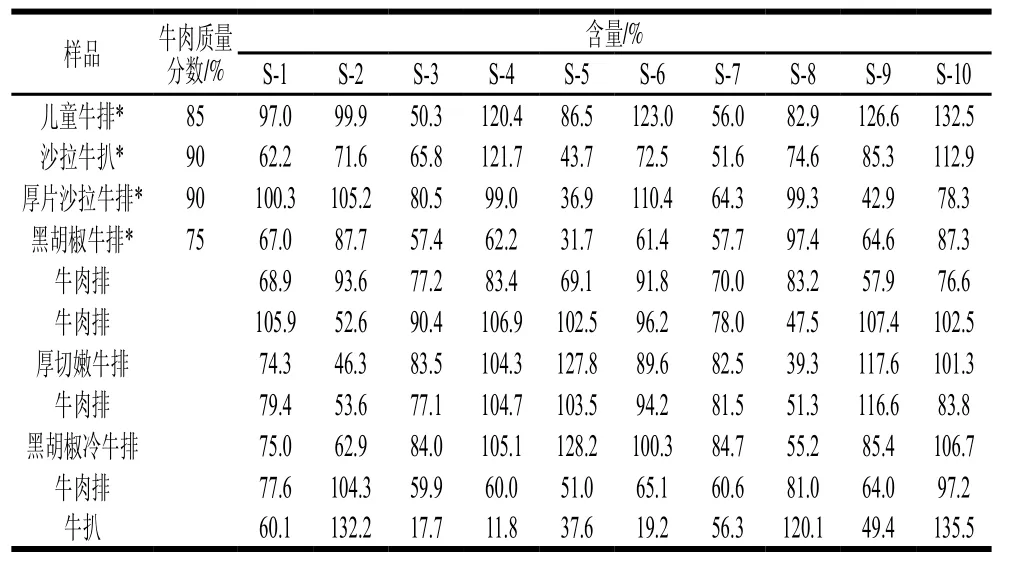
表5 牛排样品中牛肉含量的测定结果Table 5 Determination of beef contents in real steak samples

图6 牛排样品回收率分布图Fig.6 Distribution of recoveries of beef in real steak samples
本研究旨在提供一种快速筛选用于定量分析多肽的方法,因此仅查询了猪、牛的数据库并筛选了差异显著性多肽,并未对筛选多肽的物种专属性进一步筛查。通过对比测定牛排样品中牛肉含量的结果,发现不同多肽测定结果并不相同,这可能是由于不同加工工艺或是添加的辅料使蛋白质产生了不同程度的变性,从而影响了酶解过程中多肽的响应值,并且本实验是通过高分辨质谱进行定量分析,相对而言,液相色谱-三重串联四极杆质谱的多反应监测模式具有强大的抗干扰能力及准确的定量特性[32],因此后期实验会针对筛选的差异显著性定量多肽进行进一步物种唯一性分析,并使用液相色谱-三重串联四极杆质谱进行样品检测,筛选回收率好的物种专属性多肽进行日常样品定量分析。
3 结 论
本研究通过对牛排中的猪、牛多肽数据进行统计计量学分析,分别采用PCA、PLS-DA、OPLS-DA 3种模型验证牛排中猪肉、牛肉含量的可行性,结果显示,3种模型均能够实现不同含量样品之间的区分,其中通过结合参数VIP值大于1筛选的多肽数据建立的OPLS-DA模型效果最好。利用该模型分别筛选出25 条差异显著的牛、猪多肽,其中10 条牛多肽和6 条猪多肽得到的线性拟合结果较好,其线性相关系数R2均大于0.99,并开展了真实样品的验证,可以测定分析牛排中猪肉、牛肉的含量比例,实现定量分析。本研究为食品的定量分析及判别提供了一种新的多肽筛选思路。

