野生大豆GsSnRK1.1蛋白激酶在淹水胁迫下功能分析
丁晓东,张 晴,于海燕,刘圆明,李明龙,肖佳雷
(1.东北农业大学农业生物功能基因重点实验室,哈尔滨 150030;2.望奎县种子能源服务中心,黑龙江 望奎 152100)
大豆(Glycinemax)蛋白含量和出油率高,种植面积广泛,是我国重要粮食作物之一[1-2]。其中栽培大豆对淹水胁迫较为敏感,当发生涝害时,其根系淹水沤根,叶片黄化,大豆光合速率、株高、荚果数和种子产量降低[3-4]。野生大豆是栽培大豆亲缘种,在形态上具有发达根系和柔性藤蔓状枝干,在基因水平上具有遗传多样性,对环境适应性强,对涝害具有较强耐受性[5]。研究野生大豆淹水胁迫分子调控机制,可为大豆抗涝品种育种提供理论基础。同时,来自野生大豆的优良基因可直接应用于栽培大豆育种,解决栽培大豆对淹水胁迫较为敏感的问题,对实现栽培大豆品种改良具有重要意义。
植物蔗糖非发酵-1-相关蛋白激酶-1在参与生长发育、基础代谢、胁迫应答以及碳氨代谢等多项生理活动中发挥重要作用[6-8]。Lovas等最早发现植物SnRK1蛋白激酶在马铃薯高盐胁迫下敏感性,发现马铃薯反义StubGAL83基因在高盐胁迫下根细胞较小比野生型更为敏感;SnRK1蛋白激酶可通过改变淀粉合成,将其回流到植物根中,从而影响地上部分植物口感,减少食草性动物食用[9]。番茄叶片中SISNF基因在干旱胁迫下表达量升高,猜想番茄SISNF基因参与干旱胁迫调控[10]。Kudahettige等发现在淹水处理下,拟南芥中过量表达水稻SnRK1基因,促进植物体内淀粉降解,提升对植物细胞能量供给和植物存活能力[11]。但SnRK1如何响应淹水胁迫,增强植物抗逆性的分子机制尚不明确。
由于栽培大豆是重要粮食作物,其野生近缘种野生大豆对环境适应环境能力强,喜水耐湿,多生于山野以及河流沿岸、湿草地、湖边、沼泽等地,因此野生大豆是研究耐淹水基因重要植物材料。为研究GsSnRK1.1蛋白激酶在淹水胁迫中作用与功能,在野生大豆(Glycinesoja)中克隆GsSnRK1.1基因、启动子及点突变基因;在Col-0野生型拟南芥中超表达以35S启动子驱动的GsSnRK1.1、GsSnRK1.1(T176A)、GsSnRK1.1(T176E)和GsSnRK1.1(K49M)基因,获得转基因植株;通过对野生大豆不同时间淹水处理,利用RT-qPCR分析GsSnRK1.1基因在淹水胁迫条件下时空表达模式,同时也对pGsSnRK1.1::GUS转基因拟南芥植株作GUS染色,从侧面印证qRT-PCR数据正确性;将GsSnRK1.1基因在拟南芥中过量表达,测定其叶绿素、SOD和Pro等生理指标,以及ADH1(EC1.1.1.1)、PDC1(EC4.1.1.1)标记淹水应激反应基因在Col-0与转基因拟南芥中基因表达。试验对GsSnRK1.1基因功能验证分析,为探究大豆抗涝品种选育提供理论依据。
1 材料与方法
1.1 材料
野生大豆为Glycine soja(G07256),拟南芥为Columbia-0,以pCAMBIA3301和pCAMBIA2300为植物表达载体。研究所用菌株为农杆菌GV3101和大肠杆菌DH5α。以上材料均由东北农业大学植物与环境互作实验室提供。
1.2 方法
1.2.1 总RNA、总DNA提取及cDNA合成
将饱满且无病虫害野生大豆(G07256)种子用浓硫酸处理6~10 min去除种皮表面泥膜,倒掉浓硫酸,自来水冲洗5次。冲洗后种子放在含湿润滤纸培养皿上,为其提供萌发所需水分,25℃黑暗培养,培养时间为2~3 d。当种子发芽达到2 cm时,转移至人工气候室水培,设置温度为25℃,营养液每隔2~3 d更换一次。待幼苗生长至六叶期时,使用相应试剂盒提取总RNA/DNA以及反转录合成cDNA。
1.2.2 基因克隆
根据NCBI基因数据库所提供GsSnRK1.1(KHN13196)基因及其启动子CDS全长序列信息,使用Primer Premier 5.0设计引物。克隆所用模板为野生大豆cDNA和DNA,作基因及启动子序列扩增。进一步根据位点设计引物,引物序列如表1所示。通过引物重叠PCR方法获得激酶调控区域失活的转录本GsSnRK1.1(T176A)、激酶调控区域模拟磷酸化的转录本GsSnRK1.1(T176E)和催化区域失活的转录本GsSnRK1.1(K49M)基因片段。其中将GsSnRK1.1第176位苏氨酸(Thr,T)密码子替换为谷氨酸(Glu,E)与丙氨酸(Ala,A)密码子,第49位赖氨酸(Lys,K)密码子突变为甲硫氨酸(Met,M)密码子。KpnⅠ-XbaⅠ内切酶酶切pCAMBIA2300质粒使之线性化,再利用T4DNA连接酶构建超表达载体,获得p2300-35S-Myc-GsSnRK1.1、p2300-35S-Myc-GsSnRK1.1(T176A)、p2300-35S-Myc-GsSnRK1.1(T176E)、p2300-35SMyc-GsSnRK1.1(K45M)超表达载体;Eco RⅠ-NcoⅠ内切酶酶切pCAMBIA3301质粒使之线性化,利用T4DNA连接酶将GsSnRK1.1基因内源启动子替换载体原本35S启动子获得p3301-GsSnRK1.1P-GUS启动子报告表达载体;BglⅡ-BstEⅡ内切酶酶切p3301-GsSnRK1.1P-GUS,再利用同源重组试剂盒构建载体,获得p3301-GsSnRK1.1P-Myc-GsSnRK1.1
1.2.3 转基因植株获得
利用冻融法将构建成功的过表达载体和启动子驱动GUS基因重组质粒转入农杆菌,而后农杆菌侵染模式植物拟南芥,使用方法为蘸花法[12]。种植完成侵染的拟南芥种子,长出两片子叶时开始喷洒抗生素筛选转基因植株,其中利用卡纳霉素和除草剂(Basta)分别喷洒pCAMBIA2300和pCAMBIA3301载体侵染的拟南芥。筛选过后对其作PCR和Western blot鉴定,确定是否为转基因纯合体株系。
1.2.4 表型分析及生理指标测定
为模拟长时间淹水情况,将拟南芥野生型和缺失突变体种子置于灭菌EP管中,加入1 mL 30%NaClO,消毒15 min,用灭菌ddH2O清洗种子6~8次,最后向EP管中加入适量灭菌ddH2O,4℃春化处理48 h。用灭菌牙签将种子点在含0.8%琼脂和1%蔗糖的1/2MS固体培养基试管中,将试管置于人工气候箱中培养。待植物生长至四叶期时,将10 mL经高压灭菌过蒸馏水小心灌入每个试管中,黑暗中放置37 d,观察其表型并测定生理指标。
1.2.5 淹水应激反应标记基因表达分析
对淹水胁迫处理37 d拟南芥幼苗提取总RNA,利用RT-qPCR测量乙醇脱氢酶1(ADH1;At1g77120)和丙酮酸脱羧酶1(PDC1;At4g33070)标记基因对淹水胁迫的响应。
1.2.6 野生大豆GsSnRK1.1表达特性分析
将三周龄野生大豆幼苗完全浸没至水中处理2 d,分别在0、6、12、24及48 h不同时间点取样;选用Plant RNA Kit(OMEGA)提取总RNA,使用ReverTra Ace qPCRRTMaster MIX with gDNA Remover cDNA试剂盒(TOYOBO)反转录合成cDNA,使用SYBRGreen试剂盒(TOYOBO)对GsSnRK1.1基因作实时荧光定量PCR,结果分析采用比较CT法(ΔΔCT法)。
2 结果与分析
2.1 野生大豆GsSnRK1.1基因克隆与序列分析
PCR扩增结果见图1,获得GsSnRK1.1基因、启动子及点突变基因,其中基因长度为1 548 bp,启动子长度为1 663 bp,因此选择DL2000 DNA Marker。经克隆、测序确定GsSnRK1.1为所需研究基因。
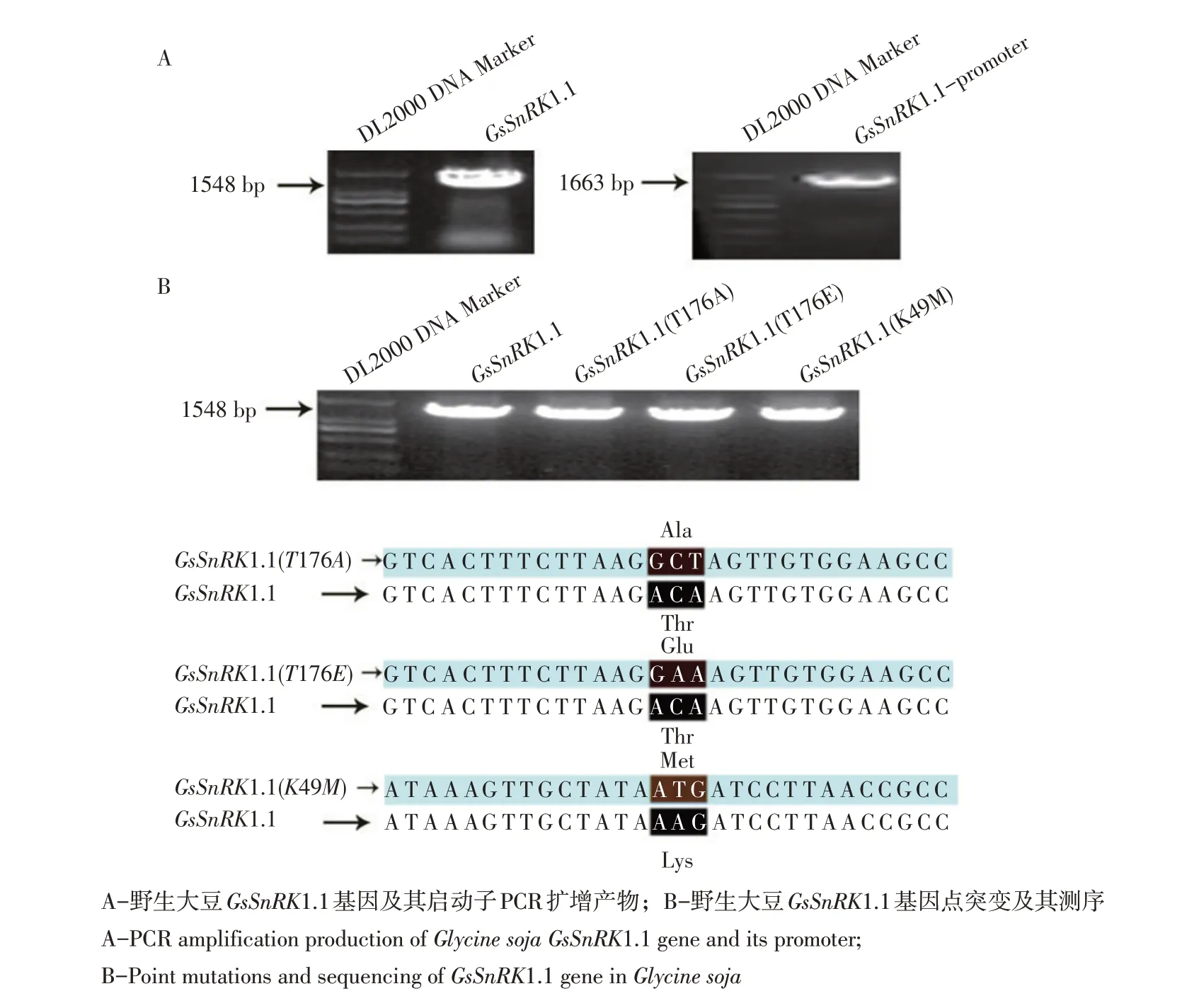
图1 野生大豆GsSnRK1.1基因克隆及点突变测序Fig.1 GsSnRK1.1 gene cloning and point mutation sequencing
2.2 野生大豆GsSnRK1.1基因表达响应淹水胁迫
SnRK1是一种广泛存在于植物体内的Ser/Thr蛋白激酶,其蛋白活性对植物体生长发育、新陈代谢及非生物胁迫抗逆具有重要作用。为探究野生大豆GsSnRK1.1基因对淹水胁迫的响应,分别对野生大豆淹水处理0、6、12、24和48 h,并对不同淹水时间处理野生大豆内源GsSnRK1.1基因作转录水平分析。结果如图2所示,随淹水处理时间增加,野生大豆中GsSnRK1.1基因转录水平随之显著升高。为进一步探究野生大豆GsSnRK1.1基因启动子对淹水胁迫的响应,构建pGsSnRK1.1::GUS稳定表达株系,GUS染色结果如图3所示,可观察到与未淹水处理对照组相比,淹水处理后染色效果更为明显,说明GsSnRK1.1基因启动子感知淹水胁迫进而促进基因转录。
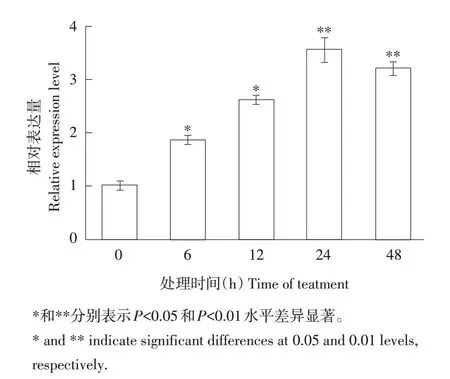
图2 GsSnRK1.1在淹水胁迫下表达模式Fig.2 Expression pattern of GsSnRK1.1 gene under waterlogging stress
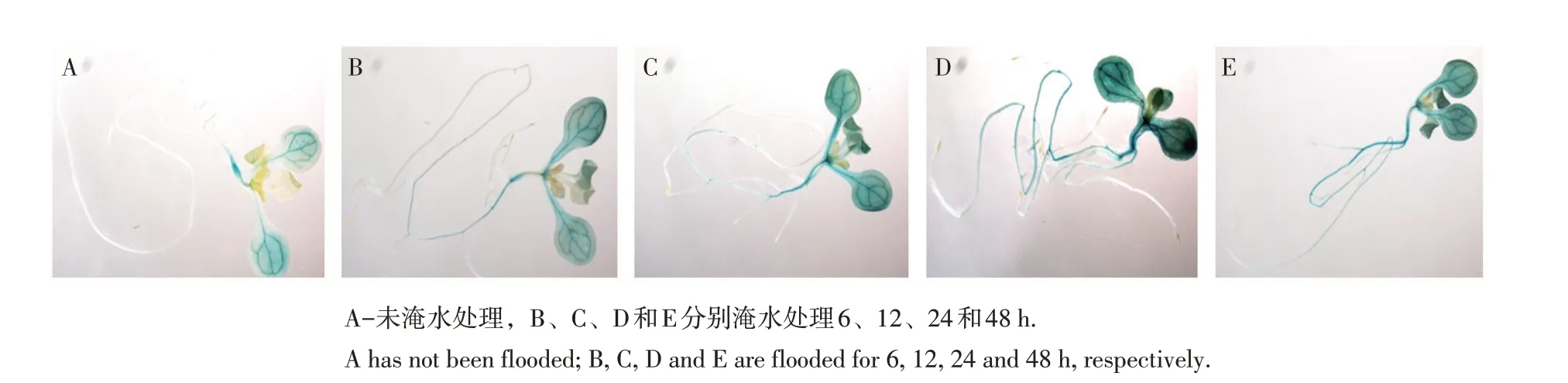
图3 淹水胁迫下proGsSnRK1.1:GUS转基因幼苗GUS染色Fig.3 GUSstaining of proGsSnRK1.1::GUS transgenic seedlings under waterlogging stress
2.3 GsSnRK1.1及其点突变在拟南芥中超表达鉴定
为研究GsSnRK1.1蛋白激酶在植物抵抗淹水胁迫中功能,将GsSnRK1.1及其3个活性位点突变基因在Col-0拟南芥中超表达。以atkin10为阴性对照,利用相应抗性筛选后获得T1代,再利用T5 Direct PCR(plant)试剂盒对初步筛选得到的转基因植株作基因型鉴定,如图4所示,Col-0超表达植株对GsSnRK1.1及其3个位点突变基因作PCR鉴定,基因长度为1 548 bp。
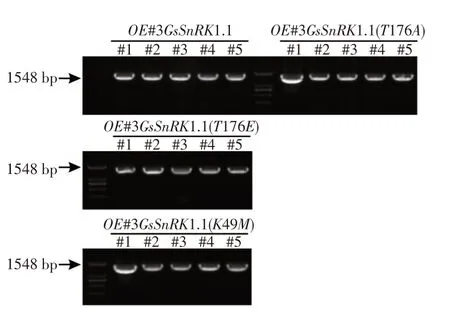
图4 GsSnRK1.1及其3个点突变基因超表达植株PCR鉴定Fig.4 PCR identification of overexpressed plantswith GsSnRK1.1 and three point mutated genes
从鉴定正确的T1代单个植株上收集种子,进一步对T2代作抗性和基因型筛选,获得纯合T3植株。为进一步确认转基因超表达和回补T3代拟南芥中蛋白表达,提取拟南芥叶片蛋白,并作Western blot蛋白水平测定,结果如图5所示,通过anti-Myc抗体验证确定35S启动子驱动的Myc-GsSnRK1.1、Myc-GsSnRK1.1(T176E)、Myc-GsSnRK1.1(T176A)、Myc-GsSnRK1.1(K49M)和空载体转化的纯合T3代中靶蛋白表达水平。
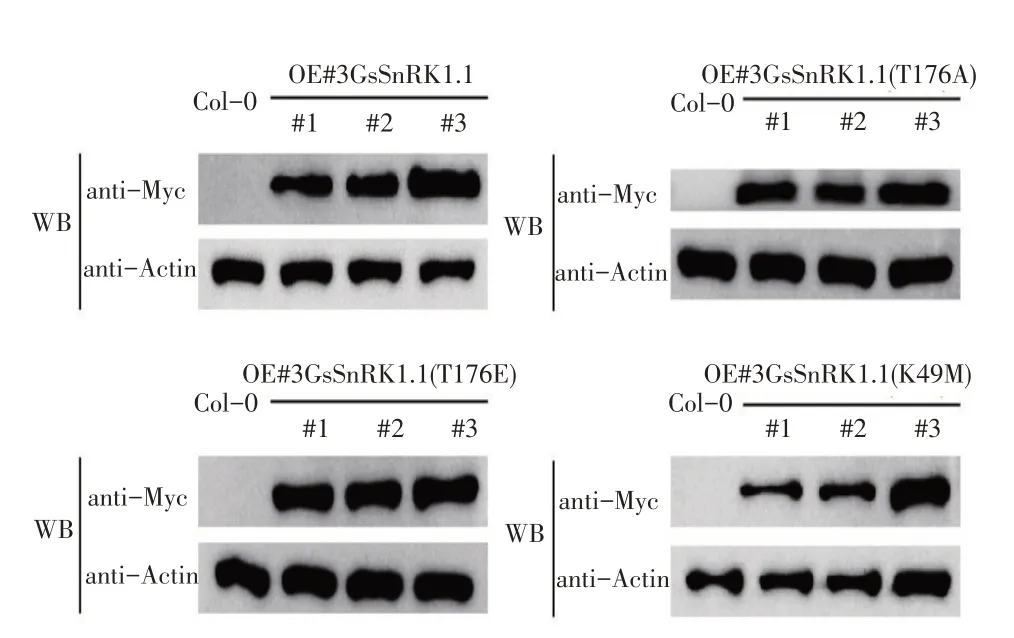
图5 转基因拟南芥Western blot蛋白水平检测Fig.5 Western blot protein level detection of transgenic Arabidopsis thaliana
2.4 GsSnRK1.1在淹水胁迫下生理指标测定
为进一步确定GsSnRK1.1蛋白激酶在淹水胁迫中生理功能,本研究利用超表达GsSnRK1.1、GsSnRK1.1(T176A)、GsSnRK1.1(T176E)和GsSnRK1.1(K49M)研究拟南芥转基因植株。如图6所示,Col-0绿化程度略高于atkin10,但并不明显;表达GsSnRK1.1蛋白激酶和模拟磷酸化GsSnRK1.1蛋白激酶(OE#T176E)的拟南芥叶片明显呈绿色,且比Col-0绿化程度更高;而表达催化区域失活的GsSnRK1.1蛋白激酶(OE#K49M)和表达调控区域失活的GsSnRK1.1蛋白激酶(OE#T176A)的拟南芥在淹水条件下明显叶片失绿,且比atkin10绿化程度更低;进一步测定各株系叶绿素含量,如图7所示,叶绿素含量与表型相符,说明GsSnRK1.1蛋白激酶活性可降低拟南芥在淹水条件下叶片失绿。

图6 淹水胁迫下转基因株系表型Fig.6 Phenotypesof transgenic linesunder waterlogging stress
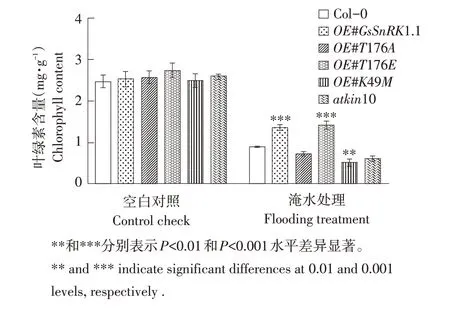
图7 淹水胁迫下转基因拟南芥叶绿素含量Fig.7 Chlorophyll content of transgenic Arabidopsis thaliana under waterlogging stress
测定经淹水胁迫处理后转基因拟南芥体内超氧化物歧化酶(SOD)和脯氨酸(Pro)含量,并计算淹水处理前后SOD和Pro含量增加倍数。如图8A所示,经淹水胁迫后,Col-0体内SOD酶活增加约20倍,OE#GsSnRK1.1和OE#T176E体内SOD酶活增加倍数约为35倍,且显著高于Col-0;OE#T176A体内SOD酶活增加约10倍,OE#K49M体内SOD酶活增加约7倍,atkin10体内SOD酶活增加约6倍,均显著低于Col-0。如图8B所示,淹水处理后,Col-0体内Pro含量增加约50倍,OE#GsSnRK1.1和OE#T176E体内Pro含量增加倍数分别约为140倍和170倍,且显著高于Col-0株系;OE#T176A体内Pro含量增加约23倍,OE#K49M体内Pro含量增加约24倍,atkin10株系中Pro含量增加约20倍,且三者均显著低于Col-0株系。由此说明,GsSnRK1.1可提高拟南芥SOD活性和Pro含量以抵抗淹水胁迫。
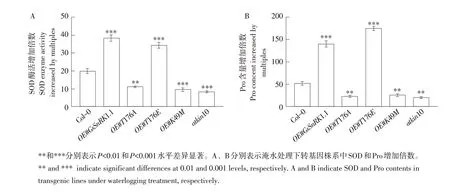
图8 淹水胁迫下转基因株系中SOD和Pro含量增加倍数Fig.8 Fold increase of SOD and Pro contents of transgenic lines under waterlogging stress
2.5 GsSnRK1.1影响淹水应激反应标记基因表达
乙醇脱氢酶1(ADH1)和丙酮酸脱羧酶1(PDC1)是标记淹水应激反应基因,为检测SnRK1活性对氧缺乏期间基因调节的影响,测定ADH1和PDC1标记基因在淹水胁迫下表达。如图9所示,ADH1和PDC1在Col中上调,并在表达OE#GsSnRK1.1(野生型GsSnRK1.1)和OE#GsSnRK(T176E)(表达模拟磷酸化GsSnRK1.1)转基因植物中进一步上调,但在OE#GsSnRK1.1(T176A)和OE#GsSnRK1.1(K49M)(表达无激酶活性的GsSnRK1.1)转基因植物中几乎未发生ADH1和PDC1基因上调。结果表明,GsSnRK1.1诱导淹水应激反应的两个标记基因表达,其依赖于GsSnRK1.1蛋白激酶活性。

图9 淹水应激反应标记基因表达模式Fig.9 Expression pattern of marker genes for stress responseto flooding
3 讨论
植物中蔗糖非发酵-1-相关蛋白激酶-1(SnRK1)与酵母SNF1和哺乳动物(AMPK)同源[11]。其中SnRK1蛋白激酶在感应新陈代谢方面具有重要意义,可抵抗各种压力并保持能量稳态[12]。SnRK1在植物抵抗生物及非生物胁迫过程中发挥作用,添加葡萄糖和ABA处理时,超表达SnRK1植株对ABA敏感程度更强。PP2C被ABA降低进一步提高SnRK1活性,通过两个互补过程相互作用增强应激反应,并为ABA和糖信号通路之间广泛遗传相互作用提供解释[14-15]。SnRK1α植物防御机制在食草动物、真菌、细菌和病毒中扮演重要角色[16-17]。首次发现植物SnRK1参与胁迫响应是反义表达StubGAL83的马铃薯对高盐胁迫极其敏感[18]。AtKIN10(SnRK1.1)在拟南芥耐淹性和抗盐信号途径调节过程中存在拮抗作用[19]。拟南芥(Arabidopsis thaliana)SnRK1活性提升拟南芥耐淹水能力,参加控制胁迫耐受的信号途径存在特异性,超表达失去激酶活性的OsSnRK1或者AtKIN10突变转录本植株,在淹水处理下其叶片明显白化[20]。本研究发现GsSnRK1.1可提高转基因拟南芥对淹水胁迫抗性,且在抵抗淹水胁迫中GsSnRK1.1蛋白激酶酶活性发挥关键作用。
植物受淹水胁迫后降低光合速率,进而影响其生长发育及产量收益,因此挖掘和利用抗逆基因资源对提高作物抗逆性非常重要。本研究选用野生大豆为植物材料,从中克隆GsSnRK1.1基因,通过多序列比对发现其蛋白结构中保守的STKc-AMPK-alpha结构域,以及对于行使蛋白激酶功能十分重要的保守氨基酸位点:K49和T176。其中第176位苏氨酸(Thr,T)是受上游激酶调控的特异性磷酸化位点,当其被上游激酶磷酸化,则激活催化区域第49位赖氨酸(Lys,K)从而磷酸化下游底物蛋白,影响植物生长发育。在Col-0野生型拟南芥中超表达以35S启动子驱动GsSnRK1.1、GsSnRK1.1(T176A)、GsSnRK1.1(T176E)和GsSnRK1.1(K49M)基因,获得转基因植株,检测GsSnRK1.1蛋白激酶活性对淹水胁迫标记基因调节的影响,并测定ADH1和PDC1标记基因在淹水胁迫下基因表达,结果表明,野生大豆GsSnRK1.1诱导淹水应激反应的两个标记基因表达,GsSnRK1.1基因对淹水胁迫响应,且参与淹水应激反应调控。有关GsSnRK1.1蛋白激酶对淹水胁迫下大豆根系乙醇脱氢酶、活性氧代谢系统及碳氮代谢等影响仍需进一步研究。

