小麦粒形QTL元分析及候选基因预测
王 鹏,田 甜,张沛沛,刘 媛,陈 涛,王彩香,程宏波,杨德龙
(1.甘肃省干旱生境作物学重点实验室,甘肃兰州 730070;2.甘肃农业大学生命科学技术学院,甘肃兰州 730070)
籽粒长度和宽度决定小麦(Triticumaestivum)种子的大小,进而影响小麦的产量和品质,是小麦育种中的重要农艺性状[1]。研究表明,小麦的粒长和粒宽是典型的数量性状,由多基因控制,其遗传基础复杂,易受环境因素的影响[2]。目前,QTL定位是深入研究复杂数量性状遗传基础的有效方法之一,已广泛应用于作物农艺性状遗传解析[3]。许多研究者利用不同遗传背景材料和遗传图谱,对小麦粒长和粒宽进行了QTL定位和遗传分析。Ramya等[4]以Rye Selection 111×中国春构建的RIL群体,检测到8个控制粒长的QTL和15个控制粒宽的QTL,分布在1A、1D、2B、2D、4B、5A、5B和5D染色体上,单个QTL可解释4.4%~11.5%的表型变异。Kumari等[5]以NW1014×HUW468构建的RIL群体,在4A和5A染色体上检测到2个控制粒长的QTL,在6A和7D染色体上检测到2个控制粒宽的QTL,单个QTL可解释7.66%~53.84%的表型变异。Xie等[6]以面包小麦×Spelt构建的RIL群体,在1D、2A、3B、4A、5A和7B染色体上检测到8个控制粒长的QTL;在2D和5A染色体上检测到2个控制粒宽的QTL,单个QTL可解释6.6%~31.8%的表型变异。然而,由于群体材料遗传背景、统计方法、标记类型和环境条件的不同,导致定位到的控制小麦粒长和粒宽的QTL数目、染色体位置和遗传效应差异较大,难以获得真实、稳定的QTL区段和位点,无法直接在育种实践中应用。因此,通过整合QTL,可提高小麦粒长和粒宽定位的精确度。
元分析是一种整合前人研究数据的统计分析方法,应用于动、植物的QTL整合分析。Arcade等[7]开发出用于元分析的BioMercator软件,该软件可将目标性状相关的所有QTL位点映射整合在一张遗传图谱上,通过比对分析,预测一致性真实QTL位点,并可进一步缩小置信区间,为精细定位奠定基础。Semagn等[8]收集183个与玉米花期和产量相关的QTL位点进行元分析,发现68个一致性QTL(meta quantitative trait loci,MQTL)。雷 蕾等[9]对344个水稻耐盐相关性状QTL进行元分析,最终获得46个MQTL,共筛选出9个与耐盐性状相关的候选基因。江培顺等[10]通过对584个控制玉米穗行数、行粒数、粒重相关的QTL进行元分析,分别发现7、22和44个与行粒数、穗行数和粒重相关的MQTL,最终得到10个与玉米产量性状相关的基因。本研究将已报道的控制小麦粒长和粒宽的QTL进行归纳整理,以小麦高密度遗传图谱作为参考图谱,利用BioMercator 4.2软件将这些QTL映射到参考图谱上,构建小麦粒长和粒宽QTL一致性图谱,并预测MQTL区间内与小麦粒长和粒宽相关的候选基因,以期为深入研究小麦粒长粒宽的遗传机制提供理论依据。
1 材料与方法
1.1 材 料
以美国农业部小麦公共数据库(http://wheat.pw.usda.gov/)和已发表的文献中控制小麦籽粒长度和宽度的QTL信息[2-4,6,11-21]为研究对象。
1.2 方 法
1.2.1 小麦粒长和粒宽QTL信息的收集整合
收集已报道的控制小麦粒长和粒宽QTL定位信息,按照BioMercator 4.2软件(https://www.mybiosoftware.com/biomercator-2-1-genetic-maps-qtl-integration.html)要求进行数据整理,包括QTL名称、染色体位置、置信区间、连锁系数、贡献率、临近标记、LOD值和群体大小等,其中,QTL位置(置信区间和QTL的最大可能位置)和遗传贡献率是进行QTL元分析的两个必要参数。
1.2.2 小麦粒长和粒宽QTL映射
确定收集到的QTL所涉及的染色体,选取Wheat composite 2004(https://wheat.pw.usda.gov/cgi-bin/GG3/report.cgi?class=mapdata;query=;name=Wheat,+Composite,+2004)高密度遗传图谱作为参考图谱,将目标QTL的位置信息按比例标注到参考图谱上,即QTL映射。将映射失败的标记先映射到Somers等[22]绘制的小麦整合图谱(即中介图谱)上,然后对共有分子标记进行映射,剔除有争议的QTL。
1.2.3 小麦粒长和粒宽QTL元分析
利用BioMercator 4.2软件将位于同一染色体相同位点附近的N个独立存在的与小麦粒长和粒宽相关的QTL进行运算,对独立来源的同一性状且位于同一座位或重叠座位的QTL计算出一个MQTL。每个MQTL给出5个模型(1、2、3、4和N),其中,赤池信息量准则值(akaike-type criteria values,AIC)最小的模型为真实QTL模型,按照最大似然函数比方法通过高斯定理计算其置信区间及物理位置。若某一QTL置信区间未知,可通过Darvasi等[23]应用的公式进行推断:
(1)C.I.=530/(N×R2)
(2)C.I.=163/(N×R2)
其中C.I.指QTL 95%的置信区间,N指作图群体的大小,R2指QTL的遗传贡献率,F2和BC群体用公式(1)进行计算,RIL、DH及NIL群体用公式(2)进行计算。若已知QTL置信区间,也可根据此公式计算其遗传贡献率[24]。
1.2.4 基于小麦粒长和粒宽MQTL范围内候选基因的发掘
利用生物信息学手段进行目标性状QTL的整合,获得粒长和粒宽MQTL区域,根据这些区域内的EST或DNA序列搜索候选基因。该方法的原理为:物种内及物种间存在序列同源性,基因的功能和序列紧密相关,当序列的相似性超过一定的范围时,它们执行的功能可能相同,通过对比未知功能序列和已知功能序列,预测序列功能。
在参考图谱Wheat composite 2004上,通过元分析所得的小麦粒长和粒宽MQTL区域由两端标记界定。对MQTL区间内相关基因位点进行整理,根据基因位点名称,在GrainGenes (https://wheat.pw.usda.gov/)网站上下载相关基因序列和各种标记的原始序列,从而确定MQTL在物理图谱上的位置。利用小麦基因组数据库(http://202.194.139.32/)中JBrowse工具检索MQTL内的基因信息,并获得该区间内所有基因的功能注释信息,利用NCBI网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi)BLAST比对相关基因序列,预测目标性状候选基因[25]。
2 结果与分析
2.1 QTL信息收集和一致性图谱整合结果
从表1可以看出,从小麦公共数据库和已发表的文献共收集到113个与粒长相关的QTL和86个与粒宽相关的QTL,它们来自于川35050×山农483、大粒饱×BYL8、Rye Selection 112×中国春、面包小麦×Spelt、波兰小麦×普通小麦、0911-46×42、Rye Selection 111×中国春、山农0431×鲁麦21、南大2149×望水白、西藏半野生Q1028×郑麦9023、0911-14×42、G18-16×Langdon、6044×01-35、人工合成小麦SHW-L1×川麦32共14个不同遗传背景的作图群体。利用BioMercator 4.2软件中的QTL projection功能将收集到的QTL映射到Wheat composite 2004小麦参考图谱上。与粒长相关的QTL LOD值范围为 2.00~12.90,遗传贡献率为0.04%~ 32.60%;与粒宽相关的QTL LOD值范围为 2.00~8.57,遗传贡献率为 0.03%~40.79%。

表1 小麦粒长和粒宽QTL数据整合Table 1 Integration of QTL data for the kernel length and width in wheat
将收集到的原始图谱上的QTL通过BioMercator 4.2软件映射到参考图谱Wheat composite 2004上,构建小麦粒长和粒宽QTL一致性图谱。结果(图1)表明,与粒长、粒宽相关的QTL在各染色体上分布不均匀,部分QTL在同一染色体上成簇存在。如在2B、2D、3A、4B和5A染色体上各有1个QTL簇。这些QTL大都出现在一段区间内,彼此之间有区间的重叠。其中,4B染色体上170.00 cM左右和5A染色体上85.00 cM左右各有3个QTL,说明该区间可能存在更为真实的QTL位点。
2.2 小麦粒长和粒宽QTL的元分析
利用BioMercator 4.2软件中的Maps-analysis程序对小麦粒长、粒宽QTL进行元分析,以每次元分析中AIC值最小的模型为准,确定1个真实QTL,最终得到18个控制粒长的MQTL,分别位于小麦的1A(4个)、2A(2个)、2B(3个)、2D(2个)、3A(1个)、4B(2个)、5A(1个)、5B(1个)、7A(1个)和7D(1个)染色体上,平均每条染色体上含有1.8个MQTL。根据其所在染色体的位置排列为MQTL1~MQTL18,其中,图距小于3 cM的MQTL位点有2个,即MQTL5(0.57 cM)和MQTL6(2.80 cM)。MQTL7(118.48~125.73 cM)和MQTL8 (119.58~139.22 cM)置信区间存在很大的重合,推测此重叠区段可能存在控制小麦粒长的重要基因(表2)。
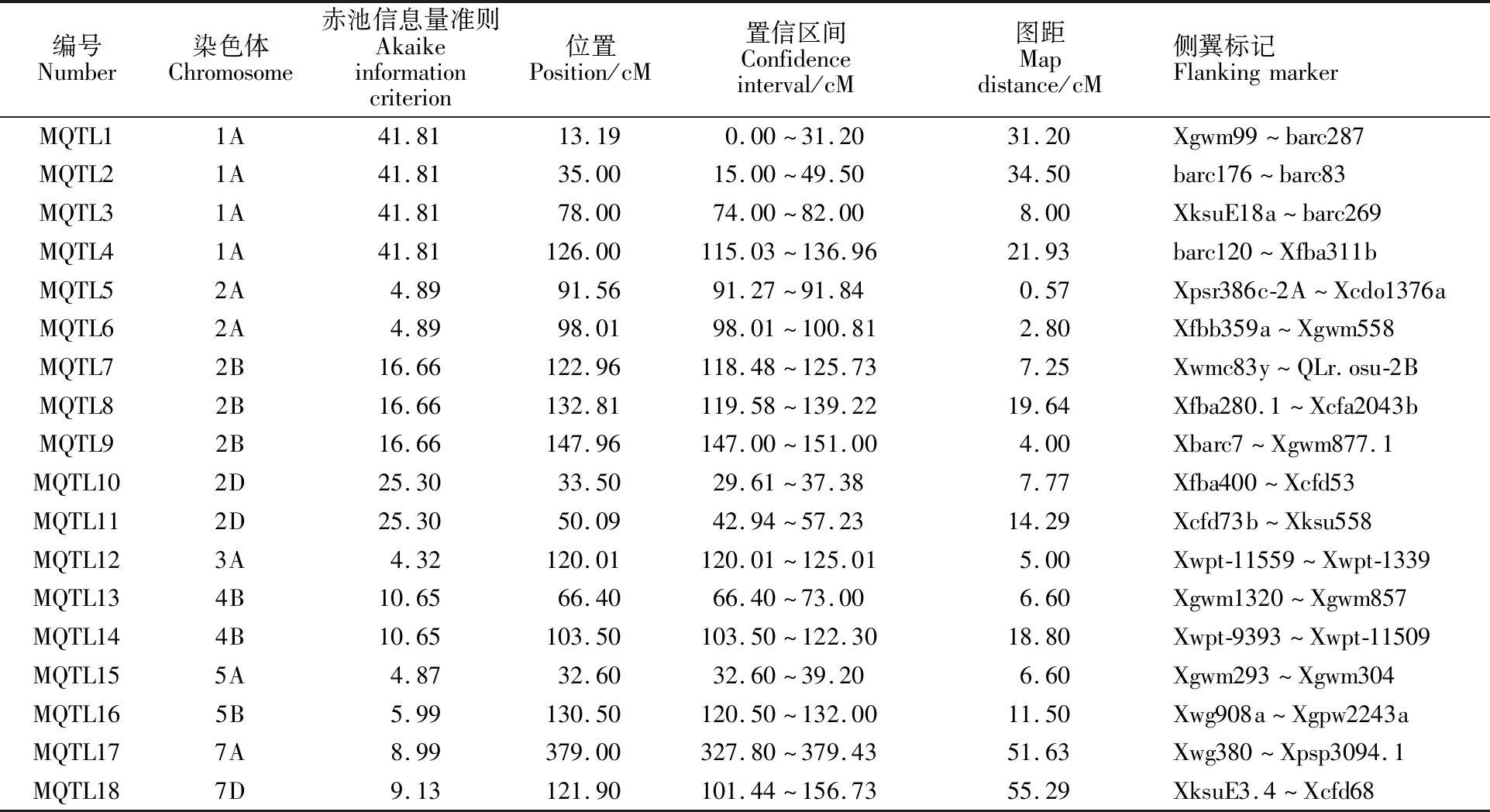
表2 小麦粒长QTL的元分析Table 2 Meta-analysis of QTLs for the kernel length in wheat
通过元分析得到了8个控制粒宽的MQTL,其中,2个MQTL定位在4B染色体上,3个MQTL定位在5A染色体上,在1A、3B和5B染色体上分别定位到1个MQTL。根据其所在染色体的位置排列为MQTL1~MQTL8,其中MQTL2和MQTL7图距小于4.00 cM,分别为3.70 cM和3.60 cM(表3)。

表3 小麦粒宽QTL的元分析Table 3 Meta-analysis of QTL for the kernel width in wheat
2.3 小麦粒长、粒宽MQTL区间内候选基因的预测结果
根据MQTL在物理图谱上的位置,利用小麦基因组数据库所提供的基因注释信息,预测小麦粒长和粒宽QTL定位区域内的候选基因。由于选取较小的MQTL AIC值和图距有利于提高QTL定位的准确度,因此,将小麦粒长和粒宽QTL定位结果中AIC值和图距均较小的MQTL进行物理图谱定位,统计MQTL区间内所包含的基因个数。结果发现,在5A染色体上,控制粒长的MQTL15其侧翼标记为Xgwm293~Xgwm304,图距为6.60 cM,包含28个基因;控制粒宽的MQTL7其侧翼标记为Xgpw2120~Xgpw2273a,遗传距离为3.60 cM,包含82个基因。在这两个MQTL区段进行Jbrowse检索,共筛选出22个候选基因(表4)。其中TraesCS5A01 G122300LC.1与小麦粒长相关,TraesCS5A01G-484900.1、TraesCS5A01G652600LC.1、Traes-CS5A01G652700LC.1、TraesCS5A01G487500.1、TraesCS5A01G651000LC.1、TraesCS5A01G651400LC.1共6个基因与小麦粒宽相关。它们通过编码光系统CP43反应中心蛋白、LEA蛋白、F-box蛋白、U-box蛋白和丝氨酸/苏氨酸蛋白磷酸酶,参与到小麦籽粒生长发育过程,如信号转导、光合作用和器官发育等途径,进而影响小麦粒长和粒宽,但具体功能有待进一步验证。

表4 小麦粒长和粒宽MQTL内预测到的候选基因Table 4 Candidate genes within MQTL related to the kernel lenqth and width in wheat

染色体左侧“点”至“横线”表示QTL所在位点的遗传贡献率大小的连续变化;“竖线”表示QTL所在置信区间。The “dot”to “transverse line”on the left side of chromosome mean the successive change in the genetic contribution rate of QTL;“Vertical line”means the confidence interval of QTL.图1 小麦粒长和粒宽QTL一致性图谱Fig.1 Map of QTLs for the kernel length and width in wheat
3 讨 论
高密度的遗传图谱可提高QTL映射的准确性和高效性。本研究选取整合了11个标准图谱的Wheat composite 2004作为参考图谱,其主要包含AFLP、RFLP和SSR标记,共涉及3 741个标记,总长度为3 236 cM。此图谱密度较大,但某些研究中原始图谱与参考图谱存在标记一致性较差的问题,导致部分目标性状QTL不能直接映射成功。因此,本研究利用Somers等[22]绘制的小麦整合图谱作为中介图谱,该图谱涉及到 1 234个SSR标记,总长度为2 540 cM,与Wheat composite 2004参考图谱存在大量共有标记,可以提高映射效果,缩短两标记距离,为精细定位奠定基础。
元分析技术可以对不同研究中定位的QTL进行比较整合,缩小其置信区间,提高QTL定位的精确性。目前,该技术已应用于多种作物目标性状QTL的遗传改良。吉海莲等[26]收集了22个抗玉米黑穗病QTL,发掘出2个抗病MQTL区间,并在其中初步确定1个抗病基因;杨鑫雷等[27]构建了含63个棉花纤维品质性状QTL的整合图谱,在12条染色体上获得15个相关性状的MQTL;李修平等[28]对43个作图群体的207个水稻抽穗期QTL信息进行整理,以Cornell 2001为参考图谱,共获得14个与水稻抽穗期相关的MQTL。利用元分析对小麦不同数量性状进行QTL整合的研究报道较少,王健维等[29]对控制小麦品质相关性状的585个QTL进行元分析,得到264个MQTL,其中34个与吸水率相关,26个与面粉蛋白质相关,84个与籽粒蛋白质含量相关,52个与籽粒硬度相关,39个与沉降值相关,29个与湿面筋含量相关,最小的置信区间为0.04 cM,表明基于元分析的 QTL 图谱整合及其在目标性状精细定位中具有重要应用价值。本研究收集14个群体113个控制小麦粒长的QTL和86个控制小麦粒宽的QTL,构建小麦粒长和粒宽QTL一致性图谱。通过元分析得到18个粒长MQTL和8个粒宽MQTL,置信区间均缩短,其中最小可缩短至0.57 cM,减小了QTL定位的误差。其中,本研究在5A染色体上Xgwm239~Xgwm304区间内获得的控制粒长的MQTL与王建维等[29]发现的控制小麦品质的MQTL(P41/M49-76~Xwmc492)有重叠区段,说明这些位点的表达可能存在遗传关联,或存在“一因多效”现象。
QTL定位的准确性和置信区间大小是定位和克隆目标性状相关候选基因的基础,利用元分析的方法确定和缩小置信区间,能够有效提高候选基因克隆效率。随着更多目标性状QTL的精细定位,借助生物信息学、模式作物的基因测序结果和比较基因组学方法,在MQTL区域内可以得到更多DNA序列,这些序列具有较完整的基因结构,根据同源对比,可以预测基因的功能,确定目标性状候选基因。本研究在控制小麦粒长和粒宽的MQTL内初步确定了7个候选基因。其中,Traes CS5A01G484900.1编码LEA蛋白,研究表明,LEA蛋白在植物种子脱水成熟过程中积累,调控种子含水量的降低速率,进而影响种子形态[30];TraesCS5A01G651000LC.1和TraesCS 5A01G651400LC.1编码丝氨酸/苏氨酸蛋白磷酸酶,可调节植物激素信号通路中关键基因的表达,影响细胞周期蛋白的磷酸化状态,最终影响种子形态[31];TraesCS5A01G 652600LC.1和TraesCS5A01G652700LC.1编码光系统CP43反应中心蛋白,CP43是一种叶绿素-蛋白质复合物,主要参与太阳能的捕获、激发能量向光化学反应中心的有效转移,调节能量流动进而影响种子形态形成[32];TraesCS5A01G122300LC.1编码U-box型泛素连接酶E3,参与蛋白降解、细胞周期调节、信号传导和形态发生等过程,从而影响植物的生长发育[33];TraesCS5A01G487500.1编码F-box蛋白,是组成E3泛素连接酶的成分,参与植物的信号转导、形态发生和器官发育等过程[34],Chen等[35]在水稻中发现了一个F-box基因OsFBK12,发现其可通过影响种子发育和籽粒灌浆调控种子大小。本研究通过对来自不同作图群体的小麦粒长和粒宽QTL进行整合,通过元分析方法获得更为精确的MQTL,在相应区段发掘候选基因,为小麦粒形QTL精细定位和基因图位克隆奠定理论基础,对小麦粒形分子标记辅助选择育种具有重要的意义。
4 结 论
借助小麦高密度遗传图谱,对199个来自不同遗传作图群体的控制小麦粒长(113)和粒宽(86)的QTL进行元分析。建立一致性图谱,共获得18个控制粒长的MQTL和8个控制粒宽的MQTL,置信区间最短缩短至0.57 cM。在5A染色体上预测到7个与小麦粒长(1)和粒宽(6)相关的基因,但具体功能有待进一步验证,这为小麦粒形分子标记辅助选择育种提供参考依据。

