睡莲转录因子bZIP家族的分子进化以及功能分析
叶方婷,潘鑫峰,毛志君,李兆伟,范凯
睡莲转录因子bZIP家族的分子进化以及功能分析
叶方婷,潘鑫峰,毛志君,李兆伟,范凯
福建农林大学农学院/作物遗传育种与综合利用教育部重点实验室,福州 350002
【】基于睡莲基因组鉴定睡莲bZIP(basic leucine zipper)家族成员,并对其进行分析,以揭示睡莲bZIP家族的分子进化和功能。从Waterlily Pond数据库获取睡莲基因组序列,利用HMMER3.0程序识别睡莲的bZIP家族成员,并使用CDD程序进一步确认其含有的保守bZIP结构域,使用IQ-tree软件构建系统进化树。利用ExPASy和SOPMA在线网站进行蛋白质结构分析,通过MEME程序进行保守基序分析,使用MCScan和Circos软件对基因复制事件进行分析以及可视化展示。从NCBI下载睡莲转录组数据(SRA Study:SRP222853),用R软件对睡莲bZIP家族成员表达数据的Pearson相关系数(PCC)进行计算和可视化分析,使用Cytoscape软件对NcbZIP成员之间的表达数据关系进行分析。从睡莲基因组中共鉴定出46个bZIP家族成员,按成员在染色体上的分布命名为—。根据系统进化分析可以将睡莲bZIP家族成员分为A、B、C、D、E、G、H、I、J和S共10个亚家族,其中A亚家族所含成员最多(11个),相同亚家族成员具有相似的保守结构域和基因结构。理化性质分析表明,睡莲bZIP家族成员蛋白质长度介于101—1 898 aa,分子量大小介于12.04—214.64 kD。染色体定位分析发现,睡莲共有14条染色体,46个bZIP家族成员不均匀地分布在其中的10条染色体上,其中1号染色体上分布最多。睡莲bZIP基因家族发生10个复制事件,其中9个片段复制事件,1个串联复制事件,A亚家族所含基因复制事件最多(3次)。对成员在不同组织下的表达进行分析,根据表达情况分为Ⅰ、Ⅱ和Ⅲ组,Ⅰ组成员在所有组织中均高度表达,Ⅱ组成员几乎在所有组织中均不表达,Ⅲ组成员在不同组织中表达水平各不相同,其中C、D和G亚家族的大部分成员集中在Ⅲ组。通过睡莲成员表达量的Pearson相关系数分析,发现与所有成员之间的相关性最高。在睡莲基因组中鉴定出46个bZIP成员,分为10个亚家族,不均匀地分布在14条染色体上,结构进化保守,组织表达模式多样。
睡莲;bZIP家族;分子进化;表达谱;功能分析
0 引言
【研究意义】睡莲是基部被子植物,对植物进化的研究具有重要意义。睡莲基因组的破译,揭示了睡莲科植物与十字花科植物之间的分化时间大约为147—185百万年前,基因组共线性分析为睡莲发生全基因组复制事件提供了证据[1],这有助于解析睡莲中基因家族的分子进化研究。【前人研究进展】在植物界中,转录因子bZIP(basic leucine zipper)是一类包含众多成员的家族[2-3]。bZIP家族包含两个保守的功能结构域,一个是由16个氨基酸残基组成的基本碱性区域,另一个是由1个七肽重复的亮氨酸或其他质量较大的疏水氨基酸组成的亮氨酸拉链二聚体基序区域[4-5],bZIP家族在植物胁迫应答、生物合成等方面发挥重要作用[6-8]。转录因子bZIP家族与植物生长发育有密切关系。在橄榄(L)中,在果实快速积油期高度表达,通过比较油分积累的动态变化,发现4个与橄榄果实发育和油脂合成密切相关的基因,说明橄榄bZIP成员可能参与果实的发育和脂质的合成[9]。转录因子bZIP家族在物质积累和胁迫应答过程中也发挥着重要作用。在花生中,发现bZIP家族中G亚家族成员和可能具有抗盐胁迫的能力[10]。对菠萝转录因子bZIP家族的全基因组分析鉴定以及表达谱分析,发现在所有组织中均表达上调;对菠萝的根和地上部进行抗盐、干旱、冷和热胁迫处理后发现,部分bZIP转录因子在植物遭受胁迫一段时间后也出现上调表达[11]。此外,bZIP成员可能参与许多生物代谢过程。通过对亚洲棉和雷蒙德士棉bZIP蛋白的功能进行GO分析预测,发现bZIP成员可能参与有机物代谢过程、初级代谢过程、细胞代谢过程的调节和氮化合物代谢过程等[12]。另外,在柳枝菊中也进行了全基因组分析,并对bZIP可能参与的表达和功能进行了预测[13]。【本研究切入点】目前未见对睡莲bZIP家族的研究,本研究利用全基因组分析的方法对睡莲bZIP家族分子进化及功能进行探索。【拟解决的关键问题】本研究通过构建系统发育树对睡莲与拟南芥bZIP家族同源关系进行比较基因组学研究,进而对睡莲bZIP成员基因结构、保守基序和基因复制事件进行分析,揭示bZIP成员的分子进化历程,同时通过分析睡莲中bZIP成员表达量、启动子元件等,为阐明睡莲转录因子bZIP家族的功能奠定一定的基础。
1 材料与方法
试验于2020—2021年在福建农林大学农学院作物遗传育种与综合利用教育部重点实验室进行。
1.1 转录因子bZIP家族成员的鉴定和系统发育分析
从Waterlily Pond数据库(http://waterlily.eplant.org)获取睡莲基因组序列,随后使用HMMER3.0程序进行HMM搜索,从Pfam数据库(https://pfam.xfam.org/)中获得bZIP结构域的HMM模型(PF00170),用以识别睡莲的bZIP成员。拟南芥的bZIP成员主要从TAIR数据库中下载获得(https://www.arabidopsis.org/)。所有的bZIP成员均使用CDD(https://www.ncbi.nlm. nih.gov/cdd)程序,进一步确保每一个候选基因均含有保守的bZIP结构域。
采用MAFFT v7.453对所有bZIP序列进行氨基酸序列比对分析,然后使用IQ-tree v2.0.7软件构建系统发育树,应用ModelFinder程序选择最佳模型,bootstrap值设置为1 000。使用MEGA v10.1.8软件对系统发育树进行可视化分析,并使用在线工具ExPASy网站(http://web.expasy.org/protparam/)和SOPMA网站(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对睡莲bZIP成员的一级结构和二级结构进行分析。
1.2 睡莲中转录因子bZIP家族序列分析
拟南芥和睡莲bZIP成员的保守基序通过在线工具MEME(http://meme-suite.org/tools/meme)进行鉴定,设置参数与前人研究类似[14]。从Waterlily Pond数据(http://waterlily.eplant.org)中下载睡莲bZIP成员的注释文件,从中提取基因结构和染色体位置信息,并通过使用TBtools v1.068软件对保守基序、基因结构和染色体定位信息进行可视化展示。
本研究从睡莲基因组数据库中提取NcbZIP成员上游的2 000 bp序列,用PlantCARE数据库(http:// bioinformatics.psb.ugent.be/webtools/plantcare/html/)确定NcbZIP成员启动子中顺式调控元件,最后使用TBtools v1.068软件进行可视化分析。
1.3 睡莲bZIP成员基因复制事件分析
使用MCScan(https://github.com/tanghaibao/jcvi/ wiki/MCscan-Python version)软件对NcbZIP成员的基因复制事件进行分析,基因复制关系使用Circos v0.67软件进行可视化展示。利用TBTools v1.068软件估计非同义替换率(Ka)、同义替换率(Ks)及其比值(Ka/Ks)。
1.4 睡莲bZIP成员的表达分析
本研究从NCBI数据库(https://www.ncbi.nlm.nih. gov/)下载了睡莲不同组织(成熟叶、幼叶、成熟茎、幼茎、花、萼片和心皮)的RNA-seq转录组数据(SRA Study:SRP222853),下载的RNA-Seq数据主要通过之前的转录组数据处理方法进行数据分析[15]。睡莲中bZIP成员的表达水平通过FPKM值进行log2(FPKM+1)转换获得。在RStudio中使用ComplexHeatmap v2.4.2软件包对NcbZIP成员的表达水平进行聚类分析,同时使用ggcorrplo v0.1.3软件包对NcbZIP成员之间表达数据的Pearson相关系数(PCC)进行计算和可视化分析,并利用Cytoscape v3.7.2软件对NcbZIP成员之间的表达数据网络图进行分析。
2 结果
2.1 睡莲中bZIP成员的鉴定与系统发育分析
在睡莲中共鉴定出46个bZIP成员,根据其在染色体上的位置,将其命名为-。根据前人对拟南芥的研究结果进行分类[13],可以将睡莲的46个bZIP成员共分为10个亚家族,并命名为A、B、C、D、E、G、H、I、J和S(图1)。在睡莲bZIP家族中,A亚家族和D亚家族的bZIP成员数量最多,分别有11个成员和7个成员;在I亚家族中有6个成员,S亚家族和G亚家族有相同的成员数量(5个);C、E、H、B和J五个亚家族分别只有4、3、2、2和1个bZIP成员,而在F亚家族中没有发现睡莲bZIP成员。
睡莲bZIP成员的蛋白质长度介于101 aa(NcbZIP43)—1 898 aa(NcbZIP13);分子量大小12.04 kD(NcbZIP43)—214.64 kD(NcbZIP13);其中,B亚家族成员的平均蛋白质长度(774 aa)和分子量(84.17 kD)最大,H亚家族成员的平均蛋白质长度(167 aa)和分子量(18.57 kD)最小。在睡莲bZIP家族成员中,有一半的pI小于7.0,最低达4.79;另一半pI值大于7.0,最高可达9.92(表1)。
2.2 睡莲bZIP成员保守基序分析
本研究在睡莲bZIP成员中共鉴定到20个保守基序,大部分睡莲bZIP成员都含有Motif 1和Motif 7(图2)。对各组所含基序进行统计分析,发现D亚家族和I亚家族所含保守基序的种类最多,有7种保守基序;H亚家族和J亚家族的保守基序种类最少,均只有2种。此外,本研究还发现一些亚家族所特有的基序,如Motif 4、Motif 5、Motif 18和Motif 20为D亚家族所特有,Motif 11、Motif 15和Motif 17只在B亚家族中发现,Motif 16和Motif 19为E亚家族和I亚家族所共有。

图1 拟南芥和睡莲bZIP家族的系统发育分析
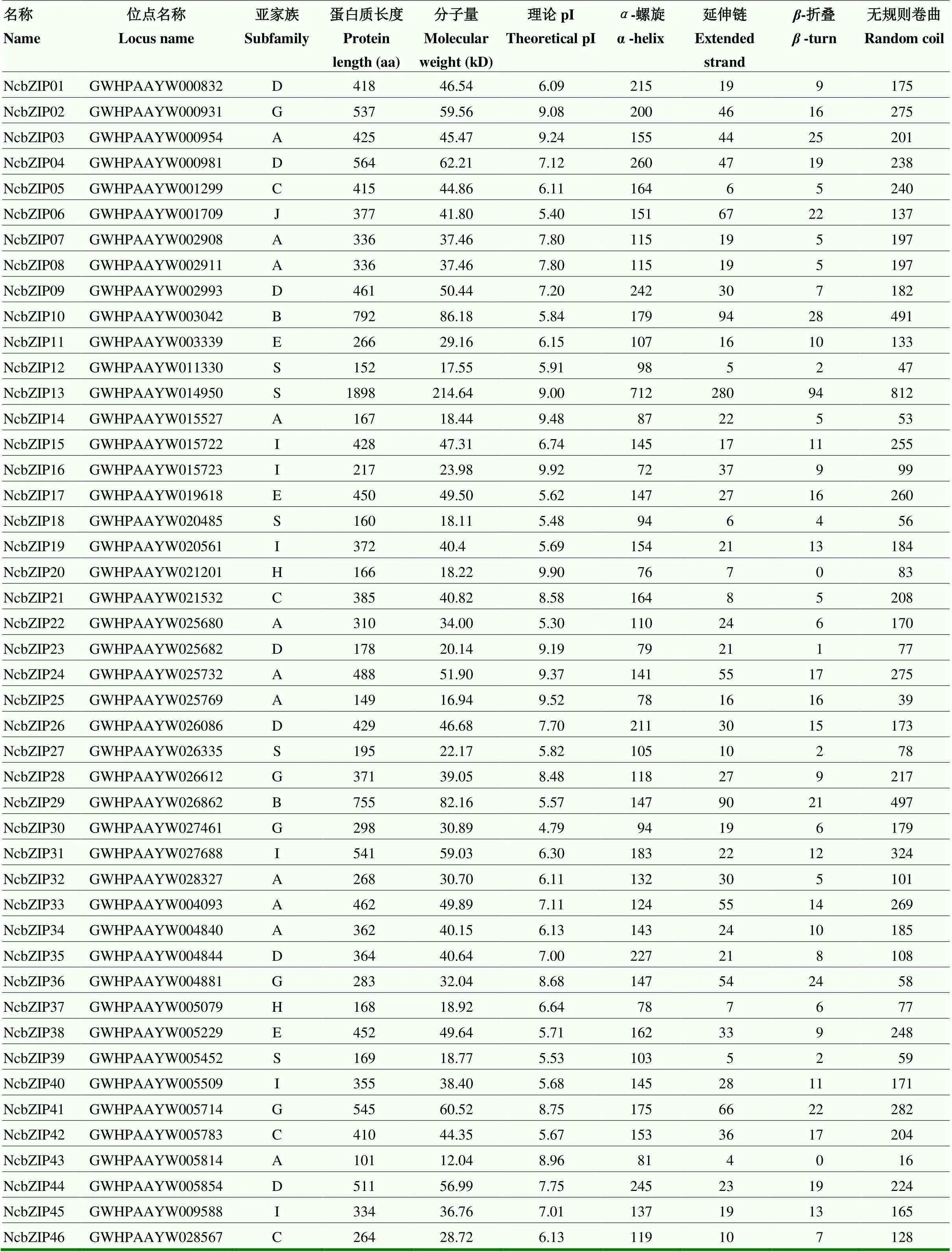
表1 本研究中NcbZIP成员的鉴定及结构分析
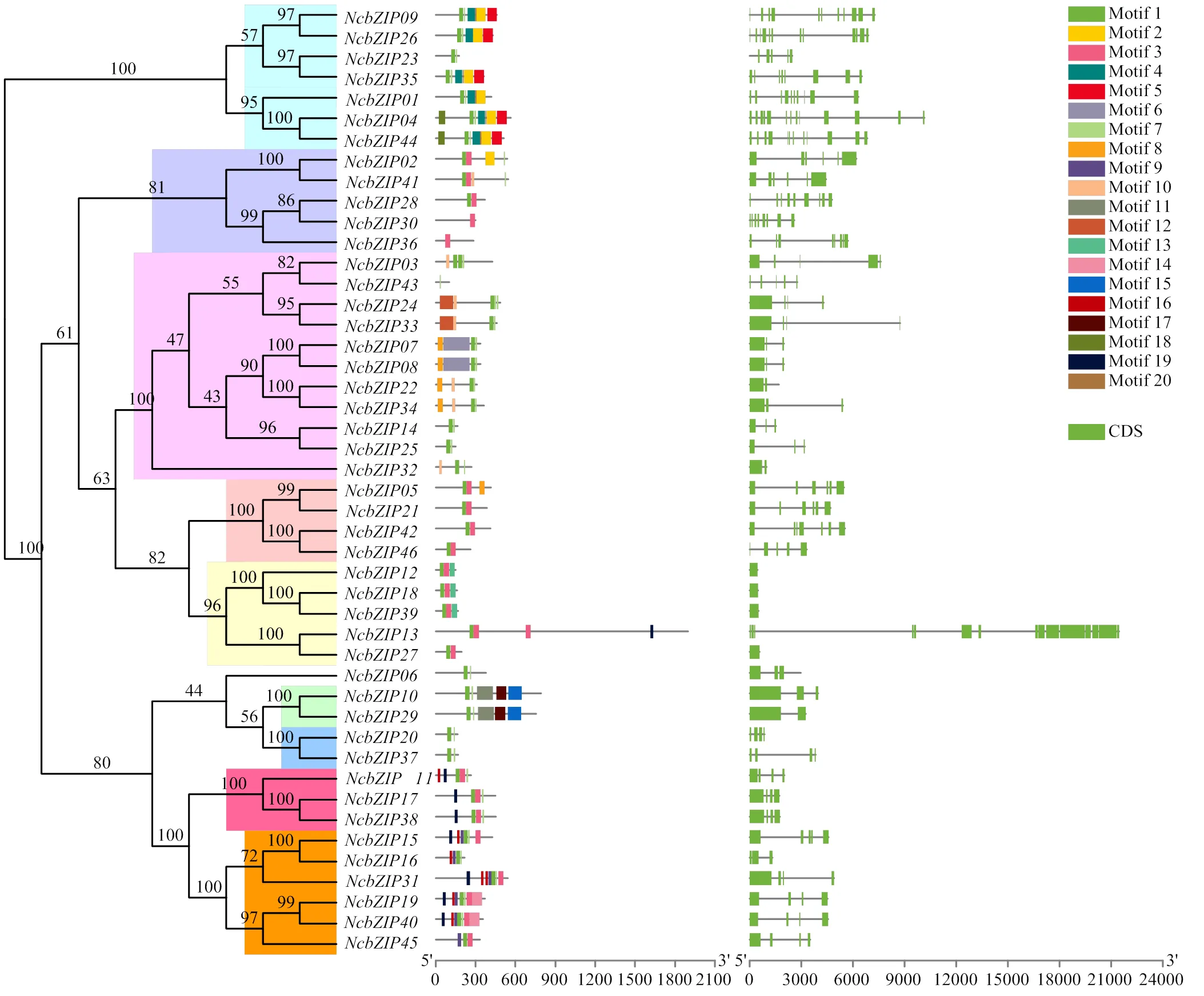
图2 睡莲中bZIP家族保守基序(左)与基因结构(右)的鉴定
2.3 睡莲bZIP成员的基因结构分析
最复杂的基因结构出现在成员(含有16个外显子和15个内含子),最简单的基因结构出现在成员(仅含有1个外显子,没有内含子)。D亚家族的基因结构最为复杂,平均每个基因含有10个外显子;其次是G亚家族,每个基因平均有7个外显子;最简单的基因结构出现在S亚家族,除了外,大部分成员仅含有1个外显子。
2.4 睡莲bZIP成员的染色体定位分析
睡莲共有14条染色体,而46个成员分布在睡莲10条染色体上,4、7、12和14号染色体上没有发现任何睡莲成员(图3)。在睡莲中,不同染色体含有不同数目的成员。睡莲1号和10号染色体上分布较多的成员,分别有11个和8个成员;在2号和13号染色体上分布较少的成员,均只有1个成员。在1号染色体的上端和下端均发现成员的聚集现象,在8号染色体的下端、10号染色体的下端和11号染色体的上端也发现成员的聚集现象。
2.5 睡莲bZIP成员的基因复制事件分析
在睡莲bZIP家族中共鉴定出10个基因复制事件,其中有9个片段复制事件(////////和/)和1个串联复制事件(/)(图4-A)。不同亚家族含有不同数目的基因复制事件,在A亚家族中有3个复制基因事件,在I亚家族中有2个复制基因事件,而在C、D、E、G和S亚家族中均只有1个复制基因事件(图4-B)。随后对这些复制基因事件的Ka和Ks进行分析,通过计算复制基因对的同义替换率分布,发现其约在1.108处Ks达到峰值(图4-C)。所有复制基因对的Ka/Ks比值均小于1(表2),因此,以上所有复制基因对都经过纯化选择作用。此外,在睡莲的bZIP家族中,发生串联复制事件基因对的Ks值基本小于发生片段复制事件的基因对。
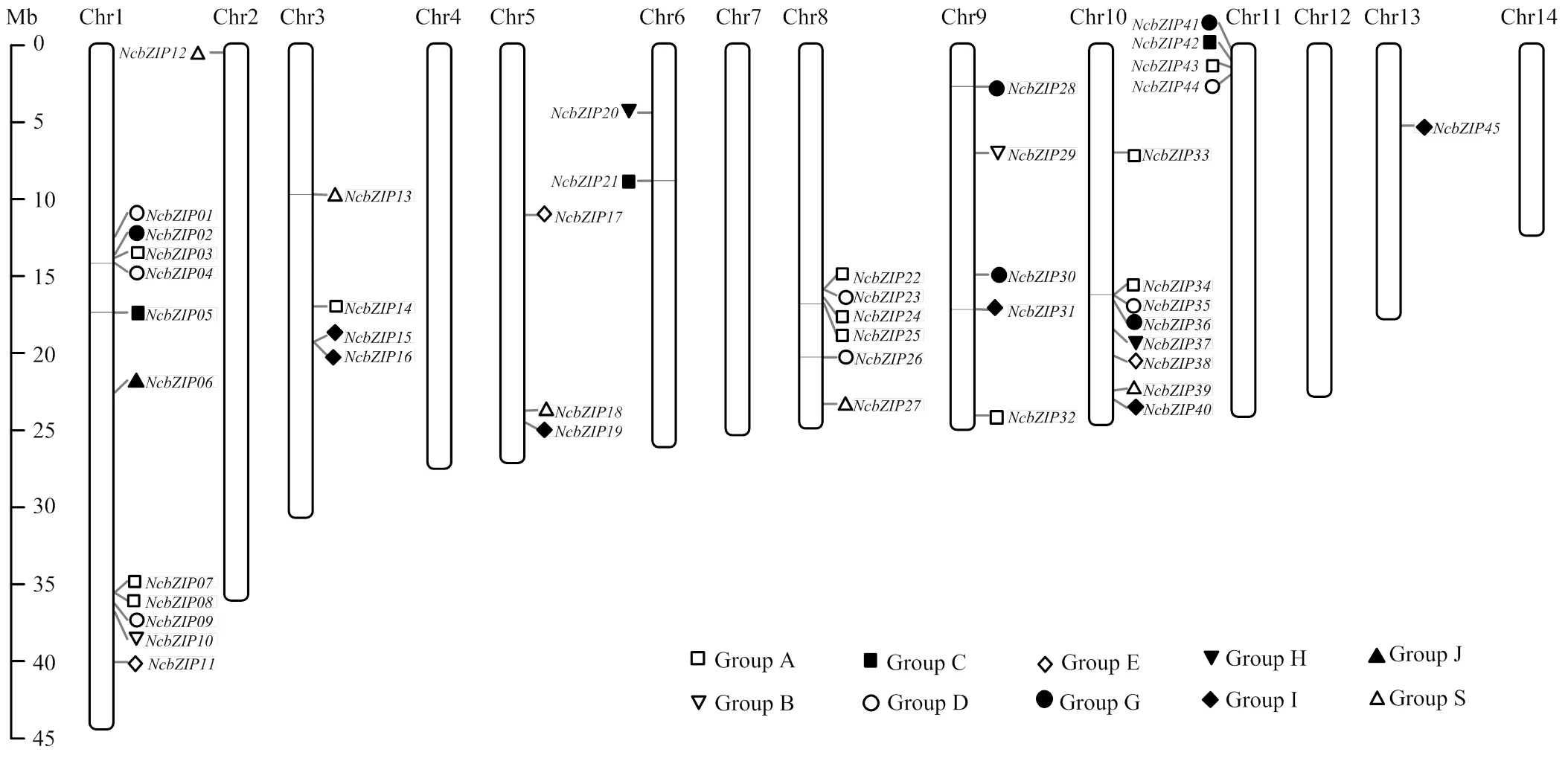
图3 睡莲bZIP成员的染色体定位

表2 睡莲复制的NcbZIP成员的Ka和Ks分析

A:睡莲bZIP复制基因的共线性分析;B:睡莲bZIP复制基因的亚家族分布;C:睡莲bZIP复制基因的Ks分布,箭头指向Ks峰值
2.6 睡莲bZIP家族的顺式调控元件分析
顺式调控元件中的光反应、植物激素和高温/低温胁迫等调控元件在植物胁迫应答方面起着重要作用[16]。本研究通过PlantCARE数据库分析获得的睡莲成员的顺式调控元件,其中包括生长素响应元件、水杨酸响应元件、赤霉素响应元件、茉莉酸甲酯响应元件、低温响应元件、ABA响应元件、防御和应激响应元件以及MYB结合位点(图5)。其中,茉莉酸甲酯响应元件数量最多,存在于每个成员中;ABA响应元件和茉莉酸甲酯响应元件几乎占所发现顺式调控元件的一半以上,其中几乎有一半分布在A、D和G亚家族中;防御和应激响应元件相对较少,在46个睡莲bZIP成员中只发现14个防御和应激响应元件。在B亚家族中生长素响应元件最多,E亚家族含有最多的顺式调控元件是ABA响应元件。
2.7 睡莲bZIP成员在不同组织中的表达分析
对睡莲成员在成熟叶、幼叶、成熟茎、幼茎、花、雄蕊、萼片和心皮中表达情况进行分析,通过基因表达聚类分析可以将睡莲成员大致分为3个组(Ⅰ、Ⅱ和Ⅲ)(图6)。在Ⅰ组中,14个成员几乎在所有组织中均高度表达。在Ⅱ组中,13个成员几乎在所有组织中均不表达;在Ⅲ组中,19个成员在不同组织中表达水平各不相同,具有一定的组织特异性,例如,在成熟叶和成熟茎的表达量最高,仅在花中高表达,而在雄蕊中具有明显的高表达。此外,大部分的D亚家族成员集中分布在Ⅲ组,该亚家族在花中均有较高的表达,C亚家族和G亚家族的大部分成员也集中分布在Ⅲ组。
本研究根据表达量计算出睡莲成员之间的Pearson相关系数(PCC)(图7),发现在10个发生基因复制事件的基因对中,有9对基因对呈正相关关系,1对基因对(和)呈负相关关系。随后对不同亚家族成员的相关性进行分析,发现E亚家族3个成员(和)之间的相关系数高达0.707,其次是D亚家族成员的相关系数较高(0.444)。此外,本研究对成员之间的相关性进行了研究,发现与所有成员间的相关性最高,其次为。对PCC大于0.5的44对成员进行分析,发现与其中21个成员均有较高的相关性(图8)。

图5 NcbZIP启动子区域的胁迫响应调节元件分析
3 讨论
bZIP家族在植物生长发育、干旱胁迫、盐胁迫、高温胁迫等生物学过程中发挥着重要的作用[17-21]。本研究在睡莲中共鉴定了46个转录因子bZIP成员,而在水稻中鉴定出89个bZIP成员[22-23],在大豆中鉴定出160个bZIP家族成员[24],其中,睡莲的基因组大小为409 Mb[1],水稻基因组大小是466 Mb[25],大豆的基因组大小为1 100 Mb[26]。睡莲是古老且保守的植物,所含的bZIP成员数量相对较少,可能与其基因组大小有关。此外,在bZIP家族系统发育分析中,睡莲bZIP家族的A亚家族和D亚家族所含成员数最多,其次为I亚家族,而在J亚家族中成员数量最少,在拟南芥、水稻、棉花中也有类似的分布[27],这表明植物中bZIP家族的亚家族分布具有一定的保守性。睡莲bZIP家族中共发现10个基因复制事件,不同亚家族发生基因复制事件数不同,因此,基因复制事件在不同亚家族中发生的频率不同可能是导致各亚家族之间成员数量产生差异的原因之一,相似的结果也出现在荞麦bZIP转录因子家族[28]。另外,在睡莲bZIP家族中,相同bZIP亚家族具有相似的保守基序分布和基因结构分布,这与bZIP家族成员的系统发育分析结果类似。

图6 睡莲bZIP成员在不同组织中的表达模式
通过MCScan软件分析bZIP成员的基因复制事件,发现A亚家族的基因复制事件发生的最多,这可能是导致其亚家族成员数量较多的原因之一,相似的研究结果也出现在番茄、土豆等植物的bZIP家族[29-30]。对基因复制事件的Ks值进行计算,发现大部分基因复制事件的Ks值集中在1.108附近,由此推测成员的基因复制事件发生在睡莲全基因组复制事件中。此外,在睡莲bZIP家族中,串联复制事件的Ks值几乎比所有片段复制事件的Ks值小,说明串联复制事件发生的时间是在片段复制事件后,这表明在睡莲中大规模发生的片段复制事件可能早于小规模发生的串联复制事件。与此同时,在睡莲bZIP家族中,有9个片段复制事件和1个串联复制事件,说明在睡莲bZIP家族的扩增中,片段复制事件是bZIP成员扩增的主要原因。此外,所有基因复制事件的Ka/Ks值均小于1,说明所有基因复制事件都经历纯化选择作用,且大部分复制基因对的PCC值都大于0,即呈正相关性,类似的现象也出现在大豆PP2C(蛋白磷酸酶2C)家族[31]、玉米NAC家族[14]、棉花KUP家族[32]等作物。

图7 睡莲bZIP家族表达谱的PCC热图
本研究对睡莲bZIP成员启动子的顺式调控元件进行分析,发现成员的启动子区域中含有最多的响应元件为茉莉酸甲酯响应元件,其次是ABA响应元件,表明成员可能受到茉莉酸甲酯和ABA的调控。在前人的研究中发现,棉花bZIP家族的A亚家族成员通过ABA介导可提高植株的抗旱性和耐盐性[33];在拟南芥A亚家族中,植株受脱落酸和非生物胁迫时,可通过ABA响应元件来诱导的表达[4];脱落酸和茉莉酸甲酯是影响植物生长发育和逆境响应的主要信号分子,可以参与植物多种生理过程[34-39]。在甘蔗中,曾发现ABA和茉莉酸甲酯调控了8个甘蔗bZIP成员的表达[40]。由此推测,睡莲的A亚家族成员在ABA或应激信号传导中可能具有重要作用。对转录组数据进行分析,发现睡莲bZIP成员在成熟叶、幼叶、成熟茎、幼茎、花、雄蕊、萼片和心皮的表达可大致分为Ⅰ、Ⅱ、Ⅲ组。其中,A亚家族一半的成员和E亚家族的3个成员集中出现在Ⅱ组中,它们在所有组织中均不表达;在甘薯转录因子bZIP家族的鉴定分析中,E亚家族的大部分成员在花中也不表达[41]。来自于D亚家族的在花和雄蕊中有较高的表达,与其相同亚家族的拟南芥bZIP成员AT1G08320和AT5G06839参与花药的发育[42];此外在S亚家族中,除外,其余成员均在雄蕊和心皮中有较高的表达,表明D和S亚家族的成员可能与花器官的发育有关。在对NcbZIP成员表达量的PCC分析发现,与其他NcbZIP成员的表达谱之间具有高度的正相关性,其在拟南芥的同源基因(AT1G43700)可以通过ABA调控植株的耐旱性[43],其在水稻中的同源基因LOC_Os11g06170可以调控茉莉酸代谢和信号传导通路的相关基因[44],而的启动子元件中含有茉莉酸甲酯响应元件最多,其次是ABA响应元件,表明可能具有通过ABA和茉莉酸甲酯介导调控睡莲非生物胁迫响应的功能。

图8 睡莲bZIP家族成员网络关系图
4 结论
本研究在睡莲中共获得46个NcbZIP成员,可以将46个NcbZIP成员进一步分为10个亚家族,相同亚家族成员具有保守的基因结构和保守的基序分布;在睡莲中,4号、7号、12号和14号染色体上没有任何睡莲bZIP成员的存在,NcbZIP成员在其他10条染色体上存在不均匀分布;片段复制事件对睡莲bZIP家族的扩增起主要作用;睡莲bZIP成员启动子区中含有最多的茉莉酸甲酯和ABA响应元件;组织特异性表达分析发现各个成员在不同的组织中表达不同,可以作为睡莲育种中一个具有潜在应用价值的重要候选基因。
[1] ZHANG L S, CHEN F, ZHANG X T, LI Z, ZHAO Y Y, LOHAUS R, CHANG X J, DONG W, HO S Y W, LIU X, SONG A X, CHEN J H, GUO W L, WANG Z J, ZHUANG Y Y, WANG H F, CHEN X Q, HU J A, LIU Y H, QIN Y. The water lily genome and the early evolution of flowering plants. Nature, 2020, 577(7788): 79-84.
[2] DRöGE-LASER W, SNOEK B L, SNEL B, WEISTE C. ThebZIP transcription factor family-an update. Current Opinion in Plant Biology, 2018, 45(Pt A): 36-49.
[3] 刘慧洁, 徐恒, 邱文怡, 李晓芳, 张华, 朱英, 李春寿, 王良超. bZIP转录因子在植物生长发育及非生物逆境响应的作用. 浙江农业学报, 2019, 31(7): 1205-1214.
LIU H J, XU H, QIU W Y, LI X F, ZHANG H, ZHU Y, LI C S, WANG L C. Roles of bZIP transcription factors in plant growth and development and abiotic stress response. Acta Agriculturae Zhejiangensis, 2019, 31(7): 1205-1214. (in Chinese)
[4] JAKOBY M, WEISSHAAR B, DRöGE-LASER W, VICENTE- CARBAJOSA J, TIEDEMANN J, KROJ T, PARCY F. bZIP transcription factors in. Trends in Plant Science, 2002, 7(3): 106-111.
[5] 王金英, 丁峰, 潘介春, 张树伟, 杨亚涵, 黄幸, 范志毅, 李琳, 王颖. 植物bZIP转录因子家族的研究进展. 热带农业科学, 2019, 39(6): 39-45.
WANG J Y, DING F, PAN J C, ZHANG S W, YANG Y H, HUANG X, FAN Z Y, LI L, WANG Y. Research progress of bZIP lineage transcription factors in plant. Chinese Journal of Tropical Agriculture, 2019, 39(6): 39-45. (in Chinese)
[6] SORNARAJ P, LUANG S, LOPATO S, HRMOVA M. Basic leucine zipper (bZIP) transcription factors involved in abiotic stresses: A molecular model of a wheat bZIP factor and implications of its structure in function. Biochimica et Biophysica Acta, 2016, 1860(1 Pt A): 46-56.
[7] 崔荣秀, 张议文, 陈晓倩, 谷彩红, 张荃. 植物bZIP参与胁迫应答调控的最新研究进展. 生物技术通报, 201, 35(2): 143-155.
CUI R X, ZHANG Y W, CHEN X Q, GU C H, ZHANG Q. The Latest Research Progress on the Stress Responses of bZIP Involved in Plants. Biotechnology Bulletin, 2019, 35(2): 143-155. (in Chinese)
[8] DAS P, LAKRA N, NUTAN K K, SINGLA-PAREEK S L, PAREEK A. A unique bZIP transcription factor imparting multiple stress tolerance in Rice. Rice (New York, NY), 2019, 12(1): 58.
[9] RONG S Y, WU Z Y, CHENG Z Z, ZHANG S, LIU H, HUANG Q M. Genome-wide identification, evolutionary patterns, and expression analysis of bZIP gene family in olive (L). Genes (Basel), 2020, 11(5): 510.
[10] WANG Z H, YAN L Y, WAN L Y, HUAI D X, KANG Y P, SHI L, JIANG H F, LEI Y, LIAO B S. Genome-wide systematic characterization of bZIP transcription factors and their expression profiles during seed development and in response to salt stress in peanut. BMC Genomics, 2019, 20(1): 51.
[11] LIU Y H, CHAI M N, ZHANG M, HE Q, SU Z X, PRIYADARSHANI S V G N, LIU L P, DONG G X, QIN Y A. Genome-wide analysis, characterization, and expression profile of the basic leucine zipper transcription factor family in pineapple. International Journal of Genomics, 2020, 2020: 3165958.
[12] AZEEM F, TAHIR H, IJAZ U, SHAHEEN T. A genome-wide comparative analysis of bZIP transcription factors inand(Diploid ancestors of present-day cotton). Physiology and Molecular Biology of Plants, 2020, 26(3): 433-444.
[13] WANG W W, WANG Y F, ZHANG S M, XIE K L, ZHANG C, XI Y J, SUN F L. Genome-wide analysis of the abiotic stress-related bZIP family in switchgrass. Molecular Biology Reports, 2020, 47(6): 4439-4454.
[14] FAN K, WANG M, MIAO Y, NI M, BIBI N, YUAN S N, LI F, WANG X D. Molecular evolution and expansion analysis of the NAC transcription factor in. PLoS One, 2014, 9(11): e111837.
[15] PERTEA M, KIM D, PERTEA G M, LEEK J T, SALZBERG S L. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown. Nature Protocols, 2016, 11(9): 1650-1667.
[16] CHAI W B, SI W N, JI W, QIN Q Q, ZHAO M L, JIANG H Y. Genome-wide investigation and expression profiling of HD-zip transcription factors in foxtail millet (L.). BioMed Research International, 2018, 2018: 8457614.
[17] YANG Y, YU T F, MA J, CHEN J, ZHOU Y B, CHEN M, MA Y Z, WEI W L, XU Z S. The soybean bZIP transcription factor gene GmbZIP2 confers drought and salt resistances in transgenic plants. International Journal of Molecular Sciences, 2020, 21(2): 670.
[18] 朱芸晔, 薛冰, 王安全, 王文杰, 周昂, 黄胜雄, 刘永胜. 番茄bZIP转录因子家族的生物信息学分析. 应用与环境生物学报, 2014, 20(5): 767-774.
ZHU Y Y, XUE B, WANG A Q, WANG W J, ZHOU A, HUANG S X, LIU Y S. Comprehensive bioinformatic analysis of bZIP transcription factors in. Chinese Journal of Applied & Environmental Biology, 2014, 20(5): 767-774. (in Chinese)
[19] 王升级, 孙赫, 党慧. 盐胁迫条件下杨树bZIP转录因子全基因组分析. 山西农业大学学报(自然科学版), 2018, 38(8): 1-7, 14.
WANG S J, SUN H, DANG H. Genome-wide analysis of the bZIP transcription factors inin response to salt stress. Journal of Shanxi Agricultural University (Natural Science Edition), 2018, 38(8): 1-7, 14. (in Chinese)
[20] 高斌, 陈娟娟, 崔顺立, 侯名语, 穆国俊, 陈焕英, 杨鑫雷, 刘立峰. 花生bZIP基因家族全基因组鉴定及抗旱表达分析. 植物遗传资源学报, 2020, 21(1): 174-191.
GAO B, CHEN J J, CUI S L, HOU M Y, MU G J, CHEN H Y, YANG X L, LIU L F. Genome-wide identification and expression analysis of bZIP gene family under drought stress in peanut. Journal of Plant Genetic Resources, 2020, 21(1): 174-191. (in Chinese)
[21] BAILLO E H, KIMOTHO R N, ZHANG Z B, XU P. Transcription factors associated with abiotic and biotic stress tolerance and their potential for crops improvement. Genes (Basel), 2019, 10: 771.
[22] E Z G, ZHANG Y P, ZHOU J H, WANG L. Mini review roles of the bZIP gene family in rice. Genetics and Molecular Research, 2014, 13(2): 3025-3036.
[23] PAN F, WU M, HU W F, LIU R, YAN H W, XIANG Y. Genome-Wide Identification and Expression Analyses of the bZIP Transcription Factor Genes in moso bamboo). International Journal of Molecular Sciences, 2019, 20(9): 2203.
[24] ZHANG M, LIU Y H, SHI H, GUO M L, CHAI M N, HE Q, YAN M K, CAO D, ZHAO L H, CAI H Y, QIN Y A. Evolutionary and expression analyses of soybean basic Leucine zipper transcription factor family. BMC Genomics, 2018, 19(1): 159.
[25] YU J, HU S N, WANG J, KA-SHU G, LI S G, LIU B, DENG Y J, DAI L, ZHOU Y, ZHANG X Q, CAO M L, LIU J, SUN J D, TANG J B, CHEN Y J, HUANG X B, LIN W, YE C, TONG W, CONG L J,. A Draft Sequence of the Rice Genome (L. ssp.). Science, 2002, 296(5565): 79-92.
[26] SCHMUTZ J, CANNON S B, SCHLUETER J, MA J X, MITROS T, NELSON W, HYTEN D L, SONG Q J, THELEN J J, CHENG J L, XU D, HELLSTEN U, MAY G D, YU Y, SAKURAI T, UMEZAWA T, BHATTACHARYYA M K, SANDHU D, VALLIYODAN B, LINDQUIST E,. Genome sequence of the palaeopolyploid soybean. Nature, 2010, 463(7278): 178-183.
[27] WEI K F, CHEN J, WANG Y M, CHEN Y H, CHEN S X, LIN Y N, PAN S, ZHONG X J, XIE D X. Genome-wide analysis of bZIP-encoding genes in maize. DNA Research, 2012, 19(6): 463-476.
[28] LIU M Y, WEN Y D, SUN W J, MA Z T, HUANG L, WU Q, TANG Z Z, BU T L, LI C L, CHEN H. Genome-wide identification, phylogeny, evolutionary expansion and expression analyses of bZIP transcription factor family in tartaty buckwheat. BMC Genomics, 2019, 20(1): 483.
[29] LI D Y, FU F Y, ZHANG H J, SONG F M. Genome-wide systematic characterization of the bZIP transcriptional factor family in tomato (L.). BMC Genomics, 2015, 16: 771.
[30] ZHAO P, YE M H, WANG R Q, WANG D D, CHEN Q. Systematic identification and functional analysis of potato (L) bZIP transcription factors and overexpression of potato bZIP transcription factor StbZIP-65 enhances salt tolerance. International Journal of Biological Macromolecules, 2020, 161: 155-167.
[31] FAN K, CHEN Y R, MAO Z J, FANG Y, LI Z W, LIN W W, ZHANG Y Q, LIU J P, HUANG J W, LIN W X. Pervasive duplication, biased molecular evolution and comprehensive functional analysis of the PP2C family in. BMC Genomics, 2020, 21(1): 465.
[32] FAN K, MAO Z J, ZHENG J X, CHEN Y R, LI Z W, LIN W W, ZHANG Y Q, HUANG J W, LIN W X. Molecular evolution and expansion of the KUP family in the allopolyploid cotton speciesand. Frontiers in Plant Science, 2020, 11: 545042.
[33] LIANG C Z, MENG Z H, MENG Z G, MALIK W, YAN R, LWIN K M, LIN F Z, WANG Y A, SUN G Q, ZHOU T, ZHU T, LI J Y, JIN S X, GUO S D, ZHANG R. GhABF2, a bZIP transcription factor, confers drought and salinity tolerance in cotton (L.). Scientific Reports, 2016, 6: 35040.
[34] LIM C W, BAEK W, JUNG J, KIM J H, LEE S C. Function of ABA in stomatal defense against biotic and drought stresses. International Journal of Molecular Sciences, 2015, 16(7): 15251-15270.
[35] NAKASHIMA K, YAMAGUCHI-SHINOZAKI K. ABA signaling in stress-response and seed development. Plant Cell Reports, 2013, 32(7): 959-970.
[36] 郭贵华, 刘海艳, 李刚华, 刘明, 李岩, 王绍华, 刘正辉, 唐设, 丁艳锋. ABA缓解水稻孕穗期干旱胁迫生理特性的分析. 中国农业科学, 2014, 47(22): 4380-4391.
GUO G H, LIU H Y, LI G H, LIU M, LI Y, WANG S H, LIU Z H, TANG S, DING Y F. Analysis of physiological characteristics about ABA alleviating rice booting stage drought stress. Scientia Agricultura Sinica, 2014, 47(22): 4380-4391. (in Chinese)
[37] 山雨思, 代欢欢, 何潇, 辛正琦, 吴能表. 外源茉莉酸甲酯和水杨酸对盐胁迫下颠茄生理特性和次生代谢的影响. 植物生理学报, 2019, 55(9): 1335-1346.
SHAN Y S, DAI H H, HE X, XIN Z Q, WU N B. Effects of exogenous methyl jasmonate and salicylic acid on physiological characteristics and secondary metabolism ofunder NaCl stress. Plant Physiology Communications, 2019, 55(9): 1335-1346. (in Chinese)
[38] YU X X, ZHANG W J, ZHANG Y, ZHANG X J, LANG D Y, ZHANG X H. The roles of methyl jasmonate to stress in plants. Functional Plant Biology, 2019, 46(3): 197-212.
[39] HO T T, MURTHY H N, PARK S Y. Methyl jasmonate induced oxidative stress and accumulation of secondary metabolites in plant cell and organ cultures. International Journal of Molecular Sciences, 2020, 21(3): 716.
[40] SCHLÖGL P S, NOGUEIRA F T S, DRUMMOND R, FELIX J M, DE ROSA V E, VICENTINI R, LEITE A, ULIAN E C, MENOSSI M. Identification of new ABA- and MEJA-activated sugarcane bZIP genes by data mining in the SUCEST database. Plant Cell Reports, 2008, 27(2): 335-345.
[41] YANG Z M, SUN J, CHEN Y, ZHU P P, ZHANG L, WU S Y, MA D F, CAO Q H, LI Z Y, XU T. Genome-wide identification, structural and gene expression analysis of the bZIP transcription factor family in sweet potato wild relative. BMC Genetics, 2019, 20(1): 41.
[42] MURMU J, BUSH M J, DELONG C, LI S T, XU M L, KHAN M, MALCOLMSON C, FOBERT P R, ZACHGO S, HEPWORTH S R.basic leucine-zipper transcription factors TGA9 and TGA10 interact with floral glutaredoxins ROXY1 and ROXY2 and are redundantly required for anther development. Plant Physiology, 2010, 154(3): 1492-1504.
[43] XU D B, CHEN M, MA Y N, XU Z S, LI L C, CHEN Y F, MA Y Z. A G-protein β subunit, AGB1, negatively regulates the ABA response and drought tolerance by down-regulating AtMPK6-related pathway in. PLoS One, 2015, 10(1): e0116385.
[44] LIU D F, SHI S P, HAO Z J, XIONG W T, LUO M Z. A homologue ofVIP1, may positively regulate JA levels by directly targetting the genes in JA signaling and metabolism pathway in rice. International Journal of Molecular Sciences, 2019, 20(9): 2360.
Molecular Evolution and Function Analysis of bZIP Family in
YE FangTing, PAN XinFeng, MAO ZhiJun, LI ZhaoWei, FAN Kai
College of Agriculture, FuJian Agriculture and Forestry University/Key Laboratory of Ministry of Education for Genetics, Breeding and Multiple Utilization of Crops, Fuzhou 350002
【】The genome-wide analysis of the bZIP family inwas used to identify the bZIP (basic leucine zipper) family members in waterlily, and then which were further analyzed. This study revealed molecular evolution and function of the bZIP family in waterlily.【】The genome sequence ofwas downloaded from Waterlily Pond database. The bZIP members in waterlily were identified by using HMMER 3.0 program, and the conserved bZIP domain was verified by using CDD program. The phylogenetic tree was constructed by the IQ-tree software. The ExPASy and SOPMA online website were performed to analyze protein structure characters. The conserved motifs were identified by using MEME program. The gene duplication events were found and visualized by the MCScan and Circos software. Transcriptome data of NcbZIP members were obtained from the NCBI website (SRA Study: SRP222853). The Pearson Correlation Coefficient (PCC) about the expression levels of the NcbZIP family members was calculated by using R software, and the network of the expression levels in the NcbZIP family was analyzed by using Cytoscape software.】46 bZIP members were identified in. colorata, and were named fromtoaccording to their chromosome distributions. The A subfamily contained the most NcbZIP members (11 NcbZIPs). There were 10 subfamilies (A, B, C, D, E, G, H, I, J and S) according to the phylogenetic analysis. The protein length in the NcbZIP family was from 101 aa to 1 898 aa, and the molecular weight ranged from 12.04 kD to 214.64 kD. The NcbZIP members from same subfamily had the similar distributions of the conserved motifs and gene structures. Waterlily had 14 chromosomes, and 46 NcbZIP members were unevenly distributed across 10 chromosomes. Chromosome 1 had the highest number of NcbZIP members. There were 10 gene duplication events in the NcbZIP family, including nine segmental duplication events and one tandem duplication event. The A subfamily had the most number of the gene duplication events (three). Based on the expression patterns in different tissues, the NcbZIP family could be divided into three groups (I, II and III). Themembers in Group I were highly expressed in all tissues, while themembers in group II were not expressed in almost all tissues. Themembers in group III had tissue-specific expression profiles, and most of NcbZIP members in C, D and E subfamilies belonged to group III. The PPC analysis about the expression levels ofmembers indicatedhad the highest connection with other members.【】46 NcbZIP members were identified in, and were unevenly distributed in 14 chromosomes. The NcbZIP family could be divided into 10 subfamilies with conserved motifs distributions and diverse expression levels. The current study could lay the foundation on the functional analysis of the bZIP family in.
; bZIP family; molecular evolution; expression profile; function analysis

10.3864/j.issn.0578-1752.2021.21.018
2021-01-18;
2021-06-28
国家自然科学基金(31701470)、中国博士后基金(2017M610388,2018T110637)、福建农林大学杰出青年科研人才计划(xjq201917)、福建农林大学科技创新专项基金(CXZX2020007A)、福建省自然科学基金(2021J01073)
叶方婷,E-mail:fangtingye@163.com。通信作者范凯,E-mail:fankai@fafu.edu.cn
(责任编辑 赵伶俐)

