二种土甲族幼虫的分子鉴定
张建英,李 彪,贾 龙,丁荣荣
(1.宁夏大学 生命科学学院,宁夏 银川 750021;2.宁夏大学 农学院,宁夏 银川 750021)
土甲族(Opatrini)隶属鞘翅目(Coleoptera)拟步甲科(Tenebrionidae)拟步甲亚科(Tenebrioninae)[1],在宁夏分布有10属35种[2]。土甲族昆虫是我国荒漠半荒漠地区主要的农、林、牧业害虫和仓储害虫[3]。四五月份成虫交配产卵,孵化成幼虫[4],幼虫一般栖息在土壤之中,口器较发达,食性杂[2],为害植物嫩芽、根部和种子,损害植物生长,甚至致其死亡。土甲族昆虫的幼虫期是取食量最大、对农林牧业为害最严重的发育阶段。因此,高效、准确地对该类昆虫幼虫进行种的鉴定已成为该类昆虫防治与利用的基础,也是该类群昆虫防治与利用研究的核心内容之一。虽然目前对于土甲族昆虫的研究很多,在成虫分类研究方面较为完善,但对于土甲族昆虫幼期种类鉴定的研究却鲜有报道,其主要原因是该族昆虫幼期标本不易获得,并且属间或种间差异小,给种类鉴定造成了很大困难。
目前,国际上对昆虫的鉴定大多采用形态学方法,形态学鉴定是将成虫的形态学特征作为依据进行鉴定,而幼期的特征差异小,仅从形态特征入手,鉴定困难,必须等到幼虫老熟时,即形态特征骨化稳定后才能鉴定。因此幼虫鉴定一直是分类学的一个难题,采取的办法往往是将幼虫在实验室内进行人工饲养,待其发育成成虫后再进行鉴定,费时费力,严重影响物种鉴定的速度。20世纪80年代以来,随着分子生物学技术和生物信息学的发展以及PCR技术的建立与完善,一系列分子生物学技术被广泛应用于生物鉴定,极大地弥补了传统形态学鉴定方法存在的缺陷,DNA条形码技术就是其中具有代表性的一种[5]。加拿大动物学家Hebert等[6]首先倡导将条形编码技术应用于生物物种鉴定。DNA在生物体生长发育过程中不会改变,以DNA序列作为检测对象,不受个体发育过程、不同发育形态和生长环境等的影响,且DNA条形码技术具有可操作性强等特点,目前已广泛应用于物种鉴定[7]。
土甲族昆虫生活史普遍在1年左右。野外获得低龄幼虫后,因其鉴别特征尚未硬化定型,专业人员很难做出准确鉴定,往往通过实验室饲养,待其发育成老熟幼虫或成虫后再进行鉴定,而这个过程短则数月,长则1年,鉴定周期很长。为此,本研究利用DNA条形码技术以线粒体COⅠ基因、线粒体16S基因为分子标签,对土甲族伪坚土甲属粗背伪坚土甲(Scleropatrum horridum horridum)和笨土甲属阿笨土甲(Penthicus alashanica)成虫、低龄幼虫和高龄幼虫等8个样品的线粒体COⅠ基因、线粒体16S基因序列进行比对分析,若同种土甲成虫(雌性和雄性)和幼虫(低龄和高龄)之间的16S和COⅠ基因序列相似度达到98%及以上,说明可以通过DNA条形码技术扩增未知幼虫的16S和COⅠ基因序列,然后与已知成虫的16S和COⅠ基因序列进行比对,根据相似度来判断未知幼虫为哪种已知成虫的幼虫,从而鉴定未知幼虫种类。Hajibabaei等[8]对鳞翅目隶属3科521种的DNA条形码进行分析鉴定,证明DNA条形码的准确性极高;Virgilio等[9]对鞘翅目、双翅目、半翅目、膜翅目、鳞翅目和直翅目进行了DNA条形码技术鉴定的效率分析,认为在无数据库参照时其物种鉴定的效率会受影响;岳巧云等[10]应用COⅠ基因鉴定形态近似的鞘翅目幼虫,结果准确、可靠。该研究的意义在于:昆虫幼虫分类研究中采用DNA条形码技术,相比传统的实验室内饲养幼虫待其鉴别特征骨化后再进行鉴定的传统幼虫鉴定方法,可以有效减少鉴定时间和成本,鉴定结果也比较准确、客观、简便。采用线粒体16S基因和线粒体COⅠ基因同时作为DNA条形编码的标记基因,突破了单分子标记的局限性。
1 材料与方法
1.1 实验材料
1.1.1 供试虫源 本研究用于检测的粗背伪坚土甲和阿笨土甲成虫采集于宁夏贺兰山国家级自然保护区;幼虫经人工室内饲养获得。
1.1.2 试剂 Taq Plus DNA聚合酶、10×PCR Buffer(含Mg2+)、dNTP(10 mmol/L)、灭菌去离子水、引物DNA、6×DNA Loading Dye、DNA Ladder Mix(100~10 000 bp)、DNA Ladder Mix (100~3 000 bp)、50×TAE、琼脂糖H、SanPrep柱式DNA胶回收试剂盒、无水乙醇、异丙醇、1×TE、BigDye Terminator v1.1、POP-7TMPolymer、HiDi Formamide、EDTA、NaAc、NaOH、Ezup柱式动物组织基因组DNA抽提试剂盒
1.1.3 仪器 冰箱、洁净工作台、PCR仪、凝胶成像仪、测序仪、冷冻离心机、台式高速离心机、电泳仪、电泳槽、微型旋涡混合仪、数显恒温水浴锅、移液器、紫外可见分光光度计等。
1.2 实验方法
1.2.1 室内人工饲养方法 室内饲养成虫时,用较大的容器盛沙土,将雌雄虫按一定比例放置其中进行饲养。沙土必须进行100~120℃高温处理24 h,目的是杀死沙土中的螨类。沙土用水拌湿,土壤含水量在14%~18%为宜。将拌好的沙土装入容器,上面留出1/3体积的空余,在湿土上覆盖薄薄一层干沙土。以燕麦片、黄瓜、麸皮、苜蓿等作为主要饲料。当卵达到一定数量时,用软毛笔将卵转移到孵卵容器中,孵化卵的沙土湿度不能过大。当卵孵化成幼虫后,将其轻轻转移到新容器中饲养。饲养幼期土甲较饲养成虫困难,幼虫和蛹对沙土水分有一定要求,容器内沙土水分不宜过大,否则会使幼虫和蛹发黑腐烂;但也不能没有水分,当沙土水分减少后,可通过吸水纸向其中滴水以保持一定湿度。预蛹期时老熟幼虫会在容器底部打造蛹室,在此期间不能摇动容器。饲养过程中若发现寄生螨需及时处理,可在漏斗中用干燥的沙土来回冲刷虫体,从而去除体表的寄生螨;寄生菌需隔离处理。除预蛹期和蛹期外,需定期更换沙土从而达到保湿、除菌的目的。用透明容器饲养需注意避光。
1.2.2 DNA提取 采用Ezup柱式动物组织基因组DNA抽提试剂盒分别提取粗背伪坚土甲雌性成虫、雄性成虫、低龄幼虫、高龄幼虫和阿笨土甲雌性成虫、雄性成虫、低龄幼虫和高龄幼虫8个样品的DNA。
1.2.3 PCR扩增条件及产物检测 选用线粒体COⅠ基因、线粒体16S基因的部分片段作为分子标记,委托生工生物工程(上海)股份有限公司设计合成引物进行PCR扩增(表2)。

表2 引物序列
PCR反应体系包括模板DNA1μL、上下游引物(10μmol/L)各1μL、Dntp(mix)(10 mmol/L)1μL、Taq Buffer(10×)2.5μL、Taq酶(5 U/μL)0.2μL、dd H2O加至25μL。扩增条件:第1步95℃预变性5 min;第2步94℃变性30 s,63℃(每循环降0.5℃)退火30 s,72℃延伸30 s共10个循环;第3步95℃变性30 s,58℃退火30 s,72℃延伸30 s共30个循环;第4步72℃修复延伸10 min;第5步4℃保温。
产物检测:取5μL PCR扩增产物,以标准DNA Marker为参照,在1%琼脂糖凝胶上进行电泳分离,150 V、100 mA条件下电泳10~20 min,在紫外检测仪上观察拍照。按纯化试剂盒操作说明将目的片段电泳条带单一且明亮的PCR产物二次纯化,用测序仪对PCR产物进行双向测序。
1.2.4 序列分析 本研究测定了土甲族2种昆虫的16S基因和COⅠ基因部分序列,用Chromas软件观察每对序列峰值图,结合Contig Express软件进行人工校阅得到所测序列。用分子进化遗传分析软件MEGA 7分别统计2个基因的序列组成,计算碱基组成,统计转换(transition,TS)和颠换(transversion,TV)数,计算转化/颠换比率,分析2种昆虫间的DNA序列差异。将测得的DNA序列输入NCBI(https://www.ncbi.nlm.nih.gov/)网站,进行Blast核苷酸序列的同源性分析,用Clustal X 1.83软件对2种基因14条序列进行多重比对。
2 结果与分析
2.1 DNA提取
通过分光光度计对测得的各样品DNA质量进行检测,得到基因组DNA溶液OD值为1.7~2.0。DNA提取检测结果显示除6号样外,其他7个样品均得到清晰明亮且单一的目的条带,满足后续PCR扩增要求。其中6号样没有得到目的条带,结果见图1,这可能因为在样品处理过程中6号样基因组DNA发生降解,从而导致未能成功提取出6号样基因组DNA。
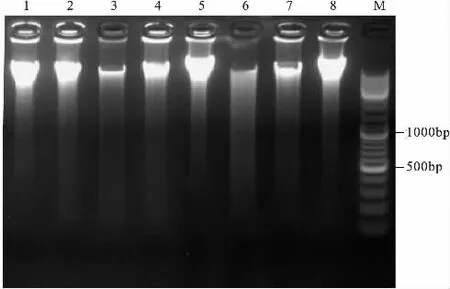
图1 DNA提取检测图
2.2 PCR扩增
对除6号样外的7个样品进行16S和COⅠ基因PCR扩增,得到的电泳图如图2、图3所示。对于16S基因片段,7个样品均得到清晰明亮的目的条带;对于COⅠ基因片段,7个样品亦均得到清晰明亮的目的条带,2个基因片段结果均为单一条带,可用于后续的基因测序分析。本实验采用特异性引物进行PCR扩增,扩增结果表明:PCR扩增产生大小相同的1条带,无其他条带干扰(图2、图3),因此所得到的序列应该是线粒体序列,而非核中线粒体假基因。

图2 16S基因PCR产物电泳图
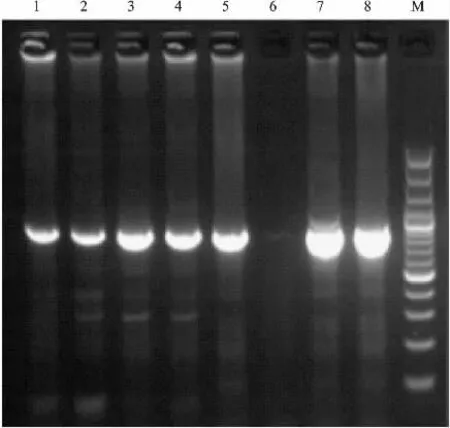
图3 COⅠ基因PCR产物电泳图
2.3 基因序列组成及变异
2.3.1 16S基因序列组成及变异 在2种土甲的16S基因序列中,有62个保守位点,935个变异位点,变异率为93.4%,645个简约信息位点,285个自裔位点,结果表明种间序列变异较大。碱基T、C、A、G的平均含量分别为32.0%、15.3%、45.1%和7.6%。A+T的平均含量为77.1%,明显高于G+C含量(22.9%)。碱基T、C、A、G在每个氨基酸密码子的不同位点差异不大,第1位点A+T含量(78.0%)高于G+C含量(22.0%),第2位点A+T含量(76.7%)高于G+C含量(23.3%),第3位点A+T含量(76.4%)高于G+C含量(23.6%),16S基因在各类昆虫中富含A、T,如鳞翅目A+T的含量为78.4%,蜉蝣目A+T的含量为66.3%[11],本实验结果与之相似,另外所有拟步甲科昆虫的序列总和中A、T碱基所占总碱基的百分比均高于G、C碱基所占总碱基的百分比,由此可知所有序列都表现出明显的AT偏向性(表3),这表明所有序列都符合昆虫线粒体的碱基组成结构。碱基替换是DNA序列碱基进化的重要衡量指标,有碱基转换和碱基替换两种形式[12]。从碱基替换的结果看,TV的发生远远多于TS,全组数据TS/TV的平均值为0.3,在第1、2、3位点的比值分别为0.3、0.4、0.4。其中转换主要发生在C←→T之间,颠换主要发生在A←→T和A←→C之间,其他类型的替换相比上述几种替换发生较少(表4)。
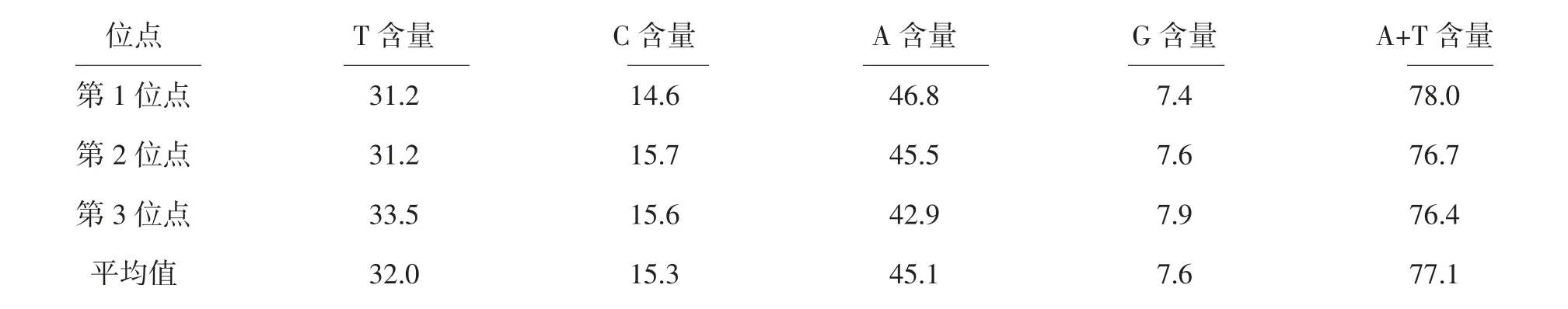
表3 土甲族2种昆虫的16S基因序列组成 %
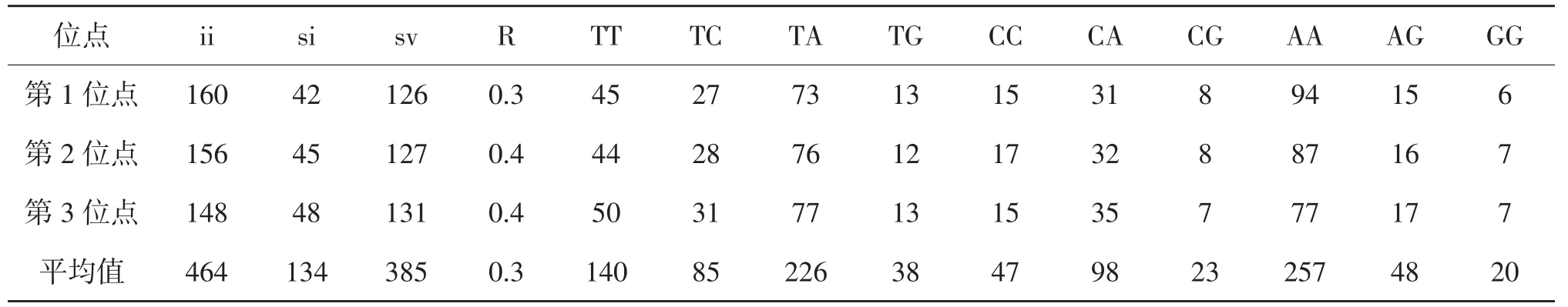
表4 土甲族2种昆虫的16S基因碱基替换统计
2.3.2 COⅠ基因序列组成及变异 在2种土甲的COⅠ基因序列中,有15个保守位点,1 021个变异位点,变异率为98.2%,698个简约信息位点,321个自裔位点,结果表明种间序列变异较大。碱基T、C、A、G的平均含量分别为35.0%、17.5%、30.8%和16.7%。A+T的平均含量为65.8%,明显高于G+C含量(34.2%)。第1位点的A+T含量为65.3%,第2位点的A+T含量为62.4%,第3位点的A+T含量为69.7%。第3位点表现出比较强的AT含量偏向性,A+T含量较前两个位点高,该位点碱基G的含量最少为12.8%。所有拟步甲科昆虫的序列总和中A、T碱基所占总碱基的百分比为63.1%[13],与本实验结果相符,都表现出明显的AT偏向性(表5)。这表明所有序列符合昆虫线粒体碱基组成结构。从碱基替换的结果看,TV的发生略多于TS,TS/TV的平均值为0.5,在第1、2、3位点的比值分别为0.5、0.5、0.5。其中转换主要发生在C←→T之间,颠换主要发生在A←→T之间,其他类型的替换相比上述几种替换发生较少(表6)。
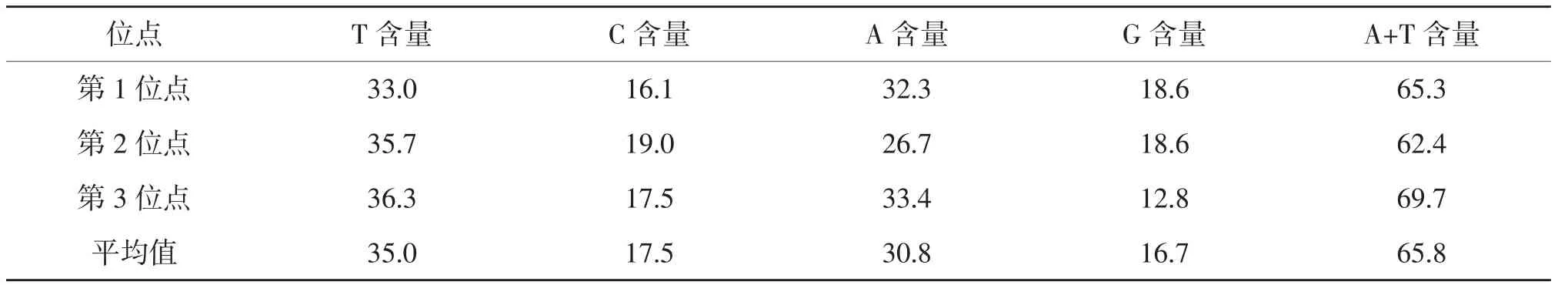
表5 土甲族2种昆虫的COⅠ基因序列组成 %
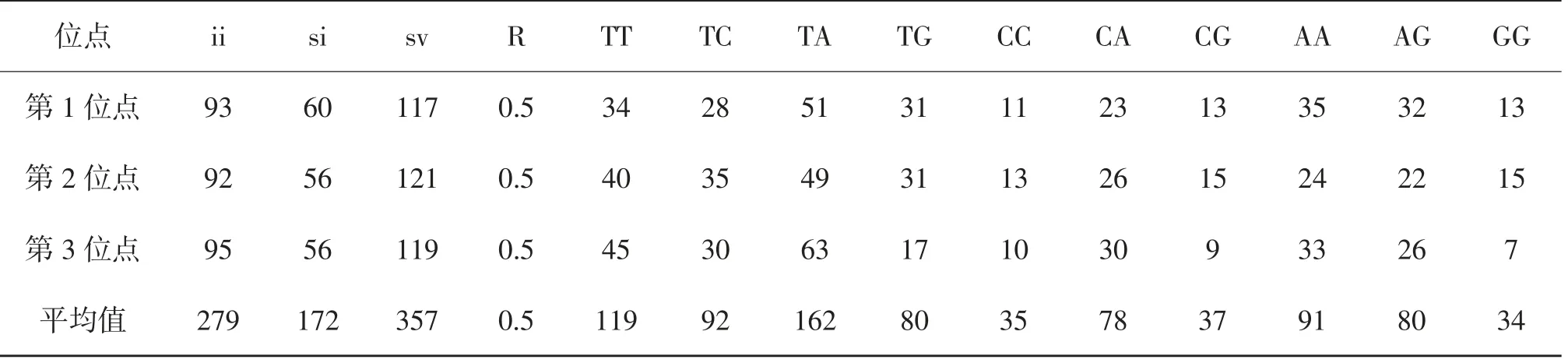
表6 土甲族2种昆虫的COⅠ基因碱基替换统计
2.4 同源性相似度比对分析
2.4.1 16S基因同源相似度 由表7和表8可知,粗背伪坚土甲雄性成虫与低龄幼虫及高龄幼虫的相似度达到100%,与雌性成虫相似度达100%,雌性成虫与低龄幼虫及高龄幼虫相似度达到100%,低龄幼虫与高龄幼虫相似度也达到100%。阿笨土甲雌性成虫与低龄幼虫相似度达99.11%,与高龄幼虫相似度达98.97%,低龄幼虫与高龄幼虫相似度达99.56%。2种土甲种间16S基因相似度:成虫之间最高为93.72%,幼虫之间最高为93.33%,成虫与幼虫之间最高为93.55%。2种土甲种内雌性成虫的16S基因与低龄幼虫和高龄幼虫的同源相似度均达到98%以上,粗背伪坚土甲雄性成虫的16S基因与低龄幼虫和高龄幼虫的同源相似度均达到98%以上。说明16S基因在不同种的各个虫态之间差异较大,在同一种内不论是雌性还是雄性成虫,它们与不同龄期幼虫都有极高的16S基因同源相似度。
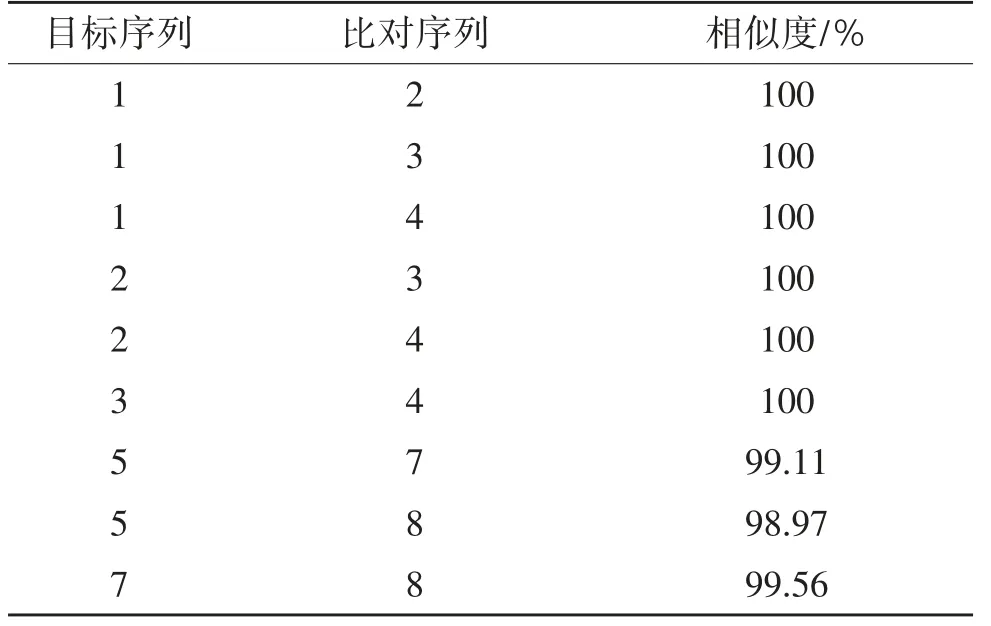
表7 种内16S基因BLAST比对结果
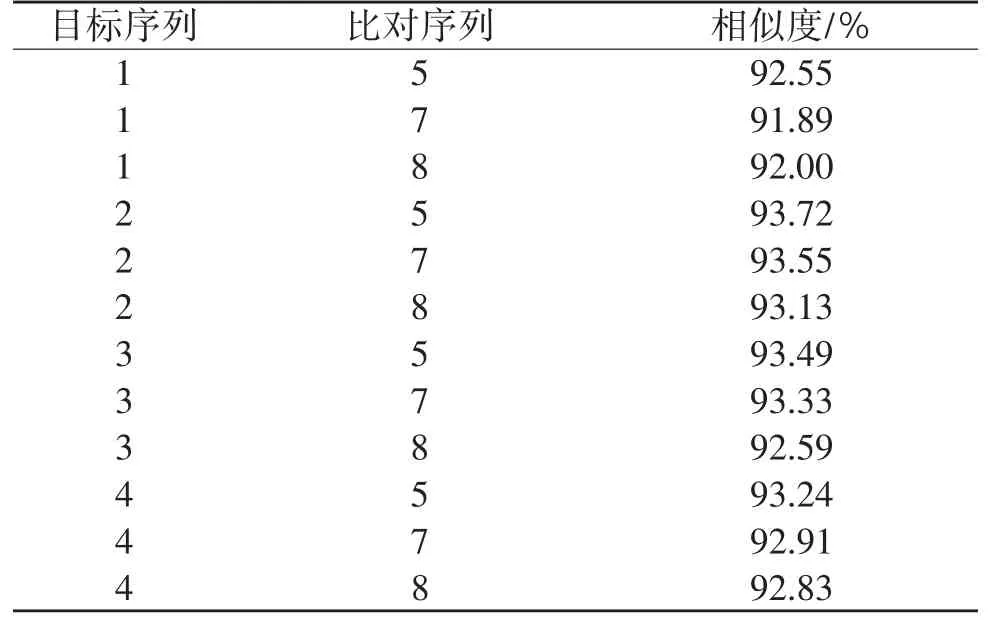
表8 种间16S基因BLAST比对结果
2.4.2 CO I基因同源相似度 由表9和表10可知,粗背伪坚土甲雌性成虫与低龄幼虫相似度达98.84%,与高龄幼虫相似度达99.50%,与雄性成虫相似度达98.49%;雄性成虫与低龄幼虫相似度达98.94%,与高龄幼虫相似度达99.39%,低龄幼虫与高龄幼虫相似度达99.24%。阿笨土甲雌性成虫与低龄幼虫相似度达98.63%,与高龄幼虫相似度达99.24%,低龄幼虫与高龄幼虫相似度达98.79%。2种土甲种间COⅠ基因相似度:成虫之间最高为91.57%,幼虫之间最高为90.53%,成虫与幼虫之间最高为91.75%。2种土甲雌性成虫的COⅠ基因与低龄幼虫和高龄幼虫的同源相似度均达到98%以上,粗背伪坚土甲雄性成虫的COⅠ基因与低龄幼虫和高龄幼虫的同源相似度均达到98%以上。说明COⅠ基因在不同种的各个虫态间差异较大,在同一种内不论是雌性还是雄性成虫,它们与不同龄期幼虫都有极高的COⅠ基因同源相似度。
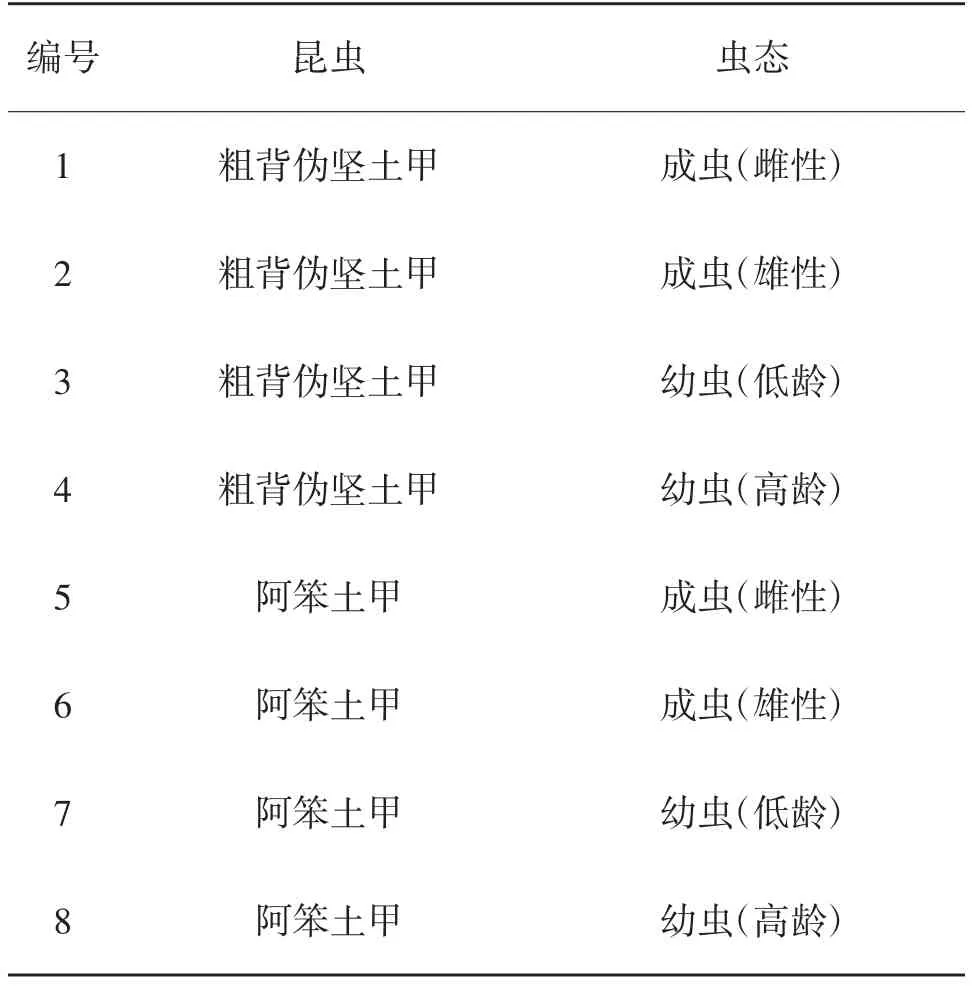
表1 样品编号及昆虫、虫态对应关系
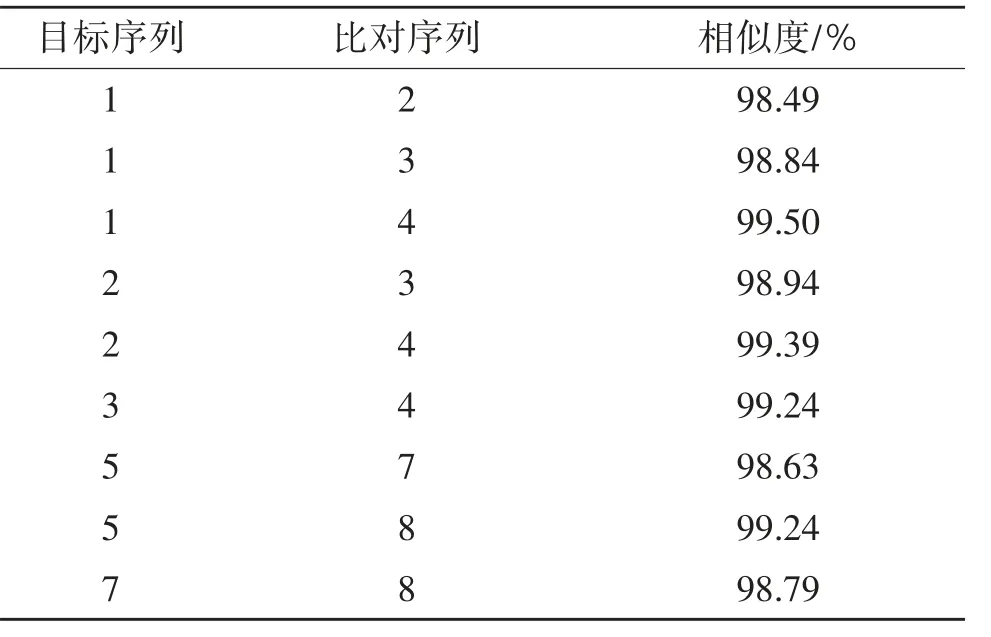
表9 种内COⅠ基因BLAST比对结果
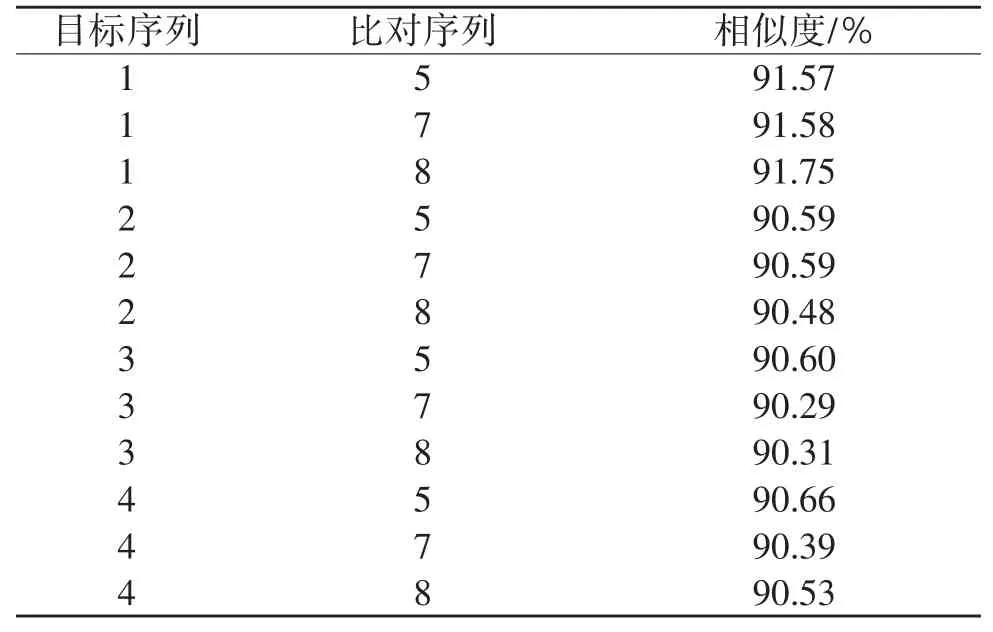
表10 种间COⅠ基因BLAST比对结果
3 结论与讨论
目前DNA条形码技术主要被用于鉴定近缘种或者建立数据库分析某个大的类群[14-15]。在昆虫研究工作中,非昆虫分类学专家很难通过形态特征对一些昆虫进行鉴定,而分子鉴定可以有效解决这一问题,DNA条形码技术就是其中的代表之一,对昆虫特定基因片段进行测序及比对,确定其分类学地位。
运用DNA条形码技术鉴定物种有一个关键点就是基因的选择,基因的选择直接关系到鉴定结果的准确性。研究表明,许多生物包括昆虫类群存在核中线粒体基因的拷贝,即线粒体假基因Numts(nuclear mitochondria DNA segments)[16]。随着线粒体假基因研究的推进,人们开始怀疑线粒体基因作为系统发育研究分子标记的准确性。因为mtDNA与线粒体假基因同源,在核基因组中进化速率很低,所以在用通用引物进行扩增时很容易被扩增出来[17],如果线粒体假基因在进行系统发育研究分析时被当作线粒体基因序列来进行分析,分析结果可能会变得不准确或者错误[18-19]。COⅠ基因因其突变速度快、容易扩增、母性遗传等特点成为分子系统发育研究中应用较为广泛的分子标记。本研究采用COⅠ基因和16S基因作为分子标记,对2种土甲进行分子分类鉴定研究。通过对2种土甲不同虫态的7条16S基因序列与7条COⅠ基因序列进行分析,根据碱基组成分析结果,2种土甲的碱基序列均表现出一定的AT偏向性,其中16S基因序列中A+T的平均含量为77.1%,G+C的平均含量为22.9%,这与刘晓丽[11]报道的拟步甲线粒体16S基因序列碱基构成的特点相符;COⅠ基因序列中A+T的平均含量为65.9%,G+C的平均含量为34.1%,这与赵亚茹[13]报道的拟步甲科昆虫COⅠ基因的A+T含量相符。这表明本研究测得的基因序列是正确的。从2种土甲不同基因序列的同源相似度分析结果来看,粗背伪坚土甲种内16S基因序列同源相似度最高达100%,阿笨土甲种内16S基因序列同源相似度最高达99.56%,粗背伪坚土甲与阿笨土甲种间16S基因序列同源相似度最高为93.72%;粗背伪坚土甲种内COⅠ基因序列同源相似度最高达99.50%,阿笨土甲种内COⅠ基因序列同源相似度最高达99.24%,粗背伪坚土甲与阿笨土甲种间COⅠ基因序列同源相似度最高为91.75%。以上结果表明,不同物种间没有完全一致的序列,同一物种不同虫态不同龄期的个体间DNA序列差异很小。2种土甲种内不同个体间16S基因序列最大差异为1.03%,小于种类鉴定界限(2%);2种土甲种间16S基因序列最小差异为6.28%,高于限值(2%)。2种土甲种内不同个体间COⅠ基因序列最大差异为1.51%,小于种类鉴定界限(2%);2种土甲种间COⅠ基因序列最小差异为8.25%,高于限值(2%)。结果显示2种土甲种内个体间与种间存在明显的差异间隔,种内最大差异小于限值(2%),种间最小差异明显高于限值(2%),与研究报道的种间差异>2%的结果一致[20]。由序列同源相似度可知1、2、3、4号为同一种,5、7、8号为同一种,这与形态学鉴定结果一致,表明DNA条形码技术可以用来进行土甲族昆虫幼期鉴定研究。
本研究采用16S与COⅠ两种基因进行测定分析,突破单分子标记的局限性,当然本研究存在一定的不足之处,如研究标本采自同一地区,标本来源单一,若有不同采集地的标本进行对比研究鉴定,结果将更有说服力;本研究只是鉴定了幼虫期的低龄和高龄幼虫,对其他龄期的研究尚未进行。贡献在于:为幼虫种类鉴定提供新途径,采用基于COⅠ基因和16S基因的DNA条形码技术与常规形态学鉴定方法相结合的鉴定方式,可以更加准确快捷地进行种类鉴定研究。本实验获得的土甲族昆虫的COⅠ基因序列可以作为该虫的条形码,进行快捷准确的鉴定,从而为害虫及时有效防治提供科学依据。DNA条形码技术除了可以弥补传统形态学分类的诸多局限性外,还可以为分类学家发现新种提供一个新的方法[21],如果DNA条形码技术能够在土甲族昆虫鉴定上得到充分利用并与常规形态学相结合,会给土甲族昆虫的幼期鉴定带来新突破。
——以Z市S社区为例

