乳腺癌基于蛋白表达的风险预后模型构建和6种关键蛋白的鉴定
李 健 梁玉娜
泰安市中心医院乳腺疾病诊疗部,山东泰安 271000
乳腺癌现在已经成为女性最常见的癌症。根据Lancet最新的乳腺癌研究综述,乳腺癌的发病率占世界女性癌症的30%,死亡率与发病率的比值为15%[1]。根据以往的研究表明,病理分期对于乳腺癌患者的预后有很大的影响。乳腺癌是一种异质性很强的肿瘤,主要分为4种分子类型:lumial A型、lumial B型、Her-2阳性和三阴性亚型,对乳腺癌的复发和转移具有重要的预测价值[2],但对于同一分子类型以及病理分期的患者,有时预后仍有非常大的差异。
随着测序技术的发展,近年来越来越多的研究表明,虽然多种mRNA、miRNA、lncRNA、ceRNA等基因标志物对乳腺癌的预后有着良好的预测作用[3-5],然而,对于乳腺癌的诊断和治疗仍然缺乏特异的、敏感的生物标志物。蛋白表达在肿瘤发生的不同阶段发挥着关键作用,但目前还没有关于乳腺癌的蛋白预后模型研究。蛋白质水平的研究比RNA水平的研究更有利于临床应用,基于此,本研究拟从蛋白质水平来分析乳腺癌,并且从蛋白质角度来构建多种蛋白质联合预后模型,以期为乳腺癌的临床诊断、治疗及预后的准确性提供重要的参考资料。
1 材料和方法
1.1 数据收集
乳腺癌患者的蛋白质表达谱和匹配的临床信息从癌症蛋白质组图谱门户(TCPA)[6](https://www.tcpaportal.org/tcpa/,包括879个乳腺癌样本和224个蛋白质)和TCGA网站(https://portal.gdc.cancer.gov/,包括901个临床病例信息)分别下载。TCPA是一个综合资源,用于访问、可视化和分析患者肿瘤样本和癌细胞系的功能蛋白质组学。本研究中所有数据均为上述官网来源的最新数据。从网站下载Perl软件(https://www.perl.org/)[7]和R软 件(https://www.rproject.org/)[8]。R软件中的impute包用于填充缺失的数据。本研究符合TCPA的出版指南。
1.2 构建蛋白质预测模型并进行生存分析
用Perl软件来整合蛋白表达数据与患者生存数据,用R软件survival包进行单因素Cox分析,按照P<0.05标准筛选与乳腺癌预后相关的蛋白,再用R软件ggplot2和ggrepel包绘制火山图以可视化结果;然后,用lasso回归去除预后相关蛋白的多重共线性,以防止模型基因之间的过度拟合;最后,对预后相关蛋白进行多因素Cox分析,筛选出与乳腺癌有关的预后相关蛋白,构建预测模型并用风险评分显示。风险评分=(蛋白1的系数×蛋白1的表达)+(蛋白2的系数×蛋白2的表达)+…+(蛋白n的系数×蛋白n的表达)。根据风险值的中位值将患者分为高危组和低危组,并对风险评分的生存率进行分析,最后,绘制生存曲线来可视化结果。
1.3 预测模型中的蛋白评估
利用R软件survival包对模型中的蛋白进行生存曲线绘制,用pheatmap包对样本进行风险评分排序,然后,根据风险评分和蛋白质表达数据来绘制风险热图,用来分析模型蛋白在高风险和低风险评分中的表达情况;绘制风险评分和患者评分的风险曲线,用来评价预测模型在评估患者生存风险中的作用;绘制风险评分和生存状态的生存状态图,用来评估模型对患者生存和预后的预测效果。
1.4 预测模型的性能评估
结合Perl软件对样本的生存时间、生存状态、风险值、年龄、分期等进行组合,然后进行单变量和多变量Cox分析,观察上述临床状态、风险值和生存状态之间的相关性,评价风险预测模型评分在预后预测中的作用,结果用可视化的森林图展示。建立预测列线图。列线图通常用于预测癌症的预后,将统计预测模型简化为根据个体患者的状况对事件(例如复发或死亡)的概率进行单一数值评估。通过R软件中的survival ROC包,绘制受试者操作特征(ROC)曲线,计算AUC值,评估乳腺癌患者预后模型的预测准确性。
1.5 共表达蛋白分析
将预测模型中的蛋白质和乳腺癌相关的223个蛋白质的表达数据作为输入数据,用R软件进行相关分析,采用相关系数cor>0.4和P<0.001作为筛选条件对结果进行筛选。使用R软件中的ggalluvial、ggplot2和dplyr包构建相关共表达蛋白的桑基图。
1.6 差异表达分析
UALCAN是一个用户友好的交互式网络资源,包含了根据TCGA数据库中31种癌症类型的3级RNA-seq数据和临床数据分析癌症OMICS数据。在UALCAN网站(http://ualcan.path.uab.edu/)[23]上分析6个中枢蛋白及其编码基因的差异表达。
1.7 统计学方法
在R软件(R 4.0.2)或Perl软件Strawberry Perl(64位)中,分析从TCPA和TCGA数据集检索得到的蛋白质数据和临床信息。所有统计分析均通过R软件进行评估。检验水准α=0.05。
2 结果
2.1 蛋白质预测模型的构建
TCPA数据库已将原始数据转换为可识别的格式。首先基于单变量Cox比例风险回归分析与总生存期(OS)相关的候选蛋白质,P值低于0.05的蛋白质被定义为显著。HR<1的蛋白质被定义为候选保护性蛋白质,HR≥1的蛋白质被认为是候选危险蛋白质。用R软件ggplot2和ggrepel包绘制火山图进行可视化(图1A);构建预后风险特征模型。通过lasso回归筛选出12种蛋白质来去除共线性(图1B,C);多因素Cox分析共筛选出6种蛋白,可作为乳腺癌预后的独立危险因素(CASPASE7-CLEAVEDD198、NFKBP65_pS536、PCADHERIN、P27、X4EBP1_pT70、EIF4G)。预后风险特征是通过组合6种中枢蛋白的表达值而构建的,这些值由其回归系数加权。将中位风险评分作为临界值,将乳腺癌患者分为高风险组和低风险组。

图1 预后相关蛋白质及lasso回归分析(红色表示高风险蛋白质,绿色表示低风险蛋白质)
2.2 预测模型中的蛋白评估
通过对6种模型蛋白的生存分析,发现CASPASE7-CLEAVEDD198、PCADHERIN和P27蛋白高表达的患者预后良好(P<0.05),可能在乳腺癌的发生发展中起到抑癌作用(图2A、B、C)。NFKBP65_pS536、X4EBP1_pT70和EIF4G蛋白高表达的患者预后较差(P<0.05),可能是乳腺癌发生发展的促癌因素(图2D、E、F)。通过预测模型风险评分的生存率分析发现,高风险组的总体生存率明显低于低风险组(P<0.001)。验证风险热图显示,蛋白CASPASE7-CLEAVEDD198、PCADHERIN和P27在高危组中较低,NFKBP65_pS 536、X4EBP1_pT70和EIF4G在高危组中较高,这与生存分析结果具有相同的趋势(图3C)。风险曲线显示,随着患者生存风险评分的增加,模型风险评分相应增加(图3A)。生存状态图显示患者的生存率随着生存风险评分的增加而降低(图3B)。

图2 6种预测模型蛋白的生存分析
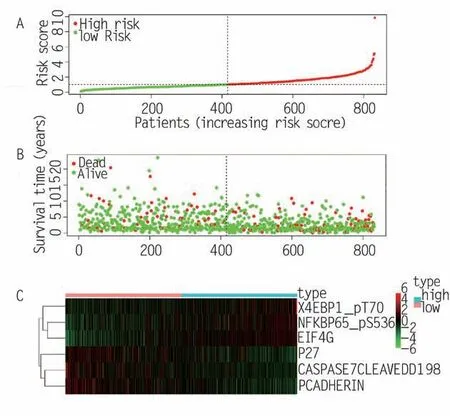
图3 乳腺癌组的详细预后模型信息
2.3 预测模型预测性能验证
我们进一步评估了预后风险模型的性能。根据风险评分分为两组后,高风险组和低风险组在OS上差异有统计学意义(P<0.05,图4)。单因素(图5A)和多因素(图5B)Cox回归分析显示,患者年龄和预测模型是影响预后的独立危险因素(P<0.05);预测列线图是通过涉及临床病理学和预后模型构建的。预后模型和临床病理数据的使用可以提高3年、5年和10年OS预测的敏感性和特异性(图6)。ROC分析可以检验预测模型评价患者预后的准确性和敏感性(图7),通过ROC分析预测模型的风险评分和临床病理特征,发现预测模型比传统的临床病理对乳腺癌患者的预后评估具有更高的准确性和敏感性。

图4 预后风险模型验证(高低风险组OS差异有统计学意义)

图5 临床病理参数和风险模型的预后意义

图6 使用临床病理数据和预后模型构建的列线图

图7 风险特征的预测准确性(ROC曲线)
2.4 6个Hub蛋白的差异表达分析
与正常样本相比,4EBP1、CASP7、CDKN1B和EIF4G1的mRNA在原发性肿瘤中的表达显著增加,而NFKB1和CDH3的表达显著降低。同样的,在蛋白质表达水平上,我们发现乳腺癌组织中CASP7、CDH3和EIF4G1的表达显著增加,而NFKB1的表达显著降低(图8,9)。

图8 乳腺癌组织和正常组织(UALCAN)之间6种编码基因的表达(*P<0.05)
2.5 蛋白质相关性分析
根据蛋白质相关性分析结果显示,蛋白CASPASE7CLEAVEDD198、PCADHERIN、X4EBP1_pT70和EIF4G与其他蛋白质有很强的相关性,尤其是EIF4G,它的相关蛋白数量最多。对相关蛋白的进一步分析发现,它们其中大多数作为促癌蛋白,参与癌症发生、PD-L1表达与PD-1检查点通路、ErbB和mTOR信号通路来促进肿瘤细胞增殖、侵袭和转移,其中CASPASE7CLEAVEDD198蛋白与癌细胞的凋亡有关,PCADHERIN与癌细胞转移有关,X4EBP1和EIF4G与癌细胞转录有关。
3 讨论
乳腺癌的发病率较高,发病机制尚未被完全阐明,并且仍然缺乏有效的治疗方法[9]。目前,虽然对乳腺癌的诊断和治疗有很多的研究,但都没有取得重大突破。因此,寻找一种可靠的预测乳腺癌预后的方法,对及时、准确地预测治疗效果,指导进一步治疗具有重要的意义。
在本研究中,通过对TCPA数据库中乳腺癌相关蛋白进行分析,构建了一个包含6种蛋白质的预测模型,结果发现,该预测模型能有效预测生存率,而高危组总生存率较低危组差。独立预后及ROC分析表明,该预测模型可作为患者预后的独立危险因素,对患者预后有较好的预测价值。Ualcan数据库分析表明CASP7、EIF4G1在乳腺癌组织中显著过表达,而NFKB1在结直肠癌组织中显著低表达。CDH3的mRNA表达与其蛋白表达不匹配。此外,通过蛋白质相关性分析,发现CASPASE7CLEAVEDD19 8、PCADHERIN、X4EBP1_pT70和EIF4G是调控乳腺癌发生发展的关键蛋白。
CASPASE7是半胱氨酸天冬氨酸蛋白酶(半胱氨酸酶)家族的一员。SREBP1通过抑制CASPASE7的表达促进乳腺癌细胞对5-Fu的抵抗[10]。在内分泌抵抗的乳腺癌细胞中,ERα的缺失通过降低ERα介导的CASPASE7的表达而影响HDAC3的稳定,从而导致CASPASE7介导的HDAC3裂解的减少[11]。同时我们还发现,ERα-HDAC3轴决定了H3K9和H4K16乙酰化的整体状态,这与ERα的表达呈正相关。
P-cadherin是细胞间粘附分子cadherin家族的一员,P-cadherin在乳腺癌中的表达与患者的生存率密切相关,是一个独立的预后预测因子。P-cadherin是比E-cadherin、N-cadherin和其他细胞粘附分子更好的临床预后指标[12-13]。本研究还发现Pcadherin的异常表达(高)与乳腺癌患者的生存率降低有关。
4EBP1是细胞mTOR信号通路的主要影响因子,它的基因扩增状态可用于预测乳腺癌的预后以及不同亚型乳腺癌中抗雌激素治疗的有效性[14]。
研究发现,NFKBP65_pS536(自噬相关蛋白NFKB p65)与高组织学分级(P=0.05)、雌激素受体(ER)阴性(P=0.01)和高Ki67指数(P=0.002)相关,核NF-κB染色患者的病理完全反应(PCR)率高于未染色患者(分别为26.5%和6.0%,P=0.004)[15]。

图9 乳腺癌组织和正常组织(UALCAN)之间6种关键蛋白的表达(*P<0.05)

图10 TCPA数据库中与6个中枢蛋白相关的所有蛋白质的桑基图(R>0.4,P<0.001)
EIF4G是真核翻译起始因子4F(eIF4F)复合物的支架成分,主要参与蛋白质合成的起始,eIF4G的两个亚型(eIF4G1和eIF4G2)与各种肿瘤密切相关。Zhang等[16]研究发现,与癌旁组织相比,eIF4G1在乳腺癌、宫颈癌、鼻咽癌、肺鳞癌、前列腺癌等恶性肿瘤中表达显著上调;eIF4G2在弥漫性大B细胞淋巴瘤和急性髓系白血病中明显高表达,而在膀胱移行细胞癌中低表达。
P27是从G1到S期进展的关键调节因子[17]。细胞周期抑制剂P27是小型(T1a、b)浸润性乳腺癌的独立预后标志物[18]。
在这项研究中,我们还鉴定了与预后特征蛋白的表达显著相关的蛋白质。我们的研究也存在一些不足。首先,TCPA数据库中仅鉴定了200多种蛋白质,缺乏许多关键蛋白质的信息;其次,缺乏外部验证导致我们的研究结果临床应用价值有限,需要进一步研究其外部验证和临床实用性;最后,分子生物学实验对于阐明蛋白质组学特征的潜在分子机制是必要的。总之,我们的研究已经建立并验证了基于蛋白质的乳腺癌患者预后风险模型,这有助于为乳腺癌患者更个性化的治疗提供经济、准确的依据。
利益冲突所有作者均声明不存在利益冲突

