重复序列对植物基因组大小进化的影响
李新玉,王希胤
(1.华北理工大学 生命科学学院,河北 唐山 063210;2.华北理工大学 基因组学与计算生物学研究中心,河北 唐山 063210)
1植物基因组大小的研究现状
基因组大小(Genome Size)普遍指细胞核中所含DNA的数量,也被人们称为C值(C-value),一般以单倍体基因组的核酸量来衡量[1]。植物C值数据库(http://data.kew.org/cvalues/)[2]中包含12 273种植物的基因组大小数据,该数据库对各类植物的基因组大小的动态变化都具有较好的记录。到目前为止,植物C值数据库中包括10 770种被子植物、421种裸子植物、303种蕨类植物、334种苔藓植物和445种藻类植物的基因组大小信息。此外,各种植物的基因组大小表现出惊人的多样性。例如,在开花植物中,食肉植物螺旋狸藻(Genlisea tuberosa)的基因组最小,只有61 Mb,而同为有花植物的日本重楼(Paris japonica)拥有最大的基因组,基因组高达148 Gb,它们二者的基因组大小差异超过2 400倍[3, 4]。但是,经过人们更加深入的研究,研究者们发现物种的基因组大小和进化的复杂程度之间没有明显的对应关系,这一现象被称为“C值悖论”[5]。例如,进化程度较高的被子植物拟南芥的基因组大小为118 Mb[6],而进化程度较低的苔藓植物小叶萍的基因组大小为790 Mb[7],是拟南芥的6倍多。因此,基因组大小与进化复杂度之间没有显著关系。
造成植物基因组大小多样性的原因主要有3种:一是多倍化(Polyploidization),也称为全基因组复制(Whole Genome Duplication,WGD),多倍体在植物界是很常见的,现存的植物中大约有70%是多倍体[8]。几乎所有的植物都经历过一次或多次的全基因组复制事件,影响基因组大小和基因含量[9]。但是,基因组大小并不会有大的变动,因为基因组经历了加倍后会发生许多重组事件进而删除掉大量的DNA序列,因此,基因组大小的增加与多倍体化的次数不成正比,多倍体基因组的大小通常比预期小,每个基本亚基因组的平均DNA含量往往随着倍性的增加而减少[10]。例如,模式植物拟南芥虽然受到额外全基因组复制的影响,但是其基因组很小,为118 Mb[6],而一些经历较少全基因组复制事件的物种,如葡萄基因组大小为504.6 Mb[11]和桃子基因组大小为265 Mb[12],二者的基因组反而比拟南芥的更大。二是种内变异。例如,研究人员对20个瓜拉尼玉米群体进行研究,结果表明,群体内基因组大小的差异为36.15%[13],这种差异可能是由于在每个旋钮染色体位置观察到的存在/不存在的杂合性和异染色质变异的百分比。对淡色羊茅(Festuca pallens)24个居群的种内基因组大小变异进行了研究,发现有16个居群的DNA含量存在种内变异,基因组大小受地理因素的影响。在二倍体物种中,基因组较大的物种生活在冰缘草原地区。四倍体的相对DNA含量也与纬度和经度显著相关[14]。二倍体或四倍体可能是高倍性的新多倍体,它们更多地出现在高纬度地区,每一次多倍体化都使冷相关基因富集[15]。以上研究表明,种内基因组大小存在较大差异,并且这种差异与地理因素、气候因素等存在相关性。但是,这种联系并不能断定这些因素就是导致种内基因组大小变异的根本原因。人们对引起种内基因组大小变异的原因仍知之甚少。三是重复序列,尤其是LTR反转录转座子的插入与删除是造成基因组大小巨大差异的主要因素。种内和种间基因组大小之间的差异都与重复序列的差异表现出了明显相关性。该项研究将详细地讨论重复序列对基因组大小进化的影响。
2植物基因组中重复序列的分类
重复序列是指在基因组中不同位置重复数百次或数千次的DNA序列基序[16],重复序列主要分为两大类:一类是串联重复序列,主要包括一些较短的重复,如卫星DNA;另一类是散在重复序列,主要指的是转座元件,包括DNA转座子和RNA转座子[17],具体分类见图1。散在重复序列也是通常所说的转座子。Thomas Wicker等人[18]提出的分类系统按照转座方式的不同,将转座子分为两大类:I类转座子通常指的是RNA转座子或者反转录转座子(Retrotransposons)。反转录转座子(或称作RNA转座子)可以进一步划分为两大类转座子,一是长末端重复(Long terminal repeat, LTR)反转录转座子,这类转座子两端存在相同的长末端重复序列,即LTR序列;二是非长末端重复(Non-long terminal repeat,Non-LTR)反转录转座子。在植物基因组中,LTR反转座子根据其内部编码序列的结构,可以进一步分为Copia和Gypsy两大类;Non-LTR转座子主要分为LINEs(Long interspersed elements)和SINEs(Short interspersed elememts)两大类。在I型转座子中,Gypsy和Copia是LTR反转座子中最丰富的两类转座子[19, 20]。二者结构特征的主要区别是LTR序列之间的编码序列的排列方式不同,如图2所示。在许多数植物中Gypsy所占基因组的比例要比Copia高[21],也有特殊情况,例如,小果野蕉(Musa acuminata)和芹菜(Apium graveolens)等植物的基因组中Copia转座子比Gypsy转座子的比例高[22, 23]。
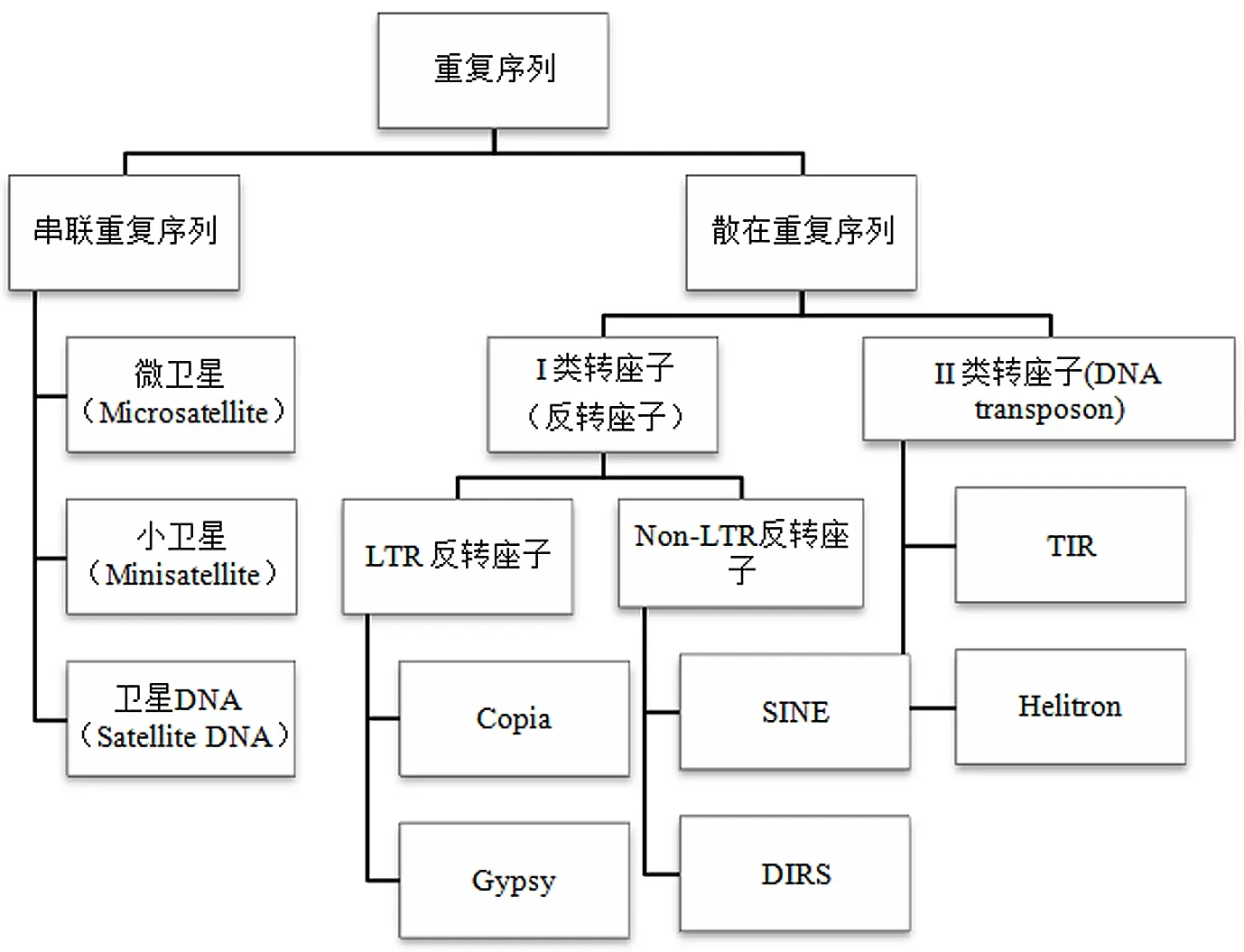
图1 植物基因组中重复序列的分类
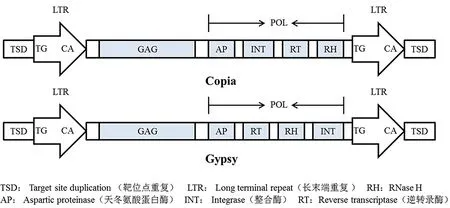
图2 Copia和Gypsy反转录转座子的结构示意图
II类转座子(Class II elements)又叫做DNA转座子。这类转座子以“剪切-粘贴”的形式进行转座,它们在转座的时候在酶的作用下从基因组某个位置剪切下来,然后在整合酶的作用下重新整合到基因组的一个新的位置,而之前位置少了一段DNA序列。DNA转座子在植物基因组中主要分为2个子类,Subclass 1(TIR)和Subclass 2(Helitron)[18]。它们的区别在于转座过程中被切割的脱氧核糖核酸链的数量。TIR类转座子的末端反向重复序列(Terminal Inverted Repeats, TIRs)的长度可变,这是这类转座子的一大特征,根据TIR序列和TSD大小可以进一步将这类转座子分为9个超家族,比较常见的包括hAT、CACTA、Mutator等超家族。Helitron转座子是近年来发现的一种新型DNA转座子,通过滚环(rolling-circle)的方式进行转座,转座的时候只有一条链被切割,并且不产生TSD。这个过程包括Helitron单链末端切口的切割,链侵入、DNA合成、链置换和通过DNA复制拆分异源双链体等[24]。
串联重复序列(Tandem Repeats, TRs)也被称为卫星DNA,是包含至少2个相邻重复单元的DNA序列基序[25],它们广泛存在于原核生物和真核生物中[26]。一般来说,根据重复单位长度和阵列大小将串联重复序列分为3类:微卫星(Microsatellite),又称短串联重复序列(Short Tandem Repeat,STR)或简单重复序列(Simple Sequence RepeatSSR),长度为2~5 bp,通常具有10~100 bp的重复阵列;小卫星(Minisatellite),长度为6~100 bp,阵列长度0.5~35 kb;卫星DNA(satellite DNA),长度一般大于100 bp,通常形成高达100 Mb的阵列[26, 27]。串联重复序列被认为是通过突变、不平等交叉、基因转换、滑移复制和/或滚圈复制等分子机制的组合作用形成的,这些机制创造并维持物种内卫星DNA序列的同质性[28, 29]。
3串联重复序列对植物基因组大小进化的影响
串联重复序列主要分布在着丝粒、着丝粒周围或近着丝粒的异染色质区域。串联重复序列最初被认为是垃圾DNA,如今被认为是进化过程中的“调节旋钮”,因为它们能够调节遗传性状,从而促进适应性进化[30]。例如,串联重复序列能维持姐妹染色单体的内聚性和正确的染色体分离[31]。串联重复序列可以从亚染色体向间质基因座扩散,导致染色体特异性位点的形成或在不同染色体的等位点积累;一些卫星重复序列被转录,可以参与异染色质结构的形成和维持以及基因表达的调节。同时,卫星DNA对染色体的形成也至关重要,卫星DNA能够被特定的DNA结合蛋白识别,形成染色体的端粒、着丝粒和近端着丝粒等[31],对染色体的形成至关重要[32]。
串联重复序列在植物基因组中的含量存在较大差异,比如,卫星DNA的基因组含量可以从几种贝母属(Fritillaria)物种中FriSAT1卫星DNA的0.1%或小果野蕉(Musa acuminata)基因组中卫星DNA的0.3%不等[33, 34]到Fritillaria falcate中FriSAT1的36%[35]。因此,基因组中存在的卫星DNA可能对基因组大小有一定影响[31]。但是,还没有发现卫星DNA含量与基因组大小变化之间存在关系的证据,赵致新博士等人对植物和绿藻全基因组范围内的串联重复序列进行了研究,分析了串联重复序列的分布和基序特征,并探讨了串联重复序列的生物学功能。在31个物种中,串联重复序列密度与基因组大小之间没有显著相关性。在紧凑基因组(如病毒)和具有大量基因间区域的基因组(如植物)中,基因组大小与串联重复序列密度之间存在弱阳性但不显著的相关性,这表明串联重复序列可能对进化中基因组大小的扩增没有显著贡献[36],基因组大小主要是由于其他种类的重复DNA,如转座元件所致[35]。
4散在重复序列对基因组大小进化的影响
4.1 反转座子对基因组大小进化的影响
散在重复序列占据了植物基因组重复序列的大部分比例,一类重复序列长末端重复反转录转座子是植物基因组中最丰富的DNA成分[37],如表1所示。许多研究表明[38, 39],一个物种的基因组大小与反转录转座子的增殖有直接关系,反转录转座子对基因组的扩增有重要贡献。比如,LTR反转座子占玉米基因组的75%以上[40],在过去的300万年之内,玉米基因组从1 200 Mb 增加到2 400 Mb,这主要是由LTR反转座子插入引起的[41]。对稻属一个古老的反转座子家族的研究表明,在稻属分化之后,由于RWG家系Gran3反转座子的爆发,水稻GG基因组(Oryza granulata)的基因组大小扩增了近25%,驯化水稻(Oryzasativa)的基因组大小与大多数祖先谱系的基因组大小存在2倍以上的差异,这可能是RWG家族的扩增差异造成的[42]。
根据反转座子的转座机制,人们称反转座子为“复制-粘贴”型转座子。反转座子在转座时,会先以DNA为模板,在RNA聚合酶II的作用下,转录成一段mRNA,然后再以这段mRNA为模板反转录成cDNA,最后在整合酶的作用下将这段cDNA整合到基因组上新的位置,每个完整的复制周期都会生成一个新的反转座子[24]。因此,反转座子通常是基因组中大量重复片段的主要贡献者[43]。
有研究指出,如果没有一个积极的DNA去除过程,所有的植物都可能走向基因组肥胖[44]。一些植物的基因组在经历了如此多繁重的大片段重复DNA的情况下存活下来,这意味着冗余DNA的负面影响可能已经被某种补偿所抵消。一方面,大多数LTR反转座子被DNA甲基化和小RNA介导的表观遗传沉默机制灭活[45]。因此,转座事件发生得非常少,很难实时观察到[46]。当植物受到外界各种刺激时[47],如病原体攻击、组织培养、损伤、环境胁迫等,反转录转座子会被激活并插入到靶基因中,例如,ONSEN反转座子获得了一个热响应调节元件,尽管有功能性沉默机制,但该反转座子在受到热应激时仍会被激活[48],同时,周边基因的表达也会受到影响。另一方面,LTR反转座子被不平等的同源重组(Unequal Homologous Recombination,UR)和非法重组(Illegitimate Recombination,IR)所删除/截断,作为抑制机制对抗LTR反转座子扩增引起的基因组肥胖[37]。不平等同源重组和异常重组是LTR反转座子的去除的主要机制[49]。不平等同源重组,通常发生在一个完整元件的2个LTR之间,这2个LTR去除了它的内部部分,重组之后的LTR形成一个solo-LTR,即只含有LTR区的序列[49],从而减少(但不是完全逆转)由反转录转座子造成的基因组大小的增加[50, 51]。非法重组不需要发生在同源序列之间,发生重组的序列之间通过产生和积累小的缺失片段来消除转座子序列[49, 50, 52]。研究表明,非法重组是拟南芥基因组大小减少的驱动力,去除的脱氧核糖核酸至少是不相等同源重组的5倍[50];还有研究表明,水稻基因组中的LTR反转录转座子序列已经至少在不平等同源重组和非法重组的作用下删除了194 Mb[49]。
4.2 DNA转座子对基因组大小进化的影响
在大多数植物中,逆转录转座子(Class I elements)构成了大部分的转座子,促成了基因组大小的扩张和收缩以及种间序列的差异[53],而不太丰富的DNA转座子(Class II elements)往往与基因组的常染色体或基因成分有关,是遗传多样性产生的主要进化力量[54]。因此,与植物寄主的基因有更密切的相互作用。有研究表明,一些DNA转座子可能会导致更高的突变率,因此可以加速基因进化,例如,DNA转座子在禾本科植物中以数万个拷贝存在[40, 55],大多数基因在其附近的某个点上都会经历转座子切除,因此随着时间的推移,可能会积累较多的突变基因,因此,DNA转座子的活性是禾本科植物进化的重要驱动力。“剪切-粘贴”型的DNA转座子在入侵新的宿主后,新到达的DNA转座子必须在宿主群体中增殖和扩散。然而,在传播之后,宿主的自我调节开始限制转座和灭活转座子,最终阻碍了该元素在宿主中的扩增。由于不能转座和增殖,不活跃的转座子会遗留在宿主基因组中,最终可能会因遗传漂变而丢失[56]。由于DNA转座子在植物基因组中的数量并不丰富,再加上各种灭活和删除机制的作用,DNA转座子通常不会获得高拷贝数[57],但MITEs类DNA转座子在拷贝数上是个例外,它们在一些基因组中以极高的拷贝数存在[46, 58],然而,MITEs类DNA转座子的长度只有200~500 bp,因此MITEs对基因组肥胖的影响有限[59]。综上所述,“剪切粘贴”型DNA转座子对宿主的基因影响较大,而对基因组大小的影响有限[57]。表1所示为一些常见植物的基因组大小与重复序列富集情况。
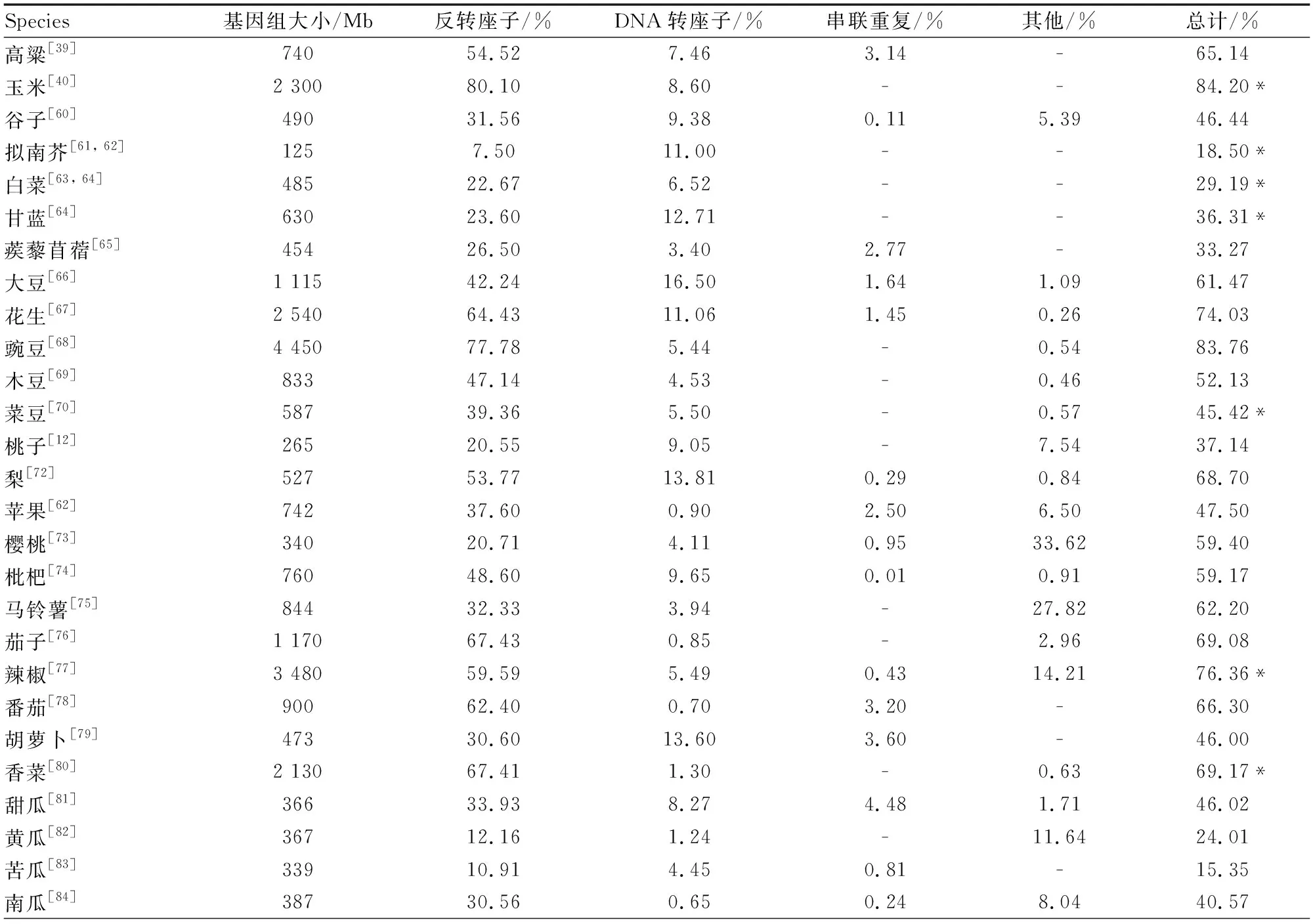
表1 常见植物的基因组大小与重复序列富集统计
Helitron转座子是近年来发现的一种新型DNA转座子,它在不同物种之间的分布存在较大差异,例如,在拟南芥中主要分布在基因贫乏的区域,尤其是近着丝粒区域[85]。水稻基因组中的Helitron分布在着丝粒周围和染色体末端[85],玉米中的Helitrons多分布在基因丰富区域[86]。Helitron是一种独特的滚动循环型转座子,与其他转座子一样,它们的激活可能涉及新基因的生成或现有基因的调节,并可能影响表型表达[87]。Helitron对基因组的影响与所有的转座元件一样,与它的转座机制、在宿主内的相互作用、与其他转座元件家族的竞争以及群体遗传特征密切相关[88]。虽然Helitron在植物基因组中广泛存在,但比例远远不及LTR反转录转座子,在不同植物中的比例也存在差异,比如在禾本科物种中,Helitron占水稻基因组的4%,高粱基因组的1%,玉米基因组的6.6%[89];在拟南芥基因组中,大约1.6%的基因组DNA由Helitron组成,而在琴叶拟南芥中Helitron占其基因组的3.6%[89],可见Helitron的含量在同一科或同一属不同物种之间都存在较大差异,Helitron丰度和基因组大小之间没有显著的相关性,Helitron转座子对基因组大小没有显著影响。图3所示为几种代表植物的基因组大小和重复序列的富集程度的散点图。
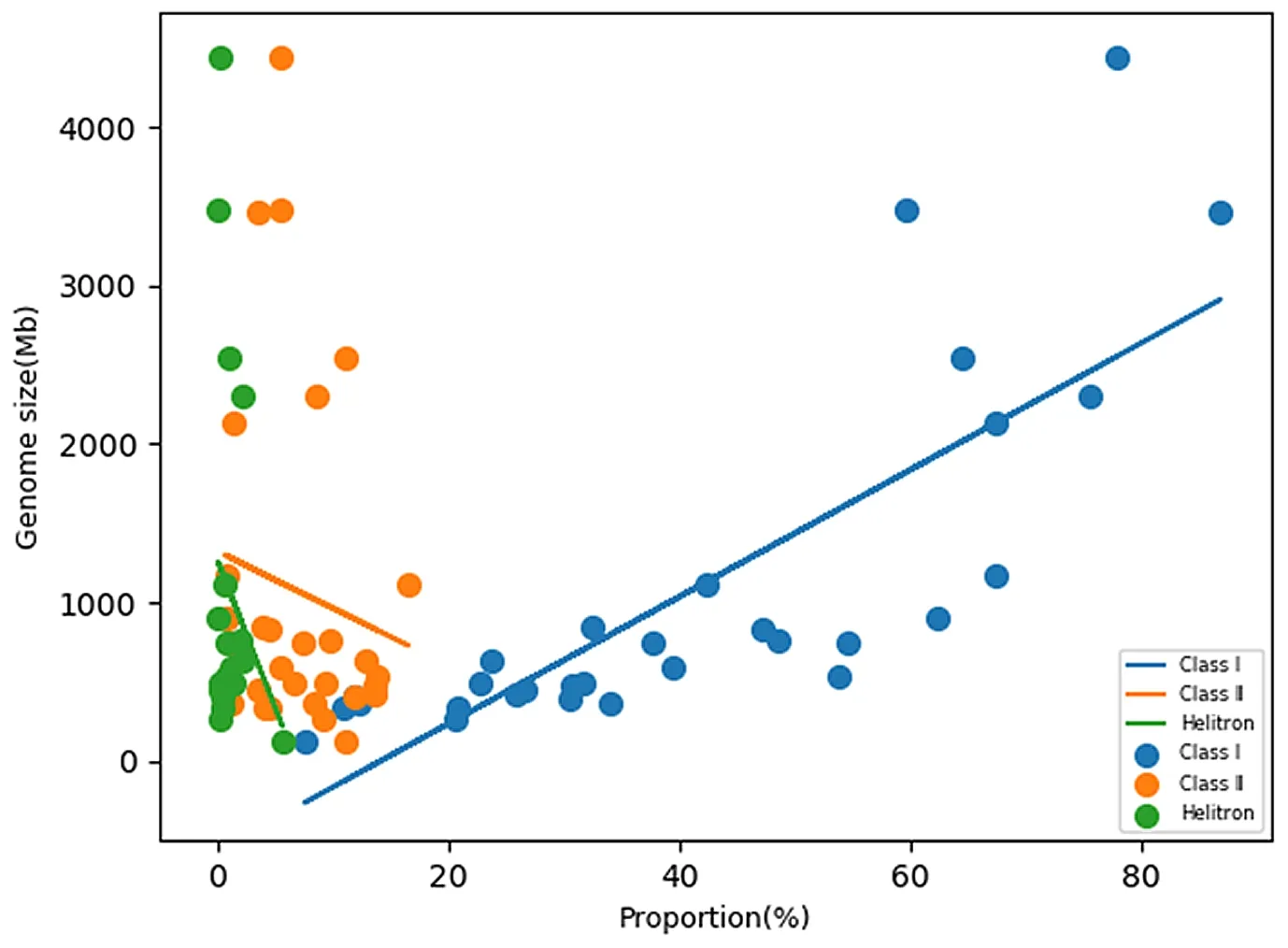
图3 几种代表植物的基因组大小和重复序列的富集程度的散点图
5存在的问题及展望
虽然已知基因组大小多样性的主要贡献者是高度重复的DNA序列尤其是反转座子,如图3所示,classⅠ,即反转座子的比例与基因组大小存在明显的正相关。但是,大量研究结果表明,重复序列尤其是转座元件对基因组大小的影响呈高度动态且很难预测[90]。它们的起源、表达、插入特异性、进化命运以及对遗传和表观遗传基因调控的潜在影响仍需进一步探索[37]。大量额外基因组的测序可能为进一步研究这种关系提供有用的信息。到底是什么原因造成不同物种之间重复序列数量差异如此之大,以及基因组大小最终的进化方向,不同物种之间基因组大小的进化差异等问题仍然有待研究。随着测序技术和生物信息学的不断发展,基因组数据分析变得越来越简便化与高效化,但是如何利用解析出来的基因组密码解决实际问题才是真正的研究重点。伴随着大数据时代的到来,期望以后能够开发出更多的优质算法与数学模型,来帮助阐明基因组增大和减小之间的动态变化以及基因组大小最终的进化方向,进而为了解重要经济作物的基因组结构以及进化机制提供重要理论支持。

